基于葉綠體atpB-rbcL序列探討猬草屬和賴草屬植物的系統(tǒng)發(fā)育和母系起源
劉靜,張海琴,凡星,沙莉娜,曾建,周永紅,4*
(1.四川農(nóng)業(yè)大學(xué)成都校區(qū)小麥研究所,四川 成都611130;2.四川農(nóng)業(yè)大學(xué)生命科學(xué)與理學(xué)院,四川 雅安625014;3.四川農(nóng)業(yè)大學(xué)成都校區(qū)資源與環(huán)境學(xué)院,四川 成都611130;4.四川農(nóng)業(yè)大學(xué)作物基因資源與遺傳改良教育部重點實驗室,四川 成都611130)
小麥族(Triticeae)是禾本科(Poaceae)植物中十分重要的類群,既包含有重要的糧食作物如小麥(Triticum aestivum)、大麥(Hordeum vulgare),也有許多優(yōu)良的牧草資源如羊草(Leymus chinensis)和老芒麥(Elymus sibiricus)等[1]。基于麥類作物和牧草資源的開發(fā)、生態(tài)保護(hù)與重建的需要,理清小麥族內(nèi)物種分類問題及系統(tǒng)發(fā)育關(guān)系尤為重要。
猬草屬(Hystrix)是小麥族的一個多年生小屬,Moench[2]根據(jù)穎強烈退化或缺失的特征以Hystrix patula為模式種建立。根據(jù)Baden等[3]的分類系統(tǒng),猬草屬有6種3變種:Hystrix patula、猬草(H.duthiei)、長芒猬草(H.duthiei ssp.longearistata)、H.duthiei ssp.japonica、東北猬草(H.komarovii)、高麗猬草(H.coreana)、H.sibirica和H.californica。L9ve[4]認(rèn)為猬草屬模式種 H.patula具有與披堿草屬(Elymus)相同的StH染色體組組成,將猬草屬作為披堿草屬的一個組處理。然而,Jensen和 Wang[5]根據(jù)染色體組分析結(jié)果認(rèn)為,高麗猬草和H.californica含有與賴草屬(Leymus)植物相同的NsXm染色體組組成,并將它們組合到賴草屬中。Zhang等[6]根據(jù)細(xì)胞遺傳學(xué)和染色體組原位雜交資料,報道H.patula含有StH染色體組,而猬草和長芒猬草含有NsXm染色體組。Ellneskog-Staam等[7]通過基因組原位雜交(GISH)和Southern雜交分析認(rèn)為,猬草、長芒猬草和高麗猬草含有Ns1Ns2染色體組,而東北猬草具有與猬草屬模式種H.patula相似的StH染色體組。因此,猬草屬不是一個單系類群,自建屬以來,屬的界限和分類地位以及物種的染色體組組成,一直處于爭論之中。
賴草屬是小麥族中重要的多年生異源多倍體屬,該屬植物約有30個種,從北海的沿岸地區(qū),越過中亞到東亞直至阿拉斯加和北美西部的廣闊地域均有分布。賴草屬物種倍性變化從四倍體(2n=4x=28)到十二倍體(2n=12x=84),由2個基本的染色體組Ns和Xm組成[1,8]。通過形態(tài)學(xué)觀察、染色體組分析及分子資料分析,認(rèn)為Ns染色體組來源于新麥草屬(Psathyrostachys)[1,4,8-12],而 Xm 染色體組的來源尚不確定[8]。迄今為止,來源于擬鵝觀草屬(Pseudoroegneria)的St染色體組[13]、冠麥草屬(Lophopyrum)的Ee染色體組[14]、Eb染色體組[4]、新麥草屬的Ns染色體組[15]、冰草屬(Agropyron)的P染色體組和旱麥草屬(Eremopyrum)的F染色體組[11-19]被推測是Xm染色體組的供體來源。
細(xì)胞核基因資料已廣泛應(yīng)用于小麥族植物的遺傳多樣性和系統(tǒng)進(jìn)化研究[11,16-18],而細(xì)胞質(zhì)基因組具有單拷貝及母系遺傳的特征,在植物系統(tǒng)與進(jìn)化研究中具有更好的利用價值。rbcL、ndhF、trnL-F等葉綠體基因或基因間隔區(qū)序列已成功應(yīng)用于研究小麥族賴草屬、披堿草屬等類群的系統(tǒng)關(guān)系、網(wǎng)狀進(jìn)化及母系起源問題[19-23]。Sha等[19]利用單拷貝核基因DMC和葉綠體trnL-F分子序列對猬草屬和賴草屬系統(tǒng)分析時發(fā)現(xiàn),猬草屬物種具有不同的母本起源,猬草、長芒猬草及歐亞分布的賴草屬植物的母本來源為新麥草屬植物,高麗猬草和東北猬草與北美分布的賴草的母本來源可能是冰草屬的P染色體組。Liu等[20]根據(jù)ITS及葉綠體trnL-F序列分析發(fā)現(xiàn):歐亞分布的賴草屬植物母本起源為新麥草屬,而北美分布的賴草屬植物母本來源仍然未知,但推測不為新麥草屬。
葉綠體atpB-rbcL序列是位于編碼ATP合成酶β亞基和核酮糖1,5二磷酸羧化酶大亞基基因之間的非編碼區(qū)域,Nishikawa等[24]利用該序列探討了大麥屬的系統(tǒng)與進(jìn)化。本研究利用葉綠體atpB-rbcL序列對猬草屬及其近緣屬植物進(jìn)行系統(tǒng)發(fā)育分析,主要目的在于:1)分析猬草屬和賴草屬植物的系統(tǒng)關(guān)系;2)探討猬草屬與賴草屬植物可能的母本起源。
1 材料與方法
1.1 供試材料
供試材料共46份,包括5個猬草屬物種、來自北美、中亞及青藏高原的22個賴草屬物種、2個披堿草屬物種,以及來自小麥族8個不同基本染色體組的17個二倍體材料,包括4個新麥草屬(Ns染色體組)、2個擬鵝觀草屬(St染色體組)、2個大麥屬(Hordeum,H染色體組)、3個冰草屬(P染色體組)、2個旱麥草屬(F染色體組)、反穎南麥(Australopyrumretrofractum,W 染色體組)、簇毛麥(Dasypyrumvillosum,V 染色體組)、長穗冠麥草(Lophopyrumelongatum,Ee染色體組)、百薩冠麥草(L.bessarabicum,Eb染色體組)等物種。旱雀麥(Bromus tectorum)作為外類群。所有材料的物種名稱、采集編號、來源及GenBank號列于表1。染色體組符號依照Wang等[8]的命名標(biāo)準(zhǔn)。所有材料均種植于四川農(nóng)業(yè)大學(xué)小麥研究所多年生種質(zhì)圃,憑證標(biāo)本藏于四川農(nóng)業(yè)大學(xué)小麥研究所標(biāo)本室(SAUTI)。
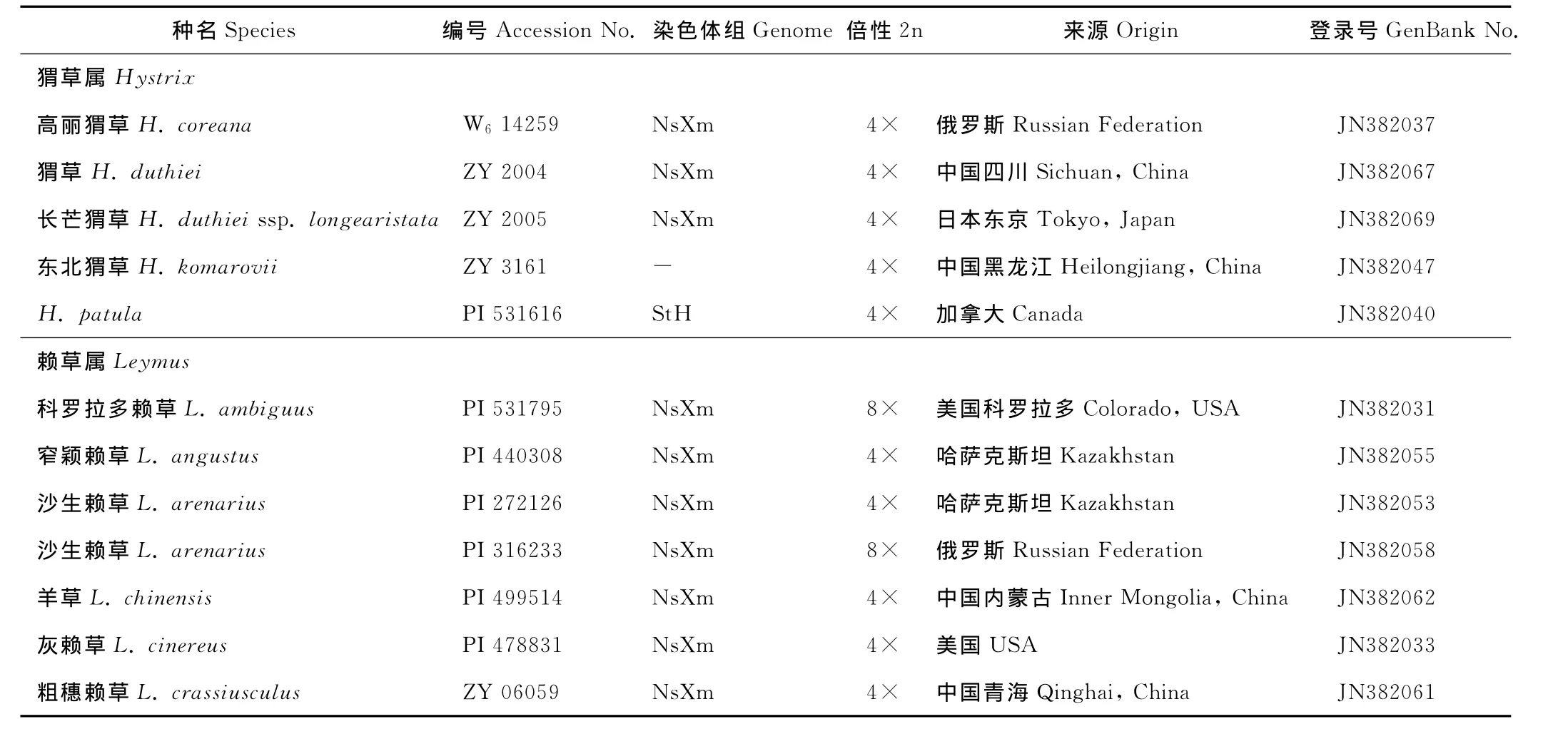
表1 供試材料Table 1 The materials used in this study

續(xù)表1 Continued
1.2 DNA提取、擴(kuò)增和測序
DNA提 取 采 用 改 良 的 CTAB 法[25]。PCR 擴(kuò) 增 引 物 序 列 為 P1:5′-ACATCGTAAGTACGTGGACCAATAA-3′和P2:5′-TTGGATTCTAAAGCTGGTGTT-3′[24]。PCR擴(kuò)增反應(yīng)在 ABI 9700型PCR儀上進(jìn)行。反應(yīng)體積為50μL,包括3μL DNA模板(約0.5μg,由Gel Doc圖像分析儀測得),10×ExTaq緩沖液(5μL),2.5mmol/L MgCl2(4.0μL)、2.5mmol/L 4種dNTP混合液(pH 8.0)(4.0μL)、15μmol/L正、反向引物(各1.5μL),5U/μL ExTaq酶(0.6μL)和ddH2O(30.4μL)。PCR反應(yīng)條件為:94℃預(yù)變性4min;94℃變性1min,50℃復(fù)性1min,72℃延伸2min,35個循環(huán);最后72℃延伸10min。PCR產(chǎn)物用1.2%的瓊脂糖進(jìn)行電泳,用OMEGA試劑盒(OMEGA Bio-Tek)進(jìn)行割膠回收。然后,以pMD18-T載體(Takara)為連接載體,DH10B作為宿主菌,對目的片段進(jìn)行克隆。每份材料選取3~5個陽性克隆用于DNA序列測序。序列測定由上海華大生物技術(shù)有限公司完成。所有序列均進(jìn)行雙向測序。DNA提取到測序完成于2009年3-11月。
1.3 基因序列分析
根據(jù)新麥草(Psathyrostachys juncea)的atpB-rbcL序列(GenBank序列號 AB078409)[24]對本研究獲得的序列進(jìn)行確認(rèn)。采用DNAMAN軟件包(version 5.2.10;Lynnon Biosoft,http://www.lynnon.com)對序列進(jìn)行多重排定后再輔以手工校正,并進(jìn)行序列長度變異、插入/缺失(Indel)分析。序列的堿基組成及替代用MEGA 4.0(Kumar S,Tamura K,Jakobsen I,Nei M,http://www.megasoftware.net)進(jìn)行統(tǒng)計。序列變異位點采用PAUP4.0b10(Swofford D L,Sinauer Associates,http://www.sinauer.com)進(jìn)行統(tǒng)計。基于轉(zhuǎn)換和顛換與遺傳距離的線性關(guān)系,采用DAMBE(V4.1.33)[26]替代飽和性分析,選用的進(jìn)化模型是F84。
1.4 系統(tǒng)發(fā)育分析
系統(tǒng)發(fā)育分析采用最大簡約法(maximum parsimony,MP)和貝葉斯推斷(bayesian inference,BI)。MP分析由PAUP4.0b10Win(Swofford,2003)軟件進(jìn)行,采用啟發(fā)式(Heuristic)搜索,gap作為缺失(missing)處理,數(shù)據(jù)隨機添加(addseq=random),重復(fù)次數(shù)(nreps)為100,樹等分與重連分之交換法(TBR)構(gòu)建系統(tǒng)樹,計算50%多數(shù)一致樹和嚴(yán)格一致樹,并采用1 000次重復(fù)抽樣(replicates)的自展分析(bootstrap,BS)來檢驗。貝葉斯推斷采用 MrBayes 3.1.2[27]軟件分析。利用 Modeltest 3.7[28]進(jìn)行模型和參數(shù)估計,基于hLRT 標(biāo)準(zhǔn)選擇的最適堿基替代模型是 K81UF。BI分析中,4條馬爾可夫鏈?zhǔn)椒磻?yīng)(Markov Chain Monte Carlo,MCMC)運行1 500 000代,以隨機樹為起始樹,每100代取樣1次,開始的250個樣本作為老化樣本(burn-in samples)舍棄,剩下的樹用于構(gòu)建50%多數(shù)一致性樹(BI樹),并以后驗概率(posterior probability,PP)來評估其拓?fù)浣Y(jié)構(gòu)的有效性。
2 結(jié)果與分析
2.1 序列分析
葉綠體atpB-rbcL基因間隔區(qū)序列長度變異為773~813bp,平均GC含量30.5%。所有序列排序后共得到831個排列位點,其中保守位點764個,可變位點46個,簡約信息位點21個。序列飽和性分析顯示轉(zhuǎn)換和顛換與遺傳距離F84呈線性關(guān)系(圖1),表明不同序列和不同位點間突變沒有達(dá)到飽和,可用于系統(tǒng)發(fā)育分析。
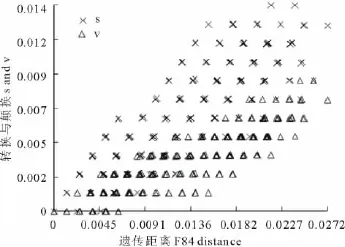
圖1 atpB-rbcL序列的飽和性檢測結(jié)果Fig.1 Substitution saturation analysis of atpB-rbcL
2.2 系統(tǒng)發(fā)育關(guān)系
采用MP和BI兩種方法構(gòu)建atpB-rbcL系統(tǒng)發(fā)育樹。MP分析獲得1 632棵最大簡約樹,樹長(tree length)為72,一致性指數(shù) CI(consistency index)為0.944 4,保持性指數(shù) RI(retention index)為0.966 7,50%多數(shù)一致性樹與通過BI分析得到的系統(tǒng)發(fā)育樹的拓?fù)浣Y(jié)構(gòu)基本一致。圖2顯示的是BI系統(tǒng)發(fā)育樹。
在構(gòu)建的BI樹中,所有供試物種分別聚為A、B兩個分支。分支A包括:2個猬草屬物種(猬草、長芒猬草)、4個新麥草屬物種、所有歐亞分布的賴草屬物種以及來自北美的濱麥(L.mollis),統(tǒng)計支持率(97%PP,65%BS),羊草位于該分支底部。分支A中的物種其atpB-rbcL序列上均含有1個5bp的ATATA插入。分支B中,猬草屬模式種H.patula與含StH染色體組的加拿大披堿草(E.canadensis)聚為姊妹支(支持率為100%PP,85%BS),再與含St染色體組的穗狀擬鵝觀草(P.spicata)、擬鵝觀草(P.strigosa),以及簇毛麥(V)、長穗冠麥草(Ee)和百薩冠麥草(Eb)聚為亞支Ⅰ;冰草屬(P染色體組)、旱麥草屬(F染色體組)物種和反穎南麥(W染色體組)聚為亞支Ⅱ;3個北美賴草無芒賴草(L.triticoides)、灰賴草(L.cinereus)、鹽生賴草(L.salinus)聚為亞支Ⅲ;剩下的2個猬草屬物種(高麗猬草、東北猬草)、2個北美分布的賴草屬物種新枝賴草(L.innovatus)、科羅拉多賴草(L.ambiguus)以及2個大麥屬物種布頓大麥(H.bogdanii)、智利大麥(H.chilense)與3個亞支形成平行分支(圖2)。
3 討論
3.1 猬草屬和賴草屬植物的系統(tǒng)關(guān)系
猬草屬因以其穎退化乃至缺失的形態(tài)特征區(qū)別于小麥族其他屬,Baden等[3]認(rèn)為猬草屬是一獨立有效的屬。然而,染色體組分析和分子系統(tǒng)學(xué)研究表明:模式種H.patula含StH染色體組,東北猬草具變異的StH染色體組,而猬草屬的猬草、長芒猬草和高麗猬草含有與賴草屬一致的NsXm染色體組[5-7,29,30]。
本研究中,猬草屬物種聚成不同的分支。猬草屬模式種H.patula與披堿草屬(StH染色體組)及擬鵝觀草屬(St染色體組)物種聚為一支,表明H.patula與披堿草屬植物有較近的親緣關(guān)系,支持細(xì)胞學(xué)及分子系統(tǒng)學(xué)研究結(jié)果[18,23,29,30],將H.patula組合到披堿草屬中。猬草、長芒猬草、高麗猬草和東北猬草則與新麥草屬(Ns染色體組)和賴草屬(NsXm染色體組)聚在一起,表明該4種猬草屬物種與新麥草屬和賴草屬植物的系統(tǒng)關(guān)系較近,支持猬草、長芒猬草、高麗猬草含NsXm染色體組,并將它們組合到賴草屬中的分類處理[5,6]。
關(guān)于東北猬草的染色體組組成,有不同的報道。Ellneskog-Staam等[7]認(rèn)為東北猬草的染色體組組成為StH;而分子系統(tǒng)學(xué)研究及染色體組特異RAPD分析表明,東北猬草與含Ns染色體組的新麥草屬和賴草屬親緣關(guān)系較近[30-32]。本研究中,東北猬草與高麗猬草及北美分布的賴草屬物種聚在一支,而沒有與含StH染色體組的披堿草屬及H.patula聚類,表明東北猬草與賴草屬物種關(guān)系較近,而與含StH染色體組的披堿草屬物種親緣關(guān)系較遠(yuǎn)。
猬草和長芒猬草與柴達(dá)木賴草(L.pseudoracemosus)等中亞分布的賴草屬物種及含Ns染色體組的新麥草屬物種聚為一支,高麗猬草、東北猬草則與北美分布的賴草屬物種處于另一分支,表明猬草、長芒猬草與中亞分布的賴草屬物種關(guān)系更為緊密,而高麗猬草和東北猬草與北美分布的賴草屬物種關(guān)系密切。單拷貝核基因DMC1和Acc1的分子數(shù)據(jù)也顯示賴草屬物種存在歐亞和北美類群的差異[11,19]。Fan等[11]對世界范圍內(nèi)的賴草屬植物分子系統(tǒng)與進(jìn)化研究推測:北美賴草屬植物很可能是從東亞經(jīng)由白令陸橋向北美擴(kuò)散的。從地理分布上看,高麗猬草、東北猬草處于歐亞大陸東端,與白令海峽接近。本研究結(jié)果也表明高麗猬草、東北猬草與北美賴草具有較近的親緣關(guān)系。
Wen[33]指出相似的生境可能產(chǎn)生相似的選擇壓,從而導(dǎo)致物種形成相近的形態(tài)變異。猬草屬植物常生長于溫暖、潮濕的自然環(huán)境中,含不同染色體組組成的物種其形態(tài)上的相似可能是自然選擇中趨同進(jìn)化的結(jié)果。
3.2 猬草屬和賴草屬植物可能的母本起源
細(xì)胞遺傳學(xué)資料表明:H.patula含有與披堿草屬植物相同的StH染色體組,其中St染色體組起源于擬鵝觀草屬,H染色體組來源于大麥屬[1,4,29]。在本研究中,H.patula與含StH染色體組的披堿草屬及擬鵝觀草屬(St)物種聚為一支,表明H.patula及披堿草屬的母本供體為擬鵝觀草屬植物。本研究結(jié)果與前人對披堿草屬母本來源的研究結(jié)果一致[21,34,35]。同時,擬鵝觀草屬物種與含Ee、Eb及V染色體組的物種聚為一支,表明擬鵝觀草屬的St染色體組和長穗冠麥草的Ee染色體組、百薩冠麥草的Eb染色體組及簇毛麥的V染色體組關(guān)系密切,這一結(jié)果與基于葉綠體trnL-F序列對相關(guān)類群進(jìn)行的聚類結(jié)果一致[20,36]。
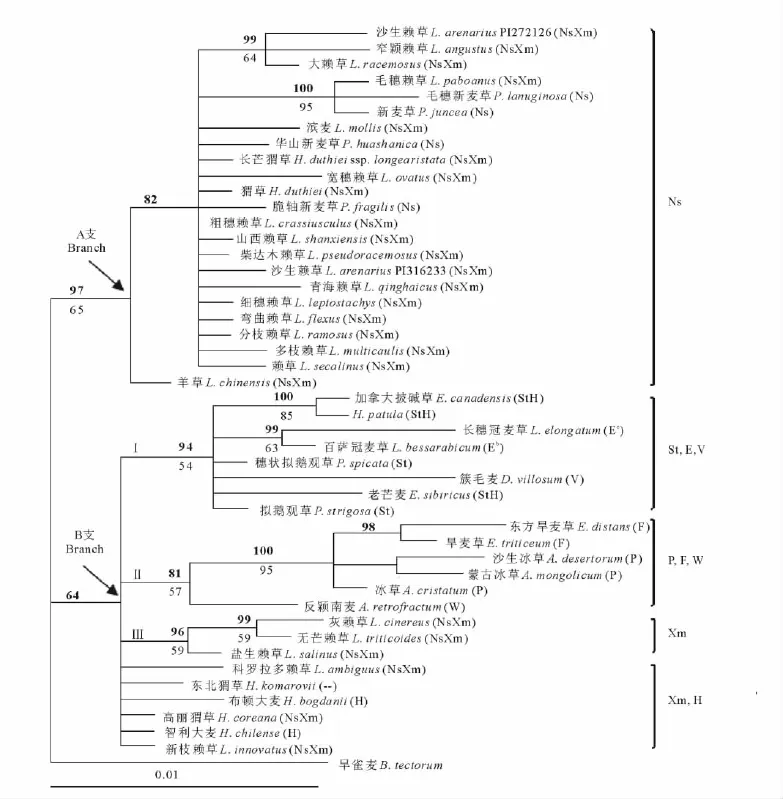
圖2 atpB-rbcL序列的BI系統(tǒng)發(fā)育樹Fig.2 BI tree inferred fromatpB-rbcL sequences
Sha等[19]和Liu等[20]基于葉綠體trnL-F序列的分析,認(rèn)為猬草屬及賴草屬物種的母本起源存在多元性。Sha等[19]認(rèn)為猬草、長芒猬草及歐亞分布的賴草屬植物的母本來源為新麥草屬植物,高麗猬草和東北猬草與北美分布的賴草的母本來源可能是冰草屬的P染色體組。Liu等[20]研究認(rèn)為歐亞分布的賴草屬植物母本起源為新麥草屬,而北美分布的賴草屬植物母本來源仍然未知,但推測不為新麥草屬。本研究中,具NsXm染色體組的物種其葉綠體atpB-rbcL序列明顯聚為2個分支,其中:猬草、長芒猬草及所有歐亞分布的賴草屬物種與4個新麥草屬植物聚為一個分支(Ns支),而高麗猬草、東北猬草和5個來自美洲的賴草屬物種處于另一分支。結(jié)果表明猬草、長芒猬草及歐亞分布的賴草屬物種以新麥草屬(Ns)作為母本供體,而高麗猬草、東北猬草及北美分布的賴草屬植物的母本來源可能為Xm染色體組。由于Xm染色體組確切的二倍體供體物種未知,所以高麗猬草、東北猬草及北美分布的賴草屬植物的母本來源還有待進(jìn)一步研究。
本研究中,來自北美的濱麥在atpB-rbcL序列上缺少其他北美賴草所共有的7bp(TGAAAAA)的插入片段,而和歐亞賴草聚在一起。從地理分布看,濱麥不僅分布于北美,也廣泛分布于中國北方沿海地區(qū)、俄羅斯遠(yuǎn)東地區(qū)、朝鮮和日本[37];從形態(tài)上看,濱麥與歐亞的沙生賴草(L.arenarius)、大賴草(L.racemosus)形態(tài)相似,甚至曾作為沙生賴草的亞種ssp.mollis處理[38]。因此,盡管濱麥來自北美,但與歐亞賴草有更近的親緣關(guān)系,其母本供體仍為Ns染色體組。
[1] Dewey D R.The genome system of classification as a guide to intergeneric hybridization with the perennial Triticeae[A].In:Gustafson J P.Gene Manipulation in Plant Improvement[M].New York:Plenum Press,1984:209-279.
[2] Moench C.Methods Plantas Horti botanici et Agri marburgensis a Staminum Situ Describendi[M].Margburgi Cattorum:in Officina Nova Libraria Academiae,1794.
[3] Baden C,F(xiàn)rederiksen S,Seberg O.A taxonomic revision of the genus Hystrix (Triticeae,Poaceae)[J].Nordic Journal of Botany,1997,17:449-468.
[4] L9ve.Conspectus of the Triticeae[J].Feddes Repert,1984,95:425-521.
[5] Jensen K B,Wang R R C.Cytological and molecular evidence for transferring Elymus coreanus and Elymus californicus from the genus Elymus to Leymus(Poaceae:Triticeae)[J].International Journal of Plant Sciences,1997,158:872-877.
[6] Zhang H Q,Yang R W,Dou Q W,et al.Genome constitutions of Hystrix patula,H.duthiei ssp.duthiei and H.duthiei ssp.longearistata (Poaceae:Triticeae)revealed by meiotic pairing behavior and genomic in-situ hybridization[J].Chromosome Research,2006,14:595-604.
[7] Ellneskog-Staam P,Bothmer R von,Anamthawat-Jónsson,et al.Genome analysis of species in the genus Hystrix (Triticeae;Poaceae)[J].Plant Systematic and Evolution,2007,265:241-249.
[8] Wang R R C,Bothmer R von,Dvorak J,et al.Genome symbols in the Triticeae(Poaceae)[A].In:Wang R R C,Jensen K B,Jaussi C.Proceedings of the 2ndInternational Triticeae Symposium[C].Utah:Logan,USA,1994:29-34.
[9] Wang R R C,Hsiao C.Morphology and cytology of interspecific hybrids of Leymus mollis[J].Journal of Heredity,1984,75:488-492.
[10] Wang R R C,Zhang J Y,Lee B S,et al.Variations in abundance of 2repetitive sequences in Leymus and Psathyrostachys species[J].Genome,2006,49:511-519.
[11] Fan X,Sha L N,Yang R W,et al.Phylogeny and evolutionary history of Leymus(Triticeae;Poaceae)based on a singlecopy nuclear gene encoding plastid acetyl-CoA carboxylase[J].BMC Evolutionary Biology,2009,9:247.
[12] Wang R R C,Jensen K B.Absence of the J-genome in Leymus species(Poaceae,Triticeae)evidence from DNA hybridization and meiotic pairing[J].Genome,1994,37:231-235.
[13] Shiotani I.Species differentiation in Agropyron,Elymus,Hystrix,and Sitanion[A].Proceedings of the 12th International Congress of Genetics[C].Tokyo:The Science Council of Japan,1968:184.
[14] Sun G L,Wu B H,Liu F.Cytogenetic and genomic relationships of Thinopyrum elongatumwith two Psathyrostachys species and with Leymus secalinus (Poaceae)[J].Plant Systematics and Evolution,1995,197:225-231.
[15] Zhang H B,Dvorak J.The genome origin of tetraploid species of Leymus(Poaceae:Triticeae)inferred from variation in repeated nucleotide sequences[J].American Journal of Botany,1991,78:871-884.
[16] 鄢家俊,白史且,張新全,等.青藏高原東南緣老芒麥自然居群遺傳多樣性的SRAP和SSR分析[J].草業(yè)學(xué)報,2010,19(4):122-134.
[17] Zhang H Q,F(xiàn)an X,Sha L N,et al.Phylogeny of Hystrixand related genera(Poaceae:Triticeae)based on nuclear rDNA ITS sequences[J].Plant Biology,2008,10:635-642.
[18] 王曉麗,凡星,張春,等.用nrDNA ITS序列探討小麥族含StH染色體組物種的系統(tǒng)發(fā)育[J].草業(yè)學(xué)報,2009,18(6):82-90.
[19] Sha L N,F(xiàn)an X,Yang R W,et al.Phylogenetic relationships between Hystrixand its closely related genera(Triticeae;Poaceae)based on nuclear Acc1,DMC1and chloroplast trnL-F sequences[J].Molecular Phylogenetics and Evolution,2010,54:327-335.
[20] Liu Z P,Chen Z Y,Pan J,et al.Phylogenetic relationships in Leymus(Poaceae:Triticeae)revealed by the nuclear ribosomal internal transcribed spacer and chloroplast trnL-F sequences[J].Molecular Phylogenetics and Evolution,2008,46:278-289.
[21] Mason-Gamer R J,Orme N L,Anderson C M.Phylogenetic analysis of North American Elymus and the monogenomic Triticeae(Poaceae)using three chloroplast DNA data sets[J].Genome,2002,45:991-1002.
[22] Mason-Gamer R J.Reticulate evolution,introgression,and intertribal gene capture in an allhexapolid grass[J].Systematic Biology,2004,53:25-37.
[23] 張春,凡星,莎莉娜,等.基于葉綠體trnL-F序列對廣義披堿草屬物種的系統(tǒng)進(jìn)化研究[J].草業(yè)學(xué)報,2011,20(3):162-173.
[24] Nishikawa T,Salomon B,Komatsuda T,et al.Molecular phylogeny of the genus Hordeumusing three chloroplast DNA sequences[J].Genome,2002,45:1157-1166.
[25] Doyle J J,Doyle J L.A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J].Phytochemical Bulletin,1987,19:11-15.
[26] Xia X,Xie Z.DAMBE:Software package for data analysis in molecular biology and evolution[J].Journal of Heredity,2001,92:371-373.
[27] Huelsenbeck J P,Ronquist F R.MrBayes:Bayesian inference of phylogenetic trees[J].Bioinformatics,2001,17:754-755.
[28] Posada D,Crandall K A.Modeltest:testing the model of DNA substitution[J].Bioinformatics,1998,14:817-818.
[29] Zhang H Q,Zhou Y H.Meiotic pairing behavior reveals differences in genomic constitution between Hystrix patulaand other species of the genus Hystrix Moench(Poaceae:Triticeae)[J].Plant Systematic and Evolution,2006,258:129-136.
[30] Fan X,Zhang H Q,Sha L N,et al.Phylogenetic analysis among Hystrix,Leymus and its affinitive genera(Poaceae:Triticeae)based on the sequences of a gene encoding plastid acetyl-CoA carboxylase[J].Plant Science,2007,172:701-707.
[31] Zhang H Q,F(xiàn)an X,Sha L N,et al.Phylogeny of Hystrixand related genera(Poaceae:Triticeae)based on nuclear rDNA ITS sequences[J].Plant Biology,2008,10:635-642.
[32] 黃燕,張海琴,劉靜,等.利用RAPD特異標(biāo)記分析東北猬草染色體組成[J].西北植物學(xué)報,2009,29:1538-1543.
[33] Wen J.Evolution of eastern Asian and eastern North American disjunct pattern in flowering plants[J].Annual Review of E-cology and Systematics,1999,30:421-455.
[34] Liu Q L,Ge S,Tang H B,et al.Phylogenetic relationships in Elymus(Poaceae:Triticeae)based on the nuclear ribosomal internal transcribed spacer and chloroplast trnL-F sequences[J].New Phytologist,2006,170:411-420.
[35] Yen N,Asamoah-Odei N,Sun G L.Maternal origin,genome constitution and evolutionary relationships of polyploid Elymus species and Hordelymus europaeus[J].Biologia Plantarum,2011,55:68-74.
[36] Zhang C,F(xiàn)an X,Yu H Q,et al.Different maternal genome donor to Kengyiliaspecies inferred from chloroplast trnL-F sequences[J].Biologia Plantarum,2009,53:759-763.
[37] Wu Z Y,Peter H R,Hong D Y.Flora of China Illustrations[M].Beijing:Science Press,2006:386-394.
[38] Hulten E,F(xiàn)ries M.Atlas of North European Vascular Plants[M].Konigstein:Koeltz Scientific Books,Germany,1986:140.

