鴨茅品種的SCoT遺傳變異分析
蔣林峰,張新全 ,黃琳凱,馬嘯,嚴德飛,胡強,付玉鳳
(1.四川農(nóng)業(yè)大學(xué)動物科技學(xué)院草業(yè)科學(xué)系,四川雅安625014;2.重慶市墊江縣畜牧生產(chǎn)站,重慶408300)
目標起始密碼子多態(tài)性(start codon targeted polymorphism,SCoT),是基于SRAP(sequence-related amplified polymorphism,相關(guān)擴增多態(tài)性)[1]的一種新型目的基因分子標記技術(shù),其產(chǎn)生的顯性多態(tài)性偏向候選功能基因區(qū)[2]。主要依據(jù)植物基因中ATG翻譯起始位點側(cè)翼序列的保守性而設(shè)計[3-4]。另外,較高退火溫度的設(shè)計有助于減少假陽性擴增的可能性[5]。與其他標記相比,SCoT標記結(jié)合了ISSR(inter simple sequence repeat,簡單重復(fù)序列間區(qū))[6-7]和RAPD(randomly amplified polymorphic DNA,隨機擴增多態(tài)性DNA)的優(yōu)點,具引物通用性,穩(wěn)定性好,重復(fù)性好等優(yōu)點,同時能有效產(chǎn)生相關(guān)性狀多態(tài),更好地反映物種遺傳多樣性和親緣關(guān)系[8]。自2009年開發(fā)以來,已有其用于葡萄(Vitis vinifera)[9]、芒果(Mangifera indica)[10]、花生(Arachis hypogaea)[11]、土豆(Solanum tuberosum)[12]等農(nóng)作物和植物的相關(guān)研究報道。但就牧草研究領(lǐng)域,目前僅有SCoT標記用于苜蓿(Medicago sativa)[4]種質(zhì)遺傳分析的少量報道。
鴨茅(Dactylis glomerata)為禾本科(Poaceae)早熟禾亞科(Pooideae)鴨茅屬(Dactylis)多年生疏叢型禾草[13-14],是鴨茅亞種中重要的同源四倍體栽培種[15],起源于歐洲、北非和亞洲溫帶地區(qū)[16],因草質(zhì)柔嫩、葉量豐富、耐蔭性強、適口性好等優(yōu)點,在世界各地被廣泛應(yīng)用于飼草栽培和干草制作[17],是溫帶和亞熱帶地區(qū)重要優(yōu)良牧草。我國是鴨茅起源地之一,已發(fā)現(xiàn)野生鴨茅生長地約26個[18],可分為二倍體(2n=2x=14)、四倍體(2n=4x=28)及稀有的六倍體(2n=6x=42)3種類型[19],近年來,鴨茅大量應(yīng)用于我國草地畜牧業(yè)及生態(tài)建設(shè),取得了良好的經(jīng)濟和生態(tài)效益。
到目前,我國審定登記鴨茅品種僅為8個,且只有“寶興”、“川東”、“古藺”3個品種通過野生栽培馴化而來,其余5個皆為引進品種,國內(nèi)鴨茅品種遺傳基礎(chǔ)相對狹窄,引進品種普遍表現(xiàn)出在當(dāng)?shù)乜剐缘汀⑦m應(yīng)性差等缺點,難以滿足我國生態(tài)治理和畜牧業(yè)發(fā)展的需求。采用不同的育種方法,綜合選育利用我國豐富的鴨茅種質(zhì)資源已經(jīng)刻不容緩。而縱觀全球,國外通過不同的選育目標和育種方式已獲得近500個鴨茅品種,品種適應(yīng)性強。采用不同的技術(shù)手段開展鴨茅種質(zhì)遺傳研究,對于鴨茅育種具有重要意義。同時,國內(nèi)外學(xué)者對鴨茅的研究已從形態(tài)學(xué)[20]、細胞學(xué)[21]、同工酶[22]深入到分子水平。分子標記因不受環(huán)境影響、效率快、分辨率高等優(yōu)點被廣泛應(yīng)用。謝文剛等[23]利用SSR(simple sequence repeat,簡單重復(fù)序列)對11份鴨茅種質(zhì)的110個單株遺傳分析表明,我國西南地區(qū)鴨茅種質(zhì)遺傳多樣性豐富,其遺傳變異主要存在于種質(zhì)內(nèi)和地理區(qū)域內(nèi);Peng等[24]利用AFLP(amplified fragment length polymorphism,擴增片段長度多態(tài)性)對32份鴨茅分析表明,鴨茅遺傳多樣性與染色體倍性、地理分布等顯著相關(guān);萬剛等[25]對23份鴨茅二倍體、四倍體SSR研究表明,四倍體較二倍體遺傳多樣性更為豐富,是鴨茅品種選育的優(yōu)良材料。這些研究主要集中于鴨茅野生材料、栽培馴化品種等的研究,但對于鴨茅國內(nèi)栽培馴化品種與引進品種的遺傳變異對比研究,國內(nèi)外均未有報道。了解我國現(xiàn)有鴨茅栽培馴化品種的遺傳背景和變異,有助于保證我國鴨茅種質(zhì)資源的合理利用和豐富我國鴨茅野生馴化栽培馴化品種的遺傳多樣性。
本研究首次利用SCoT標記技術(shù)對來自世界4大洲的32個鴨茅品種進行遺傳變異分析,比較栽培馴化品種與引進品種之間的遺傳差異,以期為進一步加快我國鴨茅育種進程和針對性提高鴨茅品種生態(tài)適應(yīng)能力及利用價值等提供理論依據(jù)。
1 材料與方法
1.1 試驗材料
供試材料為32個鴨茅品種,包括8個國內(nèi)栽培馴化品種(系)和24個國外引進品種,均為四川農(nóng)業(yè)大學(xué)草業(yè)科學(xué)系從國內(nèi)及國外采集和收集而來(表1),于2012年9月種植于四川農(nóng)業(yè)大學(xué)雅安草業(yè)科學(xué)試驗基地。
1.2 方法
1.2.1 DNA提取 每份種質(zhì)隨機選取25個單株的幼嫩葉片等量混合,采用Doyle等[26]的CTAB(cetyl trimethyl ammonium bromide,十六烷基三甲基溴化銨)法提取DNA,并用1%瓊脂糖凝膠電泳和紫外分光光度計分別檢測其DNA的純度和濃度,合格的樣品保存于-20℃低溫冰箱,實驗開始時,取出部分按照其測得濃度稀釋至10 ng/μL,4℃冰箱保存?zhèn)溆谩?/p>
1.2.2 SCoT引物篩選 所用80個SCoT引物由澳大利亞Collard和Mackill[3](SCoT1~SCoT36)及中國廣西農(nóng)業(yè)大學(xué)Luo等[5](SCoT37~SCoT80)提供,引物由上海生工生物技術(shù)有限公司合成。PCR所用Mix混合液(含有10×PCR buffer、Mg2+、dNTPs)和Taq酶均購自天根科技生化公司。采用田間形態(tài)差異較大的4個品種,即栽培馴化品種“寶興”、引進品種“安巴”、“Chinook”和新品系“YA01472”,對引物進行預(yù)先篩選,從中選取擴增條帶清晰且重復(fù)性較好的22個SCoT引物,用于供試32個鴨茅品種的進一步PCR擴增(表2)。
1.2.3 SCoT-PCR擴增 本實驗PCR程序參考植物水稻(Oryza sativa)[3]和牧草苜蓿[4]并有所改動,擴增體系優(yōu)化為15 μL:包括DNA 模板1 μL(10 ng/μL),引物1.5 μL(10 pmol/μL),Mix混合液7.5 μL,Taq酶0.4 μL(2.5 U/μL),ddH2O 4.6 μL。PCR 反應(yīng)程序為94℃預(yù)變性3 min;94℃變性50 s,50℃退火1 min,72℃延伸2 min,36個循環(huán);72℃延伸5 min,最后冷卻至4℃保存。PCR擴增產(chǎn)物在含0.05μL/mL Gelred(10000×水溶液)的1.8%瓊脂糖凝膠中電泳,待溴酚藍電泳至凝膠尾端約1 cm時停止電泳,約需2.5~3.0 h。電泳完畢觀察,再用凝膠成像系統(tǒng)拍照。
1.2.4 數(shù)據(jù)分析 SCoT是顯性標記,對獲得的清晰可重復(fù)的DNA條帶進行統(tǒng)計,在相同遷移位置上將穩(wěn)定出現(xiàn)的條帶的有或無賦值為1和0進行統(tǒng)計,構(gòu)成原始數(shù)據(jù)矩陣。利用軟件Excel 2007和POPGENE 1.31[27]計算多態(tài)性條帶百分比(percentage of polymorphic bands,PPB),Nei氏遺傳多樣性指數(shù)(Nei’s gene diversity,H)[28]和Shannon信息多樣性指數(shù)(Shannon’s information index of diversity,I)。利用WINAMOVA 1.55對國內(nèi)栽培馴化品種與引進品種的遺傳變異進行分子方差分析(analysis ofmolecular variance,AMOVA)[29]。POPGENE和AMOVA數(shù)據(jù)輸入文件均由軟件 DCFA1.1[30]生成。利用軟件 FreeTree[31]基于 Nei-Li[32]遺傳相似系數(shù)(genetic similarity,GS)的不加權(quán)成對群算術(shù)平均法(unweighted pair-group method with arithmetic averages,UPGMA)計算各品種的遺傳距離(genetic distance,GD),按公式GD=1-GS計算。采用Dendroscope 3[33]構(gòu)建各品種的聚類樹,利用NTsyspc V2.1[34]進行鴨茅品種的主成分分析(principal coordinate analysis,PCoA)。
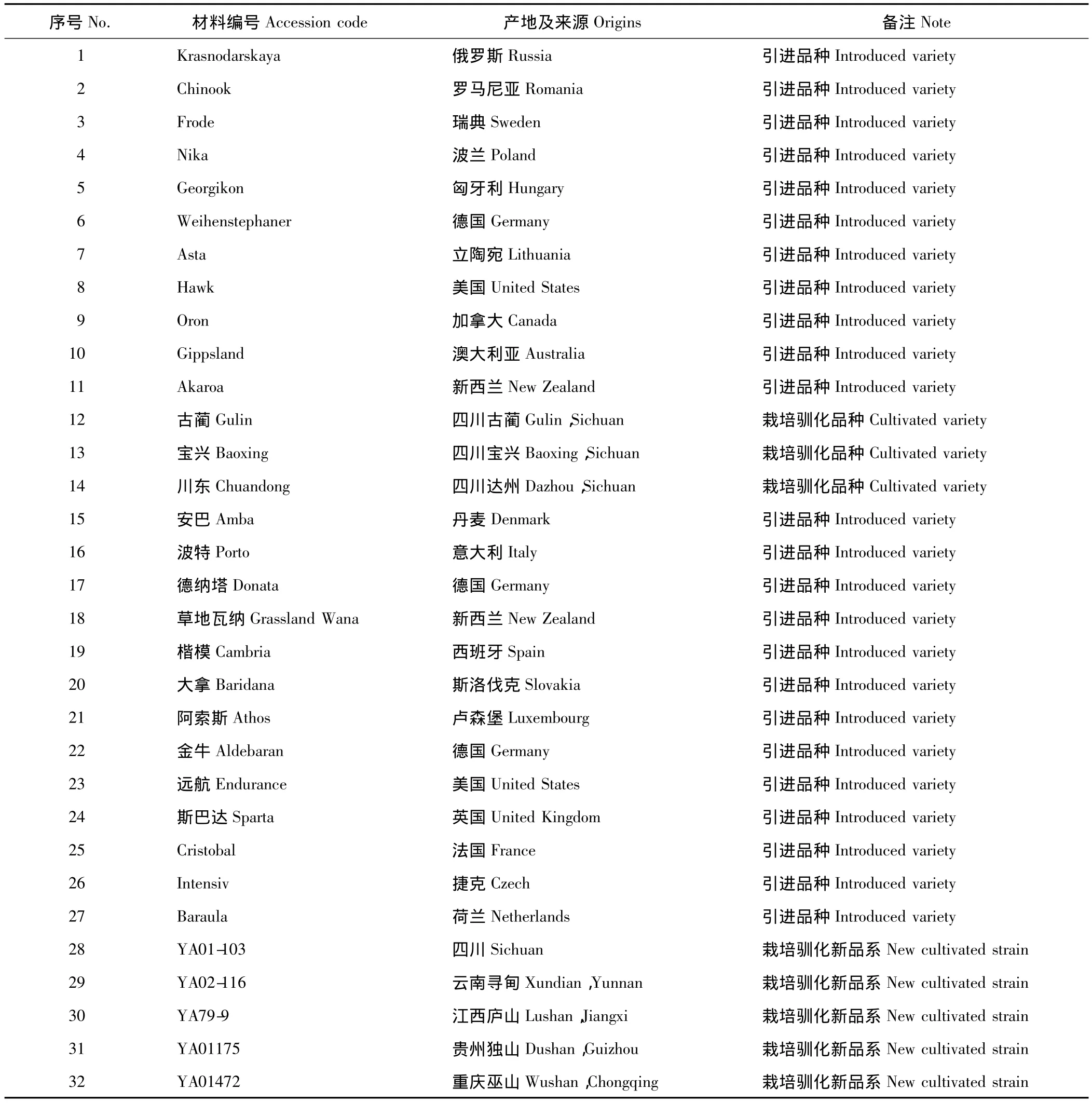
表1 供試鴨茅材料名稱及來源Table 1 Names and sources of D.glomerata used in this study
2 結(jié)果與分析
2.1 SCoT標記多態(tài)性分析
22個引物共擴增出308條清晰可辨條帶,擴增片段大小在250~2000 bp,大部分集中在250~1500 bp(圖1),平均每個引物擴增條帶數(shù)為14條,條帶變化為9條(SCoT8,SCoT9和SCoT20)到19條(SCoT40),其中,多態(tài)性譜帶245條,平均每個引物擴增多態(tài)性條帶數(shù)為11.14條,多態(tài)性比率(PPB)為79.55%,多態(tài)性條帶變化為5條(SCoT9,SCoT11和SCoT12)到18條(SCoT65),表明SCoT標記能檢測到較多的遺傳位點,獲得多態(tài)性較好的PCR結(jié)果(表3)。
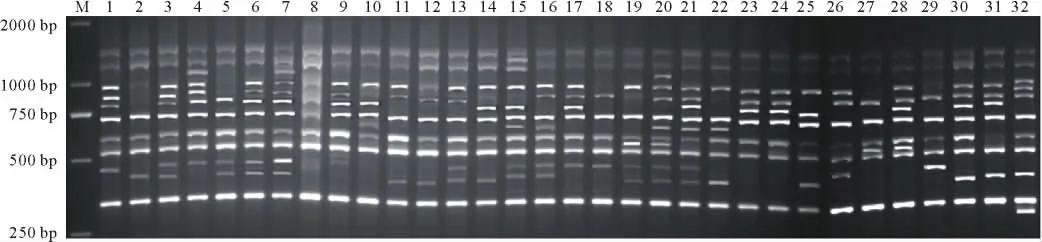
圖1 引物SCoT 23對鴨茅品種的擴增結(jié)果Fig.1 Am plified resultsw ith primer No.SCoT 23 on orchardgrass varieties
2.2 供試鴨茅品種遺傳距離(GD)分析
根據(jù)0,1原始數(shù)據(jù)表征矩陣,利用軟件Freetree[31]基于Nei-Li遺傳相似系數(shù)得到供試鴨茅品種的遺傳距離,用于分析其相互之間的親緣關(guān)系。32份鴨茅品種遺傳距離范圍為0.0251~0.3157,平均遺傳距離為0.1916,其中來自法國的Cristobal和來自捷克的Intensiv遺傳距離最大,表明其之間的親緣關(guān)系最遠,來自匈牙利的Georgikon和來自波蘭的Nika遺傳距離最小,表明其親緣關(guān)系最近。總體上遺傳距離的差異呈現(xiàn)了不同供試鴨茅品種來源地的分布差異,品種間遺傳差異較大。將所有品種按照育種背景劃分為國內(nèi)栽培馴化品種和國外引進品種,可以進一步分析2種選育背景下不同鴨茅品種之間的遺傳分化和差異,結(jié)果發(fā)現(xiàn)栽培馴化品種內(nèi)部的遺傳距離范圍為0.0601~0.1802,平均遺傳距離為0.1242,其中“寶興”和“川東”遺傳距離最小,其都為國審栽培馴化品種,“YA01472”和“YA02-116”遺傳距離最大,其為鴨茅栽培馴化新品系,表明目前國內(nèi)登記的鴨茅栽培馴化品種間遺傳相似度較高,新品系“YA01472”和“YA02-116”遺傳多樣性較已經(jīng)登記的栽培馴化品種(“寶興”、“古藺”、“川東”)有所提高;而引進品種內(nèi)部的遺傳距離范圍為0.0251~0.3157,平均遺傳距離為0.1952,可見國內(nèi)栽培馴化品種的遺傳多樣性水平低于引進品種。
2.3 鴨茅類群的遺傳變異比較分析
將32份鴨茅品種劃分為栽培馴化品種類群和國外引進品種類群,比較他們之間的遺傳變異。采用軟件AMOVA 1.55[29]分析其類群之間和之內(nèi)的方差、方差分量及貢獻率(表 4),采用 POPGENE version 1.32[27]分析其類群的遺傳多樣性和反應(yīng)其類群的等位基因豐富度和均勻程度(表5)。分子方差分析(AMOVA)結(jié)果表明,16.32%的遺傳變異存在于兩類群之間,兩類群之內(nèi)的遺傳變異較高,占有83.68%,2種品種類群之間和之內(nèi)的差異均極顯著(P<0.001)。此外,供試鴨茅品種類群間的表型預(yù)測(Фst)值為0.1630,也說明遺傳結(jié)構(gòu)的變異主要存在于品種類群之內(nèi)。由表5可知,供試鴨茅品種基因多樣性指數(shù)為0.2646,Shannon指數(shù)為0.3983。其中引進品種的多態(tài)性條帶(NPB),多態(tài)性比率(PPB),基因多樣性指數(shù)(H),Shannon指數(shù)(I)都高于栽培馴化品種,基本代表了鴨茅物種的整體遺傳水平,與2.2中分析的遺傳距離分析結(jié)果相符合,說明鴨茅引進品種較目前國內(nèi)普遍存在的栽培馴化品種有更為豐富的遺傳多樣性和變異水平,這可能是國外品種選育方法和選育材料較為多元化的結(jié)果。
2.4 基于Nei-Li遺傳相似系數(shù)的聚類分析
利用Freetree[31]軟件計算出供試各鴨茅品種間的Nei氏遺傳一致度和遺傳距離的無偏估計值,然后用Dendroscope 3[33]繪制 UPGMA 聚類圖。總體看來,供試32份鴨茅品種主要可分為6類(圖2)。類群I有8個品種(系),包括來自中國的3個栽培馴化品種(“寶興”、“古藺”、“川東”)和 5個野生馴化新品系(“YA79-9”、“YA01-103”、“YA02-116”、“YA01175”、“YA01472”);類群II包括3個品種,即來自大洋洲的“Gippsland”、“草地瓦納”和“Akaroa”;類群 VI包括 8個品種,即來自歐洲的“Georgikon”、“Nika”、“Weihenstephaner”、“金牛”、“德納塔”、“大拿”、“Asta”和“Intensiv”;類群 III包括來自南美洲的 3個品種(“Hawk”、“遠航”、“Oron”)及來自歐洲的 6 個品種(“Krasnodarskaya”、“Frode”、“安巴”、“波特”、“斯巴達”、“阿索斯”);其余2個類群(IV和V)包括了來自歐洲的其余供試鴨茅品種。由聚類分析可知,目前國內(nèi)已經(jīng)登記的鴨茅栽培馴化品種間具有一定的遺傳相似度,遺傳距離較近,3個品種(“古藺”、“寶興”、“川東”)育成登記時間分別為1995,1999和2003年,可能由當(dāng)時較為簡單的選育方法、相似的生態(tài)地理環(huán)境等原因?qū)е拢缕废怠癥A02-116”較之前栽培馴化品種有所改善,但遺傳變異仍較為狹窄,這與前面的分析結(jié)果一致,可能是由于不同生境材料混合采樣及選育過程中混合選擇等原因;而反觀國外選育的品種,其大多數(shù)為綜合品種,其同一來源的品種間存在較大的遺傳距離和遺傳差異,但總體上供試品種的聚類與地理分布存在一定的相關(guān)性,且其品種繼承了更為豐富的遺傳多樣性和更為一致的群體一致性,利于品種的推廣和應(yīng)用。
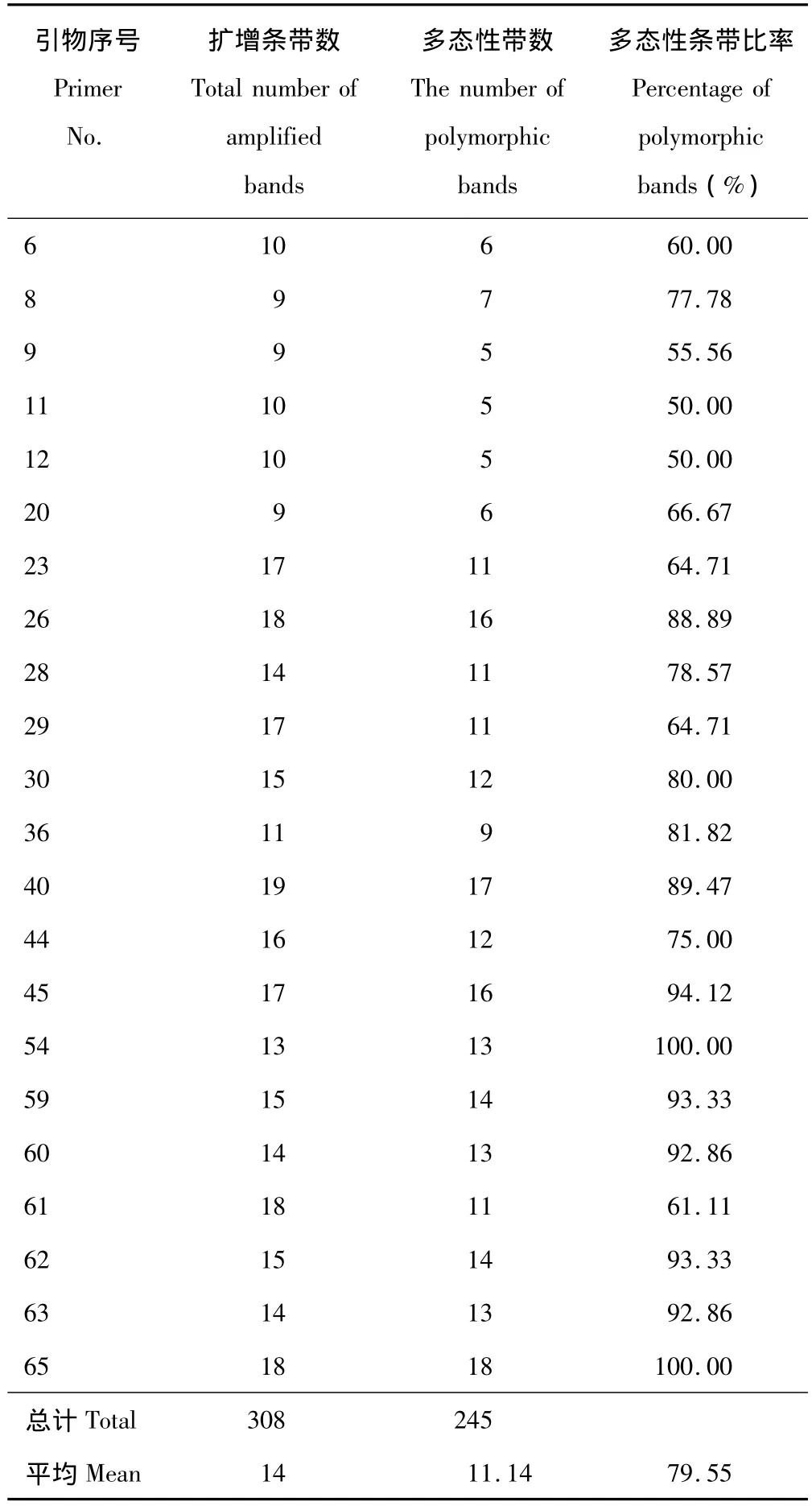
表3 SCoT標記擴增結(jié)果Table 3 Amp lified results of SCoT markers
2.5 主成分分析
主成分分析(PCoA)可作為種質(zhì)聚類分析結(jié)果的驗證和對比,同時各種質(zhì)在主成分分析二維或三維圖上的遠近關(guān)系可以更為直觀的反映它們之間的親緣關(guān)系和遺傳差異。利用原始矩陣數(shù)據(jù)基于遺傳相似系數(shù)(GS),用NTsys-pc V2.1[34]軟件對供試32份鴨茅品種進行主成分分析,前3個主成分所能解釋的遺傳變異為46.08%,并根據(jù)第一、第三主成分進行作圖(圖3)。主成分結(jié)果與聚類分析結(jié)果基本一致,整體上顯示出國內(nèi)的栽培馴化品種(系),尤其是3個國審鴨茅栽培馴化品種間親緣關(guān)系較近,遺傳豐富度和遺傳變異水平較引進品種稍差,通過栽培馴化品種和引進品種遺傳變異的對比,反思目前國內(nèi)普遍使用的選育手段是否具有代表性、品種遺傳一致度是否較好等問題,對于今后我國的鴨茅新品種選育工作和加快鴨茅育種進程具有一定的指導(dǎo)意義。

表4 鴨茅品種分子變異方差分析(AMOVA)Table 4 Analysis ofmolecular variance(AMOVA)for variety of orchardgrass
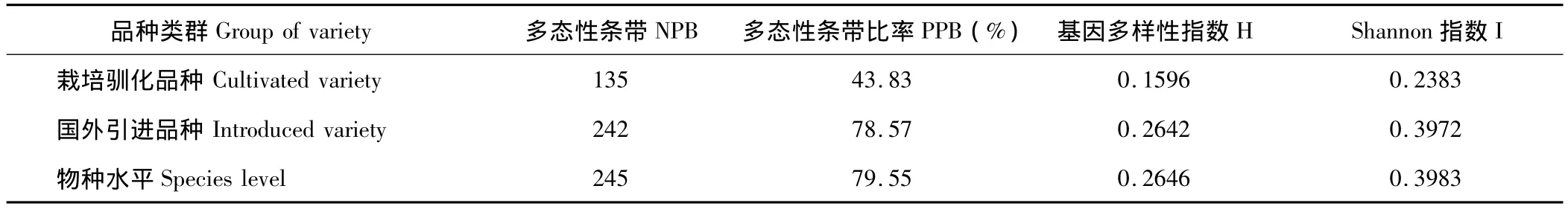
表5 2種品種類群遺傳多樣性指數(shù)Table 5 Genetic diversity index of two varieties groups
3 討論
3.1 SCoT標記的多態(tài)性
SCoT標記是一種基于翻譯起始位點(translation initiation site,TIS)的目標分子標記新技術(shù),具有操作簡單、引物通用性、多態(tài)性高、成本低、重復(fù)性好等優(yōu)點[2],其較多的基因組基因形成了較多的引物結(jié)合位點,且其單引物的設(shè)計,使得那些較近而又反向結(jié)合的引物結(jié)合位點之間的片段得以有效擴增。擴增不確定性介于外顯子與內(nèi)含子之間,大幅度地提升了其引物的多態(tài)性[8]。較其他分子標記(AFLP,ISSR,SSR,SRAP,RAPD等),其與功能基因相關(guān),更能反應(yīng)相關(guān)功能性狀多態(tài)[11],是用于農(nóng)作物、水果和牧草等植物種質(zhì)資源鑒定、保護和利用,高密度遺傳連鎖圖譜構(gòu)建,分子標記輔助育種,基因定位和克隆等研究的非常有效的一種分子標記。
本研究首次將SCoT標記技術(shù)應(yīng)用于鴨茅品種的DNA多態(tài)性鑒定,其能在鴨茅品種資源中檢測出較豐富的遺傳多態(tài)性,這與鴨茅品種資源間具有較豐富的表型性狀多態(tài)性相一致。擴增平均多態(tài)性條帶為11.14條,而謝文剛等[23]使用SSR標記對來自中國西南地區(qū)鴨茅的遺傳多樣性研究多態(tài)性條帶為6.8條,Guo等[9]采用SCoT標記對64份葡萄品種研究的多態(tài)性條帶為7.7條,可見SCoT標記能夠產(chǎn)生較多的多態(tài)位點用于鴨茅遺傳變異研究。另外,其多態(tài)性位點率為79.55%,而Luo等[5]采用SCoT標記對來自中國的50份芒果種質(zhì)研究的多態(tài)性為76.19%,Luo等[35]采用ISSR標記對23份芒果種質(zhì)分析的多態(tài)性為55.77%,熊發(fā)前等[8]對花生屬采用與功能基因相關(guān)的SCoT研究多態(tài)性為31.80%,韓國輝等[2]對柑橘(Citrus)SCoT研究多態(tài)性為54.80%,由此可知,SCoT標記可以作為研究鴨茅遺傳多樣性的有效手段,其得到的分子標記極有可能是功能基因的一部分,是進一步開發(fā)特定功能基因標記和建立分子標記輔助育種技術(shù)體系的基礎(chǔ)。
3.2 鴨茅栽培馴化品種與引進品種遺傳差異
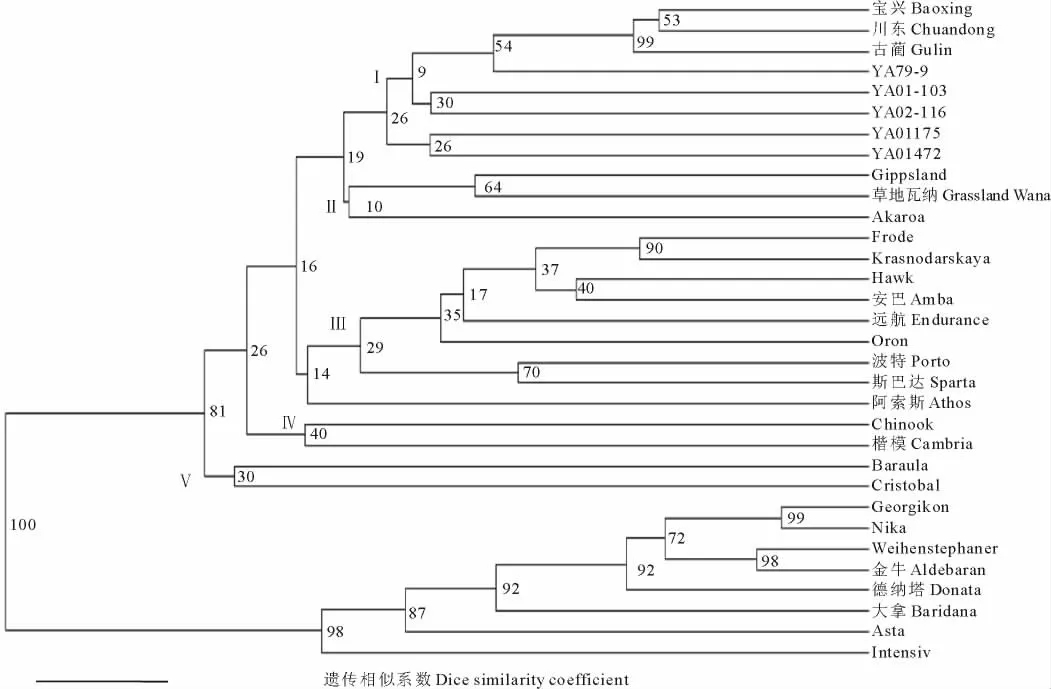
圖2 SCoT標記對32份鴨茅品種親緣關(guān)系聚類圖Fig.2 Dendrogram of the relationship UPGMA cluster of 32 orchardgrass varieties based on SCoT markers
本研究采用SCoT標記技術(shù)開展鴨茅栽培馴化品種與引進品種的遺傳變異比較,研究表明,國內(nèi)鴨茅栽培馴化品種平均遺傳距離為0.1242,多態(tài)性條帶為135條,基因多樣性指數(shù)為0.1596,Shannon指數(shù)為0.2383,而鴨茅引進品種的遺傳多樣性從以上幾個水平都遠高于栽培馴化品種,基本代表了鴨茅物種的遺傳變異水平。從側(cè)面揭示了目前國內(nèi)鴨茅品種遺傳基礎(chǔ)狹窄的現(xiàn)狀。通過分子方差分析表明,鴨茅栽培馴化品種和引進品種2個類群的遺傳變異主要還是分布于類群內(nèi)部,尤其是引進品種內(nèi)部的不同鴨茅品種間具有更為豐富的遺傳差異。聚類分析和主成分分析的結(jié)果基本一致,供試32個鴨茅品種主要可分為6類,其中來自國內(nèi)的8個栽培馴化品種聚為類群I,進一步印證了目前國內(nèi)鴨茅栽培馴化品種內(nèi)部遺傳變異較小的瓶頸。這與萬剛等[25]的研究結(jié)果一致,認為3個國內(nèi)栽培馴化品種(“寶興”、“川東”、“古藺”)間的遺傳變異較為狹窄,可能是緣于3個鴨茅栽培馴化品種采集地相似的自然氣候地理條件和較為傳統(tǒng)的育種方式等原因。
總的來說,就鴨茅栽培馴化品種和引進品種的遺傳分化而言,引進品種和國內(nèi)栽培馴化品種間差異較大,引進品種的遺傳多樣性更為豐富,而栽培馴化品種相對來說整齊度差,品種結(jié)實性差,適應(yīng)范圍小。究其原因,可能是國外鴨茅育種家在育種方法和育種材料的選擇上,較國內(nèi)科研單位更為先進和合理,其針對性強,工作延續(xù)性強,選育時間長,品種性狀更為穩(wěn)定。目前采用較多的是選擇在各方面性狀表現(xiàn)較為優(yōu)良的不同材料,讓其自由傳粉,經(jīng)過多代選擇和淘汰,并采取一定的育種手段,使優(yōu)良基因得到穩(wěn)定遺傳,這樣選育得到的品種具有較為豐富的遺傳多樣性和較大的生境適應(yīng)能力,及更為穩(wěn)定的群體一致性。
3.3 鴨茅新品種選育及其利用
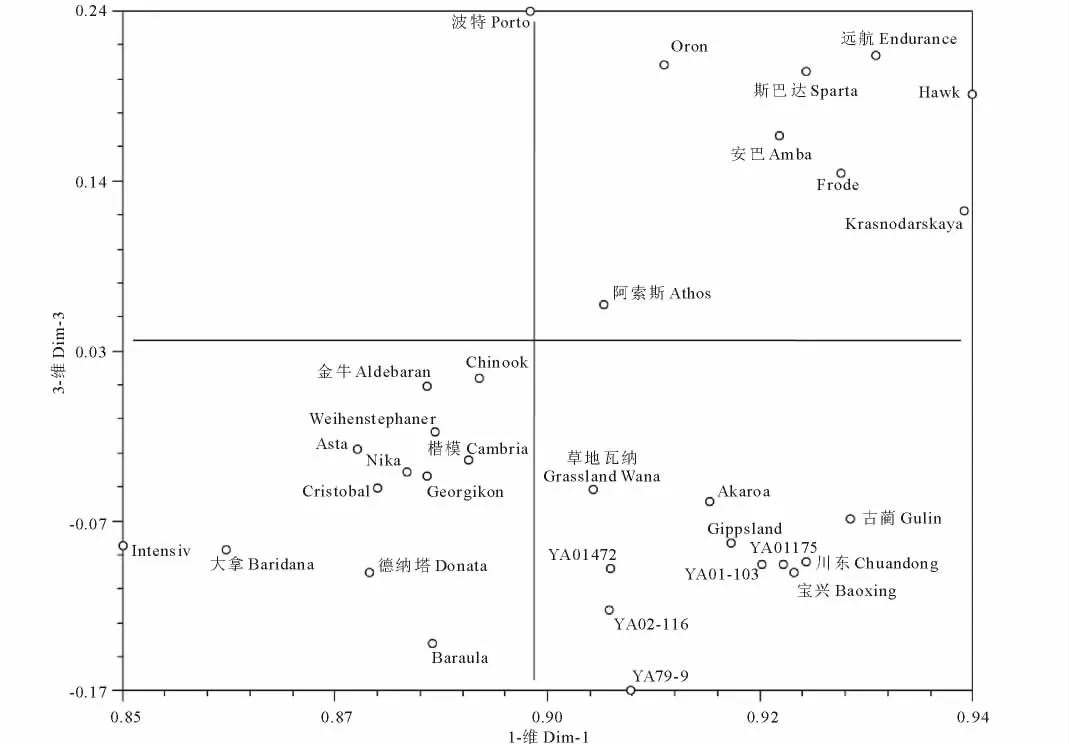
圖3 基于SCoT譜型的32份鴨茅品種主成分分析Fig.3 Principal component analysis based on SCoT patterns in 32 orchardgrass varieties
我國是鴨茅的主要起源地之一,具有包括二倍體、四倍體和六倍體的野生鴨茅分布地26個[18-19],大量的研究表明我國野生鴨茅種質(zhì)資源遺傳多樣性豐富。謝文剛等[23]對西南區(qū)鴨茅種質(zhì)研究也表明我國鴨茅野生種質(zhì)資源豐富,保持了較大的變異度和遺傳多樣性;萬剛等[25]對鴨茅栽培馴化品種與野生材料遺傳多樣性的SSR研究分析表明,我國目前推廣應(yīng)用的鴨茅栽培馴化品種少且遺傳基礎(chǔ)狹窄,而我國的野生鴨茅種質(zhì)資源具有豐富的遺傳多樣性,可通過開展相關(guān)的雜交篩選等工作,選育出能夠滿足我國不同生態(tài)條件下的鴨茅品種。Peng等[24]和鐘聲[20]的研究同樣也證明了上述結(jié)論。本研究表明,鴨茅引進品種基因多樣性指數(shù)等指標都高于栽培馴化品種。同時,目前國外已登記鴨茅品種達500個,在今后我國的鴨茅新品種選育策略上,應(yīng)更多的針對于不同倍性、抗病、抗旱、葉量、適應(yīng)性等特性進行雜交親本材料選擇,特別是在鴨茅易感的銹病等癥狀方面,選出在不同特性上表現(xiàn)突出和較弱的材料進行雜交,通過采用較為合理的育種方法,選育出適合我國不同生境生長的鴨茅新品種。另外,鴨茅為高度異花授粉植物[14],天然的雜交會引起品種內(nèi)部的分化,對于品種的有效隔離也是一個必不可少的保護措施。
[1]李杰勤,王麗華,詹秋文,等.20個黑麥草品系的SRAP遺傳多樣性分析[J].草業(yè)學(xué)報,2013,22(2):158-164.
[2]韓國輝,向素瓊,汪衛(wèi)星,等.柑橘SCoT分子標記技術(shù)體系的建立及其在遺傳分析中的應(yīng)用[J].園藝學(xué)報,2011,38(7):1243-1250.
[3]Collard BCY,Mackill D J.Start codon targeted(SCoT)polymorphism:A simple,novel DNAmarker technique for generating genetargeted markers in plants[J].Plant Molecular Biology Reporter,2009,27:86-93.
[4]何慶元,王吳斌,楊紅燕,等.利用SCoT標記分析不同秋眠型苜蓿的遺傳多樣性[J].草業(yè)學(xué)報,2012,21(2):133-140.
[5]Luo C,He X H,Chen H,et al.Analysis of diversity and relationships amongmango cultivars using start codon targeted(SCoT)markers[J].Biochemical Systematics and Ecology,2010,38:1176-1184.
[6]曾漢元,魏麟,劉鵬,等.能源草蘆竹遺傳多樣性的ISSR分析[J].草業(yè)學(xué)報,2013,22(3):266-273.
[7]曾亮,袁慶華,王方,等.冰草屬植物種質(zhì)資源遺傳多樣性的ISSR分析[J].草業(yè)學(xué)報,2013,22(1):260-267.
[8]熊發(fā)前,蔣菁,鐘瑞春,等.目標起始密碼子多態(tài)性(SCoT)分子標記技術(shù)在花生屬中的應(yīng)用[J].作物學(xué)報,2010,36(12):2055-2061.
[9]Guo D L,Zhang JY,Liu CH.Genetic diversity in some grape varieties revealed by SCoT analyses[J].Molecular Biology Reports,2012,39:5307-5313.
[10]Luo C,He X H,Chen H,etal.Genetic relationship and diversity of Mangifera indica L.:revealed through SCoT analysis[J].Genetic Resources and Crop Evolution,2012,59:1505-1515.
[11]Xiong FQ,Zhong R C,Han ZQ,etal.Start codon targeted polymorphism for evaluation of functional genetic variation and relationships in cultivated peanut(Arachis hypogaea L.)genotypes[J].Molecular Biology Reports,2011,38:3487-3494.
[12]Gorji A M,Poczai P,Polgar Z,etal.Efficiency of arbitrarily amplified dominantmarkers(SCOT,ISSR and RAPD)for diagnostic fingerprinting in tetraploid potato[J].American Journal of Potato Research,2011,88:226-237.
[13]Lindner R,Garcia A.Geographic distribution and genetic resources of Dactylis in Galicia(northwest Spain)[J].Genetic Resources and Crop Evolution,1997,44:499-507.
[14]Bushman B S,Larson SR,Tuna M,et al.Orchardgrass(Dactylis glomerata L.)EST and SSR marker development,annotation,and transferability[J].Theoretical and Applied Genetics,2011,123:119-129.
[15]Lumaret R.Cytology,genetics,and evolution in the genus Dactylis[J].Critical Reviews in Plant Sciences,1988,7:55-89.
[16]Sanada Y,Tamura K,Yamada T.Relationship between water-soluble carbohydrates in falland spring and vigor of spring regrowth in orchardgrass[J].Crop Science,2010,50:380-390.
[17]Jafari A,Naseri H.Genetic variation and correlation among yield and quality traits in cocksfoot(Dactylis glomerata L.)[J].Journal of Agricultural Science,2007,145:599-610.
[18]彭燕,張新全.鴨茅種質(zhì)資源多樣性研究進展[J].植物遺傳資源學(xué)報,2003,4(2):179-183.
[19]Stebbins G L,Zohary D.Cytogenetics and Evolutionary Studies in the Genus Dactylis L.Morphology,Distribution and Interrelationships of the Diploid Subspecies[M].California:University of California Press,1959,31:1-40.
[20]鐘聲.野生鴨茅雜交后代農(nóng)藝性狀的初步研究[J].草業(yè)學(xué)報,2007,16(1):69-74.
[21]張新全,杜逸,鄭德誠.鴨茅二倍體和四倍體PMC減數(shù)分裂,花粉育性及結(jié)實性的研究[J].中國草地,1996,(6):38-40.
[22]帥素容,張新全,白史且.不同倍性鴨茅同工酶比較研究[J].草業(yè)科學(xué),1998,(6):11-16.
[23]謝文剛,張新全,馬嘯,等.中國西南區(qū)鴨茅種質(zhì)遺傳變異的SSR分析[J].草業(yè)學(xué)報,2009,18(4):138-146.
[24]Peng Y,Zhang X Q,Deng Y L,et al.Evaluation of genetic diversity in wild orchardgrass(Dactylisglomerata L.)based on AFLP markers[J].Hereditas,2008,145:174-181.
[25]萬剛,張新全,劉偉,等.鴨茅栽培馴化品種與野生材料遺傳多樣性比較的SSR分析[J].草業(yè)學(xué)報,2010,19(6):187-196.
[26]Doyle J J,Doyle JL,Brown A H D.Analysis of a polyploid complex in glycine with chloroplast and nuclear DNA[J].Australian Systematic Botany,1990,3:125-136.
[27]Yeh F C,Yang R C,Boyle T.POPGENE VERSION 1.31.Microsoft Windows-based Freeware for Population Genetic Analysis.Quick User Guide[M].University of Alberta:Center for International Forestry Research,1999.
[28]Nei M.Analysis of gene diversity in subdivided populations[J].Proceedings of the National Academy of Sciences of the United States of America,1973,70:3321-3323.
[29]Excoffier L,Smouse PE,Quattro JM.Analysis ofmolecular variance inferred from metric distances among DNA haplotypes:application to human mitochondrial DNA restriction data[J].Genetics,1992,131(2):479-491.
[30]張富民,葛頌.群體遺傳學(xué)研究中的數(shù)據(jù)處理方法I.RAPD數(shù)據(jù)的AMOVA分析[J].生物多樣性,2002,10:438-444.
[31]Pavlicek A,Hrda S,F(xiàn)legr J.FreeTree-free ware program for construction of phylogenetic trees on the basis of distance data and bootstrap/jackknife analysis of the tree robustness.Application in the RAPD analysis of the genus Frenkelia[J].Folia Biologica,1999,45(3):97.
[32]NeiM,LiW H.Mathematicalmodel for studying the genetic variation in terms of restriction endonucleases[J].Proceedings of the National Academy of Sciences of the United States of America,1979,76:5269-5273.
[33]Huson D H,Scornavacca C.Dendroscope 3:An interactive viewer for rooted phylogenetic trees and networks[J].Systematic Biology,2012,61:1061-1067.
[34]Rohlf F J.NTSYS-pc:Numerical Taxonomy and Multivariate Analysis System,version 2.1.User Guide[M].New York:Exeter Software,2000.
[35]Luo C,He X H,Chen H,etal.Genetic diversity ofmango cultivars estimated using SCoT and ISSRmarkers[J].Biochemical Systematics and Ecology,2011,39:676-684.

