利用微衛星標記分析釀酒酵母的遺傳多樣性
張留燕,黃英子,劉延琳,2,*
(1.西北農林科技大學葡萄酒學院,陜西 楊凌 712100;2.陜西省葡萄與葡萄酒工程中心,陜西 楊凌 712100)
利用微衛星標記分析釀酒酵母的遺傳多樣性
張留燕1,黃英子1,劉延琳1,2,*
(1.西北農林科技大學葡萄酒學院,陜西 楊凌 712100;2.陜西省葡萄與葡萄酒工程中心,陜西 楊凌 712100)
為研究本土釀酒酵母的種內多態性,以及本土野生酵母與商業酵母之間的差異性,本實驗采用4個微衛星標記(ScAAT1、YOR267C、C11、YPL009C)分析18株商業酵母和28株陜西涇陽分離的野生釀酒酵母的遺傳多樣性,利用PopGen32進行遺傳參數的分析。結果表明:4個位點共檢測出66個等位基因,每個位點等位基因數為16~18個;平均多態信息含量0.862 3,均為高多態位點;46株菌表現出39種基因型,分辨率為98.94%;觀測雜合度0.478 3~0.658 5。野生酵母和商業酵母均具有豐富的遺傳多樣性,兩個群體各具有自己獨特的等位基因,且兩者在聚類圖中有較清晰的界線。
微衛星;商業酵母;本土釀酒酵母;遺傳多樣性
釀酒酵母是葡萄酒釀造中起最主要作用的微生物。它不僅決定葡萄醪發酵的成敗,而且對葡萄酒最終產品的產量、質量及產品特色的形成都起到至關重要的作用。釀酒酵母的多樣性和獨特性,對葡萄酒的感官特性有重要貢獻[1]。采用分子技術對釀酒酵母進行分類鑒定及多樣性研究是開發和利用酵母菌資源的基礎,是選育優良釀酒酵母菌種的基礎和關鍵。
微衛星是DNA短串聯重復序列(1~6個堿基重復),表現出重復次數的變異,這些序列廣泛存在于真核生物、原核生物[2]和病毒[3]基因組中。微衛星表現出相同物種的個體之間實質水平的多態性,廣泛用于父權否定測試[4]、法醫學[5]和不同真核生物的分子分型,如葡萄品種[6]和病原酵母(Candida albicans)[7]等。微衛星標記具有保守性好、高度多態、共顯性遺傳等優點,經常在遺傳圖譜或群體遺傳的研究中用做遺傳標記[8]。
Perez等[9]在釀酒酵母的基因組DNA數據庫中搜索二核苷酸和三核苷酸串聯重復,從中選取10個位點進行分析,證明微衛星是一個分析釀酒酵母種群基因組的強大且靈活的工具。進而幾位作者提出用于釀酒酵母分型的微衛星位點組合[9-12]。多個研究表明,與隨機擴增多態性DNA標記(random amplified polymorphic DNA,RAPD)、限制片段長度多態性(restriction fragment length polymorphism,RFLP)、Interdelta等相比,微衛星表現出較優的分型效果[13-15]。采用微衛星標記對釀酒酵母的分布和種群結構塑造中地理、生態以及人類所發揮的作用進行研究的報道也越來越多[16-18]。目前采用微衛星標記對我國釀酒酵母的研究極其缺乏,本實驗采用4個微衛星位點對28株本土野生酵母和18株商業酵母進行基因分型,分析其遺傳多樣性,并對野生酵母和商業酵母兩個群體表現出來的差異進行討論,為研究我國酵母資源多樣性及酵母選育奠定理論基礎。
1 材料與方法
1.1 菌株與引物
1.1.1 供試菌株
供試菌株包括商業酵母18株(BDX、CLIB2047、T306、R-HST、K1、Lalvin、Syrah、RA17、71B、W15、GRE、QA23、D47,法國Lallemand公司;F10、X16、VL1,法國Lafford公司;XR,法國Lamothe-Abiet公司;DSM,德國微生物菌種保藏中心)和2009年陜西涇陽小白玫瑰葡萄發酵過程中分離的本土釀酒酵母28株(02y14、111y3、111y9、112y1、112y4、112y14、14w11、14w14、 173y1、173y4、173y7、173y8、173y10、173y15、174y1、174y5、174y15、181y1、181y2、181y3、181y4、181y6、181y10、182y5、182y12、23w12、23w13、23w15)。
1.1.2 微衛星引物
本實驗所采用的4個微衛星位點信息見表1。
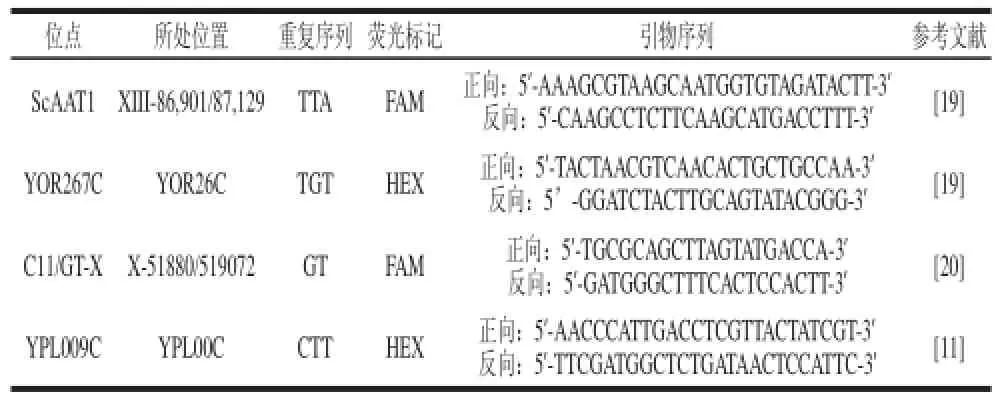
表1 微衛星引物位點基本信息Table 1 Characteristics of four microsatellite loci tested in this study
1.2 方法
1.2.1 基因組DNA提取
將菌株在YPD培養基上培養48 h后,采用石英砂破壁法提取DNA[21]。
1.2.2 基因組DNA的PCR擴增
PCR反應體系:總體積25μL,包含10mmol/L Tris-HCl,50mmol/L KCl,1.5mmol/L MgCl2,200μmol/L dNTP,0.5μmol/L引物,1.25U Taq DNA聚合酶,模板DNA 10~250 ng。
PCR擴增程序為:95℃、4 min;94℃、30 s,53℃、30 s,68℃、1 min,34個循環;68℃、10 min。
1.2.3 PCR產物檢測及分析
PCR產物經2%瓊脂糖凝膠電泳確認后,使用ABI 3730DNA測序儀以GeneScan ROX500作為內標進行毛細管電泳,GeneMapper v3.2分析PCR擴增產物大小。小數部分采取四舍五入的處理方法,在比較菌株的微衛星基因型計算等位基因時,1核苷酸的差異不被認為是不同的[22]。根據每個個體的條帶確定基因型,利用PopGen32軟件[23]統計微衛星基因座的等位基因數(number of alleles,Na)、等位基因頻率(frenquencies of alleles,Fa)、有效等位基因數(effective number of alleles,Ne)、觀測雜合度(observed heterozygosity,Ho)、期望雜合度(expected heterozygosity,He)、遺傳分化系數(coefficient of genetic differentiation,Fst)等。用NTsys軟件構建基于每對菌株的Jaccard相似性系數的非加權算術平均數(unw eighted pair group method with arithmetic mean,UPGMA)系統發育樹。
2 結果與分析
2.1 微衛星多態性和遺傳多態性
46株釀酒酵母ScAAT1、YOR267C、C11、YPL009C 4個微衛星位點分型共檢測到66個等位基因,在S288C基因組中擁有最長的TAA基元的ScAAT1,表現出最高的等位基因多樣性,檢測到18個等位基因。YOR267C、C11和YPL009C表現出稍微低一點的遺傳多態性,均有16個等 位基因。如果考慮較小和較大等位基因之間大小的差異,YOR267C有最顯著的188 bp的差異,這與前人研究相同[19]。ScATT1、YOR267C和C11最大和最小等位基因相差分別83、64、44 bp。野生酵母菌株111y9在ScAAT1和YPL009C兩個位點均未檢測到條帶,還有4株菌(174y15、181y2、181y4、181y6)在YPL009c位點未檢測到目的帶。4個位點的基因型數目分別為21、19、23、18種,46株菌共表現出39種基因型,分辨率[24]為98.94%。
ScAAT1、YOR267C、C11、YPL009C 4個位點多態信息含量分別為0.860 2、0.869 3、0.852 9、0.866 7。依PIC判斷標準[25]:PIC<0.25為低度多態,0.25≤PIC<0.5為中度多態,PIC≥0.5為高度多態,故4個位點均為高多態位點。
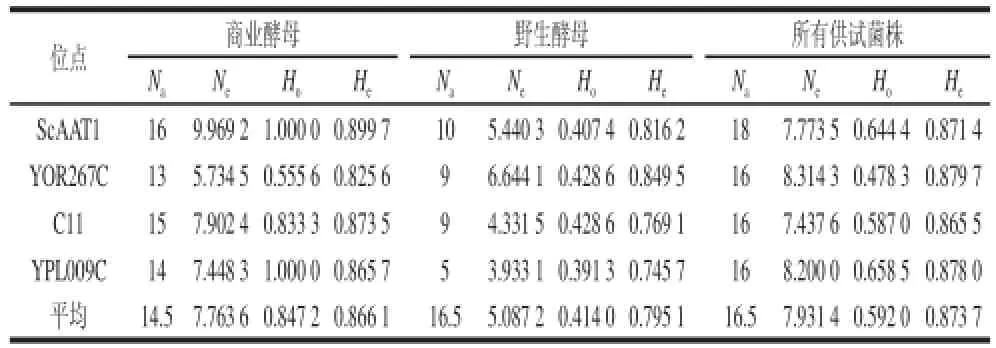
表2 46個供試菌株的多樣性分析結果Table 2 Statistical analysis for genetic diversity of 46 S. cereviase strains
如表2所示,商業酵母中,ScAAT1和YPL0 09C兩個位點雜合率高,YOR267C位點雜合率最低。野生酵母中,4個位點雜合度差異較小,與商業酵母相反,YOR267C和C11表現出雜合率稍高于另兩個位點,除C11外,3個位點的雜合度均高于Legras中[19]報道的雜合度。雜合度反應群體的遺傳一致性,雜合度越高,群體的遺傳一致性就越低,其遺傳多樣性越高,由此說明我國本土酵母包含遺傳信息的豐富程度較好。將所有菌株進行整體分析,ScAAT1和YPL009C兩個位點雜合率較高,4個位點平均雜合率為0.592 0。
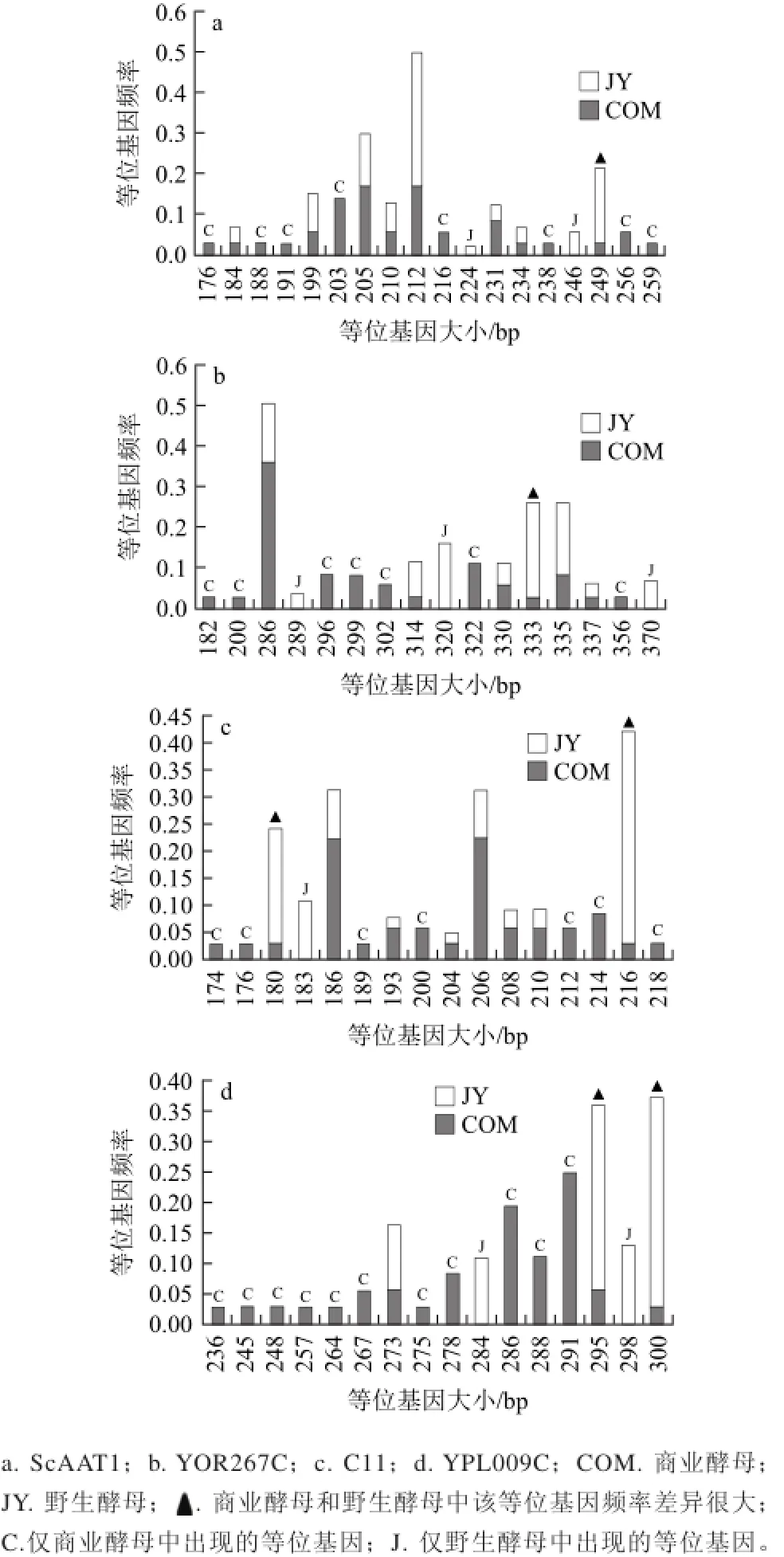
圖1 商業酵母和野生酵母4個微衛星位點的等位基因及等位基因頻率Fig.1 Alleles of four microsatellite loci and their frequencies in S. cerevisiae from commercial wine strains and indigenous yeasts
如圖1所示,每個位點均出現商業酵母和野生酵母群體特有的等位基因,商業酵母的特有等位基因較多,7~11個;野生酵母特有等位基因很少,1~3個。每個位點片段最小的一個等位基因均為商業酵母特有。如將等位基因橫軸從中分為兩部分,除二核苷酸重復C11位點外,野生酵母的等位基因傾向于出現在較大片段區;YOR267C和YPL009C位點商業酵母的特有等位基因傾向于分布在較小片段區域,ScAAT1位點較小片段區和較大片段區商業酵母特有基因數相等。等位基因頻率范圍從0.028~0.393。每個位點均出現有在商業酵母和野生酵母中表現差異較大的等位基因。每個位點每個群體中的等位基因分布不呈現明顯的規律,商業酵母YOR267C位點286 bp表現出較強的優勢,其他位點沒有主導的等位基因。
商 業酵母群體與野生酵母群體的遺傳距離無偏估計為0.828 3,遺傳一致性無偏估計[26]較低,為0.436 8,表明商業酵母與野生酵母兩者之間的遺傳差異明顯。4個位點的Fst值分別為0.025 8、0.047 3、0.066 3、0.091 2,較小,說明群體內部多樣性較高。
2.2 菌株的聚類分析
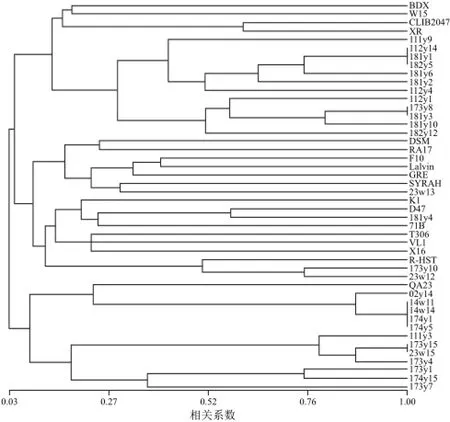
圖2 46株釀酒酵母菌的UPMGA聚類圖Fig.2 UPMGA dendrograms showinggenetic relationships among 46 yeast strains
運用NTsys進行采用UPMGA的遺傳多樣性分析,可將菌株劃分為3個群體如圖2所示。第一類12株野生酵母和1株來源于波爾多地區的商業酵母QA23,其中,173y1、174y15、173y73株菌具有相同的Interdelta基因型,微衛星基因型不同而得以區分,三者的親緣關系較近。第二類包括13株商業酵母和4株野生酵母,多數商業酵母和野生酵母與其同類聚在一起,有趣的是,181y4與來源于法國的D47關系較近,23w12、173y10與來源于奧地利的R-HST關系較近,23w12、173y10具有相同的Interdelta基因型B,且有3個微衛星位點的基因型相同。第三類包括4株商業酵母和12株野生酵母,兩者有清晰的界線,分為2支,12株野生酵母中有5株Interdelta基因型相同為M[27],4株Interdelta基因型相同為J,說明Interdelta基因型相同的菌株的微衛星聚類親緣關系也較近。
3 討 論
一些菌株在一個位點出現2個條帶,可以認定這些菌株在這個位點是二倍體且是雜合的。當一個菌株在所有位點都表現出一條帶時,那么這個菌株的倍性不能被確定。26株野生酵母中有10株4個位點中至少有一個是雜合的。本實驗所采用的微衛星標記與前人研究報道中的等位基因數的差別主要是由于研究群體的不同而引起的[20,28]。14w11、14w14、174y1、174y5這4株菌表現出相同的基因型;還有菌株112y14、181y1、182y5的基因型相同;菌株173y8和181y3的基因型相同;菌株23w15和173y15基因型相同。這些基因型相同的菌株,可能是同一株菌,也有可能是所選用的4個位點不足以將其區分開,需要進一步分析。然而這些菌株中有些菌株的Interdelta基因型不同,一些能被Interdelta PCR鑒別的菌株的微衛星基因型卻不能被區分開,這在前人的研究中也有報道[29]。
野生酵母的特有等位基因較少,原因可能為本實驗中的酵母來源于同一產區同一葡萄品種的發酵過程,如果將野生酵母菌株擴大到不同產區不同葡萄品種,應該能得到更豐富的遺傳多態性。商業酵母來源較為廣泛,表現出遺傳多樣性較豐富。對特有等位基因的分析可以認為,商業酵母特有等位基因傾向于較小的片段,野生酵母的特有等位基因傾向于較大的片段。
本研究表明ScAAT1、YOR267C、C11、YPL009C 4個微衛星位點多態性高,可作為釀酒酵母遺傳多樣性的有效遺傳標記。46株釀酒酵母菌共檢測出66個等位基因,39種基因型,商業酵母和本土酵母遺傳多樣性豐富,并具有明顯差異。本研究為釀酒酵母的多樣性研究及優良菌株選育奠定了基礎。
[1] CIANI M, MANNAZZU I, MARINANGELI P, et al. Contribution of winery-resident Saccharomyces cerevisiae strains to spontaneous grape must fermentation[J]. Antonie van Leeuwenhoek, 2004, 85(2): 159-164.
[2] FIELD D, WILLS C. Abundant microsatellite polymorphism in Saccharomyces cerevisiae, and the different distribution in eight prokaryotes and S. cerevisiae, result from strong mutation pressures and a variety of selective forces[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(4): 1647-1652.
[3] SREENU V B, RANJITKUMAR G, SWAMINATHAN S, et al. MICAS: a fully automated web server for microsatellite extraction and analysis from prokaryote and viral genomic sequences[J]. Applied Bioinformatics, 2003, 2(3): 165-168.
[4] HELMINEN P, LOKKI M L, EHNHOLM C, et al. Application of DNA “fingerprints” to paternity determinations[J]. Lancet, 1988, 331(8585): 574-576.
[5] HAGELBERG E, GRAY I C, JEFFREYS A J. Identification of the skeletal remains of a murder victim by DNA analysis[J]. Nature, 1991, 352: 427-429.
[6] THIS P, JUNG A, BOCCACCI P, et al. Development of a standard set of microsatellite reference alleles for identi cation of grape cultivars[J]. Theoretical and Applied Genetics, 2004, 109: 1448-1458.
[7] BOTTEREL F, DESTERKE C, COSTA C, et al. Analysis of microsatellite markers of Candida albicans used for rapid typing[J]. Journal of Clinical Microbiology, 2001, 39: 4076-4081.
[8] WEISSENBACH J, GYAPAY G, DIB C, et al. A second-generation linkage map of the human genome[J]. Nature, 1992, 359: 794-801.
[9] PEREZ M A, GALLEGO F J, MARTINEZ I, et al. Detection, distribution and selection of microsatellites (SSRs) in the genome of the yeast Saccharomyces cerevisiae as molecular markers[J]. Letters in Applied Microbiology, 2001, 33: 461-466.
[10] GALLEGO F J, PEREZ M A, MARTINEZ I, et al. Microsatellites obtained from database sequences are useful to characterize Saccharomyces cerevisiae strains[J]. American Journal of Enology and Viticulture, 1998, 49: 350-351.
[11] HENNEQUIN C, THIERRY A, RICHARD G F, et al. Microsatellite typing as a new tool for identification of Saccharomyces cerevisiae strains[J]. Journal of Clinical Microbiology, 2001, 39: 551-559.
[12] GONZALEZ T A, JUBANY S, CARRAU F M, et al. Differentiation of industrial wine yeast strains using microsatellite markers[J]. Letters in Applied Microbiology, 2001, 33: 71-75.
[13] SCHULLER D, VALERO E, DEQUIN S, et al. Survey of molecular methods for the typing of wine yeast strains[J]. FEMS Microbiology Letters, 2004, 231(1): 19-26.
[14] POSTERARO B, SANGUINETTI M, ROMANO L, et al. Molecular tools for differentiating probiotic and clinical strains of Saccharomyces cerevisiae[J]. International Jounal of Food Microbiology, 2005, 103(3): 295-304.
[15] OLIVEIRA V A, VICENTE M A, FIETTO L G, et al. Biochemical and molecular characterization of Saccharomyces cerevisiae strains obtained from sugar-cane juice fermentations and their impact in Cachaca production[J]. Applied and Environmental Microbiology, 2008, 74(3): 693-701.
[16] LEGRAS J L, MERDINOGLU D, CORNUET J M, et al. Bread, beer and wine: Saccharomyces cerevisiae diversity reflects human history[J]. Molecular Ecology, 2007, 16: 2091-2102.
[17] GODDARD M R, ANFANG N, TANG R Y, et al. A distinct population of Saccharomyces cerevisiae in New Zealand: evidence for local dispersal by insects and human-aided global dispersal in oak barrels[J]. Environmental Microbiology, 2010, 12(1): 63-73.
[18] ZHANG H Y, LEE S A, BRADBURY J E, et al. Yeasts isolated from New Zealand vineyards and wineries[J]. Australian Journal of Grape and Wine Research, 2010, 16(3): 491-496.
[19] LEGRAS J L, RUH O, MERDINOGLU D, et al. Selection of hypervariable microsatellite loci for the characterization of Saccharomyces cerevisiae strains[J]. International Journal of Food Microbiology, 2005, 102: 73-83.
[20] JUBANY S, TOMASCO I, de LEON I P, et al. Toward a global database for the molecular typing of Saccharomyces cerevisiae strains[J]. FEMS Yeast Research, 2008, 8: 472-484.
[21] 周小玲, 沈微, 饒志明, 等. 一種快速提取真菌染色體DNA的方法[J]. 微生物學通報, 2004(4): 89-92.
[22] RICHARDS K D, GODDARD M R, GARDNER R C. A database of microsatellite genotypes for Saccharomyces cerevisiae[J]. Antonie van Leeuwenhoek, 2009, 96: 355-359.
[23] YEH F C, YANG R C, BOYLE T. POPGENE, the user-friendly shareware for population genetic analysis[DB/OL]. Canada: Molecular Biology and Biotechnology Centre, University of Alberta, 1997[2013-01-10]. http://www.ualberta.ca/~fyeh/.
[24] HUNTER P R. Reproducibility and indices of discriminatory power of microbial typing methods[J]. Journal of Clinical Microbiology, 1990, 28: 1903-1905.
[25] BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3): 314-331.
[26] NEI M. Estimation of average heterozygosity and genetic distance from a small number of individuals[J]. Genetics, 1978, 89(3): 583-590.
[27] 張靜. 自然發酵和接種發酵的酵母菌群生態研究及對葡萄酒質量的影響[D]. 楊凌: 西北農林科技大學, 2010.
[28] 馮敏, 王春曉, 劉延琳. 利用微衛星多態性揭示釀酒酵母菌株遺傳多樣性[J]. 中國農業科學, 2012, 45(12): 2537-2543.
[29] AYOUB M J, LEGRAS J L, SALIBA R, et al. Application of multilocus sequence typing to the analysis of the biodiversity of indigenous Saccharomyces cerevisiae wine yeasts from Lebanon[J]. Journal of Applied Microbiology, 2006, 100: 699-711.
Genetic Diversity Analysis of Saccharomyces cerevisiae Strains by Microsatellite Marker Technique
ZHANG Liu-yan1, HUANG Ying-zi1, LIU Yan-lin1,2,*
(1. College of Enology, Northwest A&F University, Yangling 712100, China; 2. Shaanxi Engineering Research Center for Viti-viniculture, Yangling 712100, China)
In order to analy ze the genetic diversity of indigenous wine yeasts and the differences between indigenous isolates and commercial wine yeast strains, genetic variability of 28 isolates from Jingyang, Shaanxi Province and 18 commercial strains was analyzed using four microsatellite markers (ScAAT1, YOR267C, C11 and YPL009C). Meanwhile, genetic parameter s were also analyzed by PopGen32. Forty-six strains were amplified by four microsatellite prime pairs, and generated a total of 66 polymorphic bands with 16–18 alleles for each locus. The average polymorphism information content (PIC) of all strains at four loci was 0.862 3, suggesting that all the four SSR loci were hypervariable. Forty-six strains gave 39 different patterns with a discriminatory power of 98.94%. The heterozygosity was observed as 0.478 3–0.658 5. These results indicate that indigenous wine yeasts and commercial yeasts have high levels of genetic diversity, and both have unique alleles. Both kinds of strains have clear clustering boundary in the phylogentic tree.
microsatellite; commercial yeasts; indigenous wine yeast; genetic diversity
Q93.331
A
1002-6630(2014)01-0130-04
10.7506/spkx1002-6630-201401025
2013-01-22
國家自然科學基金項目(31271917);西北農林科技大學基本科研業務費項目(22050205);國家現代農業(葡萄)產業技術體系建設專項(CARS-30-jg-3)
張留燕(1988—),女,碩士研究生,研究方向為釀酒微生物。E-mail:forever7365@163.com
*通信作者:劉延琳(1966—),女,教授,博士,研究方向為葡萄、葡萄酒及釀酒微生物。E-mail:yanlinliu@nwsuaf.edu.cn

