單鏈環狀DNA添加劑抑制基因表達序列分析Ditag聚合酶鏈反應引物多聚體生成
劉玉慧,劉星,劉學東,鄭冬
(東北林業大學,黑龍江哈爾濱 150040)
單鏈環狀DNA添加劑抑制基因表達序列分析Ditag聚合酶鏈反應引物多聚體生成
劉玉慧,劉星,劉學東,鄭冬*
(東北林業大學,黑龍江哈爾濱 150040)
[目的]研究利用單鏈環狀DNA抑制聚合酶鏈反應(PCR)副產物的作用效果。[方法]設計并合成單鏈環狀DNA,并以具同樣序列發卡狀結構的單鏈DNA為對照組,在基因表達序列分析(SAGE)Ditag PCR反應中加入上述不同濃度的DNA,利用電泳技術檢測不同結構(環狀/單鏈)以及不同濃度單鏈DNA對PCR多聚體附產物的抑制作用。[結果]發卡狀結構的單鏈DNA無法抑制PCR引物多聚體副產物的產生;而在70~150 nmol/L終濃度范圍內,單鏈環狀DNA可以抑制SAGE Ditag PCR反應中引物多聚體的生成,并且不影響SAGE不同tag間的表達豐度差異。[結論]單鏈環狀DNA可以作為PCR反應中引物多聚體生成的有效抑制劑。
單鏈環狀DNA(sscDNA);系列基因表達分析(SAGE);引物多聚體;抑制
隨著基因測序技術的不斷完善和發展,許多模式物種和高等動物(如人)的基因組全序列的信息被一一破譯。然而,基因組計劃的研究成果只能從結構水平說明基因的組成特征和序列信息;從功能水平來如何解讀這些基因調控和相互作用成為后基因組時代一個亟待解決的問題[1]。例如,人大約有30 000個基因,但是大約只有20%的基因在同一時間被表達。基因表達的時空性已經引起廣泛的關注。針對此問題,各種組學(Omics)水平的技術方法和理論應運而生的。其中從轉錄水平闡明在特定時間和空間節點中基因組表達網絡的轉錄組學是近年來一個熱點研究領域[2]。
目前常用的研究基因差異表達的試驗方法有很多種。常見的有mRNA差顯RTPCR法(mRNA differential display RT-PCR,mRNA DDRT-PCR)、消減雜交(subtractive hybridization)等[3]。然而這些方法信息容納量不足,僅能反映有限基因的表達水平,不能很好地揭示那些大量未知基因的表達水平。1995年美國Johns Hopkins大學的Velculescu等首先提出了系列基因表達分析(Serial analysis of gene expression,SAGE)技術,用于基因轉錄表達譜的分析。SAGE技術[4]的出現是組學研究上的一個重要里程碑,該技術能同時定量分析實驗對象的成千上萬個轉錄本,克服了以前很多技術存在的缺陷,還能很靈敏地檢測出那些低豐度表達的基因。SAGE技術已經從純粹的基因差異表達研究拓展到重要基因功能研究、醫學基礎理論研究、疾病的診斷和治療等很多領域,并取得了諸多顯著的成果[5]。
眾所周知,理論上一個9個堿基的短核苷酸序列標簽能夠分辨262 144個不同的轉錄物(49),而人類基因組估計僅能編碼約80 000種轉錄物,因此一個SAGE tag包含有足夠的信息能夠確認唯一一種轉錄物。在實際運用中,SAGE技術始終在不斷改進和發展。以其中Long SAGE為例,其采用特定的錨定酶能獲得約17 bp的SAGE標簽(tag)來特異性地確定mRNA轉錄物,然后利用酶將每2個tag連接形成ditag,進行PCR擴增后,再將酶切后的將多個ditag標簽(20~60個)隨機串聯形成大量的多聯體(concatemer)并克隆到載體中;經測序并應用SAGE軟件分析,可確定表達的基因種類,并可根據標簽出現的頻率確定基因的表達豐度[6]。由于SAGE是一個高度敏感系統,可用于低豐度序列的確定。目前該技術已經廣泛應用于很多疾病相關新基因的發現,以及細胞或組織的基因表達譜的研究。
近年來,試驗嘗試利用SAGE技術研究鹿茸再生過程中基因表達網絡的時空變化動態。在試驗過程中發現,對SAGE ditag進行PCR擴增過程中有大量引物多聚體出現。由于PCR擴增SAGE ditag是SAGE技術的關鍵環節,通過這一步既要保證各tag能反映原始表達豐度的差異,同時還要獲得足夠量的DNA用于下游酶切反應;而引物多聚體對實現上述實驗目標產生了極大地干擾。Brownie等曾研究通過設計新型引物來抑制PCR引物多聚體試驗方案[7]。由于這將涉及對Ditag兩端接頭序列進行較大改動,而且需要2次PCR擴增,這顯然不符合SAGE技術中關于保持表達豐度的基本原則。但是這個技術方法有一個啟示:既然具有發卡結構的引物能夠增加PCR目標產物的特異性合成,能否設計具有發卡結構的單鏈DNA作為添加劑來抑制PCR引物多聚體的產生呢?筆者針對這個設想開展試驗研究,以期為該技術的進一步開發利用提供依據。
1 材料與方法
1.1 材料 SV Total RNA Isolation System,購自Promega公司;Long SAGE文庫構建試劑盒,購自Invitrogen公司;SYBR Premix ExTaq試劑盒、Agarose ms-6、Agarose LM SIEVE,Ex taq和hot-start taq DNA polymerase,均購自Takara公司;單鏈DNA寡聚核苷酸(C1:GCGGGCGGCG CAAAAAAGCG CCGCCCGC和C2:CCCGCAAAAA AGCGGGCGGC GCAAAAAAGC GCCG)添加物、STAT1引物(S1:TGGGGCACAA GGTGACAG和S2:TCACTCTTCTGTGTTCACTTACACTTCA)以及人GAPDH實時定量PCR引物(HG1:AGAAGGCTGG GGCTCATTTG和HG2:AGGGGCCATC CACAGTCTTC),均由上海英駿公司合成與純化,其中C1通過復性形成發卡結構;C2通過連接形成單鏈環狀結構。
1.2 鹿茸上皮組織總RNA的提取 取鹿茸新鮮上皮組織30mg,放入1.5 ml離心管中。根據SV Total RNA Isolation System試劑盒試驗手冊,加入RNA Lysis Buffer后,將組織勻漿,進行總RNA的提取。用紫外分光光度計測定RNA在260和280 nm的OD值,計算濃度與純度。取其中10μl總RNA和15μl甲酰胺65℃、5 min處理后迅速放到冰上冷卻2 min。再利用1.2%的瓊脂糖凝膠電泳分析總RNA的完整性。將獲得的總RNA分裝,保存于-70℃備用。其他總RNA保存于-70℃備用。
1.3 SAGE文庫Ditag制備 根據Long SAGE文庫構建試劑盒相關試驗手冊。取100μl oligo(dT)磁珠置于1.5 ml離心管中,在磁座上對oligo(dT)磁珠進行清洗2次后,將準備好的100μg RNA與磁珠進行充分混合。隨后室溫靜置30 min,在磁座上吸附2 min,移去上清液;再用1×First Strand Buffer清洗4次,去除上清液;然后加入87μl cDNA一鏈合成液(18.0 μl 5 × First Strand Buffer;1.0 μl RNaseOUT;54.5 μl DEPC 水;9.0 μl0.1mol/LDTT;4.5 μl dNTPMix),37 ℃溫育1 h進行cDNA第1鏈的合成。接著加入660μl cDNA 2鏈合成液(465μl DEPC Water;150μl 5×Second Strand Buffer;15 μl dNTPMix;5 μlE.coliDNA Ligase;20 μlE.coliDNA Polymerase;5μlE.coliRNase H),16℃溫育反應2 h后置于冰上,并加入45μl0.5mol/L EDTA終止反應。接著加入750μlWash Buffer C 75℃溫育12 min,滅活E.coliDNA Polymerase。在磁座上靜置2min,移去上清液,用Wash Buffer D洗脫4次。最后,吸去上清,加入200μl 1×Buffer 4,搖勻。
在磁座上靜置2 min后,去除上清,將磁珠懸浮于200μl NlaⅢ酶切溶液中(172μl LoTE;2μl 100×BSA;20μl 10×Buffer 4;6μl NlaⅢ),37℃溫育1 h后,將反應液在磁座上靜置2min,吸去上清液,用750μlWash Buffer C洗脫并滅活酶的活性,再用Wash Buffer D洗脫磁珠4次。用150μl 1×Ligase Buffer洗脫2次,隨后用100μl 1×Ligase Buffer懸浮磁珠,等量分成A、B管。
將上述2管溶液靜置在磁座上2 min,小心移去上清液,逐步加入接頭連接液(1.5μl Adapter A或B(20 ng/μl);14.0 μl LoTE;2.0 μl 10×Ligase Buffer);50 ℃加熱2 min,室溫冷卻15 min,放置于冰上。隨后加入2.5μl T4 DNA連接酶,16℃溫浴2 h。每1管都分別用500μlWash Buffer D洗脫4次,再用1×Buffer4洗2次,將磁珠懸浮在1×Buffer4溶液中。
將上面得到的2管溶液靜置在磁座上2min后移去上清液。加入含有標簽酶的反應液(70μl LoTE;10μl 100×SAM;10μl10×Buffer4);37℃溫浴2min,平衡反應液,加入10μl標簽酶MmeI,37℃溫浴1 h,反應結束后,在磁座上靜置2 min,小心吸取上清液,分別保存于新的2個離心管中。利用酚-氯仿及乙醇沉淀法處理所得到的樣品,分別用4μl LoTE溶解沉淀。將上述2管溶液混合,加入連接酶及反應液,37℃溫浴30 min后。利用酚/氯仿及乙醇沉淀法處理所得到的Ditag,最后的沉淀顆粒溶解在14μl LoTE溶液中。
1.4 SAGE Ditag的擴增 將連接產物40倍作為PCR擴增模板,進行Ditag PCR反應。試驗每個反應體系(50μl)組成均如下:5 μl 10 × PCR buffer,3 μl DMSO,5 μl dNTP,0.5 UTaqDNA聚合酶,SAGE上下游引物各2μl。其他反應成分如不同濃度單鏈DNA添加劑和DNA或cDNA模板因試驗不同目的不同均為1μl或用等體積水補足。PCR反應條件為:2min,95 ℃進行變性;94 ℃,30 s;55 ℃,1 min;70 ℃,1 min;共計27個循環進行擴增;最后70℃延伸5min。所有試驗均設陰性和/或陽性對照組。PCR擴增產物用4%瓊脂糖凝膠電泳檢測。
2 結果與分析
2.1 單鏈環狀DNA添加劑可抑制引物多聚體 試驗使用SAGE文庫構建試劑盒中的引物進行ditag擴增時,引物可自身產生2個以上的清楚可見的多聚體DNA帶,雖然使用試劑盒附帶的熱啟動DNA聚合酶,但是無法抑制上述副產物的產生(圖1A),這將影響ditag目的帶的擴增效率。試驗發現,其中一條引物多聚體帶與真正的130 bp ditag帶非常接近,對于ditag帶切膠回收具有干擾。合成單鏈DNA中,具有環狀結構的C2對于引物多聚體具明顯抑制作用,且呈濃度依賴型。結果顯示,其抑制效用在終濃度75~150 nmol/L最佳(圖1B);而具發卡結構的C1對于引物多聚體均無明顯抑制效果(圖1C)。
2.2 單鏈環狀DNA C2對DNA聚合酶濃度的影響 以篩選出來的C2的最佳濃度為基準,利用已經成功建立的STAT1的基因擴增體系研究DNA聚合酶濃度與C2的相關性,結果顯示,無論是Hot-start DNA聚合酶還是普通DNA聚合酶,在含有C2的PCR體系中,最低終濃度不得少于0.025 U/μl(圖1D)。
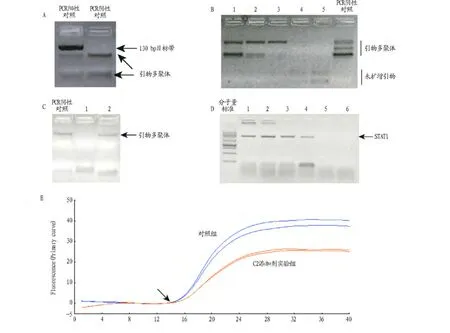
圖1 利用單鏈環狀DNA添加物抑制引物多聚體
2.3 單鏈環狀DNA C2對擴增產物產量的影響 利用實時定量PCR來研究單鏈環狀DNA C2是否會影響PCR擴增產物產量,通過比較含有或不含有C2的不同反應體系的Ct平均值,結果顯示兩者無顯著差異(圖1E),說明單鏈環狀DNA C2的添加對PCR產物影響不顯著(P>0.05)。
3 結論與討論
由于部分引物多聚體副產物與Ditag目的片段長度非常接近,不僅影響目的產物的產量,同時在經過DNA膠回收及后續處理步驟后,在導致測得的序列中將獲得大量的未知序列,有可能被誤判讀成新SAGE tag。引物多聚體的出現顯示,作為SAGE技術中最關鍵的環節,Ditag接頭序列和PCR引物在設計上存在很大的缺陷。
試驗設計的單鏈DNA添加物C1和C2主要序列和結構上非常相似,只是C2是一個封閉的單鏈環狀結構,而C1呈一端開放性的發卡結構。但是試驗顯示,發卡結構的單鏈DNA添加物C1無法抑制Ditag PCR中引物多聚體的產生;而C2對于引物多聚體的抑制呈現濃度依賴性的特點(圖1B、C)。上述結果對于多種普通或熱啟動DNA聚合酶具有一致性(數據未附)。雖然C2添加劑可以抑制引物副產物的產生,但是在試驗篩選出來的濃度范圍內,C2不會降低特異性產物的產量,這個結論利用定量PCR試驗給予了進一步證實。單鏈環狀DNA添加物C2不僅可以用于SAGE試驗,其實對于常規的PCR試驗中引物副產物的抑制也有作用,但是其最佳使用終濃度需要進一步篩選和優化。
目前,用于PCR反應的DNA聚合酶種類和來源有很大不同,但是在結構具有相似性[8-11]。試驗認為引物多聚體的產生是伴隨PCR試驗、卻同時獨立于其他目的片段擴增的過程,僅與引物和聚合酶有關。因此,從僅含有DNA聚合酶和引物的陰性對照試驗就可以發現,這也為研究引物多聚體的產生提供了一個優良的體系。試驗雖然發現了C2添加物抑制PCR引物多聚體的現象,但是對其機理還是不甚清楚。一個可能的解釋是,C2能夠優先有效地競爭性占據DNA聚合酶上對于引物多聚體形成具有關鍵性的功能域,從而抑制引物多聚體的形成。其具體分子機理尚需要進一步研究。
[1]KLOPFLEISCH R,GRUBER A D.Transcriptome and Proteome Research in Veterinary Science:What Is Possible andWhat Questions Can Be Asked?[J].The Scientific World Journal,2012,2012:25496 -254975.
[2]SCHUSTER S C.Next-generation sequencing transforms today’s biology[J].NatMethods,2008,5(1):16 -8.
[3]李靖,孔祥銀.基因表達系列分析技術的新進展[J].生物工程學報,2001,17(6):613 -616.
[4]VELCULESCU V E,ZHANG L,VOGELSTEIN B,et al.Serial analysis of gene expression[J].Science,1995,270(5235):484 -487.
[5]HORANM P.Application of serial analysisof geneexpression to the study of human genetic disease[J].Hum Genet,2009,126(5):605 -614.
[6]PATINOW D,MIAN O Y,HWANG PM.Serial analysis of gene expression:technical considerations and applications to cardiovascular biology[J].Circ Res,2002,91(7):565 -569.
[7]BROWNIE J,SHAWCROSS1 S,THEAKER J,et al.The elimination of primer-dimer accumulation in PCR[J].Nucleic Acids Research,1997,25(16):3235-3241.
[8]STEITZ T A.DNA Polymerases:Structural diversity and common mechanisms[J].JBiol Chem,1999,274(25):17395 -17398.
[9]BEESE L S,DERBYSHIRE V,STEITZ T A.Structure of DNA polymerase IKlenow fragment bound to duplex DNA[J].Science,1993,260(5106):352-355.
[10]EOM S,WANG J,STEITZ T A.Structure of Taq polymerasewith DNA at the polymerase active site[J].Nature,1996,382(6588):278 -281.
[11]LIY,MITAXOV V,WAKSMAN G.Structure-based design of Taq DNA polymeraseswith improved properties of dideoxynucleotide incorporation[J].Proc Natl Acad Sci USA,1999,96(17):9491 -9496.
Using Single Strand Circular DNA Additive to Inhibit Primer PolymersGenerated during Ditag PCR of Serial Analysisof Gene Expression
LIU Yu-hui,ZHENG Dong et al (Northeast Forestry University,Harbin,Heilongjiang 150040)
[Objective]To investigate the hypothesis thatusing single strand DNA asadditive to preventmispriming productof PCR.[Method]We designed and synthesized two kinds of single strand DNA additives;one was circular and another hairpin-like.Different concentrations of each kind of DNA additiveswere added into Ditag PCR systems of Long Serial Analysis of Gene Expression.Final productswere used for gel electrophoresis to screen DNA additives structure-or concentration-dependent inhibition of PCR primer polymer.[Results]Single strand circular buthairpin-like DNA additiveswas able to inhibit PCR primer polymer;the optimal final concentration was between 70 and 150 nmol/L,and single strand circular DNA additiveswill notaffect differential expression of SAGE tags.[Conclusion]Single strand circular DNA can be used as effective inhibitor to prevent primer polymers in PCR amplification.
Single strand circular DNA(sscDNA);Serial analysis of gene expression(SAGE);Primer polymers;Inhibition
S188
A
0517-6611(2014)15-04576-03
黑龍江省博士后科研啟動資助金(項目編號:LBH-Q07009);中央高校基本科研業務費專項資金(項目編號:2572014EA05)。
劉玉慧(1989-),女,黑龍江哈爾濱人,碩士研究生,研究方向:分子細胞生物學。*通訊作者,教授,從事分子細胞生物學研究。
2014-04-29

