不同引物擴增對東海陸架沉積物真核微型生物多樣性的影響*
俞凱成 王健鑫① 陶 詩 劉明華 蔣 然 劉雪珠 黃 備
(1.浙江海洋學院海洋微生物分子生態與應用實驗室 舟山 316022;2.浙江省舟山海洋生態環境監測站 舟山 316021)
海洋沉積物覆蓋了超過地球表面2/3的面積,是海洋生態系統的重要組成部分,更是地球上主要的微生物棲息地(Whitmanet al,1998)。由于海底沉積物的特殊地理環境,其中的微生物往往具有一些適應其環境的形態學、生理學、遺傳學上的特異性(戴欣等,2001),同時沉積物微生物還在整個海洋生態系統的生物地球化學循環過程中發揮著重要的作用(K?steret al,2008),因此了解海洋沉積物微生物群落結構和多樣性有著重要的意義。
東海是西太平洋構造活動帶中一個大型邊緣海,處于 21°54′—33°17′N,117°05′—131°03′E 之間,是西太平洋溝-弧-盆體系的典型發育地區(李家彪,2008)。東海不僅有著多樣的水文條件、顯著的物理化學梯度、顯著的季節差異,同時有很高的初級生產力,是碳和其它物質在全球生物地球化學循環的重要場所,也是研究海底各種沉積作用和微生物群落多樣性的有利場所(王健鑫等,2012)。東海陸架沉積環境微生物多樣性的研究多集中在細菌(宋志剛等,2006;Zenget al,2007;王健鑫等,2012)和古菌(張林寶等,2010)等原核生物,對于真核微型生物多樣性的研究較少(Parket al,2008),同時分析方法也比較單一,導致對于東海沉積環境真核微型生物缺乏系統科學的了解。
在真核微型生物分子多樣性研究中,18S rDNA是最常使用的靶標基因之一,既可針對保守區進行引物設計,又可根據可變區來區分不同種真核生物的差異(汪岷等,2009);另外核糖體基因間隔區(ITS區)也是常用的靶標基因,由于其進化速率較快,被認為比較適合在較小的分類階元層次進行鑒定和多樣性分析(Rodríguezet al,2005)。在對真核微型生物分子多樣性進行研究時,應根據各個靶標基因的特點來綜合分析,以增加結果準確性,雖然針對陸生環境已有部分不同引物擴增效果的比較研究(Andersonet al,2003),但在海洋沉積環境中不同引物擴增微型真核生物的相關比較研究尚未涉及。
本文以東海陸架DH-13站點沉積物為研究對象,選擇三對真核微型生物的通用引物(NS1/NS4、EF3/EF4、ITS1/ITS4),通過 18S rDNA、ITS區的基因克隆和文庫構建,進行分類學、多樣性和系統發育分析,以期為海洋沉積物真核微型生物多樣性研究的引物選擇提供科學參考。
1 材料和方法
1.1 樣品采集
本文分析所用的沉積物樣品是2007年4—5月海洋調查項目所采集的DH-13號沉積物柱樣表層樣品,采樣點坐標為(31°30.103′N,123°29.844′E),水深 40m,位于東海陸架長江古河口區。樣品采集后立即將0—10cm的沉積物柱芯以3cm為單位分為4層,分裝于無菌樣品袋中,–20°C保存,后放置于實驗室–80°C超低溫冰箱中長期保存,沉積物以中細砂為主。
1.2 樣品總DNA的提取與純化
利用 FastPrep?-24 快速核酸提取儀(MP Biomedicals公司)和FastDNA spin kit for soil試劑盒進行DNA提取,核酸蛋白檢測儀(Bio-Rad公司)測定DNA濃度和純度,純化后用于PCR擴增。
1.3 PCR擴增和產物回收
將上述總DNA在梯度PCR儀(Biometra公司)中進行擴增,擴增采用通用引物ITS1/ITS4、NS1/NS4和EF3/EF4,見表1所示。50μL PCR擴增體系:DNA模板 2μL,順反引物(10μmol/ L)各 1μL,dNTP(2.5mmol/L)4μL,10×PCR buffer(含 MgCl2,1.5mmol/L)5μL,rTaq 聚合酶(5U/μL)1μL,雙蒸水補足至 50μL。PCR擴增條件:95°C預變性4min;94°C變性30s,50°C退火 30s,72°C 延伸 60s,循環 35次;最后 72°C 延伸10min。1%瓊脂糖凝膠電泳檢測PCR擴增結果,并用DNA膠回收試劑盒(QIAGEN公司)回收產物。
1.4 克隆和測序
PCR回收產物與pMD-18T vector(TaKaRa公司)在 16°C連接過夜,將連接產物轉化到大腸桿菌(E.coli)DH5α感受態細胞,藍白斑篩選轉化子,挑選陽性克隆,重新擴增插入片斷,將含有合適大小插入片段的克隆送上海美季生物技術有限公司進行測序。
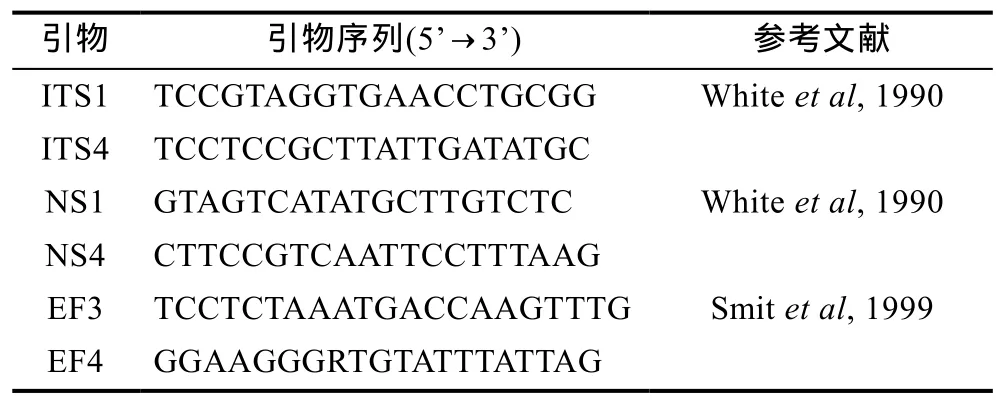
表1 三對通用引物的詳細信息Tab.1 Particular information of three universal primers
1.5 序列分析
將序列提交到RDP(ribosomal database project)Ⅱ數據庫,利用在線工具CHECK- CHIMERA檢測嵌合體;應用 BLASTN程序(www.ncbi.nlm.nih.gov/BLAST/)搜索相似性序列,進行系統發育分析。采用ClustalX(Version 1.8)對序列進行比對分析,通過MEGA 5軟件構建系統發育樹(Kumaret al,2004),采用 Neighbor-joining建樹方法,建樹結果進行bootstrap1000次系統檢驗;利用 Phylip軟件包中DNASIS程序計算距離矩陣,利用 DOTUR計算OTU(Schlosset al,2005)。引物 ITS1/ITS4、EF3/EF4、NS1/NS4擴增的部分序列在 GenBank核苷酸數據庫中的接受號分別為 KM067396—KM067409、KM067410—KM067425、KM067426—KM067449。
2 結果與分析
2.1 三對引物的真核微型生物文庫數據分析與對比
將測序返回的序列經拼接去載體后,三對引物ITS1/ITS4、EF3/EF4和NS1/NS4分別獲得94、100和129個有效克隆子。通過 DOTUR軟件將相似性大于97%的克隆子歸類為同一個OTU,ITS1/ITS4序列屬于35個OTU;EF3/EF4序列屬于61個OTU;NS1/NS4序列屬于80個OTU。三對引物克隆文庫的多樣性指數見表2 ,通過DOTUR軟件制作稀釋性曲線見圖1。
文庫分析結果表明:ITS1/ITS4、EF3/EF4、NS1/NS4三對引物克隆文庫的Chao 1指數、ACE豐富度指數和 Shannon-Weiner指數依次遞增,而Simpson優勢度指數則依次遞減,4個指數都說明三個文庫的多樣性以 NS1/NS4最高,EF3/EF4其次,ITS1/ITS4最后。從稀釋性曲線上可以看出,多樣性由高到低排列依次為NS1/NS4、EF3/EF4、ITS1/ITS4,與多樣性指數分析結果相一致;隨著克隆抽樣次數的增加,稀釋性曲線的斜率都有一定程度的變小,三對引物克隆文庫的 OTU類型都有一定程度的飽和,但ITS1/ITS4曲線相較于NS1/NS4和EF3/EF,明顯趨于平坦,說明用于ITS1/ITS4多樣性分析的取樣數量要明顯少于其它兩對引物,相對較少的克隆子可以反映多數微型真核生物的物種信息。
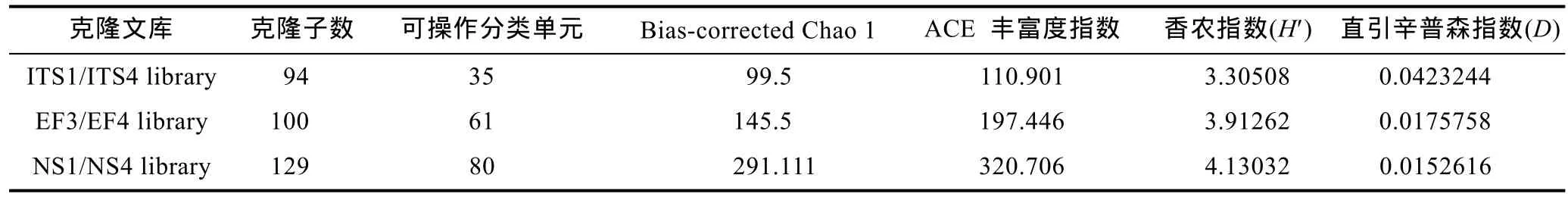
表2 東海陸架表層沉積物真核微型生物多樣性指數Tab.2 The microeukaryotic diversity from the surface layer sediments of the East China Sea
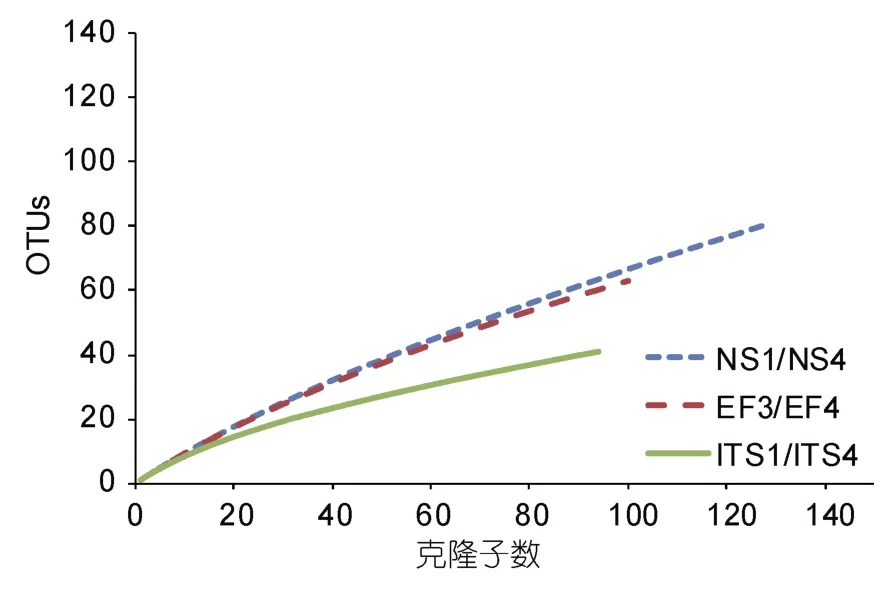
圖1 三個文庫的稀釋性曲線Fig.1 Rarefaction curves of three clone librares
2.2 三對引物的克隆文庫OTUs物種類群總述
應用 BLASTN搜索相似性序列,顯示東海陸架DH13站點176個OTU可分為4界24個類群,如圖2所示。其中原生生物界(Protist,125個OTU)具有顯著的優勢種群地位(占71%),包含絲足蟲門(Cercozoa,64個OTU)、橫裂甲藻綱(Dinophyceae,25個OTU)、不等鞭毛門(Stramenopiles,13個 OTU)、纖毛蟲門(Ciliophora,11個 OTU)、無根蟲門(Apusozoa,5個OTU)、頂復門(Apicomplexa,3個OTU)、囊泡蟲總門(Alveolata,2個OTU)、隱藻門(Cryptophyta,1個OTU)和眼蟲門(Euglenozoa,1個OTU)等9個類群。真菌界(Fungi)共有 35個 OTU(占 19.9%),包含子囊菌門(Ascomycota,13個 OTU)、壺菌門(Chytridiomycota,11個OTU)、真菌類(Fungi environmental samples,8個OTU)、毛霉亞門(Mucoromycotina,2個OTU)和擔子菌門(Basidiomycota,1個OTU)等5個類群。動物界(Metazoa,13個 OTU,占 7.4%)包含了環節動物門(Annelida,4個OTU)、腕足動物門(Brachiopoda,3個OTU)、扁形動物門(Platyhelminthes,1個OTU)、線蟲動物門(Nematoda,1個OTU)、軟體動物門(Mollusca,1個OTU)、輪形動物門(Rotifera,1個OTU)和刺胞動物門(Cnidaria,1個OTU)等8個類群。豐度最少的植物界(Plantae,3個 OTU,占 1.7%)則是包含綠藻門(Chlorophyta,1個OTU)和維管植物門(Tracheophyta,1個OTU)2個類群。
2.3 三對引物的克隆文庫OTUs物種類群比較
將三對引物ITS1/ITS4、EF3/EF4和NS1/NS4所獲得的克隆文庫(OTU分型)結果,以“門”為基準,比較三個文庫 OTU的物種多樣性,分類標準以 NCBI Taxonomy上的分類為基礎,結果見圖3、圖4、圖5。
綜合分析圖3、圖4、圖5結果表明:三對引物的克隆文庫OTUs都至少包含了原生生物界(Protist)、真菌界(Fungi)、植物界(Plantae)三大界,且原生生物界(Protist)占據了很大的比重,均超過了 50%,而特異的是 NS1/NS4同時還有相當多類群的動物界(Metazoa)OTUs。
ITS1/ITS4克隆文庫分為4界11個類群(見圖3),其中主要為原生生物界(57.1%)和真菌界(34.3%)。原生生物界涵蓋了:橫裂甲藻綱(Dinophyceae,9個OTU),纖毛蟲門(Ciliophora,7個 OTU),無根蟲門(Apusozoa,1個OTU),絲足蟲門(Cercozoa,1個OTU),不等鞭毛門(Stramenopiles,1個 OTU)以及囊泡蟲總門(1個 OTU)。真菌界分為 3類群,子囊菌門(Ascomycota,10個OTU)含量最高,另有少量擔子菌門(Basidiomycota,1個 OTU)和真菌類(Fungi,2個OTU)。除此之外,動物界的刺胞動物門(Cnidaria,1個 OTU)和植物界的綠藻門(Chlorophyta,1個 OTU)也有被檢測到類似序列的存在。
EF3/EF4克隆文庫分為3界6個類群(見圖4),同樣以原生生物界(70.5%)和真菌界(27.9%)為主。原生生物界均為絲足蟲門(Cercozoa,43個OTU)。真菌界分為4個類群,以壺菌門(Chytridiomycota,7個OTU)和真菌類(Fungi,5個 OTU)為主,另有子囊菌門(Ascomycota,3個OTU)和毛霉亞門(Mucoromycotina,2個 OTU)。植物界則是唯一的維管植物門(Tracheophyta,1個OTU)。
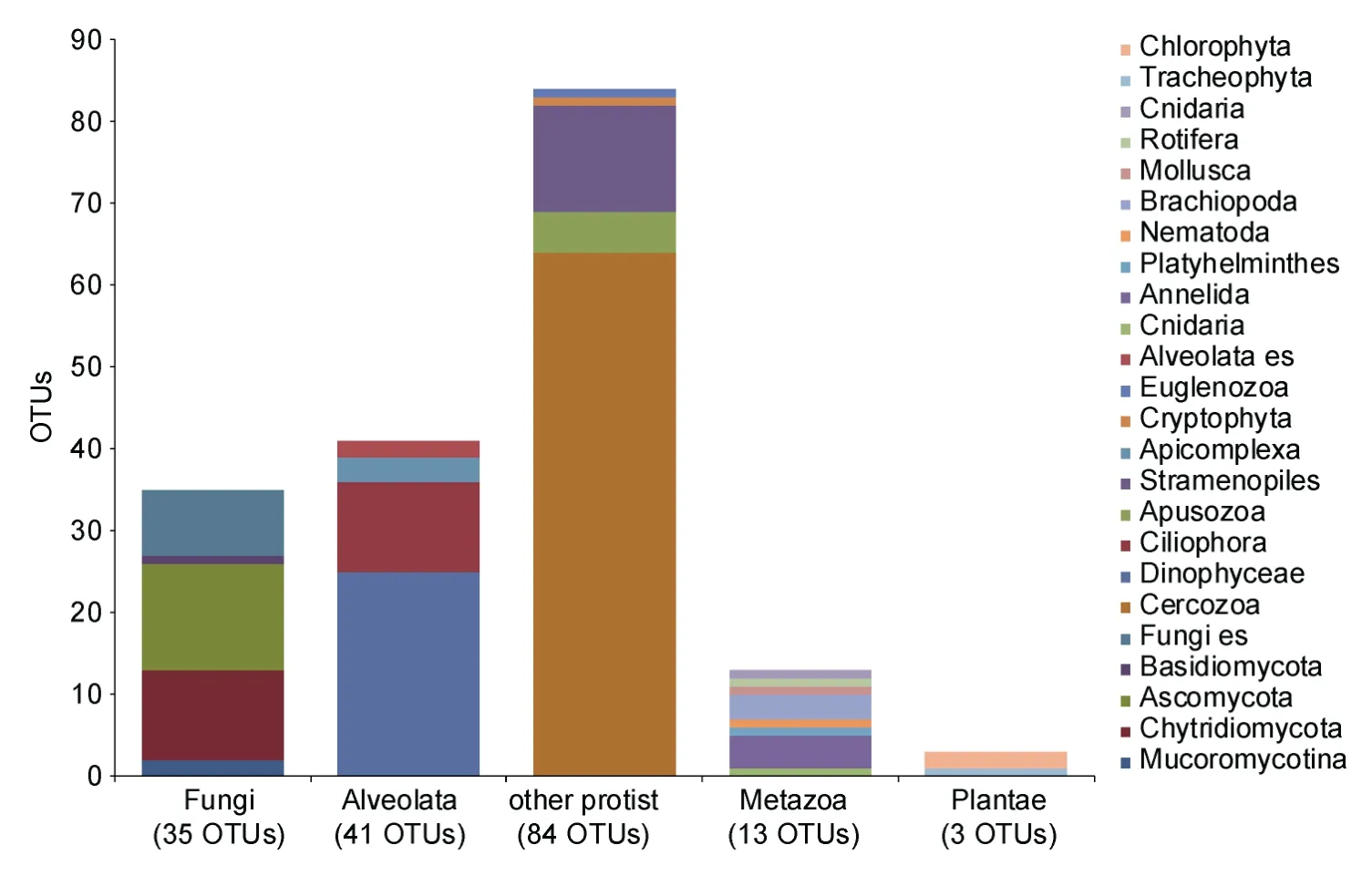
圖2 DH13站點中176個OTU的物種分類示意圖Fig.2 Species classification for 176 OTUs from station DH-13
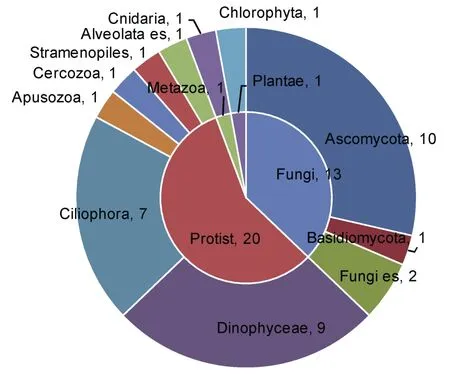
圖3 ITS1/ITS4引物克隆文庫的OTU類群比例Fig.3 Ratio of OTU groups from clone library of primer pairs ITS1/ITS4
NS1/NS4克隆文庫展現了最高的多樣性,共含4界 19個類群(見圖5),原生生物界(占 77.5%)仍是最主要的優勢種群,其中絲足蟲門(Cercozoa,20個OTU)、橫裂甲藻綱(Dinophyceae,16個OTU)和不等鞭毛門(Stramenopiles,12個 OTU)為主要的三個門,此外還有眼蟲門(Euglenozoa,1個 OTU)、隱藻門(Cryptophyta,1個 OTU)、無根蟲門(Apusozoa,4個OTU)、纖毛蟲門(Ciliophora,4個 OTU)、頂復門(Apicomplexa,3個 OTU)以及囊泡蟲總門(Alveolata environmental samples,1個OTU)。真菌界(占6.25%)以壺菌門(Chytridiomycota,4個OTU)為主。與其它兩對引物克隆文庫不同的是,NS1/NS4中動物界(15%)占到了第二位,且多樣性較高,具有 7個類群:環節動物門(Annelida,4個 OTU)、扁形動物門(Platyhelminthes,1個OTU)、線蟲動物門(Nematoda,1個OTU)、腕足動物門(Brachiopoda,3個OTU)、軟體動物門(Mollusca,1個OTU)、輪形動物門(Rotifera,1個OTU)和刺胞動物門(Cnidaria,1個OTU)。植物界則以綠藻門(Chlorophyta,1個OTU)為代表。
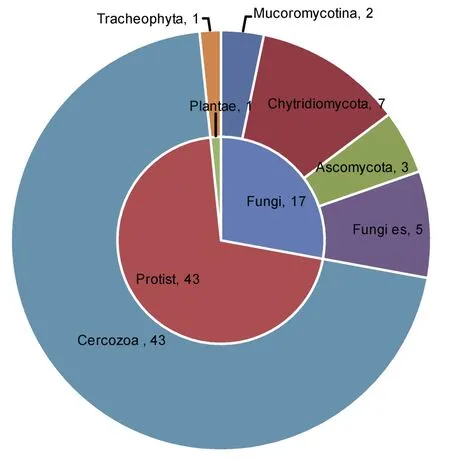
圖4 EF3/EF4引物克隆文庫的OTU類群比例Fig.4 Ratio of OTU groups from clone library of primer pairs EF3/EF4
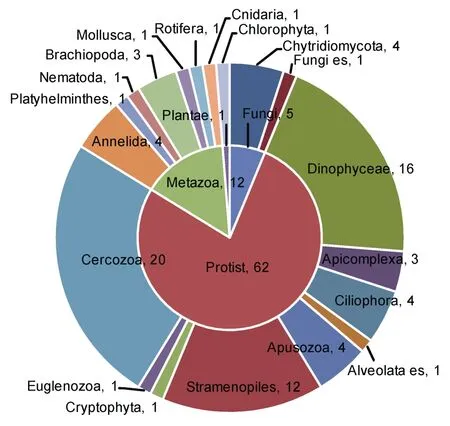
圖5 NS1/NS4引物克隆文庫OTU類群比例圖Fig.5 Ratio of OTU groups from clone library of primer pairs NS1/NS4
2.4 三對引物的克隆文庫系統發育學分析
2.4.1 三對引物的真菌克隆文庫系統發育分析分別從三對引物克隆文庫中選取代表性OTUs構建真菌系統發育樹,結果見圖6、圖7、圖8所示。
ITS1/ITS4克隆文庫構建的真菌系統發育樹主要含子囊菌門(Ascomycota)和擔子菌門(Basidiomycota)2個類群(圖6)。子囊菌門(Ascomycota)中的代表性基因型 DH132F36-A與一株來自于云南水稻根性寄生的未培養克隆子 R3-6有高同源性(99%),基因型DH132F27、DH132F14與地毯草(Axonopus compressus)根圈中分離的未培養土壤真菌克隆子 FITS_HRP1_W16具有高同源性(99%),基因型DH132F17-c與用于分解芒草和甘蔗細胞壁的Phaeosphaeriopsissp.TMS-2011有高同源性(99%),基因型 DH134F09與白黃筍頂孢霉(Acrostalagmus luteoalbusisolate L03)有高同源性(99%),此類霉菌同樣在深海環境中被發現,并用于細胞毒素的研究(Wanget al,2012)。擔子菌門(Basidiomycota)中的代表性基因型 DH132F30-B與夏威夷海域的銀耳目(Tremellales)LM477有較高同源性(95%)。
EF3/EF4克隆文庫構建的真菌系統發育樹中主要類群為壺菌門(Chytridiomycota)和子囊菌門(Ascomycota)2個類群(圖7)。壺菌門(Chytridiomycota)中的基因型DH133F27與法國湖泊Pavin和Aydat中的未培養壺菌 PA2009B6有較高同源性(95%),基因型DH131F01-a與紅根囊壺菌(Rhizophlyctis roseaJEL 318)有 93%的同源性,基因型 DH134F60、DH134F06、DH134F11與喜馬拉雅山脈、落基山脈的未培養壺菌克隆子有 90%的同源性,先前的研究顯示高海拔土壤真菌中壺菌占主導地位(Freemanet al,2009)。子囊菌門(Ascomycota)中的基因型DHE134-1與海洋中的二尖梅奇酵母(Metschnikowia bicuspidate,DQ520881)有高同源性(99%),該酵母被認為是三疣梭子蟹乳化病的致病菌(Wanget al,2007)。此外基因型DHE132-10與一株未培養Fungus clone nco32b11c1有高同源性(99%)。
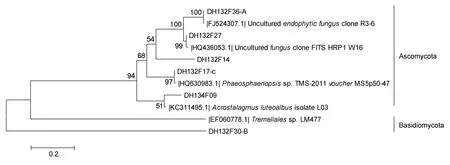
圖6 根據ITS1/ITS4克隆文庫構建的真菌系統發育樹Fig.6 Phylogenetic tree of Fungi according to clone library of primer pairs ITS1/ITS4
NS1/NS4克隆文庫構建的真菌系統發育樹中基因型都為壺菌門(Chytridiomycota),如圖8所示。基因型 DH1331F43與海洋藻類病原體菌株-壺菌polysiphoniae有 93%的同源性,基因型 DH1321F31與 EF3/EF4中的部分基因型(DH134F60,DH134F06,DH134F11)相同,與喜馬拉雅山脈、落基山脈的未培養壺菌克隆有較高同源性(94%)。
2.4.2 三對引物的原生生物克隆文庫系統發育分析分別從三對引物克隆文庫中選取代表性OTUs構建原生生物系統發育樹,結果見圖9、圖10、圖11所示。
ITS1/ITS4克隆文庫構建的原生生物系統發育樹主要分為囊泡蟲總門(Alveolata)、不等鞭毛門(Stramenopiles)、無根蟲門(Apusozoa)和絲足蟲門(Cercozoa)四大類。而囊泡蟲總門(Alveolata)中的纖毛蟲門(Ciliophora)和雙鞭毛蟲門(Dinoflagellata)下的橫裂甲藻綱(Dinophyceae),兩者占到了ITS1/ITS4克隆文庫原生生物界的絕大多數(圖9)。纖毛蟲門(Ciliophora)基因型 DH133-5和海洋擬急游蟲(Strombidinopsis Sopsis99-1)有著高相似度(98%),基因型DH131-1和急游蟲屬(StrombidiumLLK-2005)有著較高相似度(94%),而基因型DH131-2則與中國大陸海洋魚類中分離到的刺激隱核蟲(Cryptocaryon irritans)相似,但相似度不高(85%)。橫裂甲藻綱(Dinophyceae)基因型 DH134-12與異帽藻(Heterocapsa)相似(89%),基因型DH133-31與裸甲藻(Gymnodinium beii)高度相似(99%)。基因型DH131-23與 MAST-4類群的未培養不等鞭毛門克隆子 M_IND70.7有著較高相似度(94%),MAST-4被認為是一個 低 進 化 多 樣 性 的 類 群(Rodríguez-Martínezet al,2012)。無根蟲門(Apusozoa)基因型DH131-7與海洋中分離的Apusomonas proboscidea相似,但相似度不高(87%)。絲足蟲門(Cercozoa)基因型DH132F19與海洋絲足蟲類Thaumatomastix克隆子有著較高相似度(93%)。

圖7 根據EF3/EF4克隆文庫構建的真菌系統發育樹Fig.7 Phylogenetic tree of Fungi according to clone library amplificated with primer pairs EF3/EF4

圖8 根據NS1/NS4克隆文庫構建的真菌系統發育樹Fig.8 Phylogenetic tree of Fungi according to clone library amplificated with primer pairs NS1/NS4
EF3/EF4克隆文庫構建的原生生物系統發育樹顯示均為絲足蟲門(Cercozoa),這與 ITS1/ITS4和NS1/NS4顯示出的原生生物多樣性顯著不同,如圖10所示。基因型DH131F02-a與未培養絲足蟲類4-E2相似(95%),DH131F03與未培養絲足蟲類3b-F5相似(95%),兩個相似序列均來自于波羅的海的海雪(sea snow)中。基因型 DH132F33-a和 DH133F22分別與來自加拿大不列顛哥倫比亞省海洋底棲生物棲息地中的絲足蟲類CC-2009c和CC-2009e相似,相似度為96%和94%。基因型DH131F04-a與廣泛分布于國內外沿海浮游生物群落中的吞噬鞭毛蟲(Ebria tripartiteisolate 2)有94%的相似度。基因型DH133F46和基因型 DH132F15-a分別與海洋絲足蟲類的CryothecomonasAPCC MC5-1Cryo和ThaumatomastixCC002有較高相似度,相似度為97%和96%。
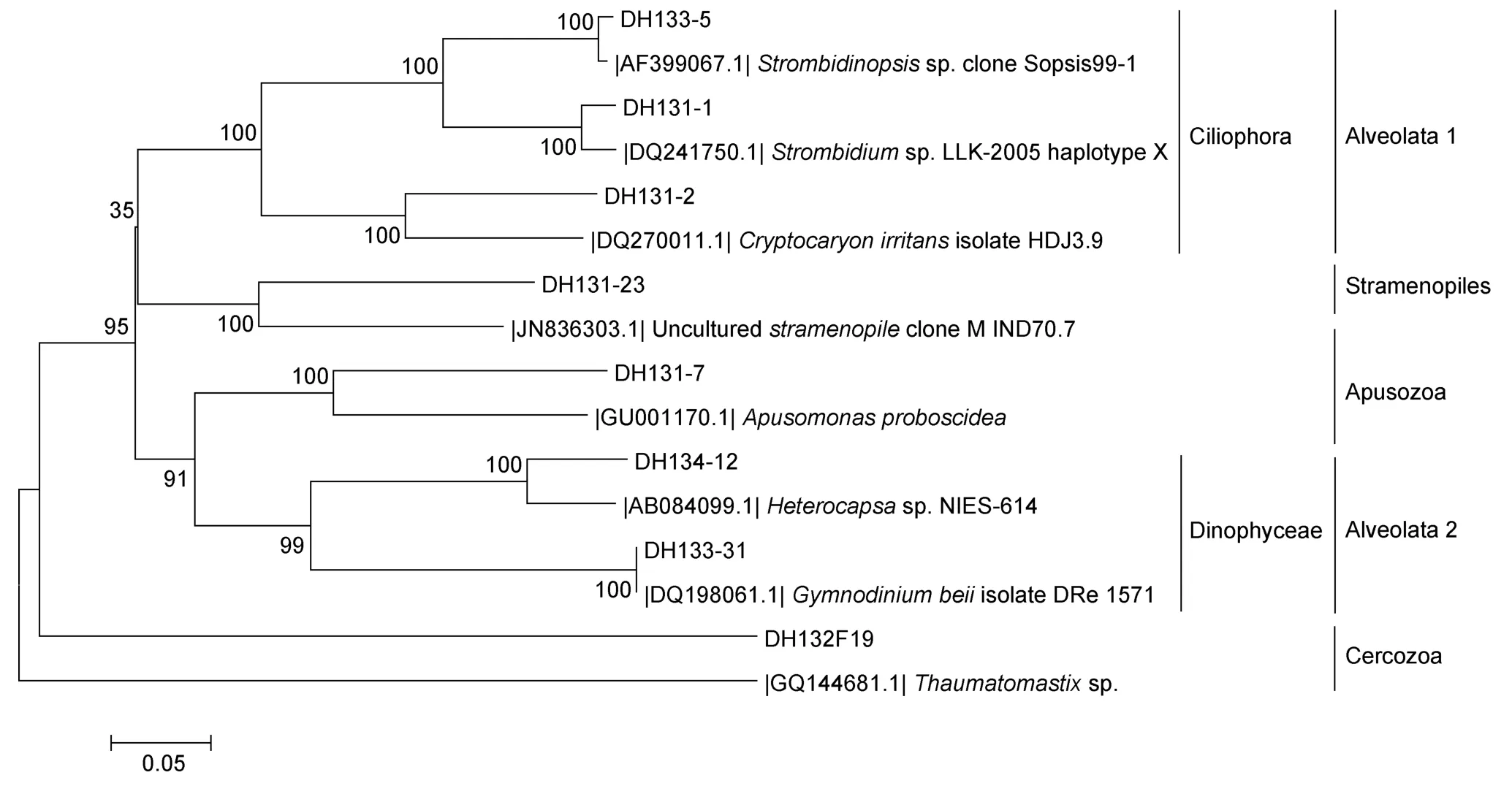
圖9 根據ITS1/ITS4克隆文庫構建的原生生物系統發育樹Fig.9 Phylogenetic tree of Protist according to clone library amplificated with primer pairs ITS1/ITS4
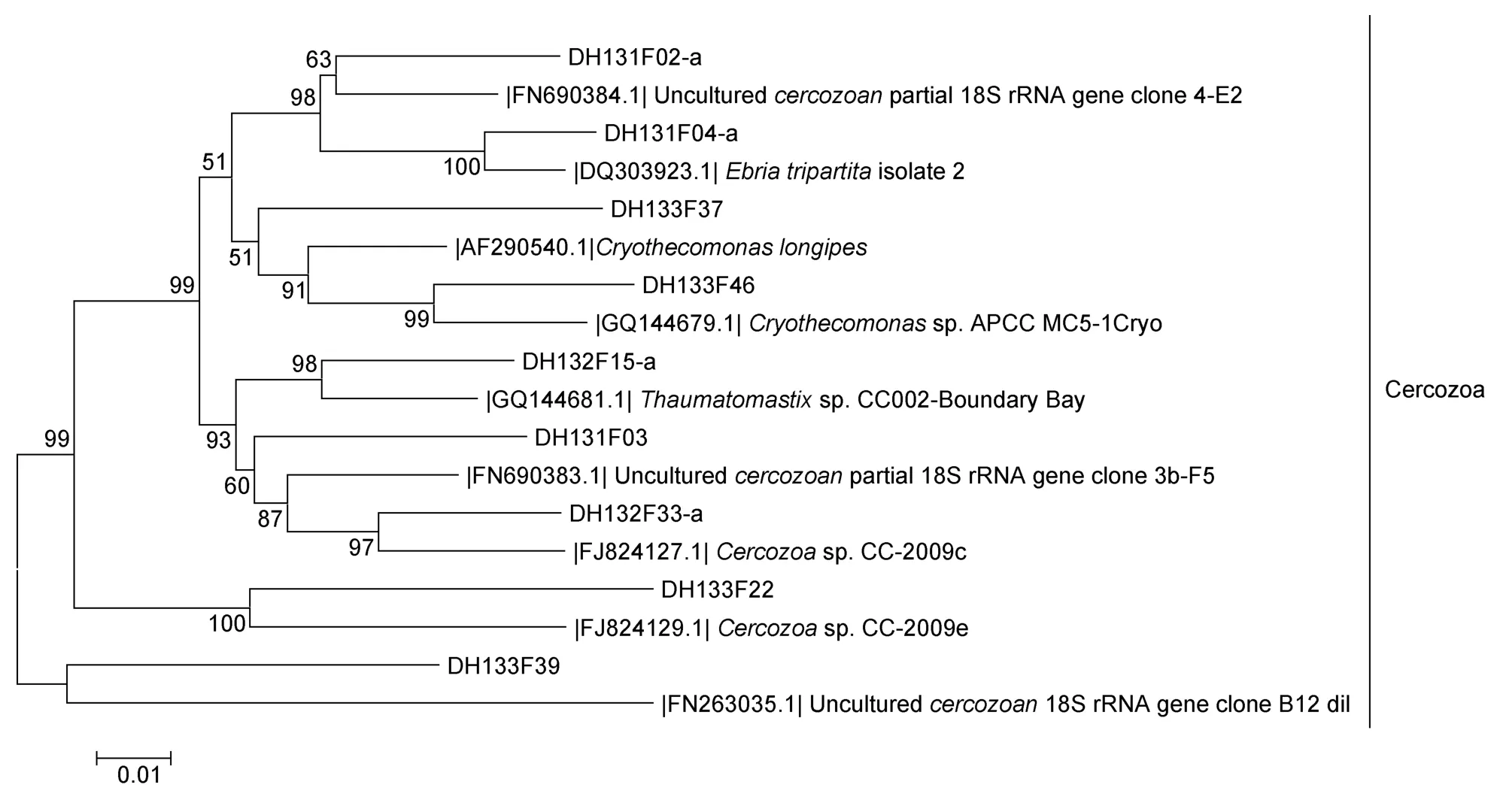
圖10 根據EF3/EF4克隆文庫構建的原生生物系統發育樹Fig.10 Phylogenetic tree of Protist according to clone library amplificated with primer pairs EF3/EF4
NS1/NS4克隆文庫表現出了豐富的原生生物多樣性,選取其中4個主要類群囊泡蟲總門(Alveolata)、不等鞭毛門(Stramenopiles)、絲足蟲總門(Cercozoa)和無根蟲門(Apusozoa)的代表性OTUs構建系統發育樹(圖11)。
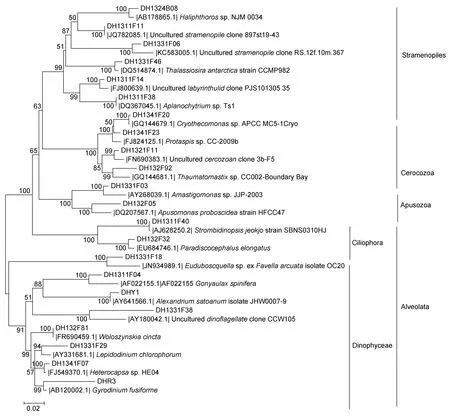
圖11 根據NS1/NS4克隆文庫構建的原生生物系統發育樹Fig.11 Phylogenetic tree of Protist according to clone library amplificated with primer pairs NS1/NS4
囊泡蟲總門(Alveolata)分為纖毛蟲門(Ciliophora)和雙鞭毛蟲門(Dinoflagellata)下的橫裂甲藻綱(Dinophyceae)兩個類群,與 ITS1/ITS4相同,纖毛蟲門(Ciliophora)基因型 DH1311F40與韓國西部海岸的Strombidinopsis jeokjoSBNS0310HJ有高度同源性(99%),基因型 DH132F32與黃海的Paradiscoce-phalus elongatus克隆子有高相似度(98%)。橫裂甲藻綱(Dinophyceae)基因型 DH1331F18與海洋環境中的Euduboscquella克隆子有較高相似度(95%),基因型DH1311F04與Gonyaulax spinifera克隆子有較高相似度(95%),基因型DHY1與Alexandrium satoanum克隆子有高同源性(99%),基因型 DH1331F38與貧氧海洋環境中的未培養雙鞭毛蟲 CCW105有 90%的相似度。基因型DH132F81與韓國西部海岸的Woloszynskia cincta克隆子有高同源性(99%),基因型DH1331F29與Lepidodinium chlorophorum克隆子有較高相似度(96%),基因型 DH1341F07與異帽藻(HeterocapsaHE04)有高相似度(99%),基因型 DHR3與Gyrodinium fusiforme克隆子有較高相似度(94%)。
不等鞭毛門(Stramenopiles)有代表性基因型OTU6個,其中基因型 DH1324B08與一株為未識別的類似Haliphthoros克隆子NJM 0034相似度為97%;基因型 DH1311F11與北太平洋海域的未培養不等鞭毛類 897st19-43有高相似度(99%);基因型DH1331F06與來自于紅海東北海岸一個貧營養站點的未培養不等鞭毛類 RS.12f.10m.367有較高相似度(95%),該站點的海洋不等鞭毛門(Marine Stramenopiles)類群多樣性豐富,是第二大類群(OTUs 16.8%)(Acostaet al,2013);基因型1331F46與南極洲海域的Thalassiosira CCMP982有高相似度(99%);基因型1311F14與美國紐約長島海域沉積物的一個未培養LabyrinthulidPJS101305_35有98%的相似度;基因型1311F38與AplanochytriumTs1有高相似度(99%)。
絲足蟲門(Cercozoa)與 EF3/EF4非常相似,基因型DH1341F20和基因型DH132F92分別與海洋絲足蟲類的CryothecomonasAPCC MC5-1Cryo和ThaumatomastixCC002相似,相似度為99%和94%,基因型DH1341F23的相似序列來自于加拿大不列顛哥倫比亞省海洋底棲生物棲息地中的絲足蟲類克隆子,相似度為98%,基因型DH1321F11與波羅的海海雪中的未培養絲足蟲類 CC-2009b,相似度為98%。
無根蟲門(Apusozoa)基因型 DH1331F03與AmastigomonasJJP-2003有較高同源性(96%),基因型DH132F05與Apusomonas proboscideaHFCC47有91%的相似度。
3 討論
3.1 真核微型生物多樣性研究中的引物選擇
隨著分子生物學的不斷發展,越來越多的引物被設計應用于真核微型生物分子生態學研究,除了本文使用的三對引物外,Euk1A/Euk516r(鮑磊等,2008)、nu-SSU-0817/nu-SSU-1196 or 1536(Bornemanet al,2000)、18S-42F or 82F/18S-1498R or 1520R(Takishitaet al,2006)、Euk-A/Euk-B(Koidet al,2012)等也同樣被應用于真核微型生物的研究。
本文使用的三對引物最初被設計用于子囊菌、小麥根系等陸地真核微型生物多樣性的研究(Whiteet al,1990;Bernieret al,1994;Smitet al,1999),后來也被用于海洋環境真核微型生物研究,如 Lai等(2007)應用ITS1/ITS4引物對南海深海甲烷水合沉積物中真菌群落結構進行了初步研究,擴增得到的大多數真菌可歸類為Ascomycota和Basidiomycota;Gao等(2008)應用ITS1/ITS4、EF3/EF4等8對引物擴增夏威夷海綿共附生真菌;Nagano等(2010)應用 ITS1/ITS4引物擴增到了深海沉積物中新的真菌類群;高遠等(2012)應用 ITS1/ITS4對鄂霍次克海冷泉沉積物真核微型生物多樣性進行研究;Singh等(2012)分別使用NS1/NS4和ITS1/ITS4對深海沉積物中可培養和不可培養兩種形式的真菌進行了擴增。
但上述多數研究僅使用單一引物對環境中真核微型生物進行分析,研究者們也逐漸發現僅用單一引物來研究復雜環境樣本中的真核微型生物多樣性存在著偏倚和局限性,如Smit等(1999)在分析了小麥根系樣本后認為引物 EF3/EF4略微偏向于擴增擔子菌和接合菌。因此采用多種引物進行比較分析的相關研究在近些年也逐漸被研究人員關注(Andersonet al,2003),但是對于海洋沉積環境的相關比較分析還未發現,本文研究結果對更深入地開展海洋沉積環境真核微型生物的多樣性研究有著借鑒作用。
3.2 不同引物的擴增特異性及其多樣性
本文三對引物擴增得到的克隆文庫均包含了沉積物中真核微型生物的主要類群:原生生物(Protist)和真菌(Fungi),NS1/NS4克隆文庫還獨特的擴增到了一些動物界(Metazoa)類群。
原生生物類群在三對引物中比例都達到了 50%以上,ITS1/ITS4克隆文庫與NS1/NS4克隆文庫均包含了橫裂甲藻綱(Dinophyceae)、纖毛蟲門(Ciliophora)、絲足蟲門(Cercozoa)、無根蟲門(Apusozoa)和不等鞭毛門(Stramenopiles)5個類群,且橫裂甲藻綱(Dinophyceae)在兩個文庫中均為優勢類群(45%和 25.8%),此外NS1/NS4擴增到了獨有的 3個類群(Apicomplexa,Cryptophyta,Euglenozoa),在三對引物對原生生物的擴增中展現了最高的多樣性。但 EF3/EF4克隆文庫中所有原生生物克隆子均為絲足蟲門(Cercozoa),這可能是由于 EF3/EF4引物對海洋沉積物中原生生物類群擴增具有局限性。
真菌方面,三對引物擴增結果各異。ITS1/ITS4克隆文庫主要是子囊菌門(Ascomycota)和擔子菌門(Basidiomycota);EF3/EF4克隆文庫是子囊菌門(Ascomycota)、壺菌門(Chytridiomycota)和毛霉亞門(Mucoromycotina);NS1/NS4克隆文庫則基本都是壺菌門(Chytridiomycota)。這些類群與國際上沉積物真菌多樣性研究中的主要類群基本一致(Laiet al,2007;Naganoet al,2010;Singhet al,2012)。值得注意的是,Smit等(1999)認為EF3/EF4引物在培養生物和環境樣本的擴增中輕微偏向于擴增擔子菌門(Basidiomycota)和接合菌門(Zygomycota),但這兩個類群在東海陸架表層沉積物中都未被擴增到,這可能是由于小麥根系與海洋沉積物的環境差異性所導致的,而 Burgaud等(2013)應用EF3/EF4引物對美國特拉華河河口沉積物的擴增所得主要類群為子囊菌門(Ascomycota)、擔子菌門(Basidiomycota)和壺菌門(Chytridiomycota),與本文結果類似。
3.3 引物選擇對海洋原生生物多樣性研究的影響
雖然三對引物在國際上的研究中多被用于真菌多樣性的研究,但根據本文的數據結果顯示,三對引物都擴增到了較多的原生生物類群,尤其是NS1/NS4引物,原生生物類群的多樣性在三對引物中是最高的。Park等(2008)應用引物18S-82F/18S- 1520R擴增東海表層沉積物的研究中顯示,90%的克隆序列為原生生物,主要類群為纖毛蟲門(Ciliophora,18%)、雙鞭毛蟲門(Dinoflagellata,19%)、不等鞭毛門(Stramenopiles,11%)和絲足蟲門(Cercozoa,20%),與本文三對引物擴增結果基本一致,且除領鞭毛蟲門(Choanozoa)外,Park等(2008)文中所擴增得到的原生生物類群本文同樣擴增得到。因此本文推斷,ITS1/ITS4、NS1/NS4引物同樣可應用于海洋沉積物原生生物多樣性的研究中,作為其進一步的補充和延伸,而 EF3/EF4則由于其在絲足蟲門(Cercozoa)擴增上的相對專一性,可能更適用于絲足蟲門(Cercozoa)下不同種屬多樣性的研究。
4 結論
本文選擇三對真核微型生物的通用引物(NS1/NS4,EF3/EF4,ITS1/ITS4),通過18S rDNA、ITS區的基因克隆和文庫構建,對東海陸架DH-13站點沉積物真核微型生物類群進行分類學、多樣性和系統發育分析,研究發現三對引物的選擇不同對真核微型生物多樣性的影響較顯著,尤其是在原生生物的擴增上。本文的研究結果再次說明了對特異性PCR引物進行嚴格檢測以適用于目的類群擴增的重要性和迫切性,同時也說明了單一引物擴增的局限性,通過多種引物綜合使用來評估復雜環境中真核微型生物多樣性是必要的,未來的研究和分析應更注重于綜合性的處理方法。
王健鑫,許賢恩,周鏈鏈等,2012.東海陸架表層沉積物微生物多樣性初步研究.海洋與湖沼,43(4):806—813
李家彪,2008.東海區域地質.北京:海洋出版社,1—105
汪 岷,江雪嬌,2009.海洋微微型真核生物分子多樣性的研究方法.中國海洋大學學報:自然科學版,39(4):627—632
宋志剛,許強芝,魯心安等,2006.中國東海海洋微生物種群多樣性初步研究.微生物學通報,33(1):63—67
張林寶,李鐵剛,黨宏月等,2010.東海內陸架泥質區沉積物古菌群落垂向分布特征.地球科學——中國地質大學學報,35(2):255—260
高 遠,李富超,姜 鵬等,2012.鄂霍次克海冷泉沉積物真核生物多樣性的初步研究.海洋科學,36(5):29
鮑 磊,陳紀新,黃邦欽等,2008.應用變性梯度凝膠電泳研究廈門西海域超微型真核浮游生物多樣性.海洋環境科學,26(6):504—509
戴 欣,蔡創華,2001.海洋沉積物中特有細菌類群的初步探討.中山大學學報:自然科學版,40(6):51—54
Acosta F,Ngugi D K,Stingl U,2013.Diversity of picoeukaryotes at an oligotrophic site off the Northeastern Red Sea Coast.Aquatic Biosystems,9(1):16
Anderson I C,Campbell C D,Prosser J I,2003.Potential bias of fungal 18S rDNA and internal transcribed spacer polymerase chain reaction primers for estimating fungal biodiversity in soil.Environmental Microbiology,5(1):36—47
Bernier L,Hamelin R C,Ouellette G B,1994.Comparison of ribosomal DNA length and restriction site polymorphisms in Gremmeniella and Ascocalyx isolates.Applied and Environmental Microbiology,60(4):1279—1286
Borneman J,Hartin R J,2000.PCR primers that amplify fungal rRNA genes from environmental samples.Applied and Environmental Microbiology,66(10):4356—4360
Burgaud G,Woehlke S,Rédou Vet al,2013.Deciphering the presence and activity of fungal communities in marine sediments using a model estuarine system.Aquatic Microbial Ecology,70:45—62
Freeman K R,Martin A P,Karki Det al,2009.Evidence that chytrids dominate fungal communities in high-elevation soils.Proceedings of the National Academy of Sciences,106(43):18315—18320
Gao Z,Li B,Zheng Cet al,2008.Molecular detection of fungal communities in the Hawaiian marine spongesSuberites zetekiandMycale armata.Applied and Environmental Microbiology,74(19):6091—6101
K?ster M,Wardenga R,Blume M,2008.Microscale investigations of microbial communities in coastal surficial sediments.Marine Ecology,29(1):89—105
Koid A,Nelson W C,Mraz Aet al,2012.Comparative analysis of eukaryotic marine microbial assemblages from 18S rRNA gene and gene transcript clone libraries by using different methods of extraction.Applied and Environmental Microbiology,78(11):3958—3965
Kumar S,Tamura K,Nei M,2004.MEGA3:Integrated software for molecular evolutionary genetics analysis and sequence alignment.Briefings in Bioinformatics,5:150—163
Lai X,Cao L,Tan Het al,2007.Fungal communities from methane hydrate-bearing deep-sea marine sediments in South China Sea.The ISME Journal,1(8):756—762
Nagano Y,Nagahama T,Hatada Yet al,2010.Fungal diversity in deep-sea sediments——the presence of novel fungal groups.Fungal Ecology,3(4):316—325
Park S J,Park B J,Pham V Het al,2008.Microeukaryotic diversity in marine environments,an analysis of surface layer sediments from the East Sea.The Journal of Microbiology,46(3):244—249
Rodríguez F,Derelle E,Guillou Let al,2005.Ecotype diversity in the marine picoeukaryote Ostreococcus(Chlorophyta,Prasinophyceae).Environmental Microbiology,7:853—859
Rodríguez-Martínez R,Rocap G,Logares Ret al,2012.Low evolutionary diversification in a widespread and abundant uncultured protist(MAST-4).Molecular Biology and Evolution,29(5):1393—1406
Schloss P D,Handelsman J,2005.Introducing DOTUR,a computer program for defining operational taxonomic units and estimating species richness.Applied and Environmental Microbiology,71(3):1501—1506
Singh P,Raghukumar C,Meena R Met al,2012.Fungal diversity in deep-sea sediments revealed by culture-dependent and cultureindependent approaches.Fungal Ecology,5(5):543—553
Smit E,Leeflang P,Glandorf Bet al,1999.Analysis of fungal diversity in the wheat rhizosphere by sequencing of cloned PCR-amplified genes encoding 18S rRNA and temperature gradient gel electrophoresis.Applied and Environmental Microbiology,65(6):2614—2621
Takishita K,Tsuchiya M,Reimer J Det al,2006.Molecular evidence demonstrating the basidiomycetous fungusCryptococcus curvatusis the dominant microbial eukaryote in sediment at the Kuroshima Knoll methane seep.Extremophiles,10(2):165—169
Wang F Z,Huang Z,Shi X Fet al,2012.Cytotoxic indole diketopiperazines from the deep sea-derived fungusAcrostalagmus luteoalbusSCSIO F457.Bioorganic &Medicinal Chemistry Letters,22(23):7265—7267
Wang X,Chi Z,Yue Let al,2007.A marine killer yeast against the pathogenic yeast strain in crab(Portunus trituberculatus)and an optimization of the toxin production.Microbiological Research,162(1):77—85
White T J,Bruns T,Lee Set al,1990.Amplification and Direct Sequencing of Fungal Ribosomal RNA Genes for Phylogenetics.PCR Protocols:a Guide to Methods and Applications.Academic Press,New York,18:315—322
Whitman W B,Coleman D C,Wiebe W J,1998.Prokaryotes:The unseen majority.Proceedings of the National Academy of Sciences,USA,95:6578—6583
Zeng Y H,Li H Y,Jiao N Z,2007.Phylogenetic diversity of planktonic archaea in the estuarine region of East China Sea.Microbiological Research,162:26—36

