利用基因芯片技術鑒別山羊絨和綿羊絨
呂雪峰 阿布力孜·阿布力米提 帕娜爾 陶衛東 邢巍婷 采復拉 鄭文新
(農業部種羊與羊毛羊絨質量監督檢驗測試中心,烏魯木齊 830000)
利用基因芯片技術鑒別山羊絨和綿羊絨
呂雪峰 阿布力孜·阿布力米提 帕娜爾 陶衛東 邢巍婷 采復拉 鄭文新
(農業部種羊與羊毛羊絨質量監督檢驗測試中心,烏魯木齊 830000)
為了建立山羊絨與綿羊絨的基因芯片鑒別方法,達到快速、高通量鑒別山羊絨與綿羊絨的目的,文章根據山羊和綿羊遺傳物質的特異性,選擇線粒體基因組細胞色素b基因(cyt b)為目的基因,在cyt b基因通用引物區間設計出一對能鑒別綿羊絨與山羊絨的探針,經過與含有不同比例綿羊源性的PCR產物雜交,結果顯示當山羊源性成分中綿羊源性含量為3% 時,仍然可見雜交信號,說明基因芯片鑒別毛絨的方法具有非常高的特異性,能夠定性的鑒別山羊絨和綿羊絨。
山羊絨 綿羊絨 線粒體DNA Cyt b基因 基因芯片
山羊絨是一種珍稀而昂貴的動物纖維,其制品的品質極高,而且非常舒適,深受消費者青睞。為了追求利潤,有些生產者將與山羊絨外觀相似的綿羊毛或其它纖維混入織物中,嚴重損害了消費者的利益,因而準確鑒別山羊絨與綿羊毛及其它動物纖維顯得尤為重要。以前常用的鑒別方法主要有顯微鏡法[1]、溶液法[2,3]和光譜分析法[4,5]等,但由于山羊絨與綿羊毛同屬蛋白質纖維,化學性質差異不大,所以存在誤判率較高、操作復雜、主觀因素強等缺點[6]。特別是粗毛羊毛被中的底絨,又稱綿羊絨[7],因其細度、光澤、鱗片都與山羊絨及其相似,故使用傳統的方法很難區別。綿羊和山羊為不同種的動物,遺傳物質的區別才是物種間根本差別,因此檢測DNA的種間差異性不失為鑒定毛絨的理想途徑[8]。
基因芯片具有高通量、自動化、檢測速度快、特異性強的特點,目前已經廣泛用于動物源性成分檢測[9]、病原微生物、細菌檢測[10-12]、基因多態性[13-15]、轉基因食品檢測[16]等領域。我國是毛絨生產大國,但毛絨摻雜檢測技術相對比較滯后,檢測手段單一,專業人員少,國內也沒有成熟的毛絨鑒別技術來支撐毛絨市場以及進出口檢驗檢疫,因此急需一項新型檢測技術來彌補這方面的缺陷。基因芯片技術在高通量檢測方面有絕對的優勢。文章根據毛絨DNA的特點和基因芯片核酸分子雜交原理,在通用引物擴增區間設計出一對能鑒別山羊和綿羊源性的探針,旨在建立能同時檢驗山羊和綿羊源性的基因芯片方法,并確定檢驗的最低限量。
1 材料與方法
1.1 材料
從綿羊和山羊體上剪下的毛絨,剪毛時盡量貼近皮膚。
PCR擴增儀(德國Biometra公司)、凝膠成像系統(美國Bio-Rad公司)、基因芯片點樣儀、芯片掃描儀、雜交儀、芯片清洗儀(北京博奧生物芯片公司),其余為實驗室常用儀器。
Chelex-100(SIGMA公司)、蛋白酶K(Genviw公司)、TaqDNA 聚合酶、dNTPs、SSC、Denhardt’s液(上海生工生物公司)、Cy5-dCTP(Health Care公司)、醛基化基片及蓋片(北京博奧生物公司)。
1.2 方法
1.2.1 引物和探針設計與合成 根據文獻選用了一對可擴增綿羊和山羊線粒體基因cyt b的通用引物[3],在引物的擴增區間設計出數對探針,將這些探針序列用NCBI的 Blast進行比對分析,結合試驗驗證,最終選擇特異性較好的序列作為鑒別綿羊絨與山羊絨的基因探針,陽性定位探針和陰性質控探針見石豐運報道[9]。引物和探針的合成、修飾由上海生工生物技術有限公司完成,具體序列見表 1。
1.2.2 毛干DNA提取 改進的chelex-100法:稱取毛發樣本10 mg左右,液氮研磨粉碎后置于2 mL離心管中。先后用無水乙醇,去離子水各浸泡30 min,每管中依次加入1720 μL 20% Chelex-100(SIGMA)、80 μL 1 mol/L DTT、200 μL 20 mg/mL 蛋 白 酶K,56℃溫浴5-6 h,劇烈振蕩30 s,100℃,8 min,使蛋白質變性。振蕩30 s。12 000 r/min 離心15 min,取上清。
1.2.3 PCR產物制備 利用上述引物,分別對綿羊毛和山羊絨DNA進行擴增,PCR 反應體系為:10×PCR buffer 5 μL、Mg2+2.5 μL、dNTPs(2.5 mmol/L)4 μL、Cy5-dCTP 0.5 μL、正向引物和反向引物各1 μL(10 μmol/L)、DNA 模板(10 ng/μL)5 μL、Taq酶 1 μL(5 U/μL)、ddH2O 補足總體積到50 μL。PCR擴增條件為:94℃預變性 4 min;94℃變性45 s,55℃退火 45 s,72℃ 延伸 1 min,共35個循環;72℃延伸7 min。
1.2.4 芯片的制備 將合成的探針用 1×TE 溶解至終濃度為 40 μmol/L,與 50% DMSO 等體積混勻后作為探針點樣混合液,加入384 孔板中,空白對照為 50% DMSO,按預先設計的探針點陣點至基片上。將芯片點有探針的一面在65℃水合10 s兩次,自然晾干,紫外交聯5 min。將芯片在42℃預熱,0.5% SDS清洗10 min,然后用42℃預熱的去離子水清洗2 min,離心甩干,4℃避光保存備用。
1.2.5 芯片雜交與清洗 將Cy5熒光標記的PCR產物和雜交液(25%甲酰胺,3×SSC,5×Dehartdt液,0.2% SDS)等量混合,總體積為80 μL;將其轉移到PCR管中,在PCR儀上95℃熱變性5 min,冰浴驟冷5 min;將雜交混合液點到芯片后蓋上蓋玻片,放入雜交儀中,42℃雜交2 h。雜交完成后分別預熱洗液A(2×SSC,0.2% SDS)和洗液B(0.2% SDS)至42℃,洗液A洗兩次,各2 min;洗液B洗一次,2 min。
2 結果
2.1 毛絨DNA提取
分別剪下山羊絨和綿羊毛毛根部分毛樣,通過改進的chelex-100法提取毛絨mtDNA作為模板,進行 PCR 擴增,電泳檢測結果見圖1。
從圖1可以看出,通用引物能有效擴增出山羊絨和綿羊毛cytb部分基因,大小在500 bp左右,與預期擴增產物大小相同。
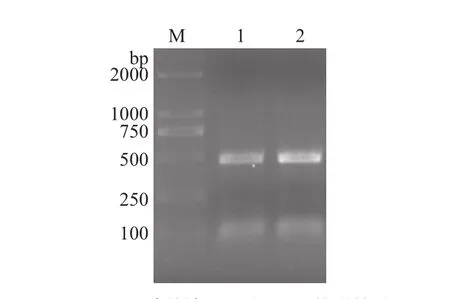
圖 1 毛絨cytb基因擴增結果
2.2 探針雜交特異性試驗
基因芯片雜交及掃描結果顯示,兩條探針與目的產物有較好的雜交信號,綿羊毛和山羊絨PCR產物均可與芯片內相應的探針進行雜交,而與其它的探針無特異性交叉反應,陽性質控探針及陰性質控探針雜交信號正常,表明這兩條探針有很好的特異性,探針點樣和雜交結果分別見圖2和圖3。
圖2 探針點樣矩陣圖
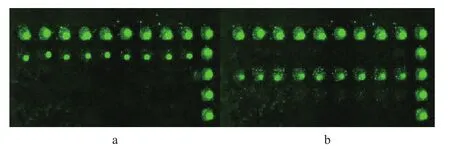
圖 3 基因探針特異性雜交結果圖
2.3 芯片最低檢測限量試驗
分別以在山羊源性PCR產物中摻入50%、10%、3%、1% 比例的綿羊源性產物,然后與綿羊源性探針雜交。根據芯片雜交掃描分析后的圖像,得出能檢測出綿羊源性最小摻入量,這也間接的反應出混合毛絨樣品中的山羊絨和綿羊絨的檢出限量。試驗結果(圖4,圖5)表明,該芯片能夠檢出到3%的綿羊絨摻雜量。
圖4 基因芯片點樣矩陣圖

圖5 不同比例綿羊源性成分與芯片雜交信號圖
3 討論
動物毛纖維中 DNA 含量極其微量,核 DNA 主要存在于毛囊部位,毛干中幾乎沒有核 DNA,僅含有微量 mtDNA,且取材部位隨著與毛囊距離的增加而呈減少趨勢[17],另外,毛干外層的角質蛋白對酸、堿和很多化學物質都具有較好的抗性,裂解非常困難,所以毛絨DNA提取一直非常困難。有關動物毛絨DNA提取方法有一些報道,如PCR緩沖液法、SDS-蛋白酶K法,試劑盒法等都容易從毛囊部位提取DNA[6],提取毛干DNA比較成功的方法有chelex-100法、堿裂解法和 QIAamp@DNA Investigator試劑盒法,從安全性和價格上考慮,chelex-100法仍是毛絨DNA提取的首選。無論何種方法,關鍵是消化毛干外層的角質層,液氮研磨毛干,chelex -100結合蛋白酶K有助于增加蛋白酶K與毛絨的接觸面積,破壞毛干保護層,從而提高DNA的抽提效率。
線粒體DNA是高等動物唯一的核外遺傳物質,所有組織細胞中均含有大量的線粒體,mtDNA 主要以編碼序列構成,種內的異質基因很少[18],而在不同的物種間具有高度的變異性[19]。山羊、綿羊都屬于牛科山羊亞科,說明它們之間存在著很近的親緣關系[20]。 而Cyt b基因是線粒體自身編碼的為數不多的蛋白質之一,其進化速度適中,進化模式清楚,加上能用一些通用引物擴增,是鑒別親緣關系較近品種的理想工具之一[21-23]。
基因芯片檢測的關鍵之一在于設計出特異性探針,探針的特異性和引物的通用性是成功建立基因芯片檢測技術的關鍵[24-26]。由于山羊和綿羊mtDNA基因組的相似度達到了90%,在通用引物的擴增區間設計特異性探針,具有一定困難。本研究所用的通用引物擴增長度為500 bp,山羊和綿羊的相似度達到了96%,探針的設計除了使用軟件外,還需手動增減堿基,然后用 NCBI的blast驗證特異性,最后經試驗雜交驗證才能最終確定出特異性強的探針。芯片檢測過程中的各項條件也是影響檢測結果的因素,合適的反應條件可以降低雜交背景、提高雜交特異性,嚴格控制溫度和試劑的離子強度是避免假陽性和假陰性結果的先決條件,在雜交過程中,溫度太低或離子強度太高都會降低雜交的嚴格性,還會影響檢測信號的特異性。相反,提高溫度或降低離子強度會增加雜交的嚴格性。研究發現雜交溫度應在42℃為宜,雜交時間2 h 即可,與雜交12 h 相比無顯著差異。
目前,國內用于羊絨羊毛鑒別依然采用傳統常規的國標方法[27],而分子鑒別手段有 SNP 分型檢測和 DNA 分析。SNP 操作復雜、所需時間長、步驟分散、易出錯、價格昂貴、專業要求高、難以實現高通量快速檢測等。而 DNA 分析常常是通過PCR法來實現,對通過 PCR 產物進行凝膠電泳完成檢測,必要時需進行酶切鑒定[28,29]或測序鑒定。基因芯片檢測最大的優點在于高通量,對于從事毛絨檢測的機構來說是非常必要的,因為毛絨檢測機構和進出口檢驗機構常常面臨成百上千的樣品,利用基因芯片進行檢測,可同時并行檢測大批量的樣品,大大地縮短了檢測的時間,節約了人力,提高了檢測的準確性。石豐運[9]等利用 mtDNA 16S rRNA 基因為目標基因,在通用引物擴增區間設計了16條特異性基因芯片檢測探針對16種動物源性成分進行檢測,體現出了基因芯片客觀、準確、高通量的優點。
4 結論
通過對山羊絨和綿羊絨DNA提取和基因芯片鑒別方法的研究,初步建立了一種快捷、特異、靈敏且穩定地鑒別綿羊絨和山羊絨的基因芯片方法,可以應用于山羊絨與綿羊絨的定性判斷。
[1] 楊桂芬, 付妍, 紅霞, 等. 掃描電子顯微鏡與光學顯微鏡鑒別山羊絨纖維的技術探討[J].中國纖檢, 2006, 6:15-20.
[2] 倪廣菊, 張毅. 細羊毛與羊絨纖維的鑒別[J]. 現代紡織技術, 2004, 12(4):40-42.
[3] 胡志宇. 多參數毛絨類纖維圖像識別系統研究[M]. 北京:北京服裝學院, 2005.
[4] 趙國, 徐靜. 利用近紅外光譜技術進行羊毛、羊絨鑒別[J].毛紡科技, 2006(1):42-45.
[5] 吳桂芳, 朱登勝, 何勇, 等. 可見-近紅外光譜用于鑒別山羊絨與細支綿羊毛的研究[J]. 光譜學與光譜分析, 2008, 28(6):1260-1263.
[6] 金美菊, 阮勇, 石東亮, 等. 羊絨與羊毛纖維的鑒別檢測綜述[J]. 山東紡織科技, 2007, 48(4):28-30.
[7] 鄭文新, 王樂, 趙曉麗. 關于“綿羊絨”纖維分類以及名稱的考證研究[J]. 草食家畜, 2009, 2:4-9.
[8] 王曉梅, 楊秀榮.DNA分子標記研究進展[J]. 天津農學院學報, 2000, 7(1):21-24.
[9] 石豐運. 應用基因芯片技術鑒別檢測動物源性成分[D].蘭州:甘肅農業大學, 2010.
[10] 丁耀忠, 劉永生, 張杰.基因芯片技術在口蹄疫檢測中的應用[J]. 浙江農業科學, 2009, 2:423-426.
[11] Wang LC, Pan CH, Severinghaus LL, et al. Simultaneous detection and differentiation of Newcastle disease and avian influenza viruses using oligo nucleotide microarrays[J]. Vet Microbiol, 2008, 127(34):217-226.
[12] 楊忠蘋, 王秀榮, 石霖, 等.區分禽流感病毒亞型診斷芯片的構建[J]. 中國動物檢疫, 2008, 25(10):29.
[13] Cowell JK, Hawthorn L. The application of microarray technology to the analysis of the cancer genome[J]. Curr Mol Med, 2007, 7(1):
103-120.
[14] Ito T, Tanaka E, Kadowaki T, et al. An ultrasensitive new DNA microarray chip provides gene expression profiles for preoperative esophageal cancer biopsies without RNA amplification[J]. Oncology, 2007, 73(5):366-375.
[15] 賴銘裕. 基因芯片及其在腫瘤研究中的應用[J]. 醫學綜述, 2009, 13:1960-1962.
[16] 于鳳池. 基因芯片技術及其在植物研究中的應用[J]. 中國農學通報, 2009, 6:64-65.
[17] 華育平, 張瓊, 徐艷, 等. 細胞色素b基因序列虎物種特異性鑒定的PCR方法研究[J]. 獸類學報, 2004, 24(2):103-108.
[18] Momcilovic D, Rasooly A. Detection and analysis of animal materials in food and feed[J]. Journal of Food Protection, 2000, 63:1602-1609.
[19] Tartaglia M, Saulle E, Pestalozza S, et al. Detection of bovine mitochondrial DNA in ruminant feeds:a molecular approach to test for the presence of bovine-derived materials[J]. Journal of Food Protection, 1998, 61:513-518.
[20] 李均祥. 巖羊染色體核型研究[J]. 甘肅畜牧獸醫, 1999, 29, 4:13-15.
[21] Kouch TD, Thomas WKA, Meyer SV, et al. Dynamics of mitochondrial DNA evolution in animal:amplification and sequencing with conserved primers[J]. Proc Acad Sci USA, 1989, 86:6190-6200.
[22] Irwin DM, Kocher TD, Wilson AC, et al. Evolution of cytochrome b gene of mammals[J]. J Mol Evol, 1991, 32:128-144.
[23] Vladimir OA, Poltoraus LA, Zhivotovsky V. Mitochondrial DNA sequence diversity in Russians[J]. Federation of European Biochemical Societies Letter, 1999, 197-201.
[24] Dorris DR, Nguyen A, Gieser L, et al. Oligo deoxyribose nucleotide probe accessibility on a three-dimensional DNA microarray surface and the effect of hybridization time on the accuracy of expression ratios[J]. BMC Biotechnol, 2003, 3:6.
[25] Chou CC, Chen CH, Lee TT, et al. Optimization of probe length and the number of probes per gene for optimal microarray analysis of gene expression[J]. Nucleic Acids Res, 2004, 32(12):99.
[26] Hans B, Stephan P. Specific and nonspecific hybridization of oligonucleotide probes on microarrays[J]. Biophys J, 2005, 89:337-352.
[27] 曹渭芳, 楊桂芬, 邱桂芬, 等. GB/T 14593-2008 山羊絨、綿羊毛及其混合纖維定量分析方法掃描電鏡法[S]. 北京:中國標準出版社, 2008:11.
[28] Subramanian S, Karthik T, Vijayaraaghavan NN. Single nucleotide polymorphism for animal fiber identification[J]. Journal of Biotechnology, 2005, 116(2):153-158.
[29] 金美菊, 阮勇, 石東亮, 等. 單核苷酸多態性技術在羊絨、羊毛纖維鑒別中的應用[J]. 毛紡科技, 2007, 11:10-14.
(責任編輯 李楠)
Identification of Cashmere and Sheep down by Microarray Technology
Lü Xuefeng Abliz Ablimit Pa Naer Tao Weidong Xing Weiting Cai Fula Zheng Wenxin
(Department of Agriculture-Breeding Sheep and Wool and Cashmere Quality Supervision Test Center,Urumqi 830000)
This study is to explore the method for identification of cashmere and sheep down by gene chip, thereby enabling rapid, highthroughput identification of cashmere and sheep down. According to the specificity of the genetic material of cashmere and sheep down, cyt b was selected as the target gene, a pair of probes that can identify cashmere and sheep down was designed in the universal primers interval of cyt b gene. Then PCR products of different proportions derived from sheep were hybridized with probe. Results showed that in goat derived PCR products, when content of PCR products derived from sheep was 3%, hybridization signal was still visible. It indicates that the gene chip method can be used to qualitatively identify the cashmere and sheep down, and has a very high specificity.
Cashmere Sheep down Mitochondria DNA Cyt b gene Gene chip
2013-10-07
新疆維吾爾自治區公益性科研院所基本科研業務經費
呂雪峰,男,中級,研究方向:動物遺傳育種;E-mail:lxf00700@163.com
鄭文新,男,研究員,研究方向:絨山羊育種與毛絨標準化質量控制技術;E-mail:zwx2020@126.com

