凡納濱對蝦(Litopenaeus vannamei)Runt轉錄因子的基因克隆及其功能分析*
王曉雯 梁 艷 邢 婧 劉旭雅 黃 倢①
(1. 中國海洋大學水產學院 青島 266003; 2. 農業部海洋漁業可持續發展重點實驗室 中國水產科學研究院黃海水產研究所青島 266071)
Runt轉錄因子家族(RUNX)是一種在細胞增殖、分化和維持干細胞方面起著重要作用的細胞轉錄因子(Braunet al, 2009; Cohen, 2009), 普遍存在于哺乳動物、無脊椎動物, 具有進化保守性。據文獻報道, 第一個Runt基因是從果蠅中被發現的, 它編碼一個Runt蛋白的一個結構域(Gergenet al, 1988)。對不同物種的Runx基因研究顯示, 其編碼的蛋白產物都由α、β亞單位構成異二聚體。α亞單位含有一個128個氨基酸組成的RD (Runt domain)保守域, 可介導RD與DNA結合及蛋白之間的相互作用(Bartfeldet al,2002; Miyazonoet al, 2004)。目前在哺乳動物體內發現了3種Runx基因(runx1, runx2, runx3), 其中Runx1在造血系統中起著重要作用, Runx2與骨生成作用有關, Runx3則控制胃上皮細胞的增殖(Specket al,1999)。現在對哺乳動物(尤其是人類)Runt基因功能的研究已經很詳細, 更多的研究轉向無脊椎動物方面, 以了解Runt在分化過程中更為原始的蛋白功能。在果蠅的研究中發現晶體細胞(crystal cells)區別于漿血細胞(plasmatocytes), 不參與細胞吞噬, 而是通過發生黑化來參與免疫反應。晶體細胞可以表達果蠅分化必需的細胞因子Lz蛋白(一種Runt家族蛋白), 而在缺乏Lz表達的生物體內則缺乏晶體細胞(Rizkiet al, 1959; Canonet al, 2000; Lebetskyet al, 2000)。
無脊椎動物沒有特異性免疫系統, 當體外受到刺激或者造成對自身健康機體威脅時, 血細胞會急劇減少。迅速地恢復血細胞, 發揮吞噬作用、黑化作用等清除異物的功能, 對機體來說很關鍵(S?derh?llet al, 1998; Johanssonet al, 2000)。因此造血作用和釋放血細胞到循環系統在無脊椎動物體內發揮著最重要的免疫功能。Runt結構域蛋白被證實參與了哺乳動物和果蠅的造血作用和促進血細胞分化作用。在軟尾太平蝲蛄(Pacifastacus leniusculus) Runt蛋白的研究中發現注射外源葡聚糖(β-1,3-glucan), 可在短時間內顯著提高造血細胞中Runt的轉錄量, 而proPO卻幾乎檢測不到。另外, Runt轉錄發生在顆粒細胞和半顆粒細胞中, 可能是造血細胞成熟為顆粒細胞的指示基因(S?derh?llet al, 2003), 在調節和維持血細胞數量上有著重要作用。目前尚未有關于對蝦類Runt家族蛋白的相關研究報道。
在本研究中, 作者克隆得到了凡納濱對蝦Runt基因的cDNA全長, 分析了其在不同組織中的轉錄分布, 以及人工注射造血激素(Astakine, AST)和感染病毒后對其轉錄水平的影響, 以期為進一步了解Runt轉錄因子在無脊椎甲殼類動物中的功能提供分析數據。
1 材料與方法
1.1 凡納濱對蝦
凡納濱對蝦于2012年7月20日購自青島寶榮水產公司, 體長約8cm, 25尾/組, 暫養于50L水體中,鹽度25, 溫度26°C, 每天換水1/2, 投喂一次飼料,暫養一周。按國標方法經PCR檢測無WSSV感染(GB/T 28630.2-2012)。
1.2 白斑綜合征病毒(WSSV)毒種液
取100g本實驗室-80°C保存的感染WSSV的凡納濱對蝦去除肝胰腺的頭胸甲, 剪碎, 加入預冷的TESP緩沖液(50mmol/L Tris-HCl, 5mmol/L EDTA,500mmol/L NaCl, pH 8.5, 使用時加入1×蛋白酶抑制劑PMSF), 10000r/min高速勻漿5s, 8000r/min 4°C下離心5min, 取上清過篩絹, 用0.45μm濾膜過濾除菌,分裝并保存于-80°C冰箱待用。
1.3 凡納濱對蝦血細胞RNA的提取
取凡納濱對蝦血淋巴400μL與檸檬酸鈉抗凝劑(27mmol/L檸檬酸鈉, 336mmol/L NaCl, 115mmol/L葡萄糖, 9mmol/L EDTA.2Na)按1︰1比例混合, 1200r/min離心10min, 收集細胞沉淀, 加入800μL TRIzol (Takara),按照其附的操作方法采用氯仿抽提法提取總RNA,1%瓊脂糖凝膠電泳檢測。
1.4 凡納濱對蝦Runt(lvrunt)全長cDNA的克隆
1.4.1 同源克隆lvruntcDNA片段 根據NCBI數據庫中9個已知物種Runt基因序列(軟尾太平蝲蛄,AJ506096; 果蠅, AF217651和X56432; 紫色球海膽,U41512; 薩氏九杯蛛, AJ272529; 爪蟾, AF035446;原雞, Z36530; 小家鼠, D26532; 人, L34598), 設計1對簡并引物runt-F和runt-R (表1)。以凡納濱對蝦血淋巴細胞cDNA為模板進行同源克隆, 獲取Runt cDNA的部分片段。25μL反應體系中含10μL 10×Buffer, 8μL 2.5mmol/L dNTP, 各1μL的10mmol/L runt-F和runt-R, 0.2μL 5U/μL Ex Taq酶(Takara), 1 μL cDNA模板, 于94°C預變性5min, 94°C 30s, 55°C 30s, 72°C 30s, 35個循環, 72°C延伸10min。將擴增產物經過膠回收后進行TA克隆(Frolanmanet al, 1988),并將陽性克隆送北京六合華大基因科技股份有限公司進行序列測定。
根據同源克隆獲得的cDNA片段序列設計特異性引物GSPI、NP3、GSPII和NP5, 分別用于RACE實驗, 引物序列見表1。

表1 實驗中用到的引物序列Tab.1 Primers used in the experiment
1.4.2lvrunt基因的5′-RACE 5′-RACE擴增使用了商品試劑盒SMARTerTMRACE cDNA Amplification Kit (Clontech, 美國), 以提取的總RNA(濃度為200μg/μL)為模板, 按其使用手冊方法合成5′- RACEReady cDNA。以基因特異性引物GSPII (5′)和NP5分別與UPM(通用接頭引物, 試劑盒提供)為引物,5'-RACE-Ready cDNA為模板進行套式PCR擴增。20μL體系中含10μL 10×Buffer, 8μL 2.5mmol/L dNTP,各1μL的10mmol/L GSPII(5′)(NP5)和UPM, 0.2μL 5U/μL Ex Taq酶(TaKaRa), 1μL cDNA模板。擴增產物經TA克隆后送華大基因公司測序, 獲取lvruntcDNA的5′末端序列。
1.4.3lvrunt基因的3′-RACElvrunt基因的3′- RACE擴增使用了商品試劑盒Trans Script First-Strand cDNA Synthesis Super Mix Kit(TransGen, 北京), 以提取的總RNA(濃度為200μg/μL)為模板, 用接頭引物3′CDs-primerA取代試劑盒中的Oligo(dT)18, 合成3′-RACE-cDNA。20μL體系中含500ng RNA模板,10pmol 3′CDs-primerA, 10μL 2×TS Reaction Mix,1μL Enzyme Mix, 42°C保溫30 min后, 于85°C終止反應5min, 冰上冷卻。以基因特異性引物GSPI(3′)和UPM(試劑盒提供)為引物, 3′-RACE-cDNA為模板,進行第一輪PCR; 再以此擴增產物為模板, 用基因特異性引物NP3和接頭引物UPM, 進行套式PCR, 2次PCR反應程序為: 94°C預變性5min; 94°C 30s, 68°C 30s, 72°C 40s循環數為10; 94°C 30s, 65°C 30s, 72°C 40s, 循環數為25; 72°C延伸10min。擴增產物經TA克隆后并測序, 以獲得lvruntcDNA3′端序列。
1.5 lvrunt全長cDNA序列及其編碼的多肽序列分析
用NCBI數據庫和在線SMART軟件(http://smart.embl-heidelberg.de)分析全長lvruntcDNA序列及其編碼的多肽序列, 利用Clustaxl軟件對該基因編碼氨基酸和其它種類的runt-family編碼的氨基酸序列進行比對, 利用MEGA5.1軟件建立進化樹。
1.6 lvrunt基因的組織表達分析
隨機選取凡納濱對蝦3尾, 分別采集其鰓、腸、肝胰腺、類淋巴、心臟、肌肉組織和血淋巴, 按1.4方法用氯仿抽提法提取總RNA。以提取的總RNA (濃度為300μg/μL)為模板, 采用Trans Script First-Strand cDNA Synthesis Super Mix試劑盒(TransGen, 北京),以Oligo(dT)為反轉錄引物, 按試劑盒操作手冊合成cDNA。根據已得到的lvrunt基因序列設計引物(Lvrunt-F, Lvrunt-R), 并以凡納濱對蝦18S rRNA (引物設計為18S-F, 18S-R)作為內參, 使用Bio-Rad熒光定量PCR儀, 對各組織lvrunt表達量進行分析, 熒光定量PCR反應條件為: 95°C預變性5min, 95°C 15s,61°C 30s, 40個循環。設定腸中該基因的表達量為“1”,結果采用2–ΔΔCt法計算, 用SPSS軟件進行數據分析。
1.7 注射重組表達的凡納濱對蝦造血激素(rLvAST)對lvrunt基因轉錄水平的影響
選用凡納濱對蝦225尾(25尾/箱)分為三個實驗組, 每組3個平行, 各25尾, 分別于第二腹節肌肉注射50μL的PBS、BSA(50μg)和BSA與rLvAST(各25μg)混合物, 每組3個平行。分別于注射后0、3、6、12、24、36、48和72h采集2尾對蝦的血淋巴, 按1.6方法測定lvrunt表達量。
軟件定義天地一體化網絡:架構、技術及挑戰…………………………許方敏,仝宗健,趙成林,秦智超 24-2-59
1.8 白斑綜合征病毒感染后凡納濱對蝦Runt基因的表達分析
選取凡納濱對蝦30尾, 在對蝦第二腹節肌肉內注射20μL稀釋了1000倍的WSSV毒種液, 感染凡納濱對蝦, 于感染后0、1、7、12h和3、5和10d抽取對蝦血淋巴, 按1.6方法檢測lvrunt表達量。
2 結果
2.1 凡納濱對蝦Runt基因(lvrunt)全長的擴增
通過在線軟件CODEHOP設計了不同種類Runt基因的保守區上下游簡并引物, 對從凡納濱對蝦血淋巴細胞mRNA反轉錄所得的cDNA進行擴增, 得到174bp產物(圖1a), 經測序和初步分析, 序列所編碼的氨基酸序列與Runt基因保守區編碼的氨基酸序列相似, 確定所獲產物為lvrunt片段的部分序列。
根據已知的lvrunt部分序列進行5′-RACE, 設計外側引物GSPII (5′), 與接頭引物進行第一次擴增,得到了一條大約500bp的條帶(圖1b)。將第一次擴增所得產物稀釋10倍作為模板, 用內側引物NP5進行套式PCR, 獲得約500bp的特異條帶(圖1c), 經測序獲得核苷酸序列466bp。
根據已知的lvrunt部分序列進行3′-RACE, 設計外側引物GSPI (3′), 與接頭引物UPM一起對凡納濱對蝦血淋巴來源的cDNA進行第一次擴增, 獲得彌散分布的產物(圖1d), 再以第一次擴增產物的1/50稀釋度為模板, 用內側引物NP3進行套式PCR, 獲得約1000bp的特異條帶(圖1e), 經測序, 得到1040bp核苷酸序列。
根據5′-RACE和3′-RACE所得序列, 拼接得到lvrunt的全長序列。
2.2 lvrunt全長序列特征
拼接所得的凡納濱對蝦runt的cDNA全長序列共1754bp(圖2), 經分析, 該全長序列中含有1個663bp開放閱讀框(ORF), 編碼221aa, 理論分子量為23.6kDa, 5′-UTR (非編碼區)為466bp, 3′-UTR(非編碼區)為625bp, 其中包含1個PolyA尾, 并出現一加尾信號。
2.3 凡納濱對蝦轉錄因子(LvRunt)蛋白結構特征分析
凡納濱對蝦轉錄因子(LvRunt)通過估算, 分子量為23.6kDa, 預測的等電點為7.57。通過SMART、NCBI、OMIGA的分析表明LvRunt蛋白序列的第4—148個氨基酸范圍內是一個runt-family結構域(圖3),53—139aa為IG_FLMN域(細絲蛋白型的免疫球蛋白域), 62—89aa為KOW模體, 104—111aa為ATP/GTP結合模體, 46—50aa為DNA結合區域, 210—219aa是低復雜性區域(LCR)。

圖1 凡納濱對蝦Runt基因全長的分段擴增結果Fig.1 Amplification of the fragments of L. vannamei Runt gene full-length

圖2 凡納濱對蝦Runt基因cDNA全長序列及編碼氨基酸序列Fig.2 The full-length nucleotide sequence and its deduced amino acid sequence of L. vannamei runt gene cDNA
2.4 不同種類Runt保守性分析
從GenBank上獲得了分別屬于扁形動物門、節肢動物動物、半索動物和脊椎動物的16個不同物種的Runt蛋白氨基酸序列, 通過Mega 5.05軟件進行保守性分析(圖4)。結果表明凡納濱對蝦Runt氨基酸序列與軟尾太平蛄(Pacifastacus leniusculus)(CAD 44570.1)的相似度為88.1%, 與麗蠅蛹集金小蜂(Nasonia vitripennis)(XP_001603414.2)有74.3%的相似度, 與埃及伊蚊(Aedes aegypt)(XP_001657548.1)有61%的相似度, 與其他種類的氨基酸序列相似性在30%—52.4%范圍內, 該蛋白表現較高的種間保守性。另外,這些Runt蛋白均有一個runt-family結構域runt domain(RD)。凡納濱對蝦Runt蛋白的RD與軟尾太平蝲蛄的相似度高達97.7%, 與其他物種蛋白的相似度在72.3%—97.7%(圖5)。
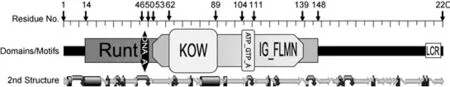
圖3 凡納濱對蝦runt氨基酸序列的SMART蛋白功能分析及二級結構預測Fig.3 The SMART analysis for the protein function and the secondary structure prediction on the ammonia acid sequence of the L.vannamei runt
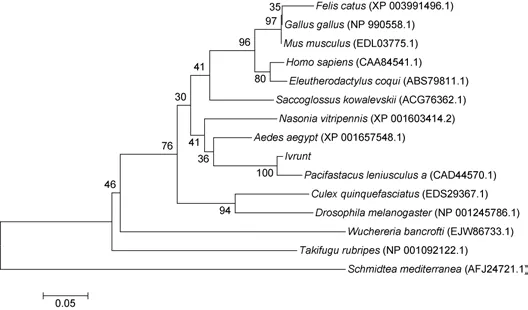
圖4 利用NJ法繪制的不同動物的Runt同源蛋白進化樹Fig.4 Neighbor-joining phylogenic tree of amino acid sequences of runt genes and its homologous gene runx from different species
2.5 lvrunt基因在凡納濱對蝦體內的不同組織表達差異
以18S rRNA為目的基因, 通過實時熒光定量PCR對lvrunt在凡納濱對蝦不同組織的表達分析, 檢測到這7種組織均有lvruntmRNA的表達。該實驗經過溶解曲線驗證了擴增的特異性, 不存在非特異性擴增及引物二聚體的干擾。根據2-ΔΔCt方法對結果進行分析, 以腸組織作為校正樣品(Calibrator), 表達量設為1, 比較不同組織中lvrunt在凡納濱對蝦組織中的表達(圖6)。結果顯示lvruntRNA在血淋巴中表達量最大, 顯著高于其他組織, 是其他組織中表達量的20—346倍。由于對蝦是開放的循環系統, 各組織中均有血淋巴細胞的存在, 推測其他組織中lvrunt的表達很可能都是由于組織中血淋巴細胞的表達所致,換而言之,lvrunt基因的表達可能作為組織中血淋巴細胞量的一種指示。
2.6 注射對蝦造血激素(Astakine)后lvrunt基因轉錄水平變化
對凡納濱對蝦注射PBS、BSA和BSA+rLvAST的三個組的lvrunt轉錄量均在注射6h達到峰值(圖7),BSA+AST組的lvrunt表達量急升到原來的接近600倍, 12h內下降并基本維持穩定, 但72h內仍然高于初始基本水平。其中BSA和BSA+AST注射組Runt基因表達量明顯高于PBS注射組, 而BSA+AST注射組lvrunt基因表達量約為BSA注射組的3.35倍, 差異顯著(P<0.05)。

圖5 不同種類的Runt結構域氨基酸序列比對Fig.5 Sequence alignment of the Runt domain in different species
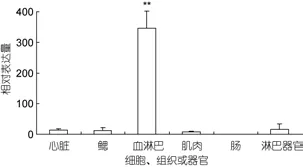
圖6 實時熒光RT-PCR對lvrunt在凡納濱對蝦不同組織中的相對表達量分析Fig.6 Quantitative analysis of the lvrunt mRNA in different tissues of L. vannamei by real-time RT-PCR
2.7 注射WSSV后lvrunt基因的表達變化
凡納濱對蝦經注射WSSV毒種液后, 其血淋巴中的lvrunt表達經熒光定量PCR分析(圖8), 顯示在注射后12h, 凡納濱對蝦血細胞的lvrunt轉錄量達到最大值, 達到正常值的25倍左右。但隨著時間延長, 在感染后第3天顯著下降, 但仍比初始水平高約5倍左右, 隨著感染進程的延伸,lvrunt的表達量又逐漸升高, 在10d時達到初始水平的10倍左右。
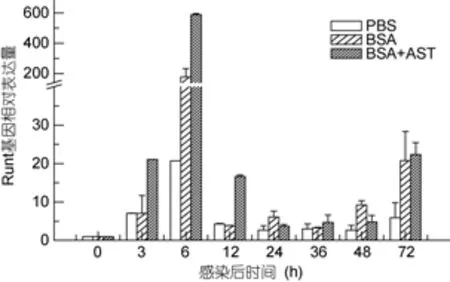
圖7 注射造血激素后凡納濱對蝦血淋巴中Runt轉錄水平情況Fig.7 Expression levels of lvrunt in hemocytes after injection of rLvAST
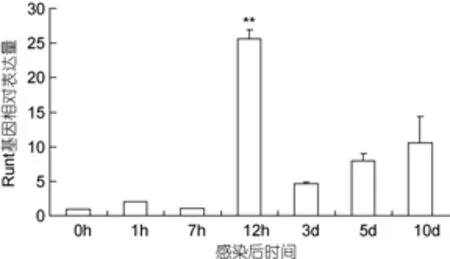
圖8 感染WSSV后凡納濱對蝦血淋巴Runt基因轉錄量分析Fig.8 Temporal expression patterns of lvrunt in hemocytes after challenged with WSSV
3 討論
Runt基因是普遍存在于多種生物的一種轉錄因子, 含有一個DNA結合域(Runt Domain), 這個結合域從昆蟲到哺乳動物都高度保守(Kagoshimaet al,1993; Wolffet al, 1999; Kataokaet al, 2000)。本研究克隆得到的凡納濱對蝦的Runt蛋白序列中就包含了這個runt family功能區域, 并與軟尾太平喇姑有高達97.7%的相似度, 與其他種類也是相對保守的, 這可能與Runt在不同生物的生命活動中發揮的作用相似有關。據報道, 最早發現的果蠅體內的Runt結構域基因runt和lozenge可以在生長過程中發揮多種作用,并且lozenge可以促進晶體血細胞的分化(Canonet al,2000)。對哺乳動物的研究發現了三類Runt基因, 證明是造血作用和骨生成必不可少的因子(Westendorfet al, 1999; Linggiet al, 2002; Ottoet al, 2002)。還有研究還表明, Runt基因的缺失會影響細胞周期蛋白B(cyclin B)與細胞周期蛋白依賴性激酶cdc 2(cell division cycle)復合物的活性, cyclin B-cdc 2復合物不僅決定細胞是否進入M期, 也決定細胞是否能退出M期。如果細胞不進入M期它將不進行分裂, 如果在M期退不出來會使細胞過度增殖, 產生腫瘤等有害影響(翟中和等, 2002; 曾亞, 2004)。本文克隆結果表明, 凡納濱對蝦僅存在一種Runt基因(lvrunt), 這與兩棲動物和海膽中只存在一種基因類型的報道是一致的(葛輝等, 2007)。lvrunt序列與軟尾太平蝲蛄相比, 在Runt結構域相似度很高, 但是RD下游的序列差異較大, 并且沒有Runt基因常見的VWRPY序列作為C末端, 這一motif不會或者很少會影響轉錄活性(Okudaet al, 2000), 可能會在促進DNA結合的功能上產生一些差異(Kagoshimaet al, 1993)。
對凡納濱對蝦Runt基因的不同組織表達分析顯示,lvrunt幾乎只在血淋巴有表達, 在其他組織的表達量遠遠低于血淋巴, 可以認為是分析其他組織時候有部分血細胞存在的影響, 因此Runt的表達量也可能作為組織中血細胞數量的一個分子指標。表達組織分布結果也揭示了lvrunt在血液系統中發揮著主要作用, 這與Runt在果蠅內促進血細胞分化, Runx1在高等哺乳動物體內具有造血功能的研究結果相吻合(Specket al, 1999)。
已有研究表明, 造血激素在螯蝦體內有促進造血和血細胞分化的作用, 而Runt也有促進轉錄和血細胞分化作用。實驗中注射了造血激素后, 對蝦血淋巴中lvrunt轉錄水平顯著增加, 可見造血激素促進了Runt基因轉錄。尤其是在注射后6hlvrunt轉錄水平達到最高值, 這樣可能會通過細胞信號傳導, 進一步促進細胞進入分裂狀態。對凡納濱對蝦注射WSSV后12h,lvrunt轉錄水平達到峰值。有研究顯示, 斑節對蝦感染WSSV后12h總血淋巴數(THC)達到一個最低值, 但這時WSSV在體內的感染應該還處于潛伏期, 病毒含量沒有達到很高水平(張濤等, 2012), 而在3—10d的時期是WSSV感染的高峰期,lvrunt的表達卻比12h時低得多, 這提示lvrunt的表達不是因為WSSV感染所致, 12h時lvrunt的高表達可能是由于血淋巴細胞對異物刺激發生反應, 數目急劇下降, 通過活化lvrunt的轉錄, 來促進細胞的分裂和分化, 以使機體有能力參與免疫反應。
本文通過RACE方法克隆得到了凡納濱對蝦的Runt基因的序列, 是首個關于對蝦屬的相關研究。本文還通過該基因的組織表達分布特征分析, 揭示其主要在血淋巴細胞表達, 這可能與其作用有關。并且采用熒光定量 PCR的方法分析了凡納濱對蝦在注射rLvAST和經過WSSV感染后的轉錄變化, 初步了解lvrunt的表達狀況, 為更深入了解對蝦的造血機制以及參與對蝦免疫作用奠定了基礎。
張 濤, 郭志勛, 黃建華等, 2012. 低劑量對蝦白斑綜合征病毒粗提液對斑節對蝦體內潛伏期病毒及血細胞的影響.水產學報, 36(10): 1544—1552
葛 輝, 王秀利, 仇雪梅等, 2007. 海膽Runt基因保守序列的克隆與初步分析. 生物技術通報, 4: 125—127
曾 亞, 2004. 國外醫學——生理、病理科學與臨床分冊, 22(2):103—105
翟中和, 王喜忠, 丁明孝, 2000. 細胞生物學. 北京: 高等教育出版社, 358—372
Bartfeld D, Shimon L, Graeme C Coutureet al, 2002. DNA recognition by the RUNX1 transcription factor is mediated by an allosteric transition in the RUNT domain and by DNA bending. Structure, 10: 1395—1407
Braun T, Woollard A, 2009. RUNX factors in development:lessons from invertebrate model systems. Blood Cells,Molecules, and Diseases, 43: 43—48
Canon J, Banerjee U, 2000. Runt and Lozenge function inDrosophiladevelopment. Semin Cell Dev Biol, 11(5): 327—336
Cohen M M Jr, 2009. Perspectives on RUNX genes: an update.American Journal of Medical Genetics Part A, 149A: 2629—2646
Gergen J P, Butler B A, 1988. Isolation of theDrosophilasegmentation gene runt and analysis of its expression during embryogenesis. Genes & Development, 2: 1179—1193
Johansson M W, Keyser P, Sritunyaluksana Ket al, 2000.Crustacean hemocytes and haematopoiesis. Aquaculture, 191:45—52
Kagoshima H, Shigesada K, Satake Met al, 1993. The runt domain identifies a new family of heterometric transcriptional regulators. Trends in Genetics, 9(10): 338—341
Kataoka H, Ochi M, Enomoto Ket al, 2000. Cloning and embryonic expression patterns of the zebrafish Runt domain genes, runxa and runxb. Mechanisms of Development, 98:139—143
Lebetsky T, Chang T, Hartenstein Vet al, 2000. Specification ofDrosophilahematopoietic lineage by conserved transcription factors. Science, 288: 146—149
Linggi B, Muller-Tidow C, Van de Locht Let al, 2002. The fusion protein, AML1 ETO, specifically represses the transcription of the p14(ARF) tumor suppressor in acute myeloid leukemia. Nature Medicine, 8: 743—750
Miyazono K, Maeda S, 2004. Imamura T coordinate regulation of cell growth and differentiation by TGF-β superfamily and Runx proteins. Oncogene, 23: 4232—4237
Okuda T, Takeda K, Fujita Yet al, 2000. Biological characteristics of the leukemia-associated transcriptional factor AML1 disclosed by hematopoietic rescue of AML1-deficient embryonic stem cells by using knock-in strategy. Molecular and Cellular Biochemistry, 20: 319—328
Otto F, Kanegane H, Mundlos S, 2002. Mutations in the RUNX2 gene in patients with cleidocranial dysplasia. Human Mutation, 19: 209—216
Rizki M T M, Rizki R, 1959. Functional significance of the crystal cells in the larva ofDrosophila melanogaster.Cytology, 5: 235—240
S?derh?ll I, Bangyeekhun E, Mayo Set al, 2003. Hemocyte production and maturation in an invertebrate animalproliferation and gene expression in heatopoietic stem cells ofPacifastacus leniusculus. Developmental and Comparative Immunology, 27: 661—672
S?derh?ll K, Cerenius L, 1998. Role of phenoloxidase activating system in invertebrate immunity. Current Opinion in Immunology, 10: 23—28
Speck N A, Stacy T, Wang Qet al, 1999. Core-binding factor: a central player in hematopoiesis and leukemia. Cancer Research, 59: 1789—1793
Westendorf J J, Hiebert S W, 1999. Mammalian runt-domain proteins and their roles in hematopoiesis, osteogenesis, and leukemia. Journal of Cellular Biochemistry-Supplement,32/33: 51—58
Wolff C, Pepling M, Gergen Pet al, 1999. Structure and evolution of a pair-rule interaction element: runt regulatory sequences inD. melanogasterandD. virilis. Mechanisms of Development, 80(1): 87—99
- 海洋與湖沼的其它文章
- 大菱鲆(Scophthalmus maximus)、星斑川鰈(Platichthys stellatus)及其雜交種的形態學分析*
- 半滑舌鰨(Cynoglossus semilaevis Günther)生長激素體外重組表達及活性分析*
- 香港牡蠣(Crassostrea hongkongensis)與長牡蠣(Crassostrea gigas)種間雜種遺傳力評估*
- 三疣梭子蟹(Portunus trituberculatus)胞內氯離子通道蛋白基因克隆及其表達分析*
- 褪黑激素對香港細首紐蟲(Cephalothrix hongkongiensis)性腺發育的影響及羥基吲哚-O-甲基轉移酶(HIOMT)的測定*
- 飼養空間大小對單體筐養養殖系統中三疣梭子蟹(Portunus trituberculatus)的攝食行為與生長特性的影響研究*

