三疣梭子蟹(Portunus trituberculatus)胞內氯離子通道蛋白基因克隆及其表達分析*
王 渝 呂建建 劉 萍① 高保全 李 健 陳 萍
(1. 農業部海洋漁業可持續發展重點實驗室 中國水產科學研究院黃海水產研究所 青島 266071;2. 上海海洋大學水產與生命學院 上海 201306)
三疣梭子蟹(Portunus trituberculatus), 俗稱梭子蟹, 隸屬于甲殼綱(Crustacea)、十足目(Decapoda)、梭子蟹科(Portunidae)、梭子蟹屬(Portunus), 是中國沿海的重要經濟蟹類, 其可以存活于鹽度為13.7—47.7的水環境中(隋延鳴等, 2012), 屬典型廣鹽性蟹類(孫穎民等, 1984)。在三疣梭子蟹的育苗過程中, 鹽度微小的變化都可能導致變態的推遲、活力變差, 乃至病害的發生(季東升, 2005)。在三疣梭子蟹養成過程鹽度也起到了重要作用, 對其存活、攝餌和生長均具有重要影響(呂富等, 2010; 路允良等, 2012)。然而, 在三疣梭子蟹養殖過程中, 降雨會改變水體的鹽度, 因此養殖過程中產生的鹽度脅迫是不可避免的。一般認為造成鹽度脅迫的主要物質是NaCl, 在其脅迫下三疣梭子蟹體內的Cl–含量會超出正常需要, 進而對機體產生脅迫作用(康敏華等, 2011)。然而, 目前在三疣梭子蟹中滲透壓相關的研究主要集中在酶活、免疫、蛻皮和能量收支等方面(鄭萍萍等, 2010; 江山等, 2011),涉及Cl–的滲透壓調節分子機理的研究則相對較少。因此, 從分子水平研究三疣梭子蟹氯離子通道對Cl–運輸、吸收和排除顯得尤為重要(康敏華等, 2011)。
氯離子通道是分布于細胞膜或細胞器質膜上的一類能夠轉運氯離子及其它陰離子的通道蛋白(陳麗娥等, 2010)。研究表明, 氯離子通道可調控不同的生理過程和細胞功能, 如: Cl–運輸、滲透壓調節、離子穩態、胞內pH、細胞容積調節、電興奮性、金屬耐性和信號識別與轉導(Uchidaet al, 1993; Adachiet al,1994; Hechenbergeret al, 1996), 甚至在細胞遷移、細胞增殖和分化等過程中也起到一定作用(Jentschet al,1999)。氯離子通道分為三種類型: 氯通道(chloride channel, CLC)、囊性纖維化跨膜傳導調解因子(cystic fibrosis transmembrane conductance regulator, CFTR)和胞內氯離子通道(chloride intracellular channel,CLIC) (戴寒晶, 2006)。CLIC家族成員大多數為單次跨膜蛋白, 與谷胱甘肽-S-轉移酶(glutathione-S-transferase, GST)具有一定的同源性(Dulhuntyet al,2001)。Valenzuela等(1997)從人類單核細胞中克隆了第一個CLIC1基因。現在已經發現了六個CLIC家族成員, 包括CLIC1 (Valenzuelaet al, 1997)、CLIC2(Heisset al, 1997)、CLIC3 (Qianet al, 1999)、CLIC4(Edwards, 1999)、CLIC5 (Berrymanet al, 2000)和CLIC6 (Perluigiet al, 2002), 各成員之間高度保守。Berry等(2003)在線蟲中發現了編碼一種CLIC樣蛋白的基因EXC-4。目前關于氯離子通道蛋白的研究主要集中在哺乳動物、兩棲類、昆蟲和植物(Cornejoet al,2001; Shorninget al, 2003; Donget al, 2013), 在水產動物中研究較少, 而在甲殼動物中更是尚未見報道。
本研究從本實驗室構建的三疣梭子蟹轉錄組文庫(Lvet al, 2013)中篩選到胞內氯離子通道蛋白基因EST序列; 采用RACE技術克隆獲得該基因全長cDNA序列; 借助相關生物信息學軟件分析PtCLIC基因及其翻譯的氨基酸序列的結構; 通過Blast程序比對其與其它物種的CLIC基因氨基酸序列同源性;利用實時熒光定量技術研究其組織表達模式、低鹽脅迫進程中的表達規律以及低鹽脅迫下低鹽耐受和低鹽敏感家系中表達規律的差異。本研究為PtCLIC基因的生物學功能研究及其在滲透壓調節機理方面的研究奠定理論基礎, 同時也為三疣梭子蟹耐鹽新品系的選育提供理論指導。
1 材料與方法
1.1 材料
健康三疣梭子蟹取自昌邑市海豐水產養殖有限公司, 體質量(5.78±1.11)g。暫養于3m3的室內水泥池中, 每池90只, 共6池, 暫養3d。養殖水溫26°C, 鹽度33, pH 8.7, 持續充氧, 養殖用水每天更換1/3, 于18:00定時喂食藍蛤。
Trizol Reagent購自Invitrogen公司; SMARTTMRACE Amplification Kit和Advantage 2 PCR Kit購自Clontech公司; DNA膠回收試劑盒購自生工生物工程(上海)股份有限公司; SYBR Premix Ex Taq Ⅱ熒光定量試劑盒、PrimeScript RT reagent Kit、PMD18-T載體和DH5α感受態細胞均購自TaKaRa公司。
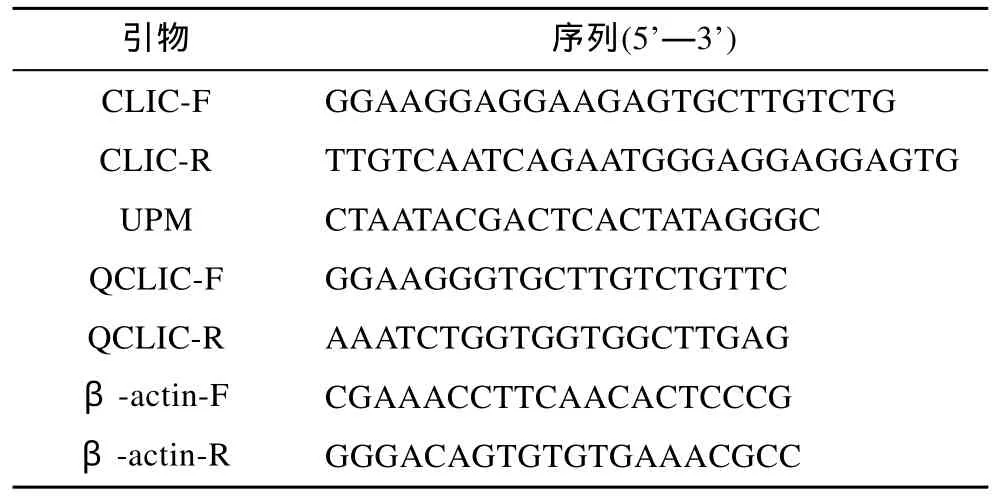
表1 實驗所用的引物序列Tab.1 All sequence of primers for the experiment
1.2 總RNA的提取及cDNA第一鏈的合成
Trizol試劑分別提取鰓、肝胰腺和肌肉組織的總RNA, 核酸定量儀與1.0%的瓊脂糖凝膠電泳檢測RNA的質量及完整性; 分別取等量的上述3種組織的總RNA均勻混合, 按照SMARTTM RACE Amplification Kit說明書上的方法分別合成3’和5’RACE的cDNA第一鏈。
1.3 PtCLIC基因全長cDNA的克隆及測序
3’RACE和5’RACE特異性引物利用從三疣梭子蟹轉錄組文庫中獲得的一段胞內氯離子通道蛋白基因EST序列和Primer Premier 5.0軟件設計, 并且委托生工生物工程(上海)股份有限公司合成。3’和5’末端的快速擴增使用試劑SMARTTMRACE Amplification Kit, Advantage 2 PCR Kit和兩對特異性引物對CLIC-F和UPM、CLIC-R和UPM進行(表1)。反應程序: 94°C 30s, 68°C 30s, 72°C 3min, 30個循環。
利用1.0%的瓊脂糖凝膠電泳分別檢測3’RACE和5’RACE的擴增產物, 使用膠回收試劑盒回收目的片段, 并連接入PMD18-T載體, 轉化入大腸桿菌DH5α感受態細胞, 挑取陽性克隆進行菌落PCR鑒定, 將含有目的條帶的菌液送往上海桑尼生物科技有限公司進行測序。
1.4 PtCLIC基因的生物信息學分析
利用Vector NTI 11.0軟件去除所測序列中的載體, 并與已知序列拼接。利用DNAStar的EditSeq程序預測開放閱讀框(ORF)并將其翻譯為氨基酸序列。使用BLAST (http://www.blast.ncbi.nlm.nih.gov/Blast.cgi)程序分別進行PtCLIC基因的核苷酸序列和推導的氨基酸序列同源性比對。利用ProtParam tool軟件進行蛋白質理化性質預測, 利用InterproScan軟件進行蛋白質功能結構域分析, 利用TMHMM 2.0軟件進行蛋白質跨膜結構域分析。使用Clustal X軟件對三疣梭子蟹PtCLIC氨基酸序列與其它物種的CLIC氨基酸序列進行多序列比對, 然后使用MEGA 4.0軟件,采用鄰接法構建系統進化樹。
1.5 鹽度脅迫實驗
隨機挑取暫養3d的三疣梭子蟹分為2組, 對照組(33)和低鹽組(11), 每個處理組設3個平行, 每個平行90只蟹。使用地下鹵水、自然海水和淡水配制各鹽度實驗用水, 使用YSI鹽度儀進行鹽度校準, 具體方法見隋延鳴等(2012)。各組分別于脅迫0、3、6、12、24、48和72h取鰓和肝胰腺, 放置液氮中冷凍保存, 用于后續RNA的提取, 每個時間點取3只。另取3只健康三疣梭子蟹的鰓、肝胰腺、肌肉、心臟、眼柄和血細胞, 放置液氮中冷凍保存, 用于后續RNA的提取。
1.6 家系低鹽脅迫實驗
從本實驗室構建的低鹽耐受家系和低鹽敏感家系中隨機選取三疣梭子蟹各90只, 脅迫鹽度為三疣梭子蟹半致死濃度(11), 每個家系設置3個平行, 每個平行30只。各組分別于脅迫0h、12h、72h取鰓組織, 放置液氮中冷凍保存, 用于后續RNA提取。
1.7 PtCLIC基因表達分析
使用Trizol提取不同實驗組三疣梭子蟹鰓和肝胰腺組織的總RNA, 使用PrimeScript RT reagent Kit反轉錄合成cDNA。
根據已獲得的三疣梭子蟹內參基因β-actin和PtCLIC基因全長序列, 分別設計兩對引物(β-actin-F和β-actin-R、QCLIC-F和QCLIC-R)(表1), 對不同時間點鹽度脅迫的三疣梭子蟹鰓和肝胰腺中PtCLIC基因的相對表達量進行檢測。使用SYBR Premix Ex TaqⅡ試劑在ABI 7500 Real Time PCR儀上利用Real-time PCR方法對各個組織中以及脅迫進程中PtCLIC基因的表達情況進行分析。熒光定量PCR反應體系為20μL, 包括10μL SYBR Premix Ex TaqTMⅡ(2×), 0.8μL 10μmol/L的引物QCLIC-F, 0.8μL 10μmol/L的引物QCLIC-R, 0.4μL ROX Reference DyeⅡ(50×), 2.0μL cDNA模板, 6.0μL無菌ddH2O。反應程序為: 95°C 30s; 95°C 5s, 60°C 34s, 40個循環;95°C 15s, 60°C 1min, 95°C 15s。采用2–△△CT方法計算PtCLIC基因的相對表達量, 使用SPSS 17.0軟件進行單因素方差分析。
2 結果
2.1 總RNA提取
利用Trizol試劑提取獲得三疣梭子蟹鰓、肝胰腺和肌肉組織的總RNA, 經核酸定量儀檢測, 其OD260/OD280均在1.9—2.0之間, 表明總RNA純度較高; 經1.0%瓊脂糖凝膠電泳檢測, 28S、18S、5S rRNA條帶清晰, 表明總RNA完整性較好, 質量較高。
2.2 PtCLIC基因的全長cDNA克隆
以三疣梭子蟹的鰓、肝胰腺和肌肉組織的等量總RNA混合物為模板, 反轉錄得到3’RACE和5’RACE的cDNA第一鏈。以特異引物CLIC-F和CLIC-R分別與通用引物UPM配對, 進行3’和5’RACE擴增。將測序獲得的3’和5’片段與已知EST序列進行拼接,得到三疣梭子蟹CLIC基因的全長cDNA序列, 命名為PtCLIC。該基因全長為2000 bp (GenBank登錄號為KJ186099), 包括76 bp的5’端非編碼區(UTR),1162 bp的3’端非編碼區和762 bp的開放閱讀框(ORF)。3’端含有一個PolyA尾和一個多聚腺苷酸加尾信號(AATAAA) (圖1)。
2.3 PtCLIC基因序列分析及結構域預測
DNAstar軟件分析表明,PtCLIC基因編碼一個由253個氨基酸組成的蛋白質, 分子量為29.2kDa, 理論等電點為5.93。ProtParam tool軟件分析表明,PtCLIC蛋白帶有負電的氨基酸殘基為36個(Asp和Glu), 帶有正電的氨基酸殘基為31個(Arg和Lys), 不穩定系數為46.34, 親水性平均數為-0.449, 屬于不穩定蛋白。InterProScan和TMHMM 2.0軟件分析表明,PtCLIC基因翻譯的蛋白質結構中無跨膜結構域。
2.4 PtCLIC基因的同源性分析
利用BLAST程序對三疣梭子蟹PtCLIC基因的氨基酸序列進行同源性比對, 發現三疣梭子蟹PtCLIC基因與蚤狀溞(Daphnia pulex)、點蜂緣蝽(Riptortus pedestris)、地中海實蠅(Ceratitis capitata)、埃及伊蚊(Aedes aegypti)和果蠅(Drosophila virilis)的同源性分別為83%、84%、82%、83%和82%。通過與上述物種的CLIC氨基酸序列比對發現,PtCLIC氨基酸序列保守性較高(圖2)。

圖1 三疣梭子蟹PtCLIC基因cDNA全長及其編碼的氨基酸序列Fig.1 Complete cDNA sequence and deduced amino acids sequence of P. trituberculatus PtCLIC gene
利用MEGA 4.0軟件進行系統進化分析表明, 所有物種的CLIC共聚為兩大類群: 脊椎動物和無脊椎動物。在無脊椎動物中, 三疣梭子蟹與擬穴青蟹緊密聚為一支, 然后與埃及伊蚊、蚤狀、捕食螨聚為一大支; 在脊椎動物中, 野豬與白眉翁聚為一支(圖3)。
2.5 PtCLIC基因表達分析
2.5.1PtCLIC基因的組織表達分析 采用實時熒光RT-PCR分析了三疣梭子蟹PtCLIC基因在不同組織中的表達分布特征。結果顯示,PtCLIC基因在鰓、肝胰腺、肌肉、眼柄、血液和心臟中均有表達。其中,在肝胰腺中的表達量最高, 且顯著高于其它組織(P<0.05)(圖4)。
2.5.2PtCLIC基因在低鹽度脅迫進程中的表達分析低鹽脅迫后PtCLIC在鰓和肝胰腺中的相對表達量分別如圖5、圖6所示。在鰓中, 與對照組相比, 低鹽脅迫0—6h后,PtCLIC出現顯著下調現象, 6h的相對表達量為對照組的0.61倍(P<0.05), 12—48hPtCLIC的相對表達量出現顯著上調, 并于48h達到最大值,相對表達量為對照組的4.18倍(P<0.05), 48—72h又出現顯著下調趨勢(圖5)。在肝胰腺中, 與對照組相比, 低鹽脅迫0—6h后,PtCLIC的相對表達量組間差異不顯著, 6—24hPtCLIC的相對表達量出現顯著上調, 并于24h達到最大值, 相對表達量為對照組的4.48倍(P<0.05), 24—72h又出現顯著下調(圖6)。
2.5.3PtCLIC基因在低鹽耐受與低鹽敏感家系中的表達分析 實時熒光定量RT-PCR結果顯示, 在低鹽耐受家系中, 0—12hPtCLIC基因相對表達量出現上調, 隨后12—72h顯著下調, 三個時間點低鹽耐受家系的表達量分別為低鹽敏感家系的2.31、2.53和1.90倍。低鹽敏感家系0—72h之間PtCLIC基因相對表達量差異不顯著, 保持初始水平幾乎沒有變化,且顯著低于低鹽耐受家系各時間點的相對表達量。見圖7。

圖2 三疣梭子蟹PtCLIC氨基酸序列與其它物種的CLIC氨基酸序列比對Fig.2 The amino acid sequence of P. trituberculatus PtCLIC alignment with other species’ CLIC
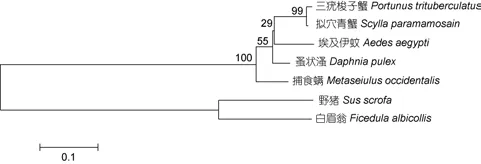
圖3 利用MEGA 4.0軟件NJ法構建的系統進化樹Fig.3 NJ phylogenetic tree made by MEGA 4.0
3 討論
CLIC家族成員在體內分布非常廣泛, 主要存在于真核細胞質膜和各種細胞器上。研究發現(Heisset al, 1997; Wartonet al, 2002), 人類CLCI1和CLIC2分別編碼241和243個氨基酸, 隨后Qian等(1999)發現小鼠CLIC3編碼208個氨基酸, Berryman等(2000)發現人類CLIC5編碼251個氨基酸。本研究采用RACE技術首次克隆得到762 bp的PtCLIC基因cDNA開放閱讀框, 預測蛋白質由253個氨基酸組成。蛋白質結構域分析表明, 在PtCLIC基因編碼的氨基酸中不存在跨膜結構域, 且分子量較小, 推測該蛋白本身并不是氯通道, 而僅僅起到氯通道活化蛋白的功能(Valenzuelaet al, 1997)。另外, 在與PtCLIC同源關系較近的蚤狀(Daphnia pulex)和地中海實蠅(Ceratitis capitata)等物種的CLIC氨基酸序列上亦不存在跨膜結構域, 這與本實驗結果一致。同源性分析表明,PtCLIC與蚤狀溞等物種CLIC的同源性均高于80%,確認該基因為三疣梭子蟹CLIC基因。多氨基酸序列比對分析表明,PtCLIC氨基酸序列保守性較高, 與NCBI數據庫中氨基酸序列比對結果未能確認本研究所得PtCLIC基因具體亞型, 因此PtCLIC基因的分型工作有待于深入研究。系統進化分析表明,PtCLIC與同屬的擬穴青蟹親緣關系較近與野豬等脊椎動物的親緣關系較遠。驗證了親緣關系近的同源性高, 親緣關系遠的同源性低的規律, 符合遺傳進化規律。
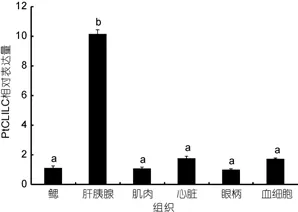
圖4 三疣梭子蟹不同組織中PtCLIC基因的表達Fig.4 Expression of PtCLIC gene in different tissues of P.trituberculatus
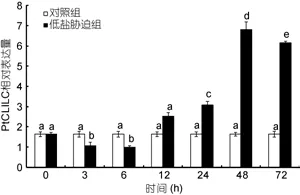
圖5 低鹽脅迫下三疣梭子蟹PtCLIC基因在鰓中的表達情況Fig.5 Expression of PtCLIC gene in P. trituberculatus gill tissue under low salinity stress
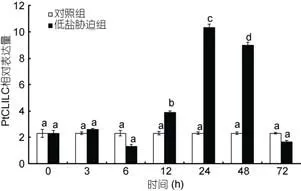
圖6 低鹽脅迫下三疣梭子蟹PtCLIC基因在肝胰腺中的表達情況Fig.6 Expression of PtCLIC gene in P. trituberculatus hepatopancreas tissue under low salinity stress
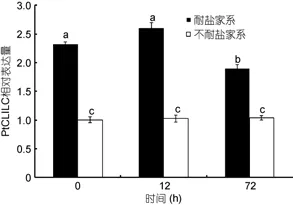
圖7 低鹽脅迫下低鹽耐受家系和低鹽敏感家系中PtCLIC基因在鰓中的表達情況Fig.7 Expression of PtCLIC gene in low salinity tolerant family and low salinity susceptive family in P. trituberculatus gill tissue under low salinity stress
實時熒光RT-PCR證實PtCLIC基因在檢測的所有組織中均有表達。在肝胰腺中的表達量最高, 且顯著高于其它組織中的表達量。這表明三疣梭子蟹PtCLIC基因的表達具有組織特異性。Liu等(2011)的研究表明, 肝臟是甲殼動物離子儲存和代謝的中心,由此推測三疣梭子蟹的肝胰腺在氯離子轉運中起到了重要作用。
目前關于甲殼類動物胞內氯離子通道蛋白對鹽度脅迫的應答機制還未見報道。鰓是甲殼動物與外界環境進行氣體和離子交換的介質(Freireet al, 2008),而肝臟又是離子儲存和代謝的中心(Liuet al, 2011),因此研究低鹽脅迫進程中PtCLIC在鰓和肝胰腺中的表達規律十分必要。Freire等(2008)研究了甲殼動物中NaCl的吸收和排出過程, 證實了Cl–的平衡是甲殼動物滲透壓調節的關鍵。在Cl–的吸收過程中,首先Cl–與HCO3–交換通過鰓上皮頂部質膜, 繼而在基底側質膜上通過Cl–通道進入血淋巴中。而在Cl–的排出過程中, Cl–首先以Na+/K+/2Cl–形式協同轉運通過基底側質膜, 繼而在鰓上皮頂部質膜上通過Cl–通道排到水環境中。本實驗結果顯示低鹽脅迫下PtCLIC基因在鰓和肝胰腺中的表達總體呈現先上調然后下調的規律。低鹽脅迫下PtCLIC基因的表達量上升, 可增強基底側質膜上Cl–通道蛋白的活性(Valenzuelaet al, 1997), 進而增強三疣梭子蟹對Cl–的吸收能力。然而當體內的Cl–水平被調節到正常水平時,PtCLIC基因又基本恢復到初始水平, 出現下調表達。
低鹽脅迫下低鹽耐受家系和低鹽敏感家系差異顯著, 低鹽耐受家系中PtCLIC的相對表達量存在上調和下調的過程, 表達量最大值為低鹽敏感家系的2.53倍, 而低鹽敏感家系中PtCLIC的相對表達量基本不變且維持在較低水平。此差異證明在低鹽耐受家系中PtCLIC存在一個積極的滲透壓調節過程, 而低鹽敏感家系幾乎被動接受應激。該研究結果可輔助選育三疣梭子蟹耐低鹽品系。
本研究首次成功克隆了三疣梭子蟹PtCLIC基因全長cDNA序列, 通過分析低鹽度脅迫下三疣梭子蟹鰓和肝胰腺中PtCLIC基因的表達規律以及低鹽耐受和低鹽敏感家系中表達規律的差異, 進一步認定PtCLIC基因在滲透壓調節中發揮了重要作用, 為三疣梭子蟹耐鹽新品系的選育提供了理論指導。在后續的研究工作中, 關于三疣梭子蟹PtCLIC基因在滲透壓調控方面的功能研究以及相關SNP位點篩選等將重點進行展開。
呂 富, 黃金田, 於葉兵, 2010. 鹽度對三疣梭子蟹生長、肌肉組成及蛋白酶活性的影響. 海洋湖沼通報, 4: 137—142
江 山, 許強華, 2011. 鹽度脅迫對三疣梭子蟹鰓Na+/K+-ATPase酶活的影響. 水產學報, 35(10): 1475—1480
孫穎民, 閆 愚, 孫進杰, 1984. 三疣梭子蟹幼體發育. 水產學報, 8(3): 219—226
陳麗娥, 謝 浩, 2010. ClC型氯離子通道的研究. 生命的化學,30(4): 545—549
季東升, 2005. 三疣梭子蟹池塘養殖技術. 特種經濟動植物, 3:12—13
鄭萍萍, 王春琳, 宋微微, 2010. 鹽度脅迫對三疣梭子蟹血清非特異性免疫因子的影響. 水產科學, 29(11): 634—638
康敏華, 陳 暖, 杜喜玲等, 2011. 水稻氯離子通道蛋白基因的克隆及表達分析. 西北植物學報, 31(4): 0647—0653
隋延鳴, 高保全, 劉 萍, 2012. 三疣梭子蟹“黃選1號”鹽度耐受性及適宜生長鹽度分析. 大連海洋大學學報, 27(5):398—401
路允良, 王 芳, 趙卓英, 2012. 鹽度對三疣梭子蟹生長、蛻殼及能量利用的影響. 中國水產科學, 19(2): 237—245
戴寒晶, 2006. 胞內氯離子通道蛋白功能的初步研究. 合肥:安徽醫科大學碩士學位論文, 49—50
Adachi S, Uchida S, Ito Het al, 1994. Two isoforms of a chloride channel predominantly expressed in thick ascending limb of Henle’s loop and collecting ducts of rat kidney. Journal of Biological Chemistry, 269: 17677—17683
Berry K L, Bulow H E, Hall D Het al, 2003. AC. elegansCLIC-like protein required for intracellular tube formation and maintenance. Science, 302(5653): 2134—2137
Berryman M, Bretscher A, 2000. Identification of a novel member of the chloride intracellular channel gene family(CLIC5) that associates with the actin cytoskeleton of placental microvilli. Molecular Biology of the Cell, 11(5):1509—1521
Cornejo I, Niemeyer M I, Sepulveda F V, 2001. Cloning cellular distribution and functional expression of small intestinal epithelium guinea pig ClC-5 chloride channel. Biochimica et Biophysica Acta-Biomembranes, 1512(2): 367—374
Dong Y X, Chen Y, Wei Qet al, 2013. Cloning and polymorphism analysis of glutamate-gated chloride channel gene ofLaodelphax striatellus(Hemiptera: Delphacidae).Florida Entomologist, 96(3): 1168—1174
Dulhunty A F, Gage P W, Curtis Set al, 2001. The glutathione transferase structural family includes a nuclear chloride channel and a ryanodine receptor calcium release channel modulator. Journal of Biological Chemistry, 276(5): 3319—3323
Edwards J C, 1999. A novel p64-related Cl–channel: subcellular distribution and nephron segment-specific expression.American Journal of Physiology, 276(3): 398—408
Freire C A, Onken H, McNamara J C, 2008. A structure-function analysis of ion transport in crustacean gills and excretory organs. Comparative Biochemistry and Physiology Part A,151: 272—304
Hechenberger M, Schwappach B, Fischer W Net al, 1996. A family of putative chloride channels from A rabidopsis and functional complementation of a yeast strain with a CLC gene disruption. Journal of Biological Chemistry, 271(52):33632—33638
Heiss N S, Poustka A, 1997. Genomic structure of a novel chloride channel gene, CLIC2, in Xq28. Genomics, 45(1):224—228
Jentsch T J, Friedrich T, Schriever A, 1999. The CLC chloride channel family. European Journal of Applied Physiology,437: 783—795
Liu H, Peatman E, Wang W Qet al, 2011. Molecular responses of calreticulin genes to iron overload and bacterial challenge in channel catfish (Ictalurus punctatus). Developmental and Comparative Immunology, 35: 267—272
Lv J J, Liu P, Wang Yet al, 2013. Transcriptome analysis ofPortunus trituberculatusin response to salinity stress provides insights into the molecular basis of osmoregulation.PloS One, 8(12): 1—15
Perluigi S, Addabbo D, Luca P Let al, 2002. Segmental paralogy in the human genome: a large-scale triplication on lp, 6p and 21q. Mammalian Genome, 13(8): 456—462
Qian Z, Okuhara D, Abe M Ket al, 1999. Molecular cloning and characterization of a mitogen-activated protein kinaseassociated intracellular chloride channel. Journal of Biological Chemistry, 274(3): 1621—1627
Shorning B Y, Wilson D B, Meehan R R, 2003. Molecular cloning and developmental expression of two Chloride Intracellular Channel (CLIC) genes inXenopus laevis.Development Genes and Evolution, 213: 514—518
Uchida S, Sasakl S, Furukawa Tet al, 1993. Molecular cloning of a chloride channel that is regulated by dehydration and expressed predominantly in kidney medulla. Journal of Biological Chemistry, 268: 3821—3824
Valenzuela S M, Artin D K, Por S Bet al, 1997. Molecular cloning and expression of a chloride ion channel of cell nucleus. Journal of Biological Chemistry, 272(19): 12575—12582
Warton K, Tonini R, Fairlie W Det al, 2002. Recombinant CLIC1 (NCC27) assembles in lipid bilayers via a pH-ependent two-state process to form chloride ion channels with identical characteristics to those observed in Chinese hamster ovary cells expressing CLIC1. Journal of Biological Chemistry, 277(29): 26003—26011
- 海洋與湖沼的其它文章
- 大菱鲆(Scophthalmus maximus)、星斑川鰈(Platichthys stellatus)及其雜交種的形態學分析*
- 半滑舌鰨(Cynoglossus semilaevis Günther)生長激素體外重組表達及活性分析*
- 香港牡蠣(Crassostrea hongkongensis)與長牡蠣(Crassostrea gigas)種間雜種遺傳力評估*
- 褪黑激素對香港細首紐蟲(Cephalothrix hongkongiensis)性腺發育的影響及羥基吲哚-O-甲基轉移酶(HIOMT)的測定*
- 飼養空間大小對單體筐養養殖系統中三疣梭子蟹(Portunus trituberculatus)的攝食行為與生長特性的影響研究*
- 凡納濱對蝦(Litopenaeus vannamei)Runt轉錄因子的基因克隆及其功能分析*

