限制性內切酶酶切確證河豚魚成分PCR檢測結果
曲良苗,陳文炳*,繆婷玉,邵碧英,彭 娟,江樹勛
(1.福建出入境檢驗檢疫局檢驗檢疫技術中心,福建 福州 350001;2.福建農林大學食品科學學院,福建 福州 350003);3.福建省檢驗檢疫技術研究重點實驗室,福建 福州 350001)
自1980年代聚合酶鏈式反應(polymerase chain reaction,PCR)技術發明以來,已被廣泛應用于食品中的動植物成分鑒別[1-8],在魚制品魚類成分的鑒別中的應用也越來越多[9-11],DNA分子標記技術在河豚魚種類鑒別及遺傳分析方面也有不少報道[12-22]。但普通PCR方法存在擴增結果出現假陽性的可能,如果PCR擴增所使用的引物序列與非目的物種的某段DNA的序列有部分互補性,就會擴增出與目的物種大小相似的PCR產物,僅靠凝膠電泳圖譜判斷,可能因假陽性結果而產生誤判[23],因此有必要采取確證措施加以排除。目前常用的PCR檢測結果的確證方法主要有DNA限制性內切酶技術與DNA測序方法[24-25]。
我國海域廣闊,河豚魚種類繁多,魚類加工食品中因混有河豚魚,使得消費者誤食而中毒的事件時有發生,2007年發生了由于出口美國的鮟鱇魚混有河豚魚,導致顧客誤食而中毒的事件,這些事件對我國食品出口貿易造成負面影響。同年日本食品檢驗部門在我國南方某食品有限公司出口的馬面鲀魚干中檢出河豚魚成分。為了保證消費者的健康與權益,保障水產加工制品的質量與安全,急需一種能夠在食品中快速檢測出河豚魚成分的方法。本研究基于作者于2011年建立的河豚魚成分檢測PCR方法[22]檢測河豚魚與其他樣品,發現在金槍魚與鱸魚中也能擴增出與河豚魚DNA片段大小(423 bp)接近的PCR產物,產生了假陽性結果。因此,本實驗應用限制性內切酶與DNA測序方法,分析了13 種河豚魚樣品與2 種非河豚魚(金槍魚與鱸魚)的PCR結果的差別,建立了有效的排除假陽性結果的方法,并將PCR產物測序結果在GenBank中進行DNA同源性查詢比對(BLAST)加以確證,為準確鑒定河豚魚成分提供科學依據。
1 材料與方法
1.1 材料
9 個已知物種名稱的河豚魚樣品、4 個未知種類的河豚魚樣品、5 個非河豚魚樣品(表1)搜集自福建省莆田、廈門、福州等地水產養殖場、農貿市場和超市。
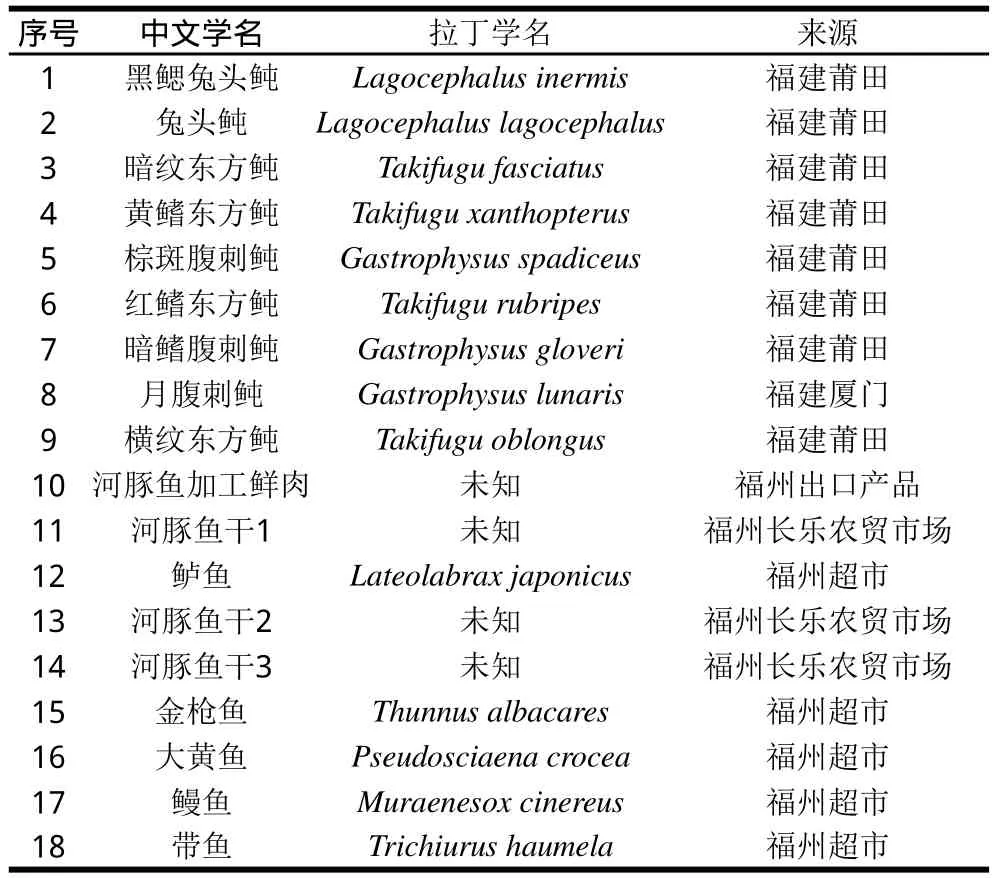
表 1 18 個河豚魚及其他魚類供試樣品Table 1 Eighteen samples of puffer fish and other fishes
1.2 試劑與引物
限制性內切酶NmeA Ⅲ(2000 U/mL) 北京紐英倫生物技術有限公司。其他參照文獻[22]。
按照文獻[21],由上海生工生物工程有限公司合成魚源性成分檢測引物(FISH)與河豚魚成分引物(HT-1),引物堿基序列、PCR產物片段長度與適合的退火溫度(Tm值)見表2。

表 2 魚源性成分與河豚魚成分的PCR檢測用的引物序列與退火溫度Table 2 Primer sequences and annealing temperatures for PCR detection of fish components and puffer fish component
1.3 方法
1.3.1 DNA提取、PCR擴增、限制性內切酶消化
參照文獻[21]優化確立的DNA提取CTAB方法與PCR引物、擴增體系和溫度程序,進行DNA提取、PCR擴增、PCR產物凝膠水平電泳與凝膠成像,每個PCR重復3 次。按照產品使用說明書規定,在10 L的PCR產物中,加入2.0 L 10× Cutsmart Buffer、1.0 L限制性內切酶NmeA Ⅲ、0.05 L S-腺苷甲硫胺酸(S-adenosylmethionine,SMA)溶液,再加純凈水6.95 L使總體積達到20 L,在37 ℃條件下消化16~18 h,然后進行瓊脂糖凝膠電泳與成像拍照。
1.3.2 DNA堿基序列測定
PCR產物通過凝膠電泳后,檢出陽性結果的PCR擴增產物委托上海生工生物技術有限公司進行克隆、測序。
1.3.3 序列比對與酶切位點確定
應用DNAMAN 5.2.2版軟件對各個樣品PCR產物序列進行整理分析,結果以MASED Document/DNAMAN1格式輸出,同時在GenBank中進行BLAST分析。應用NEBcutter 2.0軟件進行限制性內切酶NmeA Ⅲ酶切位點分析。
2 結果與分析
2.1 魚類內源基因PCR擴增

圖 1 18 種河豚魚與非河豚魚樣品的魚成分內源基因PCR擴增電泳圖Fig.1 PCR amplification results of 18 fish samples using primers targeting FISH components
通過樣品的魚類內源基因PCR擴增,監控DNA質量。13 個河豚魚樣品與5 個非河豚魚樣品的魚類特異性基因(線粒體DNA)的PCR擴增結果見圖1。18 個魚類樣品均能擴增出大小為224 bp的魚成分PCR產物,說明各樣品的DNA提取是成功的,適合于PCR擴增。
2.2 河豚魚特異基因PCR擴增
應用河豚魚PCR檢測引物(HT-1)對13 個河豚魚樣品與5 個非河豚魚樣品提取的DNA進行河豚魚線粒體特異性基因(Cyt b)PCR擴增,結果見圖2,13 個河豚魚樣品與2 個非河豚魚樣品(金槍魚與鱸魚)共15 個樣品檢出PCR產物,為疑似陽性結果。另外3 種非河豚魚類的大黃魚、鰻魚、帶魚與空白對照(ddH2O)均未擴增出DNA條帶,判為陰性結果。

圖 2 引物HT-1對河豚魚成分的PCR擴增結果Fig.2 PCR amplification results of 18 fish samples with HT-1 primers targeting puffer fishes component
2.3 PCR產物酶切圖譜分析
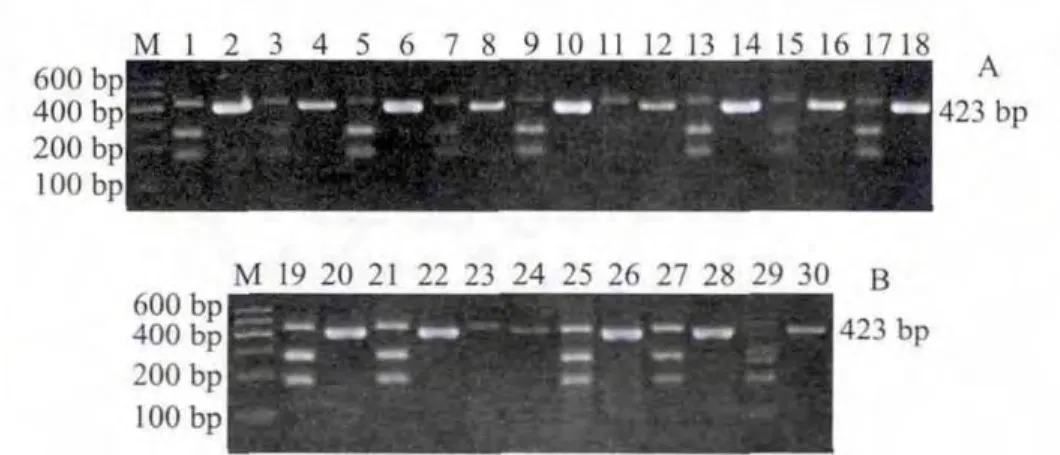
圖 3 PCR產物及其酶切結果電泳圖譜Fig.3 Electrophoresis patterns of PCR products of puffer fishes with and without restriction endonuclease digestion
15 個檢出河豚魚成分疑似陽性結果的樣品PCR產物及其經酶切消化后的產物電泳圖譜見圖3。圖3A與圖3B中9 個已知學名與4 個未知學名的的河豚魚樣品,在奇數號泳道均檢出酶切產物,各樣品圖譜相同,均有2 條新的片段較小的條帶,分別在300 bp與200 bp的下方。由圖3B可知,23、24號泳道分別為鱸魚酶切與未經酶切的PCR產物,前者無新的條帶出現,29、30號泳道為金槍魚的酶切與未經酶切的PCR產物,酶切的電泳圖譜出現4 條新的條帶,圖譜與河豚魚明顯不同。可見,應用限制性內切酶NmeA Ⅲ酶切可以區別河豚魚與非河豚魚,進而排除2 個非河豚魚的假陽性結果。圖3A與圖3B中所有疑似陽性樣品的PCR產物酶切后殘留PCR產物片段條帶位置都高于未經酶切處理的原PCR產物條帶,出現該現象的原因是加入的限制性內切酶(蛋白)與DNA結合,使得分子質量比原來純DNA片段更大。毛細管電泳結果(本文略)表明,經內切酶消化酶切后切斷的與未切斷殘留的DNA片段,因結合了內切酶,其大小都比實際的DNA片段大。
2.4 PCR產物的DNA序列與酶切位點分析
本實驗所用的河豚魚成分PCR檢測正向引物25 個堿基,反向互補20 個堿基(表2),結果正向引物有20 個連續堿基、反向互補有10 個連續堿基與7 個連續堿基分別與金槍魚Cyt b部分片段序列兩端互補,鱸魚中也存在類似情況,于是在2 個非河豚魚樣品中也能擴增出PCR產物,出現假陽性現象.

圖 4 河豚魚(兔頭鲀)Cyt b部分片段PCR產物堿基序列與限制性內切酶NmeAⅢ酶切位點NEB cutter分析圖Fig.4 Partial sequences of the Cyt b gene of puffer fish and NmeAⅢ cleavage sites
應用NEB cutter 2.0軟件對13 種河豚魚與2 種非河豚魚(鱸魚及金槍魚)樣品的PCR產物堿基序列進行限制性內切酶酶切位點分析,結果表明,NmeA Ⅲ酶切位點為5’...GCCGAG(N)21N...3’或3’... CGGCTC(N)19N...5’。河豚魚樣品的PCR產物均為423 個堿基(僅出示兔頭鲀序列,其他種類河豚魚DNA序列資料略),NmeA Ⅲ酶切位點都只有1個(圖4),在5’端起的第256/254堿基位置,被切成2段,即電泳圖出現的2 條新帶(圖3),大小為256/254 bp與167/169 bp。金槍魚PCR產物為422 個堿基,NmeA Ⅲ酶切位點有2 個(圖5),分別在5’端起的第339/337 堿基與252/250堿基的位置,被切成4 段,大小為339/337、83/85 bp與252/250、170/172 bp(圖3B 第29泳道)。鱸魚PCR產物為423個堿基,其序列無NmeA Ⅲ限制性內切酶酶切位點(圖6),NEB cutter 2.0軟件無法生成酶切分析圖,電泳結果無新條帶出現(圖3B),二者結果吻合。
2 個非河豚魚樣品中也能擴增出與河豚魚相似的PCR產物,出現假陽性現象,原因是,河豚魚成分PCR檢測所用的正向引物25 個堿基,反向互補20 個堿基(表2),結果正向引物有20 個連續堿基、反向互補有10 個連續堿基與7 個連續堿基分別與金槍魚Cyt b部分片段序列兩端互補,鱸魚中也存在類似情況。
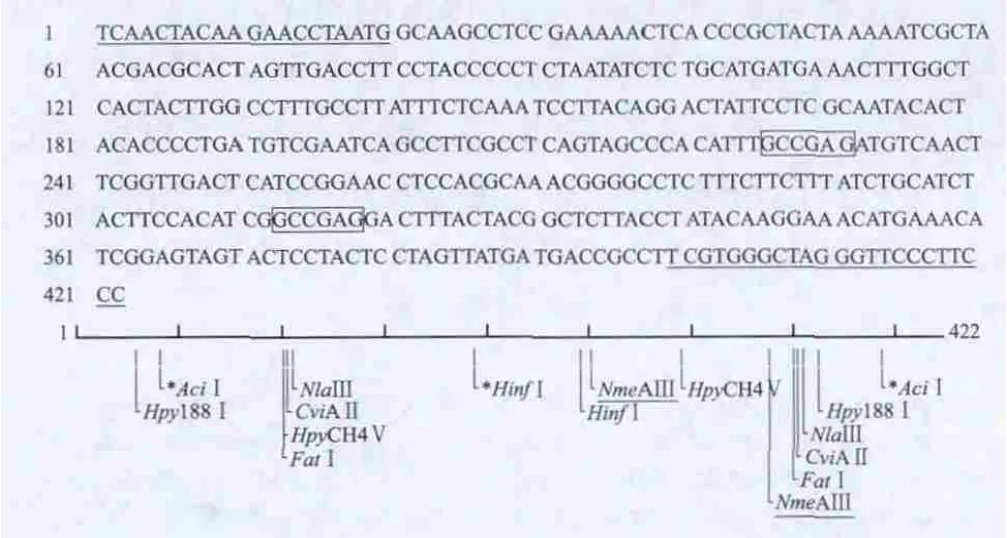
圖 5 金槍魚Cyt b部分片段PCR產物堿基序列與限制性內切酶NmeAⅢ酶切位點NEB cutter分析圖Fig.5 Partial sequences of the Cyt b gene of tuna and NmeAⅢ cleavage sites

圖 6 鱸魚Cyt b部分片段PCR產物堿基序列Fig.6 Partial sequences of the Cyt b gene of weever
2.5 PCR產物堿基序列的GenBank查詢比對與同源性分析
13 個河豚魚樣品與2 個非河豚魚(金槍魚與鱸魚)樣品的PCR產物堿基序列在GenBank數據庫中進行查詢比對(BLAST)結果表明,13 個河豚魚的序列與GenBank數據庫中河豚魚線粒體b基因(Cyt b)的部分序列(序列號有多個,略)同源性在98%~100%之間,其中有9 個樣品為100%,3 個為99%,1 個為98%。金槍魚與鱸魚的序列BLAST結果表明,金槍魚序列422 個堿基中有408個與數據庫中的Thunnus albacares(金槍魚)Cyt b部分序列(JN086153.1)同源性為99%(408/410,圖7),鱸魚的序列423 個堿基中有391 個與數據庫中的Lateolabrax japonicus(鱸魚)Cyt b部分序列(DQ351530.1)同源性為99%(391/392,圖8),可見2 個非河豚魚PCR產物不是河豚魚成分,驗證了限制性內切酶NmeA Ⅲ的酶切結果,可有效排除假陽性誤判。
3 結論與討論

圖 7 金槍魚樣品序列(Query)與GenBank數據庫中的Thunnus albacares(金槍魚)Cyt b基因部分序列(JN086153.1)比對結果圖Fig.7 BLAST results between sequences (Query) of tuna sample and partial sequences (JN086153.1)of the Cyt b gene of Thunnus albacares in GenBank

圖 8 鱸魚樣品序列(Query)與GenBank中的鱸魚(Lateolabrax japonicus Cyt b基因部分序列(DQ351530.1)比對結果圖Fig.8 BLAST results between sequences (Query) of weever sample and partial sequences (DQ351530.1) of the Cyt b gene of Lateolabrax japonicus in GenBank
普通PCR方法的缺陷之一是有時會出現假陽性現象,除了實驗環境與試劑可能受污染以及PCR反應退火溫度不恰當外,引物特異性不夠強是主要原因,而引物的特異性也不是絕對的,只要所使用的引物序列與非目的物種的某段DNA的序列有部分互補性,就會擴增出與目的片段大小相似的PCR產物,凝膠電泳圖譜與陽性結果接近或相同,肉眼無法判別而出現假陽性現象[23],導致結果誤判。本實驗應用文獻[21]建立的河豚魚成分(部分Cyt b基因特異序列)PCR檢測方法檢測13 個河豚魚樣品與5 個非河豚魚樣品,結果發現,在金槍魚與鱸魚2 個非河豚魚樣品中也能擴增出片段大小與河豚魚接近或相同的PCR產物,出現假陽性現象。目前常用的排除PCR檢測假陽性結果的確證方法主要有DNA限制性內切酶技術[24-25]與DNA測序方法[22],本研究應用這兩種方法對河豚魚成分PCR檢測結果加以確證。金槍魚及鱸魚的PCR產物堿基序列經與GenBank數據庫DNA序列比對,分別確認為Thunnus albacares(金槍魚)與Lateolabrax japonicus(鱸魚),表明2個非河豚魚PCR產物不是河豚魚成分,驗證了NmeA Ⅲ限制性內切酶酶切結果,可見該方法可用于河豚魚成分PCR檢測結果的確證。為了驗證本實驗建立的NmeA Ⅲ限制性內切酶方法是否具有廣泛的適用性,將進一步在更多的其他魚類中可能出現的假陽性結果進行確證應用,為該方法的推廣應用提供更豐富的科學依據。
[1]陳文炳, 邵碧英, 廖憲彪, 等. 加工食品中若干動物成分的PCR檢測技術應用研究[J]. 食品科學, 2005, 26(8): 338-342.
[2]陳文炳, 邵碧英, 江樹勛, 等. 食品中若干植物源性成分的PCR檢測[J].食品科學, 2006, 27(11): 404-408.
[3]陳穎, 錢增敏, 徐寶梁, 等. 保健品中牛羊源性成分的PCR檢測[J].食品科學, 2004, 25(10): 215-218.
[4]才華, 欒鳳俠, 關學佳, 等. 馬鈴薯、番茄內源基因PCR檢測引物設計及特異性分析[J]. 食品科學, 2011, 32(22): 192-195.
[5]張馳, 邱皓璞, 張筠. 熒光定量PCR檢測肉制品中鴨源性成分[J]. 食品科學, 2013, 34(18): 154-157.
[6]李富威, 張舒亞, 葉軍, 等. 實時熒光聚合酶鏈式反應檢測食品中香蕉成分[J]. 食品科學, 2013, 34(12): 243-246.
[7]范麗麗, 李培, 傅春玲, 等. 食品中雞源性成分實時熒光PCR檢測方法的建立[J]. 食品科學, 2014, 35(2): 248-251.
[8]李富威, 張舒亞, 葉軍, 等. 食品中木瓜成分實時熒光PCR檢測方法[J]. 食品研究與開發, 2013, 34(11): 51-56.
[9]REHBEIN H, KRESS G, SCHMIDT T. Application of PCR-SSCP to species identification of fishery products[J]. Journal of the Science of Food and Agriculture, 1997, 74: 35-41.
[10]LIN Wenfeng, HWANG D F. A multiplex PCR assay for species identification of raw and cooked bonito[J]. Food Control, 2008, 19:879-885.
[11]李進波, 李想, 諶鴻超, 等. 實時熒光PCR法鑒定食品中鮭亞科魚成分[J]. 食品科學, 2013, 34(20): 194-198.
[12]陳超, 石拓, 孫曙光, 等. 應用RAPD標記對東方豚屬進行種類鑒別及其聚類分析[J]. 海洋水產研究, 2001, 22(3): 32-36.
[13]邵愛華, 鄭峰, 吳勝, 等. 暗紋東方豚mtDNA的分離與純化及其細胞色素b基因的分子克隆[J]. 中國水產科學, 2005, 24(5): 4-7.
[14]邵愛華, 朱江, 陳葵, 等. 暗紋東方豚粒體細胞色素b及其側翼tRNA基因的克隆與序列分析[J]. 中國水產科學, 2005, 12(6): 675-681.
[15]邵愛華, 朱江, 陳葵, 等. 暗紋東方豚線粒體COⅡ及兩側tRNA基因的克隆和序列分析[J]. 動物學雜志, 2005, 40(6): 1-8.
[16]邵愛華, 朱江, 陳葵, 等. 暗紋東方豚線粒體CO Ⅰ及其側翼tRNA基因的克隆與序列分析[J]. 遺傳, 2006, 28(8): 963-971.
[17]邵愛華, 朱江, 史全良, 等. 暗紋東方豚線粒體COⅢ克隆及序列分析[J].水產科學, 2006, 25(8): 391-396.
[18]邵愛華, 薛峰, 陳葵, 等. 暗紋東方豚線粒體ND1及其側翼tRNA基因的克隆及序列分析[J]. 蘇州科技學院學報: 自然科學版, 2007,24(4): 61-66.
[19]邵愛華, 杜建, 陳葵, 等. 暗紋東方豚線粒體DNA 16S rRNA基因克隆、測序與在分子系統發育分析中的應用[J]. 江蘇農業科學,2009(2): 15-19.
[20]邵愛華 杜建, 陳葵, 等. 暗紋東方豚線粒體ATPase8和ATPase6基因的克隆與序列分析[J]. 蘇州科技學院學報: 自然科學版, 2010,21(7): 41-46.
[21]陳文炳, 趙晨, 邵碧英, 等. PCR方法檢測河豚魚的引物篩選及反應體系的優化[J]. 食品科學, 2011, 31(20): 376-381.
[22]陳文炳, 林少華, 邵碧英, 等. 河豚魚Cyt b基因的部分DNA序列分析與應用[J]. 食品科學, 2012, 33(20): 227-232.
[23]梁國棟. 最新分子生物學實驗技術[M]. 北京: 科學出版社, 2001:131-133.
[24]王波, 郭勇, 鮮靈. 一種野生蘑菇的鑒定[J]. 西南農業學報, 2013,26(2): 672-674.
[25]郝旭光, 孫寓嬌, 鮮靈. PCR-酶切技術在石油烴降解菌篩選鑒定中的應用[J]. 環境工程學報, 2010(10): 449-452.

