利用RFLP分析DO對附積床系統(tǒng)中AOB群落結(jié)構(gòu)的影響
張 巖,朱 敏,劉煥光,孫鳳俠,甘志明,陳 敬,史 楊,謝杭冀 (北京工業(yè)大學,北京市水質(zhì)科學與水環(huán)境恢復工程重點實驗室,北京 100124)
利用RFLP分析DO對附積床系統(tǒng)中AOB群落結(jié)構(gòu)的影響
張 巖*,朱 敏,劉煥光,孫鳳俠,甘志明,陳 敬,史 楊,謝杭冀 (北京工業(yè)大學,北京市水質(zhì)科學與水環(huán)境恢復工程重點實驗室,北京 100124)
為了解析DO濃度對附積床反應器脫氮系統(tǒng)中COD、NH4+-N、TN去除效率的影響,以及對氨氧化菌群(AOB)結(jié)構(gòu)及多樣性的影響,分析了DO分別為1.0~2.0,2.0~3.0,3.0~4.0mg/L時COD、NH4+-N、TN去除效率,并采用針對AOB功能基因氨單加氧酶(amoA)的限制性內(nèi)切酶片段長度多態(tài)性技術(shù)(RFLP)分析了三組DO濃度下反應器中AOB的群落結(jié)構(gòu)及多樣性.結(jié)果表明,不同DO條件下,系統(tǒng)均取得較高的COD和NH4+-N的去除效果, NH4+-N的去除效率隨著DO的增加而提高.不同DO濃度下反應器生物膜上AOB菌群多樣性豐富,且與DO對AOB菌群的多樣性影響較小相比,DO對AOB的菌群結(jié)構(gòu)及種類的影響較大.
氨氧化細菌(AOB);amoA基因;RFLP;生物膜
同時硝化反硝化(SND)的硝化過程首先在氨氧化細菌(AOB)的作用下,NH4+被氧化成 NO2-,繼而通過亞硝酸氧化菌(NOB)將NO-氧化為NO-.23由于AOB細菌是影響硝化過程穩(wěn)定性的主要微生物[1],且氨氧化作用成為硝化過程的起始和限速步驟[2],因此深入了解氨氧化細菌種類、種群結(jié)構(gòu)及數(shù)量變化,有針對性地對系統(tǒng)進行優(yōu)化調(diào)控,是確保脫氮處理系統(tǒng)穩(wěn)定、高效運行的關(guān)鍵.
AOB生長速度極為緩慢,傳統(tǒng)分離純化方法限制了對硝化細菌的研究與應用.由于熒光原位雜交技術(shù)(FISH)、熒光實時定量 PCR、PCR-DGGE、T-RFLP等分子生物學技術(shù)避開了傳統(tǒng)微生物培養(yǎng)分離環(huán)節(jié),采用直接從樣品中提取所含微生物總DNA,對DNA進行分析,這些技術(shù)已經(jīng)成為現(xiàn)代環(huán)境微生物學研究的重要手段.如Dong等[3]利用技術(shù)研究了濕地處理豬廢水系統(tǒng)中氨氧化細菌的種類. Winkler等[4]利用FISH、定量PCR技術(shù)分析了好氧顆粒污泥中AOB與NOB細菌比例不同的原因.Park等[5]利用 FISH技術(shù)研究了溶解氧對活性污泥中AOB菌群結(jié)構(gòu)的影響.但是目前對于SND反應器中DO對AOB菌群結(jié)構(gòu)以及AOB細菌的種類組成影響的研究報道較少.
附積床系統(tǒng)是在反應器中懸掛Bio-fix填料的生物膜與活性污泥并存的系統(tǒng),Bio-fix填料可以形成厚度適中的生物膜.本課題組在前期研究中,采用 PCR-DGGE、FISH技術(shù)研究了不同C/N[6]、碳源[7]、HRT[8]、DO[9]等條件下附積床系統(tǒng)中微生物菌群群落結(jié)構(gòu)及空間分布,但對附積床系統(tǒng)中AOB菌群結(jié)構(gòu)、多樣性及種類組成還未有研究.
DO是 SND的重要影響因素之一,研究表明
[10-12],SND系統(tǒng)中DO濃度為1~3mg/L時,系統(tǒng)脫氮效果較好.本研究將反應器 DO分別設定為1.0~2.0mg/L、2.0~3.0mg/L、3.0~4.0mg/L,對不同DO濃度下附積床反應器的COD、NH4+-N、TN去除效果進行研究,并利用限制性內(nèi)切酶段長度多態(tài)性技術(shù)(RFLP)技術(shù)分析不同 DO條件下反應器中AOB的群落結(jié)構(gòu)、多樣性及種類的組成和變化.
1 材料與方法
1.1 實驗裝置與填料
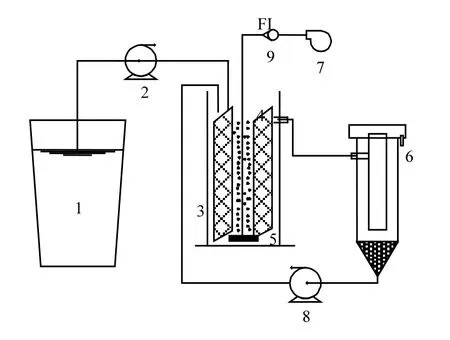
圖1 附積床生物膜反應器Fig.1 Schematic diagram of catching bed biofilm reactor
如圖 1所示.反應器裝置主體包括原水箱、SND反應器、二沉池.反應器主體及二沉池有效容積分別為12L和16L.SND反應器內(nèi)部為單槽結(jié)構(gòu),進水由蠕動泵供入底部,中間懸掛兩片bio-fix填料,上部設出水口,依靠重力作用流入二沉池.二沉池為豎流式二沉池中間進水,外圍出水,池底污泥依靠蠕動泵回流到反應器中.實驗中采用的高親水性生物填料 Bio-fix(BX) 主要材質(zhì)為丙烯酸樹脂纖維,具有很高的孔隙率和比表面積,填料的特征參數(shù)如表1所示.其表面粗糙的紋理及填料內(nèi)部所撐開的空間結(jié)構(gòu),使填料能很好地固定生物膜,其上黏附的生物膜不易大面積脫落.同時,在填料中氣水可以在其中自由流動,可有效防止污泥在填料上堵塞結(jié)塊,保證生物膜內(nèi)部微生物活性.
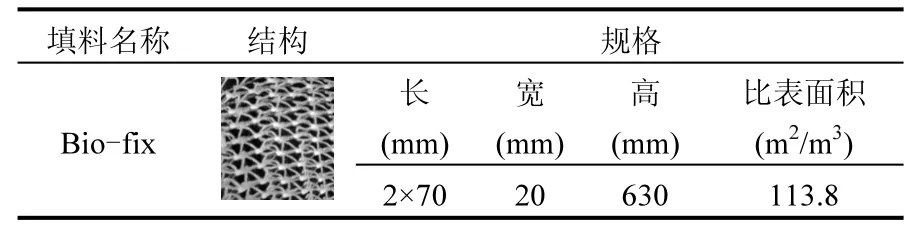
表1 填料技術(shù)參數(shù)Table 1 Characteristics of the carrier
1.2 原水水質(zhì)、運行參數(shù)及分析方法
實驗用原水為生活用水,取自北京工業(yè)大學西區(qū)教工生活小區(qū)化糞池上清液,ρ(COD)=159.5~675.7mg/L,ρ(NH4+-N)=55.4~116.4mg/L,ρ(TN)=61.0~119.6mg/L.
運行參數(shù)為 HRT=8h,污泥回流量為 100%,三組平行反應器 DO 分別為 1.0~2.0, 2.0~3.0, 3.0~4.0mg/L.
COD采用5B-3型快速測定儀;NH4+-N采用納氏試劑光度法測定;NO-2-N采用N-(1-萘基)-乙二胺光度法測定;NO3--N采用紫外分光光度法測定;DO、溫度采用WTW DO測定儀測定;pH值測定采用METTLER TOLEDO LE438pH計.
1.3 DNA提取
將取自生物膜上的污泥樣品在5000g下離心3min,取離心后樣品0.3g置于2mL離心管中,DNA樣品采用土壤DNA提取試劑盒(生工生物,上海)提取,最終DNA提取液為80μL.電泳前用核酸染料Gelred將瓊脂糖染色,130V,30min電泳完成后用去離子水漂洗,在254nm的紫外下觀察.
1.4 PCR擴增
對于AOB的擴增,選用針對功能基amoA的引物amoA-1F(5’-GGGGTTTCTACTGGTGGT-3’)和 amoA-2R (5’-CCCCKCKGSAAAGCCTTCTTC-3’;K表示G或T;S表示C或G)[13].PCR反應體系為50μL,包括2μL模板DNA,5μL 10× PCR buffer,4μL dNTP混合液(各2.5mmol/L),上、下游引物(10pmol/μL)各 2μL,0.25μL Tap酶(5U/ μL,TaKaRa,日本),加雙蒸滅菌水至50μL.PCR擴增程序如下:94℃預變性5min;94℃變性60s,50℃退火 90s,72℃延伸 90s,共 40個循環(huán);72℃延伸10min.擴增產(chǎn)物采用 Agarose Gel DNA purification kit A劑盒(生工生物,上海)進行純化,純化過程按照試劑盒說明書進行.
1.5 amoA基因克隆及RFLP分析
將純化后的PCR產(chǎn)物鏈接到pMD18-T載體(生工生物,上海)上,再轉(zhuǎn)化到 Escherichia coli JM109(TaKaRa,日本)細胞中,質(zhì)粒的轉(zhuǎn)化、克隆方法參考pMD18-T Simple Vector說明書.藍白斑篩選分別挑取80個白色菌落轉(zhuǎn)接到另一培養(yǎng)基平板上.以挑選的克隆菌落為模板,用pMD18-T載體引物RV-M和M13-47作為PCR引物,檢驗篩選的克隆是否插入正確長度的片段.片段大小正確的amoA基因PCR產(chǎn)物用MboI (TaKaRa,日本)限制性內(nèi)切酶進行酶切.酶切體系為10μL,包括PCR產(chǎn)物5μL, MBoI 1μL,ddH2O 4μL,酶切反應于 37°C條件下進行 1h.酶切產(chǎn)物用濃度為2.0%的瓊脂糖凝膠電泳檢驗,在凝膠成像系統(tǒng)中凝膠成像.凝膠成像結(jié)果中 RFLP分型相同的劃分為一個操作單元(OUT).
1.6 測序與序列系統(tǒng)發(fā)育學分析
每個OTU選取一個代表菌株送往上海生工生物公司進行測序.獲得的 amoA基因序列與GenBank 進行比對(http://www.ncbi.nlm.nih. gov/blast),選取具有代表性且同源性較高的序列,利用MEGA4.0構(gòu)建系統(tǒng)進化樹.
1.7 物種多樣性分析
基于限制性酶切分析結(jié)果,對3組反應器中群落的α多樣性指數(shù)進行分析.3種α多樣性指數(shù)Shannon Wiener指數(shù)、Simpson指數(shù)和均勻度指數(shù)的計算公式分別為:H=-∑(Pi)(log2Pi);D=1-∑Pi2,Pi=Ni/N;E=H/lnS.式中H:Shannon Wiener指數(shù),D:Simpson指數(shù),E:均勻度指數(shù),Ni:為第i種酶切類型所包含的克隆數(shù),N:總克隆數(shù),Pi=Ni/N,S:群落中總物種數(shù).
2 結(jié)果與討論
2.1 反應器運行情況
如表 2所示,DO=1.0~2.0mg/L時,反應器中COD去除率為86.4%,NH4+-N去除率為89.21%, TN去除率為 33.37%;當 DO=2.0~3.0mg/L時, COD、NH4+-N和 TN的去除率均有所增加;當DO增加至3.0~4.0mg/L時,COD、NH4+-N和TN去除率分別為 87.3%、99.31%和 29.67%.在反應器運行過程中,系統(tǒng)受到持續(xù)性的沖擊負荷,期間進水 COD最高達 675.1mg/L,NH4+-N最高達116.4mg/L,這種情況持續(xù)了17d,造成系統(tǒng)COD、NH4+-N、TN處理能力有所降低,但在沖擊負荷消失后,系統(tǒng)處理能力恢復較快,說明該系統(tǒng)承受沖擊負荷能力強,且調(diào)節(jié)能力強.從整體上看,不同DO條件下系統(tǒng)都取得了良好的有機物及氨氮去除效果,氨氮去除效率隨著DO的增加而提高.
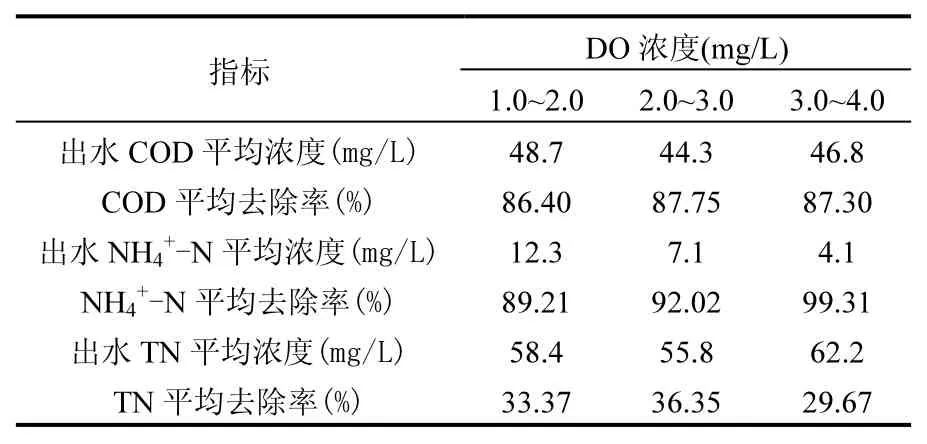
表2 不同DO條件下COD、NH4+-N、TN去除效果Table 2 COD, NH4+-N and TN removal efficiency under different DO concentrations
2.2 amoA基因的RFLP分析
在3組反應器的187個陽性克隆子中,通過酶切分型,一共得到 29個 OTUs,其中 DO=1~2mg/L時:61個陽性克隆子,8個OTUs; DO=2~3mg/L時:63個陽性克隆子,12個OTUs; DO=3~4mg/L時:60個陽性克隆子,9個OTUs.
從圖2可以看出,同時存在于3組反應器的OTU只有1個,同時存在于兩個反應器中的OTU有 7個.此外,每個反應器中都存在獨有的 OTU類型, DO=1~2mg/L(圖中M1)、DO=2~3mg/L(圖中M2)、DO=3~4mg/L(圖中M3)反應器中獨有的OTU分別為2個、7個和5個,這些數(shù)據(jù)表明,不同 DO條件下微生物群落構(gòu)成有較大的變化.3組反應器中獨有 OTU分別占各組反應器 OTU總數(shù)的25%(M1)、58.3%(M2)和55.6%(M3),這些獨有 OTU的克隆數(shù)占其文庫的 41.0%(M1)、25.4%(M2)和35.0%(M3),說明3組反應器在氨氧化細菌構(gòu)成方面相差較大,每組反應器都含有其特有的操作單元,然而從克隆數(shù)來看,這些特有的操作單元并沒有成為系統(tǒng)中的優(yōu)勢氨氧化菌群.對每組反應器中含有陽性克隆子數(shù)目前三位的OTU進行分析,其陽性克隆子數(shù)總和分別占各組反應器中總克隆子數(shù)的70.5%(M1)、69.8%(M2)、71.7%(M3),其中,M1反應器中30.2%的克隆子為獨有OTU,M3反應器中僅有 23.3%的克隆子數(shù)屬于獨有OTU,其余克隆子同時存在于兩個反應器中或同時存在于 3個反應器中,說明本實驗中各組反應器均存在優(yōu)勢氨氧化菌群,DO對氨氧化菌群有一定的篩選功能.
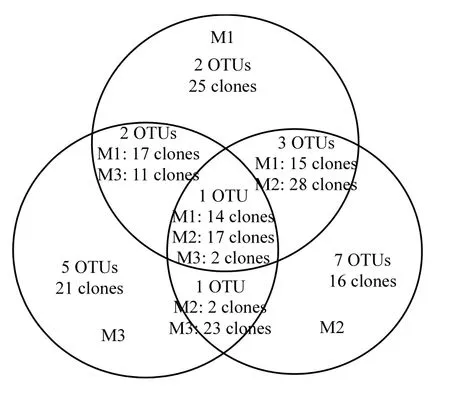
圖2 不同DO濃度下反應器中氨氧化細菌amoA基因酶切類型關(guān)系的文氏圖Fig.2 Venn diagram showing relationships between three different DO concentrations in RFLP pattern-based amoA gene OTUs
2.3 amoA基因文庫的多樣性分析
評價生物多樣性的高低時,同時要考慮到物種的豐富度與均一度.環(huán)境種類豐富度越高均一度越高,則物種的多樣性越高.Shannon-Wiener指數(shù)和Simpson指數(shù)既考慮到了物種數(shù),同時也考慮到每個物種的相對多度.

表3 不同DO濃度下樣品amoA基因文庫α多樣性指數(shù)Table 3 α diversity index of the amoA gene libraries from three samples collected at different DO concentrations
由表 3可見,DO=2.0~3.0mg/L時,Shannon-Wiener指數(shù)和Simpson指數(shù)均為最高,但是三組反應器變化不明顯,說明不同DO濃度下,反應器中的物種多樣性均較高,DO對物種多樣性影響較小.該結(jié)果與在現(xiàn)有的研究報道相似, Magdalena等[14]采用 PCR-DGGE研究發(fā)現(xiàn)當DO從0.5mg/L提高到1.5mg/L,反應器中AOB菌群相似度基本不變,Princic等[15]通過對菌群rDNA進行限制性酶切分析發(fā)現(xiàn)1%、7%、21%的氧飽和度下反應器中AOB生物多樣性基本相同.從均勻度指數(shù)看,DO為2.0~3.0mg/L時,均勻度指數(shù)最高,DO=1.0~2.0mg/L 和 DO=3.0~4.0mg/L時均勻度指數(shù)差異不大,說明AOB群落結(jié)構(gòu)能較好地適應 DO為 2.0~3.0mg/L的環(huán)境,物種較為均勻.
通過比較不同DO條件下NH4+-N的去除效果發(fā)現(xiàn),NH4+-N的去除效率隨著DO的增加而提高,說明氨氧化作用基本不受氨氧化細菌多樣性的影響,有研究表明氨氧化作用主要與氨氧化細菌在活性污泥中占的比例、活性以及種類有關(guān)[14,16].
2.4 AOB系統(tǒng)發(fā)育分析
將測序后的克隆子基因片段在 NCBI網(wǎng)站上進行 BLAST比對,發(fā)現(xiàn)大部分克隆子比對后與最近物種相似度在 90%以上.比對得到的菌群均屬于Nitrosomonas,這是因為Nitrosomonas有較快的增長速度,如 N.europaea,其μmax為0.088h-1,而 Nitrosospira 的μmax 為 0.033~0.035h-1[17],這種生長優(yōu)勢可能使Nitrosomonas更容易成為活性污泥和生物膜系統(tǒng)中優(yōu)勢AOB[18],這與已有的研究報道結(jié)論相似[19-21].由圖 3~圖 5可以看出不同DO濃度下,系統(tǒng)中AOB細菌種類發(fā)生了較大的變化,當DO=1.0~2.0mg/L時,OTU經(jīng)對比找出7種AOB細菌,當DO=2.0~3.0mg/L時,OTU經(jīng)對比找出9種AOB細菌種類,而當DO提高到3.0~4.0mg/L時,OTU經(jīng)對比只找出4種AOB細菌.這可能是因為DO=1.0~2.0mg/L的低溶解氧濃度環(huán)境限制了部分 AOB菌群的生長,不適應環(huán)境的菌群被淘汰,DO為2.0~3.0mg/L是AOB菌群最佳生長環(huán)境,更多的AOB菌群逐漸適應這種生長環(huán)境,AOB菌群種類豐富度提高,而當DO為3.0~4.0mg/L時,AOB菌群之間的內(nèi)部競爭激烈,不適應外界環(huán)境的菌種被逐漸淘汰,其空出的生存空間被其他種類的AOB菌群所替代,AOB種類減少.
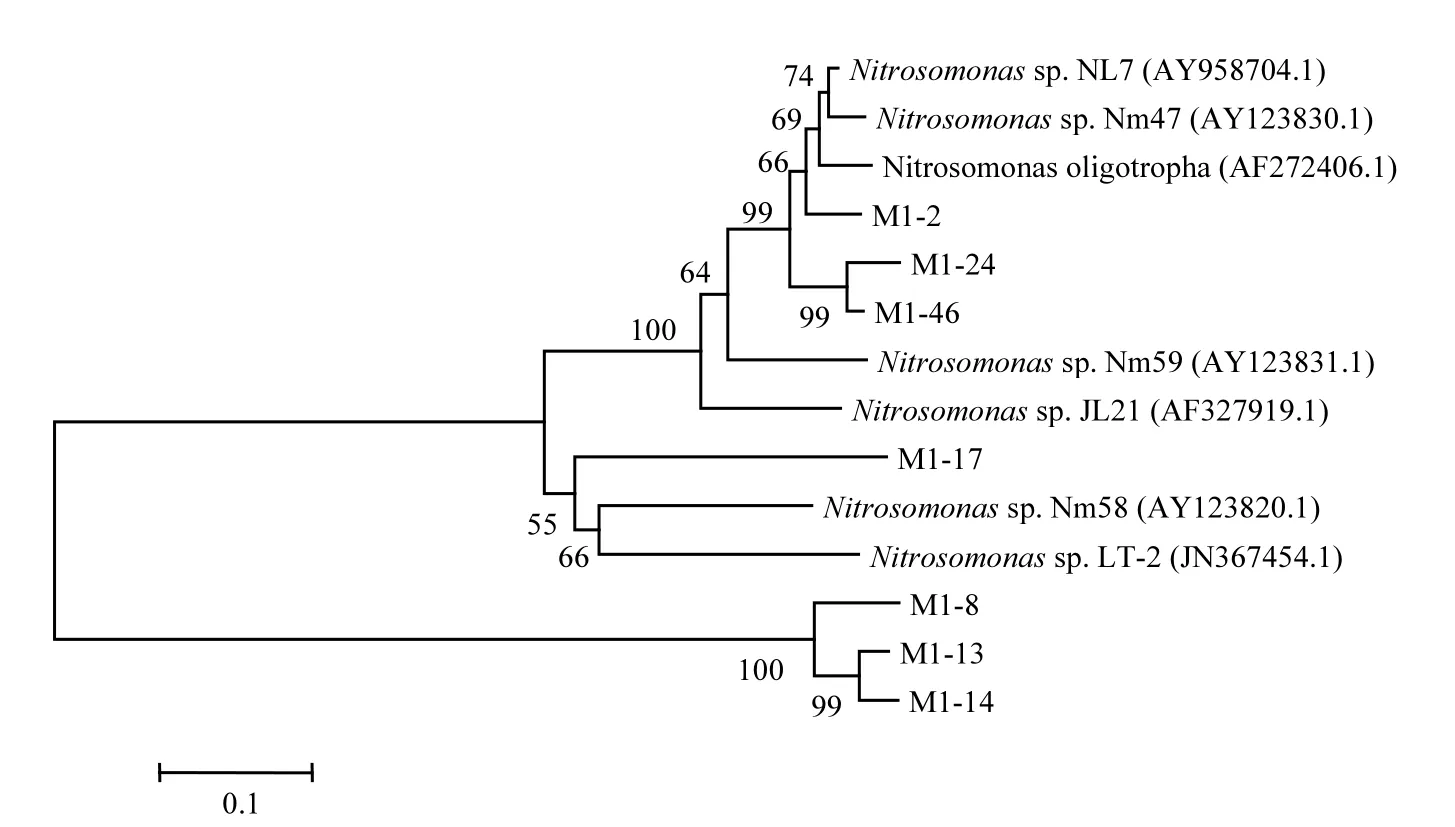
圖3 DO=1.0~2.0mg/L amoA基因的系統(tǒng)發(fā)育樹Fig.3 Phylogenetic tree based on amoA gene at DO =1.0~2.0mg/L
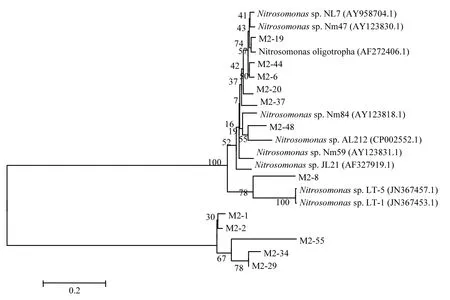
圖4 DO=2.0~3.0mg/L amoA基因的系統(tǒng)發(fā)育樹Fig.4 Phylogenetic tree based on amoA gene at DO =2.0~3.0mg/L
3組反應器中,AOB組成存在一定的差別,DO為1.0~2.0mg/L時,主要由N. oligotropha cluster(約占 88.5%)組成,同時還存在 N. communis cluster(約 占 4.9%)和 unknown Nitrosomonas cluster(約占6.6%),在DO為2.0~3.0mg/L的反應器中,N. oligotropha cluster(約占73.8%)占絕對優(yōu)勢,同時存在部分 unknown Nitrosomonas cluster(約占 16.2%),而在 DO 為3.0~4.0mg/L的反應器中,僅存在 N. oligotropha cluster,這種差異是由 DO濃度引起的,說明 N. communis cluster具有在低DO環(huán)境中生存的優(yōu)勢,這與已有的研究的結(jié)論相似[22],同時也表明,在 3組反應器中,N. oligotropha可能是優(yōu)勢菌群.有文獻指出,N. oligotropha cluster對NH4+-N的Ks 較低,在0.027~0.059mg/L之間[23],此類AOB細菌適宜在低NH4+-N濃度的污水處理系統(tǒng)中,但本實驗進水平均 NH4+-N濃度達 85.5mg/L,說明 N. oligotropha cluster的AOB細菌對NH4+-N濃度的耐受度不同,部分N. oligotropha cluster的AOB細菌能適應高濃度NH4+-N環(huán)境,具體種類和原因還需進一步的研究.
3組反應器均有較高的氨氮去除效率,且各反應器中AOB菌群主要由N. oligotropha cluster的AOB組成(3組反應器分別占總陽性克隆子數(shù)的 88.5%、73.8%、100%),說明 N. oligotropha cluster的AOB可能是去除氨氮的主要菌群.
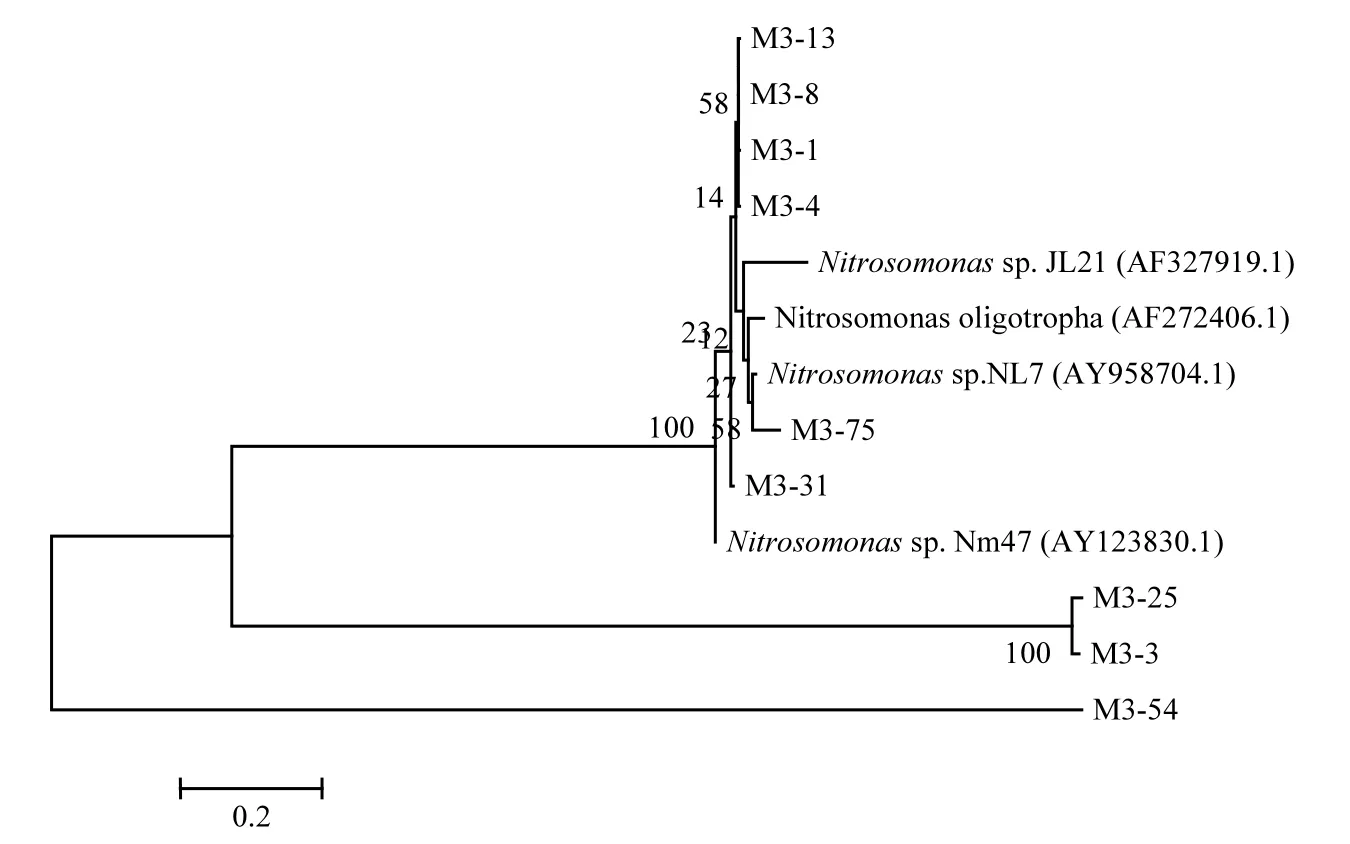
圖5 DO=3.0~4.0mg/L amoA基因的系統(tǒng)發(fā)育樹Fig.5 Phylogenetic tree based on amoA gene at DO =3.0~4.0mg/L
3 結(jié)論
3.1 DO濃度為1.0~2.0,2.0~3.0和3.0~4.0mg/L的反應器中,COD去除效率分別為86.4%,87.5%和 87.3%,NH4+-N 去除效率分別達 89.21%, 92.02%和99.31%,說明不同DO濃度下,系統(tǒng)均取得較高的COD和NH4+-N的去除效果, NH4+-N的去除效率隨著DO的增加而提高.
3.2 對不同DO條件下反應器中AOB細菌進行多樣性分析,由α多樣性指數(shù)可以看出,3組反應器生物膜上AOB菌群多樣性豐富,DO對AOB菌群的多樣性影響較小.
3.3 DO對AOB的菌群結(jié)構(gòu)和種類組成產(chǎn)生了較大的影響.在 3組反應器中,均存在 N. oligotropha cluster,且為優(yōu)勢菌群,在低濃度 DO條件下,還存在N. communis cluster和unknown Nitrosomonas cluster,均屬于Nitrosomonas.
[1]Satoh H, Yamakawa T, Kindaichi T, et al. Community structures and activities of nitrifying and denitrifying bacteria in industrial wastewater-treating biofilm [J]. Biotechnology and Bioengineering, 2006,94(4):762-772.
[2]Hallin S, Lydmark P, Kokalj S, et al. Community survey of ammonia-oxidizing bacteria in full-scale activated sludge processes with different solids retention time [J]. Journal of Applied Microbiology, 2005,99(3):629-640.
[3]Dong Xiuli, Redd G B. Ammonia-oxidizing bacterial community and nitrif i cation rates in constructed wetlands treating swine wastewater [J]. Ecological Engineering, 2012,40:89-197.
[4]Winkler M K, Bassin J P, Kleerebezem R. Unravelling the reasons for disproportion in the ratio of AOB and NOB in aerobic granular sludge [J]. Applied Microbiology and Biotechnology, 2012,94(6):1657-1666.
[5]Park H D, Noguera D R. Evaluating the effect of dissolved oxygen on ammonia-oxidizing bacteria communities in activated sludge [J]. Water Res., 2004,38:3275-3286.
[6]Zhang Yan, Yang Zhengyang, Wang Lili, et al. Influence of C/N ratio on SND and microbiological analysis in catching bed biofilm reactor using acrylic resin fiber as carrier materials in civil engineering [C]//Advanced Materials Research, 2012,568: 89-93.
[7]Zhang Yan, Wang Lili, Yang Zhengyang, et al. A PCR-DGGE approach to evaluate the influebce of environment material for civil engineering-different carbon sources on a catching bed biofilm reactor in start-up phase [C]//Advanced Materials Research, 2012,568:260-264.
[8]張 巖,郭 巖,白玉華,等.附機床生物膜反應器同步硝化反硝化脫氮特性 [J]. 環(huán)境科學, 2010,31(1):134-139.
[9]Zhang Yan, Zhu Min, Liu Huanguang, et al. Influence of DO conentration on SND and microorganisms under catching bed biofilm reactor [C]//International conference on Material Science and Environmental Engineering [MSEE 2013].
[10]徐偉鋒,孫力平,古建國,等.DO對同步硝化反硝化影響用動力學 [J]. 城市環(huán)境與城市生態(tài), 2003,16(1):8-10.
[11]阮文權(quán),卞慶榮,陳 堅.COD與 DO對好氧顆粒污泥同步硝化反硝化脫氮的影響 [J]. 應用與環(huán)境生物學報, 2004,10(3):366-369.
[12]王學江,夏四清,陳 玲,等.DO對MBBR同步硝化反硝化生物脫氮影響研究 [J]. 同濟大學學報(自然科學版), 2006,34(4): 514-538.
[13]Hornek R, Pommerening-Roser A, Koops H P, et al. Primers containing universal bases reduce multiple amoA gene specif i c DGGE band patterns when analyzing the diversity of betaammonia oxidizers in the environment. [J]. Microbiol. Meth., 2006,66(1):147-155.
[14]Magdalena Z, Katarzyna B, Agnieszka C K, et al. Nitrogen removal from wastewater and bacterial diversity in activated sludge at different COD/N ratios and dissolved oxygen concentrations [J]. Journal of Environment Sciences, 2012,24(6): 990-998.
[15]Princic A, Marne I, Megusar F, et al. Ef f ects of pH and oxygen and ammonium concentrations on the community structure of nitrifying bacteria from wastewater [J]. Applied and Environmental Microbiology, 1998,64(10):3584-3590.
[16]Ebie Y, Matsumura M, Noda N, et al. Community analysis of nitrifying bacteria in an advanced and compact Gappei-Johkasou by FISH and PCR-DGGE [J]. Water Science and Technology, 2002,46(11/12):105-111.
[17]王曉慧,文湘華,楊寧寧,等.污水處理工藝對氨氧化菌及細菌群落的影響 [J]. 中國環(huán)境科學, 2009,29(6):622-628.
[18]Siripong S, Rittmann B E. Diversity stuy of nitrifying bacteria in full-scale municipal wastewater treatment plants [J]. Water Research, 2007,41(5):1110-1120.
[19]Xiaohui W, Xianghua W, Craig C, et al. Community analysis of ammonia-oxidizing bacteria in activated sludge of eight wastewater treatment systems [J]. Journal of Environment Sciences, 2010,22(4):627-634.
[20]Lapara T M, Ghosh S. Population dynamics of the ammonia-oxidizing bacteria in a full-scale municipal wastewater treatment facility [J]. Environment Engineering Science, 2006, 23(2):309-319.
[21]Rowan A K, Snape J R, Fearnside D, et al. Composition and diversity of ammonia-oxidizing bacteria community in wastewater treatment reactors of different design treating identical wastewater [J]. FEMS Microbiology Ecology, 2003, 43(2):195-206.
[22]Limpiyakorn T, Shinohara Y, Kurisu F, et al. Communities of ammonia-oxidizing bacteria in activated sludge of various sewage treatment plants in Tokyo [J]. FEMS Microbiology Ecology, 2005,54(2):205-217.
[23]Lydmark P, Almstrand R, Samuelsson K, et al. Effects of environmental conditions on the nitrifying population dynamics in a pilot wastewater treatment plant [J]. Environmental Microbiology, 2007,9(9):2220-2233.
Analysis of ammonia-oxidizing bacteria in catching bed reactor at different DO concentrations by RFLP
Z HANG Yan*, ZHU Min, LIU Huan-guang, SUN Feng-xia, GAN Zhi-ming, CHEN-Jing, SHI-Yang, XIE Hang-ji (Key Laboratory of Water Quality Science and Water Environment Recovery Engineering, Beijing University of Technology, Beijing 100124, China). China Environmental Science, 2014,34(9):2387~2393
To investigate the influence of DO concentration on the characteristics of catching bed reactor, the removal efficiency of COD, NH4+-N and TN were analyzed under the DO concentrations at 1.0~2.0mg/L, 2.0~3.0mg/L and 3.0~4.0mg/L, respectively. Meanwhile, ammonia-oxidizing bacteria (AOB) community and diversity in each reactor were examined using restricted fragment length polymorphism (RFLP), and sequencing of amoA genes. The results showed that higher COD and NH4+-N removal efficiency were
under different DO concentrations, NH4+-N removal efficiency increased with the DO concentration increase. The diversity of AOB was abundant in different DO concentrations, but the communities and species of AOB were effect greatly by DO concentration.
ammonia-oxidzing bacteria (AOB);amoA gene;RFLP;biofilm
X172
A
1000-6923(2014)09-2387-07
張 巖(1962-),女,山東煙臺人,副教授,博士,主要從事污水處理與資源化方面的研究.
2014-01-22
國家自然科學基金資助項目(50978004)
* 責任作者, 副教授, yzhang@bjut.edu.cn

