大黃魚cyclin B 1和cdc 2 cDNA序列特征及組織表達分析
蔡明夷,周 鵬,韓坤煌,謝芳靖,張子平,王藝磊*
(1.集美大學 水產學院,農業部東海海水健康養殖重點實驗室,福建 廈門361021;2.福建省國家級大黃魚原種場,寧德市富發水產有限公司,福建 寧德352103;3.美國西東大學生物學系,新澤西州 南奧蘭治07090)
魚類的卵子發生通常要經歷減數分裂的中斷、暫停與再啟動等步驟,而精子發生則是一個減數分裂連續的過程.因而,魚類的性腺被認為是研究減數分裂周期調控的理想模型[1].許多基因參與了魚類配子發生減數分裂過程的調控.其中,成熟促進因子(maturation promoting factor,MPF)是誘導減數分裂暫停的卵子重新進入成熟過程的關鍵因子[2].MPF包括2個亞基:調節亞基細胞周期蛋白B(cyclin B,CB)和周期蛋白依賴性蛋白激酶(cyclin-dependent kinase 1,CDK1,由cdc2基因編碼)[3].CB和CDK1結合形成 MPF前體,經過磷酸化/去磷酸化作用成為具有活性的MPF.活化的MPF可以催化一系列靶蛋白的磷酸化,誘導G2期向M期轉換;在卵子發生過程中,則誘導發生卵泡破裂(GVBD)、停滯減數分裂啟動等細胞行為的發生[4-6].現有研究資料表明,MPF的有序表達、活化及降解是保證兩性生殖生物配子發生、成熟與分化等生物過程有序進行的重要條件[7].
大黃魚(Larimichthys crocea)是我國特有海水魚類,也是最重要的海水養殖魚類之一[8].其生長具有明顯的雌雄二態性,雌性生長速度明顯快于雄性[9].研究大黃魚性腺發育與性別分化分子機制,可為開發高效大黃魚性別控制方法、提高大黃魚生殖控制技術水平奠定基礎.然而,目前關于大黃魚性腺發育和配子發生的知識主要集中于細胞水平[10-13],分子水平的研究才剛剛起步,研究報道較少[14-15].因此,本研究利用cDNA 末端快速擴增法(rapid-amplification of cDNA ends,RACE)克隆了大黃魚cyclin B1 基因(Lc-cb1)和cdc2 基因(Lccdc2)的cDNA序列,并利用實時熒光定量PCR(qRTPCR)技術分析了這2個基因在大黃魚性腺及其他組織的mRNA水平,為解析MPF在大黃魚性腺發育以及配子發生中的作用機理奠定基礎.
1 材料與方法
1.1 材料收集
實驗大黃魚取自寧德市富發水產有限公司,雌雄各5尾,魚體質量(524.7±118.2)g.活魚尾椎取血后迅速解剖取精巢、卵巢、肌肉、肝臟、腦、腎臟等組織,立即放入液氮速凍,取出置-80℃冰箱中保存備用.
1.2 總RNA的提取和cDNA合成
按Zhang等[16]描述的方法從組織中提取總RNA,然后用DNA酶Ⅰ(DNaseⅠ)37℃處理30 min,去除可能混雜的基因組DNA,用瓊脂糖凝膠電泳驗證RNA的完整性.按照SMART-RACE cDNA擴增試劑盒說明書(Clontech,美國)所描述的方法,以3μg 總 RNA 為模板、Oligo(dT)為引物,并在SMARTⅡOligo的介導下,利用 M-MLV反轉錄酶(Promega,上海)在37℃下反應90min,合成RACE適用的cDNA第1鏈.合成產物在70℃下處理15 min滅活酶,然后貯存于-20℃冰箱中備用.
1.3 Lc-cb1 和Lc-cdc2 cDNA的克隆與測序
利用SMART-RACE方法獲得Lc-cb1 和Lc-cdc2 cDNA 5′端和3′端擴增產物.第1輪擴增以cDNA第1條鏈為模板,正反向引物分別為基因特異外引物(GSP)和通用引物組合(UPM);第2輪擴增的模板為第1輪擴增產物,正反向引物為GSP內引物和巢式通用引物(NUP).其中,GSP內引物和外引物是基于前期實驗構建的EST數據庫中的Lc-cb1和Lc-cdc2 cDNA部 分 序 列[17]、采 用 Primer 5.0 軟 件 設 計 的 (表1).UPM 和 NUP 序列參見SMARTer RACE cDNA擴增試劑盒說明書(Clontech,美國).25μL PCR反應體系包括:0.5μL 模板 DNA,0.5μL 10mmol/L GSP,2.5μL 10mmol/L UPM(或 NUP),2.5μL 10×PCR 緩沖液,0.5μL 10mmol/L dNTP 混合液,0.5μL Taq DNA聚合酶和18μL雙蒸水.PCR反應條件為:95℃預變性3min;然后95℃ 變性30s,60~68℃(詳見表1)退火30s,72℃90s,32個循環;72℃繼續延伸10min.
上述第2輪PCR產物經質量分數為1%的瓊脂糖凝膠電泳后,目標片段經回收純化,克隆到pMD18-T質粒(Takara,日本),進一步轉化大腸桿菌(Escherichia coli)JM109并增殖.從陽性克隆中分離質粒,送上海捷瑞生物工程有限公司測序.序列結果用VecScreen(http:∥www.ncbi.nlm.nih.gov/VecScreen/VecScreen.html)去除載體序列,再用 BLAST2(http:∥www.ncbi.nlm.nih.gov/blast/bl2seq/wblast2.cgi)比對拼接出cDNA全長序列.
1.4 qRT-PCR
大黃魚精巢、卵巢、肌肉、血、脾、肝、腎和腦等8個組織的樣品用于qRT-PCR,β-actin為內參基因,以檢查Lc-cb1 和Lc-cdc2 mRNA 組織表達特征.反應體系包括:cDNA 第1鏈9μL,10mmol/L qRT-PCR正反向引物各0.5μL(表1),SYBR Green Realtime PCRMaster Mix(Life Technology,美國)10μL.反應體系混勻后,在ABI 7500PCR儀器上完成熱循環反應和熒光檢測.PCR反應條件為:50℃保溫2min,95℃變性1min;95℃變性15s、60℃退火1min,重復循環40次.每種組織檢測了取自不同個體的4個樣品,每個樣品做4次重復.

表1 本研究所用引物Tab.1 Primers used in this study
mRNA表達水平用相對定量(relative quantification,RQ)平均值±標準差來表示.mRNA表達量平均值的組織間差異的顯著性,用SPSS 15.0統計軟件的Bonferroni模型作多重比較進行單因素方差分析,p<0.05為差異顯著.
1.5 基因序列的生物信息學分析
推導的氨基酸序列的比對和保守區域結構比較利用GenBank網站的BLAST算法(http:∥www.ncbi.nlm.nih.gov)在線完成.蛋白的分子質量和等電點預測利用 Compute pl/Mw tool(http:∥ca.expasy.org/tools/pi_tool.html)完成.用InterProScan軟件4.8(ftp:∥ftp.ebi.ac.uk/pub/software/unix/iprscan/index.html)分析氨基酸序列的結構域.氨基酸序列的多重比較利用BioEdit軟件(http:∥www.mbio.ncsu.edu/BioEdit/bioedit.html)完成.蛋白進化樹利用 Mega 5.1軟件(http:∥www.megasoftware.net/)的鄰接算法構建.
2 結 果
2.1 Lc-cb1 和Lc-cdc2 cDNA序列特征
將3′RACE獲得片段、5′RACE獲得片段和文庫插入cDNA片段以BLAST 2軟件進行序列拼接后,獲得Lc-cb1 基因的全長cDNA 序列1 882bp(Gen-Bank登錄號FJ195327),如圖1.其中,編碼區(ORF)長度為1 194bp,可編碼397個氨基酸的蛋白.5′-非翻譯區(UTR)長度為63bp;3′-UTR的長度為580bp(去除poly A),含有2個加尾信號(AUUAAA),和4個多聚腺苷酸化元件(cytoplasmic polyadenylation elements,簡稱為 CPEs;A/UUUUUAU/A).推導的LC-CB1蛋白預測分子質量約為44.56ku,等電點約為8.76,序列中含有一個典型的周期蛋白盒(166~313aa),其中272~276aa為CB特有的蛋白酶K位點(RRxSK),N端附近有一個周期蛋白A和CB共有的毀壞盒(RxALGxIxN,32~40aa).氨基酸序列同源性比較結果顯示,該序列與爪哇青鳉(Oryzias javan-icus,84%)、青 鳉 (Oryzias latipes,83%)、斑 馬 魚(Danio rerio,67%)、非 洲 爪 蟾 (Xenopus laevis,64%)、小鼠(Mus musculus,61%)、智人(Homo sapiens,63%)等動物的CB1均有較高的同源性.
將3′RACE獲得片段、5′RACE獲得片段和文庫插入cDNA片段以BLAST 2軟件進行序列拼接后,獲得Lc-cdc2基因全長cDNA序列 1 151bp(GenBank登錄號 FJ800565),如圖2.其中,ORF長度為 912bp,5′-UTR長度為72bp,3′-UTR的長度為150bp(去除poly A).在poly A上游21bp處有一個典型的加尾信號(AATAAA ).Lc-cdc2 cDNA 可編碼303個氨基酸的蛋白,預測分子質量約為34.58ku,等電點約為8.80.推導的蛋白序列中包含一個CDK1典型的絲氨酸/蘇氨酸激酶催化結構域(4~287aa),內含絲氨酸/蘇氨酸蛋白激酶活性位點(124~136aa)、與ATP結合相關的保守序列GxGxxGxV(11~17aa)、與周期蛋白結合相關的保守序列PSTAIRE(45~51aa)等.氨基酸序列同源性比較結果顯示,該序列與龜殼攀鱸(Anabas testudineus,97%)、爪哇青鳉(95%)、虹鱒(Oncorhynchus mykiss,95%)、斑馬魚(90%)、大西洋鮭(Salmo salar,88%)、胡瓜魚(Osmerus mordax,92%)、金魚(Carassius auratus,90%)、非洲爪蟾(85%)、褐家鼠(Rattus norvegicus,81%)和智人(82%)等物種的CDK1均有較高的同源性.
2.2 同源性分析和系統發育分析
將推導的LC-CB1氨基酸序列與爪哇青鳉(登錄號Q9DGA0)、斑馬魚(登錄號 BAA92876)、青鳉(登錄號Q9IBG1)、非洲爪蟾(登錄號 AAH41302)、智人(登錄號P14635)和小鼠(登錄號CAA45968)等6個物種的CB1氨基酸序列進行多序列比對.結果表明,大黃魚及其他6個物種都具有CB1特征結構域(詳見圖3).根據Lc-cb1 cDNA序列推導的氨基酸序列與上述6個物種的CB1氨基酸序列一致性在46%~84%之間,N端近中部的氨基酸序列變化較大,其他區域的保守性較高.
將推導的大黃魚CDK1氨基酸序列與爪哇青鳉(登錄號BAB17220)、龜殼攀鱸(登錄號AAS59851)、胡瓜魚(登錄號 AC0008896)、大西洋鮭(登錄號 NP 001134623)、虹鱒(登錄號 NP 001118132)、金魚 (登錄號AFV07384)、斑馬魚(登錄號 NP 997729)、非洲爪蟾(登 錄 號 NP 001080093)、鴨 嘴 獸 (Ornithorhynchus anatinus,登 錄 號 XP 001509674)、原 雞 (Gallus gallus,登 錄 號 NP 990645)、智 人 (登 錄 號 NP 001777)、褐家鼠(登錄號 NP 062169)和果蠅(Drosophila melanogaster,登錄號 NP 476797)等13個物種CDK1氨基酸序列進行多序列比對.結果表明,大黃魚CDK1與其他13個物種一樣,都具有CDK1特征結構域(詳見圖4).根據Lc-cdc2 cDNA序列推導的氨基酸序列與上述的6種魚類的CDK1氨基酸序列一致性在88%~95%之間,與果蠅的CDK1氨基酸序列一致性也有71%.

圖1 Lc-cb1 cDNA全長及推導的氨基酸序列Fig.1 The nucleotide and deduced amino acid sequences of Lc-cb1 cDNA
通過鄰接法構建的CB系統發育樹(圖5(a))顯示,大黃魚CB1與青鳉、爪哇青鳉和斑馬魚等魚類的CB1首先聚在一個小枝上,再與非洲爪蟾、小鼠和智人等其他脊椎動物CB1所聚的一小枝聚為一個較大分枝;與斑馬魚、非洲爪蟾、小鼠和智人等物種的CB2關系較遠,與麗新桿線蟲(Caenorhabditis elegans)的CB3的關系更遠.通過鄰接法構建的CDK1系統發育樹(圖5(b))顯示,大黃魚首先與龜殼攀鱸聚為一枝,并與爪哇青鳉等其他6種魚類聚為一個二級分枝;其他5種非魚類脊椎動物聚為另一個二級分枝;果蠅這種無脊椎動物的CDK1與其他13種脊椎動物距離最遠.

圖2 Lc-cdc2基因cDNA全長及推導的氨基酸序列Fig.2 The nucleotide and deduced amino acid sequences of Lc-cdc2 cDNA
2.3 Lc-cb1 和Lc-cdc2 基因在大黃魚各器官中的表達
qRT-PCR分析大黃魚不同組織中Lc-cb1和Lccdc2基因mRNA水平如圖6所示.Lc-cb1和Lc-cdc2基因在大黃魚各組織均有表達.2個基因的表達組織譜具有相似特征,并呈現出較強的組織特異性:在卵巢的表達量均為最多,在精巢的表達量次之,其他組織僅有微量表達,表達量最少的是腦組織.Lc-cb1卵巢表達量約為腦表達量的2 145倍,Lc-cdc2卵巢表達量約為腦表達量的349倍.
3 討 論

圖3 大黃魚及其他物種CB1氨基酸序列的多重比較Fig.3 Multiple alignment of the CB1amino acid sequence between L.croaker and other species
本研究獲得了大黃魚 Lc-cb1 和Lc-cdc2 cDNA全長,GenBank登錄號分別為FJ195327和FJ800565.根據所獲得的 Lc-cb1 cDNA 序列(FJ195327)推導的氨基酸序列與其他已登錄GenBank的魚類的CB1氨基酸序列有較高相似性(67%~84%),具有周期蛋白家族的特征,如周期蛋白盒和RxALGxIxN序列;周期蛋白A和CB所特有的毀壞盒以及CB特征的蛋白酶K位點.本研究獲得的Lc-cdc2 cDNA (FJ800565)推測的氨基酸序列與其他已登錄GenBank的魚類的CB1氨基酸序列也有較高相似性(88%~97%),且具有CDK1的特征序列,如絲氨酸/蘇氨酸激酶催化結構域、ATP結合相關的保守序列GxGxxGxV和與周期蛋白結合相關的保守序列PSTAIRE等.可見,生物信息學分析結果證明,本研究所獲得的2條序列分別具備了cb1和cdc2基因cDNA的基本特征.
CB是MPF的調節亞基.脊椎動物中,涉及M期調控的周期蛋白主要有4種亞型.各亞型的分布和功能存在差異.例如,在智人培養細胞中,CB1定位在微管,CB2主要分布在高爾基器上[18].在非洲爪蟾卵子中,CB2在第一次減數分裂雙極紡錘體的形成中發揮作用[19-20].小鼠中,CB1是胚胎發育和性腺發育的關鍵基因并能夠補償CB2的缺失[21].在雞10日齡胚胎中,CB3兼具周期蛋白A和CB的特點,既能與CDK1也能與CDK2聯合作用[22].本研究基于氨基酸序列比對,構建了CB進化樹.結果表明,大黃魚CB1與其他魚類CB1關系最近;與親緣關系較遠的哺乳動物(如智人類和小鼠)CB1的關系要近于與近緣種(如斑馬魚)CB2的關系.這與虹鱒、青鳉、日本鰻鱺(Anguilla japonica)等其他魚類中得到的結果一致[23-25].CB 的mRNA通常有一條較長的3′-UTR,例如智人(NM031966)cb1 mRNA 3′-UTR 的長度為615bp.3′-UTR可能通過影響mRNA的多聚腺苷化/去腺苷化、翻譯效率、翻譯定位和mRNA穩定性來實現基因表達調控.cb1 3′-UTR上含有參與mRNA翻譯調控的順式元件,其中研究最為深入的是CPE元件.CPE為富含U的序列(A/UUUUUAU/A),在卵子成熟過程中通過幫助聯接加尾信號活化CB的翻譯[26].在卵子中顯微注射CPE能夠誘導CB1蛋白合成[27].Lccb1 mRNA 3′-UTR長度為580bp,初步分析發現其中含有4個CPE元件、多個富U類的CPE序列和2個加尾信號,與斑馬魚、非洲爪蟾、中華絨螯蟹(Eriocheir sinensis)等生物cb1 3′-UTR 結構基本相似,但CPE元件的數目和分布在不同物種間有所變化.cb1 mRNA 3′-UTR的總體結構、CPE元件的數目和分布均可能影響翻譯效率,并可能與生物的具體功能需求相關[28].
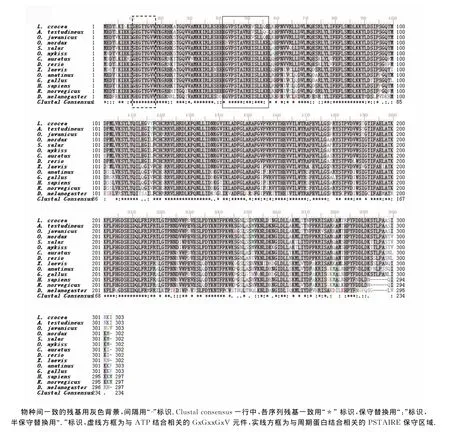
圖4 大黃魚及其他物種CDK1氨基酸序列的多重比較Fig.4 Multiple alignment of the CDK1amino acid sequence between L.croaker and other species

圖5 基于大黃魚和其他物種的CB1(a)和CDK1(b)氨基酸序列構建系統發育樹Fig.5 Phylogenetic tree based on the amino acid sequences of CB1(a)and CDK1(b)between L.crocea and other species
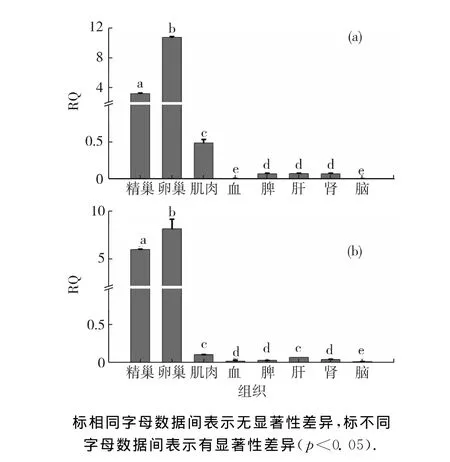
圖6 大黃點Lc-cb1(a)和Lc-cdc2(b)mRNA的組織表達圖譜Fig.6 Tissue expression profiles of Lc-cb1(a)and Lc-cdc2(b)mRNA transcripts in L.crocea
CDK1是一個高度保守的小蛋白,分子質量約34 ku.CDK1參與了細胞周期的全部進程,如細胞周期啟動、從G1期向S期轉換、從S期向G2期轉換,以及G2期向M期轉換等.CDK1能夠有序行使功能主要依靠三方面的調控:調節亞基周期蛋白的結合、保守殘基的磷酸化、以及CDK抑制物(CKI)結合.調節亞基周期蛋白的類型,通過影響CDK1活性位點的位置與結構,決定周期蛋白-CDK復合物的適合底物和特殊功能[29].其中,CB1-CDK1復合物主要調控 G2期向M期轉換過程.CB1和CDK1結合形成MPF前體,需要活化才能行使功能.在魚類卵子成熟過程中,目前已發現2種形式的MPF活化機制.在第1種機制中,CB1在即將發生GVBD時,在激素作用下開始合成,并立即與CDK1結合形成MPF前體;之后通過CDK1上Thr161的磷酸化,即可完成MPF的活化,如金魚[5].在第2種機制中,未成熟卵子已存在完整的MPF前體,MPF的活化既需要磷酸化,還需要CDC25催化的去磷酸化作用,如龜殼攀鱸[30].基于氨基酸序列的進化樹顯示,大黃魚與龜殼攀鱸的CDK1相似性最高(圖5(b)).那么,大黃魚MPF的活化機制是否與龜殼攀鱸一致,MPF的活化機制的分化是否與物種的分化相符,尚需要進一步研究.
qRT-PCR分析結果顯示,Lc-cb1 和Lc-cdc2 mRNA表達具有較強的組織特異性,性腺中Lc-cb1和Lc-cdc2 mRNA水平極顯著地高于其他組織.這與前人在虹鱒、青鳉和鋸緣青蟹等生物中觀察到的結果一致[23-24,31].CB1和CDK1組成的MPF是細胞周期調控因子,Lc-cb1和Lc-cdc2在大黃魚性腺中高水平表達,與性腺中有絲分裂和減數分裂活動活躍的特點相符合.
4 結 論
大黃魚是我國最重要的海水魚類.本研究克隆大黃魚Lc-cb1和Lc-cdc2基因全長cDNA序列,并分析了這2條序列及推測蛋白序列特征,發現大黃魚與龜殼攀鱸CDK1序列的相似性高達97%;同時,研究還揭示大黃魚中Lc-cb1和Lc-cdc2 mRNA的表達具有不嚴格的組織特異性,在性腺中優勢表達,表明MPF在大黃魚性腺發育以及配子發生中具有重要作用.本文研究結果為進一步揭示MPF在大黃魚性腺發育以及配子發生中的作用機理奠定了基礎.
[1]Yamashita M. Molecular mechanisms of meiotic maturation and arrest in fish and amphibian oocytes[J].Semin Cell Dev Biol,1998,9(5):569-579.
[2]Masui Y,Markert C L.Cytoplasmic control of nuclear behavior duringmeiotic maturation of frog oocytes[J].J Exp Zool,1971,177(2):129-145.
[3]Lohka M J,Hayes M K,Maller J L.Purification of maturationpromoting factor,an intracellular regulator of early mitotic events[J].Proc Natl Acad Sci USA,1988(9),85:3009-3013.
[4]Kondo T,Yanagawa T,Yoshida N,et al.Introduction of cyclin B induces activation of the maturation-promoting factor and breakdown of germinal vesicle in growing ze-brafish oocytes unresponsive to the maturation-inducing hormone[J].Dev Bio,1997,190(1):142-152.
[5]Katsu Y,Yamashita M,Kajiura H,et al.Behavior of the components of maturation-promoting factor,cdc2kinase and cyclin B,during oocyte maturation of goldfish[J].Dev Biol,1993,160(1):99-107.
[6]Chesnel F,Bazile F,Pascal A,et al.Cyclin B2/cyclin-dependent kinase1dissociation precedes CDK1Thr-161dephosphorylation upon M-phase promoting factor inactivation in Xenopus laevis cell-free extract[J]Int J Dev Biol,2007,51:297-305.
[7]虞晉晉,葉海輝,黃輝洋.水生動物細胞周期蛋白研究進展[J].廈門大學學報:自然科學版,2006,45(增刊2):185-189.
[8]劉家富,劉招坤.福建閩東大黃魚Larimichthys crocea (Richardson)產業展望[J].現代漁業信息,2008,23(12):3-5.
[9]王清印.海水養殖研究新進展[M].北京:海洋出版社,2008:123-130.
[10]林丹軍,張健,駱嘉,等.人工養殖的大黃魚性腺發育及性周期研究[J].福建師范大學學報:自然科學版,1992,8(3):81-87.
[11]尤永隆,林丹軍,陳蓮云.大黃魚的精子發生[J].動物學研究,2001,22(6):461-466.
[12]方永強,翁幼竹,周晶,等.大黃魚性早熟的機制:精巢中間質細胞和足細胞的顯微與亞顯微結構[J].臺灣海峽,2002,2(3):276-279.
[13]游秀容,蔡明夷,姜永華,等.大黃魚性腺性別分化的組織學觀察[J].水產學報,2012,36(7):1057-1064.
[14]Pu L L,Han K H,Xie F J,et al.Molecular cloning,characterization,and gene expression of the androgen receptor in the large yellow croaker,Larimichthys crocea[J].Fish Physiol Biochem,2013,39(2):309-324.
[15]周鵬,張子平,王藝磊,等.大黃魚ubc9基因的克隆和組織表達[J].生物技術通報,2009(8):76-82.
[16]Zhang Z,Wu R S,Mok H O,et al.Isolation,characterization and expression analysis of a hypoxia-responsive glucose transporter gene from the grass carp,Ctenopharyngodon idellus[J].Eur J Biochem,2003,270(14):3010-3017.
[17]Zhou P,Zhang Z P,Wang Y L,et al.EST analysis and identification of gonad-related genes from the normalized cDNA library of large yellow croaker,Larimichthys crocea[J].Comp Biochem Physiol Part D Genomics Proteomics,2010,5(2):89-97.
[18]Jackman M,Firth M,Pines J.Human cyclins B1and B2 are localized to strikingly different structures:B1to microtubules,B2primarily to the Golgi apparatus[J].EMBO J,1995,14(8):1646-1654.
[19]Ihara J,Yoshida N,Tanaka T,et al.Either cyclin B1or B2is necessary and sufficient for inducing germinal vesicle breakdown during frog (Rana japonica)oocyte maturation[J].Mol Reprod Dev,1998,50(4):499-509.
[20]Kotani T,Yoshida N,Mita K,et al.Requirement of cyclin B2,but not cyclin B1,for bipolar spindle formation in frog(Rana japonica)oocytes[J].Mol Reprod Dev,2001,59(2):199-208.
[21]Brandeis M,Rosewell I,Carrington M,et al.Cyclin B2-null mice develop normally and are fertile whereas cyclin B1-null mice die in utero[J].Proc Natl Acad Sci USA,1998,95(8):4344-4349.
[22]Gallant P,Nigg E A.Identification of a novel vertebrate cyclin:cyclin B3shares properties with both A-and B-type cyclins[J].EMBO J,1994,13(3):595-605.
[23]Qiu G F,Ramachandra R K,Rexroad C E 3rd,et al.Molecular characterization and expression profiles of cyclin B1,B2and CDC2kinase during oogenesis and spermatogenesis in rainbow trout (Oncorhynchus mykiss)[J].Anim Reprod Sci,2008,105(3/4):209-225.
[24]Mita K,Ohbayashi T,Tomita K,et al.Differential expression of cyclins B1and B2during medaka (Oryzias latipes)spermatogenesis[J].Zool Sci,2000,17(3):365-374.
[25]Kajiura-Kobayashi H,Kobayashi T,Nagahama Y.The cloning of cyclin B3and its gene expression during hormonally induced spermatogenesis in the teleost,Anguilla japonica [J].Biochem Biophys Res Commun,2004,323:288-292.
[26]Huarte J,Stutz A,O′Connell M L,et al.Transient translational silencing by reversible mRNA deadenylation[J].Cell,1992 69:1021-1030.
[27]de MoorC H,Richter J D.Cytoplasmic polyadenylation elements mediate masking and unmasking of cyclin B1 mRNA[J].EMBO J,1999,18:2294-2303.
[28]Zhang Y,Sheets M D.Analyses of zebrafish and Xenopus oocyte maturation reveal conserved and diverged features of translational regulation of maternal cyclin B1mRNA[J].BMC Dev Biol,2009,9:7.
[29]Nigg E A.Cyclin-dependent protein kinases:key regulators of the eukaryotic cell cycle[J].Bioessays,1995,17(6):471-480.
[30]Basu D,Navneet A K,Dasgupta S,et al.Cdc2-cyclin B-induced G2to M transition in perch oocyte is dependent on Cdc25[J].Biol Reprod,2004,71(3):894-900.
[31]Han K,Dai Y,Zou Z,et al.Molecular characterization and expression profiles of cdc2 and cyclin Bduring oogenesis and spermatogenesis in green mud crab (Scylla paramamosain)[J].Comp Biochem Physiol B Biochemmol Biol,2012,163(3/4):292-302.

