海馬齒根際降解可溶性蛋白質的研究
楊 芳,李 凱,黃凌風,3*,朱小明
(1.廈門大學海洋與地球學院,福建 廈門361102;2.廈門大學深圳研究院,廣東 深圳518057;3.濱海濕地生態系統教育部重點實驗室(廈門大學),4.廈門大學環境與生態學院,福建 廈門361102)
植物修復(phytoremediation)是指利用植物及其根際微生物的代謝活動來吸收、積累或降解轉化環境中的污染物以達到凈化、修復環境的目的.植物修復方式包括植物萃取、根際過濾、植物固定、植物揮發、植物降解和根際降解.其中,根際降解又稱為植物-微生物協同修復,是指綠色植物及其共存微生物體系,通過促進植物的根系和根系周圍微生物的活性和生化反應,使污染物釋放、吸收和轉化而降低或去除其毒性,達到凈化環境的一種污染治理技術.
植物根際環境存在著胞外酶(extracellular enzymes),胞外酶可能由植物根系釋放,也可能由根際微生物分泌.在海洋生態系統中,只有小部分的溶解有機物(DOM)可以直接通過細胞膜被異養微生物所利用,但這遠遠無法滿足微生物的營養需求.約80%~90%的DOM以高聚物(如蛋白質等)的形式存在,但無法被直接吸收利用,只有通過胞外酶的解聚作用才能使它們轉變為小分子有機物,從而被異養微生物吸收[1-3].大部分已知的亮氨酸氨基肽酶能以胞外酶的形式出現,并能通過水解那些不能直接穿透進入有機體的多肽和蛋白質,釋放出游離的氨基酸,從而供細菌利用.已有的研究表明,胞外酶的分解作用是有機質降解的關鍵因素,胞外酶活性的大小能從一定程度上反映有機物的降解速率.胞外酶在有機質的礦化過程中發揮著關鍵作用,控制著生態系統的物質循環,在一定程度上決定著生態系統的凈化功能[4].胞外酶活性還可以作為反映水體富營養化程度的指標[5].因此,胞外酶的研究對于全面理解生態系統的物質循環、評估水域環境質量和探討生態修復機制等都具有重要意義.
海馬齒(Sesuvium portulacastrumLinn.)又名濱水菜,為番杏科(Aizonaceae)海馬齒屬(Sesuvium)植物,是生長在海邊沙地或鹽堿地的多年生匍匐性肉質草本植物,在我國主要分布于福建、廣東、海南、臺灣及澎湖列島的海岸[6],浮床種植海馬齒可在海水環境中正常生長.海洋灘涂耐鹽植物海馬齒作為一種應用于海水生態浮床技術的重要修復植物,其對富營養化水體的氮、磷營養鹽和有機質的去除能力已得到了一定的認識和肯定[7],但關于其修復機制,尤其是根圈系統對海水環境中蛋白質等有機質降解的相關酶學機制的研究報道還很少.牛血清白蛋白(bovine serum albumin,BSA)是常用的蛋白質標準品,在可溶性蛋白質含量的測定等蛋白質研究領域中有廣泛的應用[8-10].本文報道了水培海馬齒對以BSA為代表的可溶性蛋白質的根際降解作用及其與根際微生物胞外水解酶活性有關的影響因素,旨在探討海馬齒根系及其根際微生物在蛋白質等有機質降解中所發揮的重要作用,為更好地利用海馬齒生態浮床修復海水環境中的有機氮提供理論依據.
1 材料與方法
1.1 材 料
實驗用海馬齒植株為本實驗室從西沙群島引種并在溫室中通過水培方法建立的植株,培養用水為廈門西海域海水.單株平均鮮質量為(15.0±0.5)g,根系發育良好.
1.2 實驗設置
取同一批預培養的海馬齒植株,剔除枯黃葉片并清洗后稱量,選擇長勢均勻、鮮質量為每株(15.0±0.5)g的植株,在經0.22μm濾膜過濾并滅菌的海水中暫養7d,以獲得含根際微生物的預培養水.
設置了抑菌和不抑菌2組實驗,每組又分為種植海馬齒的植物組、不種植海馬齒的細菌組和空白對照組.每組分別設置3個平行樣.
抑菌實驗根據 Weinberger[11]的方法采用3種抗生素,分別是氨芐霉素(ampicillin)、頭孢噻肟(cefotaxime)和萬古霉素(vancomcin),添加質量濃度均為100mg/L.這3種抗生素既可以同時抑制G+和G-菌形成細胞壁,又不會對植物的生理活動產生影響[12].預實驗表明該抗生素濃度能抑制水中微生物的繁殖,但不影響植物的正常生長.不抑菌實驗不添加抗生素.
每個樣品的培養液初始體積是500mL.植物組和細菌組的培養用水是400mL經0.22μm濾膜過濾的滅菌海水和100mL根際預培養水混合而成.對照組的培養用水是400mL經0.22μm濾膜過濾的滅菌海水和100mL經0.22μm濾膜過濾并滅菌的根際預培養水混合而成.每組加入營養鹽以滿足植物生長的需要,各種營養鹽按照Hoagland配方添加.
不抑菌實驗中,對照組用于反映非生物因素在蛋白質降解中的作用,細菌組用于反映根際細菌和非生物因素在蛋白質降解中的作用,植物組用于反映植物、根際細菌和非生物因素在蛋白質降解中的作用.抑菌實驗中的植物組用于反映植物根系和非生物因素在蛋白質降解中的作用.利用差減法計算的結果可以分別顯示植物與根際細菌聯合作用、植物根系以及根際細菌對蛋白質根際降解的貢獻率.
實驗裝置由柱狀玻璃容器(Ф:5.0cm,L:30 cm)、日光燈光源和取樣管組成.柱狀玻璃容器外表面和瓶口緊密包裹鋁箔,以防止外源微生物污染、蛋白質的光解和藻類的生長.日光燈置于柱狀玻璃容器上方約50cm 處,在室溫(28±2)℃,12h光照(6 000lx)和12h黑暗交替條件下培養.為了減少取樣時根際水體的擾動,用一次性無菌注射管作為取樣管,放置于柱狀玻璃瓶中,外用橡皮筋固定,并用止水夾保持培養體系密閉以防止外源微生物污染和工作液中溶解氧的改變.
每隔12h取樣一次,每次取樣時用一次性注射管吸取10mL水樣用于BSA濃度、細菌密度和亮氨酸氨基肽酶活性的測定.分別在36和72h取樣后,再次添加BSA(質量濃度為10mg/L)、Hoagland營養鹽和3種抗生素(質量濃度均為100mg/L,僅在抑菌實驗組中添加).
1.3 主要分析指標和方法
1.3.1 BSA含量的測定
采用考馬斯亮藍法.取125mg考馬斯亮藍G-250充分溶解在100mL 95%(體積分數)的乙醇中,另取15mL 37%(質量分數)的 HCl和5.06g NaCl溶解在適量水中后,與考馬斯亮藍乙醇液混合并定容至1 000mL,用0.45μm 濾膜過濾,即為顯色液.測定時,取5mL顯色液與1mL水樣混合,放置5min后用Spectum 756PC型紫外可見分光光度計在波長595nm下測定吸光值.
同時,配制質量濃度為0,2,4,6,8,10,12,14,16,18,20μg/mL的BSA溶液,繪制標準曲線.
1.3.2 亮氨酸氨基肽酶活性的測定
采用熒光模擬底物法(fluorogenic model substrate,FMS)[13].如圖1所示,熒光模擬底物包含一個熒光生色團和一個小分子,熒光生色團采用4-甲基-7-氨基香豆素(MCA),小分子為氨基酸,兩者之間通過肽鍵相連.在氨基肽酶的作用下,復合分子發生斷裂,熒光基團顯色.胞外酶的活性越高,復合分子斷裂的越多、越快,因此熒光強度越高.這種模擬底物的胞外酶水解作用符合米氏方程,檢測限低,適合短時間條件下的胞外酶測定.該方法以其高度的靈敏度已經廣泛應用于各種水體的胞外酶活性測定中[14-17].
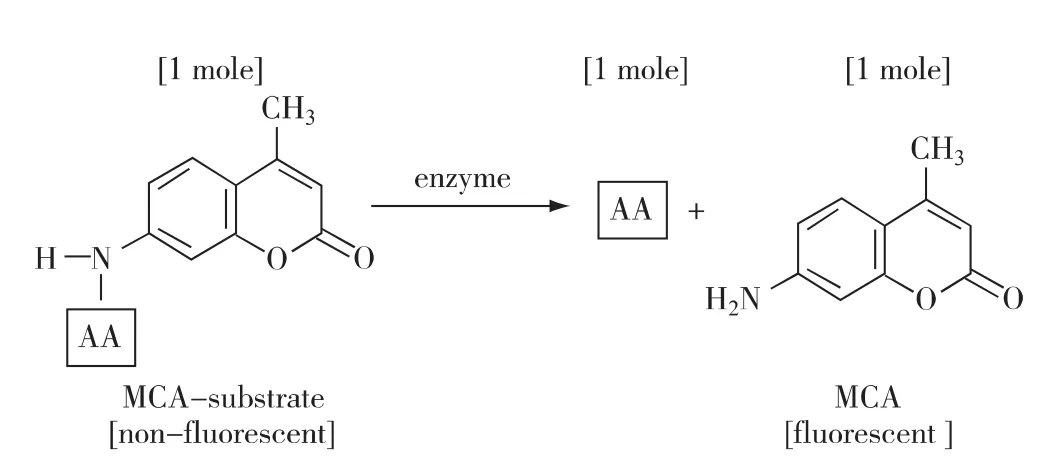
圖1 熒光模擬底物水解方程式Fig.1 Reaction equator of model substrate
標準曲線的繪制:配制100μmol/L MCA的乙二醇單甲醚溶液,再用過濾滅菌海水稀釋成0.000 5,0.001,0.002 5,0.005,0.007 5,0.01,0.025,0.05,0.075,0.1,0.25,0.5,0.75,1μmol/L 14個濃度的溶液.用島津RF5301PC熒光分光光度計迅速測定各溶液的熒光強度.MCA激發光和發射光的波長分別為380和440nm.狹縫寬Ex=3nm,Em=3nm.
酶活性的測定方法如下:移取3mL水樣到5個5 mL的凍存管中(2個空白樣和3個平行樣),立即往平行樣中加入0.5mmol/L熒光模擬底物工作液120 μL,使其終濃度為20μmol/L;同時在空白樣中加入80μL濃度為100mmol/L的HgCl2溶液使胞外酶失活,在20℃下避光培養2h.培養結束后在空白樣中加入等量的熒光模擬底物工作液,并用HgCl2溶液終止平行樣中的反應.用島津RF5301PC熒光分光光度計迅速測定樣品的熒光強度.按下式計算水樣中的胞外酶活性:
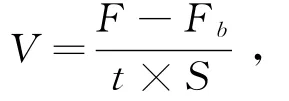
式中,V 為胞外酶水解底物的速率(μmol/(L·h));F為平行樣品的熒光強度(平均值,μmol/L);Fb為空白樣品的熒光強度(平均值,μmol/L);S為單位濃度標準熒光物質的熒光強度(S=79.807),即標準工作曲線的斜率;t為培養時間(h).
1.3.3 細菌密度測定
采用DAPI染色的熒光顯微計數法[18],測定前將孔徑為0.22μm的硝酸纖維濾膜用蘇丹黑B染液(500mg Sudan Black B溶于200mL 2%(質量分數)乙酸中)染色4h以上,使之變黑.取1mL水樣加入2 mL滅菌離心管中,加入40μL 25%(體積分數)戊二醛溶液固定,再加入20μL DAPI染液(質量濃度為20 μg/mL),避光染色5min后,在小于7 093Pa負壓下過濾,濾干后取濾膜制片,用Leica DM 4500B型熒光顯微鏡進行熒光顯微計數.按下式計算水樣中的細菌密度
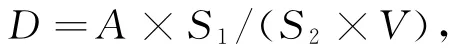
式中,D為細菌密度(mL-1);A 為10個視野的細菌平均數(cell);S1為視野面積(cm2);S2為濾膜的有效過濾面積(cm2);V 為過濾水樣體積(mL).
1.4 數據分析
測量數據取3次重復的平均值,數據用Origin 8.1和SPSS 16.0軟件進行分析,處理組間差異的顯著性采用最小顯著差異(least significant difference,LSD)檢驗法進行檢驗.
2 結果與分析
2.1 BSA的降解
不抑菌實驗中,每次添加蛋白質后,植物組的蛋白質濃度下降最快(圖2).細菌組的變化趨勢與對照組相似,并且其水體中蛋白質濃度都是隨著添加次數的增多而逐漸積累升高.36h后,植物組、細菌組和對照組蛋白的平均降解率分別是97%、41%和26%,細菌組的蛋白降解率低于植物組.利用差減法扣除非生物因素作用(26%),海馬齒與根際微生物聯合作用下的降解率(71%)是根際微生物作用(15%)的4.73倍.表明單獨的根際微生物可以降解蛋白質,但是其降解率不高,要實現蛋白質的高效降解必須以海馬齒根系為依托.
在抑菌實驗中,植物組、細菌組和對照組的變化趨勢一致,都是隨著添加次數的增多,蛋白質濃度逐漸升高(圖3).植物組并沒有表現出明顯高于其他組的降解效率.表明單獨的海馬齒根系無法實現蛋白質的高效降解.蛋白質的濃度在每次添加后期有所升高,呈現“V”型的變化特點,推測原因可能是部分吸附在瓶壁上的蛋白質由于室溫變化等原因發生了解吸,導致后期濃度有所上升.
以上結果說明,單獨的根際微生物或海馬齒根系都無法實現蛋白質的高效降解.海馬齒根系與根際微生物聯合在蛋白質的根際降解中發揮著極為重要的作用.
2.2 BSA降解過程中細菌密度的變化
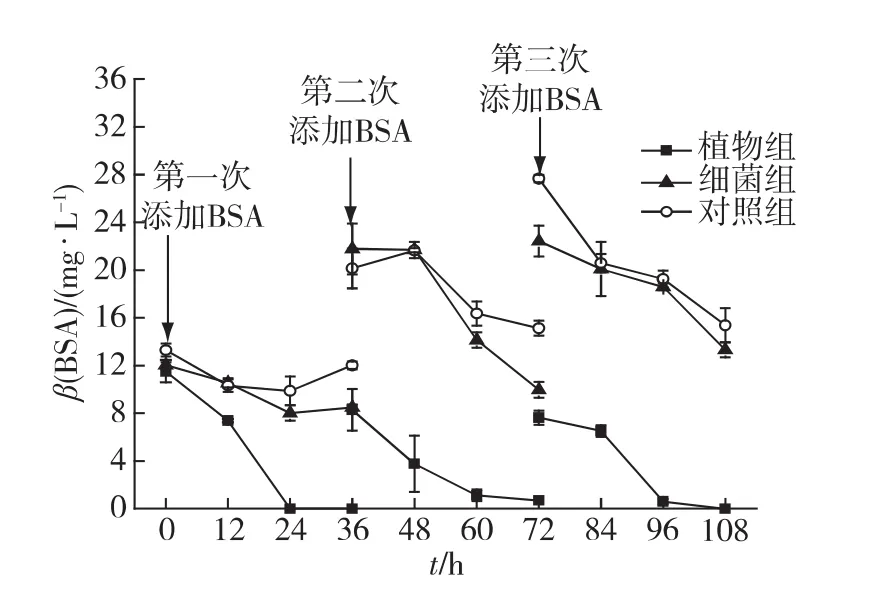
圖2 不抑菌實驗中BSA質量濃度隨時間的變化Fig.2 Changes of BSA concentration in the experiment groups without antibiotic addition
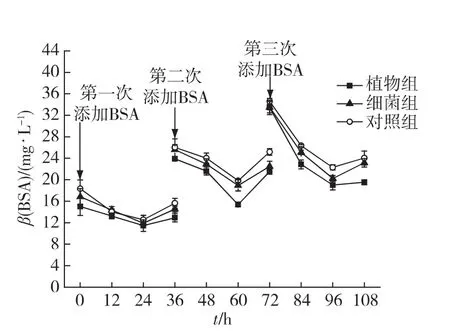
圖3 抑菌實驗中BSA質量濃度隨時間的變化Fig.3 Changes of BSA concentration in the experiment groups with antibiotic addition
在不抑菌實驗中,在0到36h內,植物組蛋白質的高效降解為細菌生長提供營養,其細菌密度的增長速度明顯快于細菌組.植物組和細菌組的細菌密度基本都是呈現初期快速增長、中期下降和后期基本穩定的單峰型變化趨勢(圖4).而整個實驗過程中,對照組沒有檢測到細菌的存在,可以有效反映非生物因素的作用.
在抑菌實驗中,對照組沒有檢測到細菌的存在.植物組和細菌組的細菌密度基本都呈下降趨勢(圖5),細菌密度分別從開始時的3.0×105和2.8×105mL-1逐漸下降到結束時的0.7×105和0.5×105mL-1,其數量與不抑菌實驗相比,可以忽略不計.表明實驗所采用的抗生素可以有效抑制細菌數量的增長,抑菌實驗中的植物組可以較好地反映單純海馬齒根系在蛋白質降解中的作用.
2.3 BSA降解過程中亮氨酸氨基肽酶活性的變化
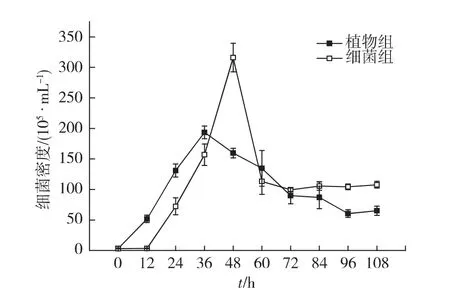
圖4 不抑菌實驗中細菌密度隨時間的變化Fig.4 Changes of bacterial density in the experiment groups without antibiotic addition
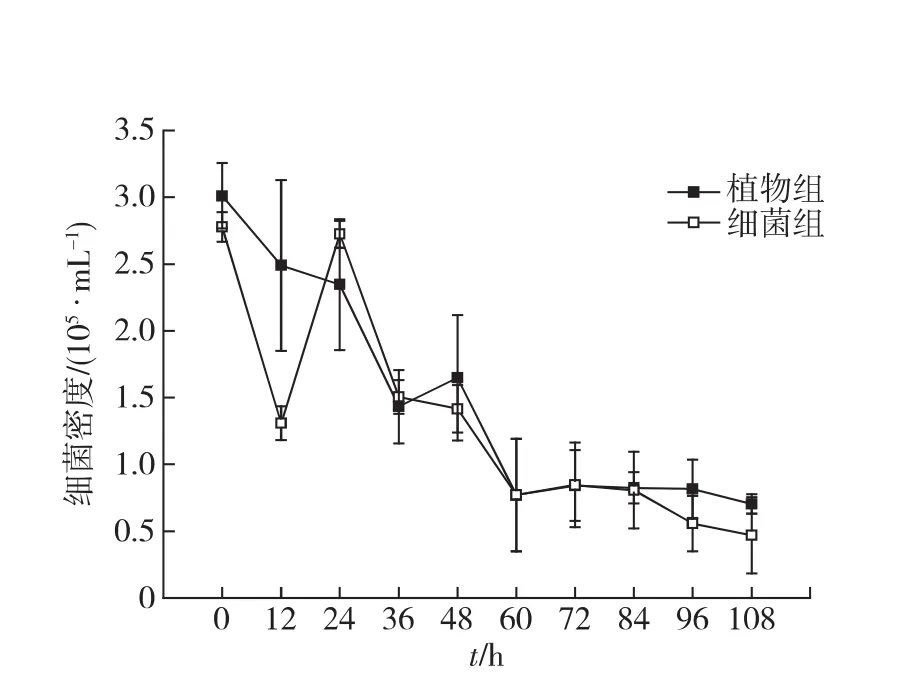
圖5 抑菌實驗中細菌密度隨時間的變化Fig.5 Changes of bacterial density in the experiment groups with antibiotic addition
在抑菌實驗中,雖然細菌組和植物組的細菌密度在0~12h時可達105mL-1,但是對照組、細菌組和植物組都沒有檢測到亮氨酸氨基肽酶活性,表明抑菌實驗的抗生素抑菌效果良好,細菌活性受到了全面抑制,而單純的海馬齒根系不分泌亮氨酸氨基肽酶.由此可以推斷,在不抑菌實驗中檢測到較高活性的亮氨酸氨基肽酶完全是由根際微生物所分泌.
不抑菌實驗中,對照組沒有檢測到亮氨酸氨基肽酶活性,只有植物組和細菌組有明顯的酶活(圖6).這2組的酶活性變化趨勢較為相似,而植物組的酶活性始終高于細菌組.實驗開始的24h內,植物組的酶活性急劇上升,24h時達到最高值0.75μmol/(L·h),此后酶活性呈下降趨勢,到實驗結束時,酶活性下降到0.22μmol/(L·h).細菌組的酶活性最高值出現在60 h,達到0.43μmol/(L·h),在實驗結束時,酶活性檢測不到.從整體上看,植物組和細菌組的酶活性也是基本呈現先升高后降低的趨勢.
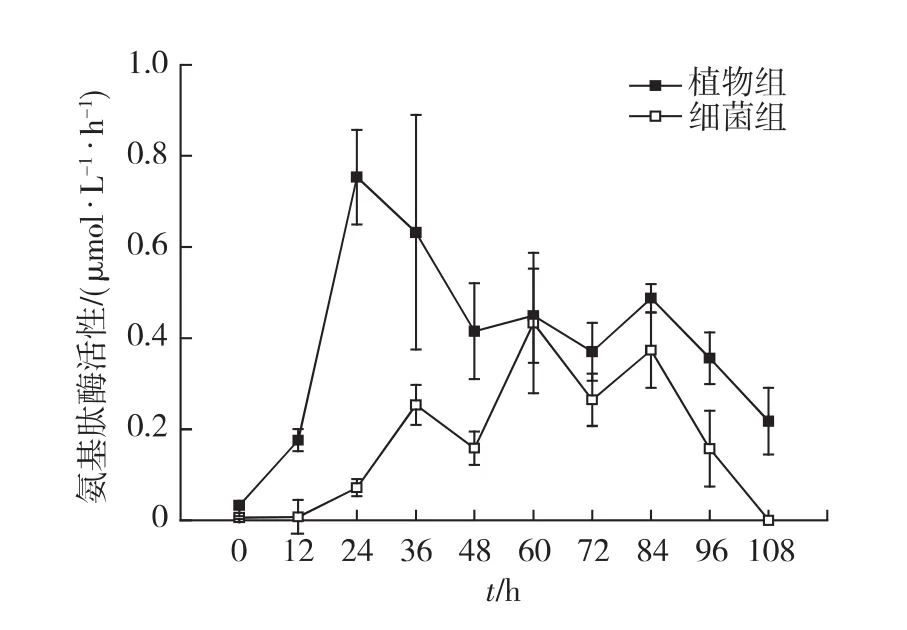
圖6 不抑菌實驗中亮氨酸氨基肽酶活性隨時間的變化Fig.6 Changes of leucine amino peptidase activity in the experiment groups without antibiotic addition
與細菌組相反,植物組的酶活性最高值出現時間(24h)早于細菌密度最高值(36h).說明在海馬齒根系和根際微生物聯合作用下,植物組蛋白質的高效降解過程伴隨著酶活性的升高和細菌密度的增加,且酶活性的升高先于細菌數量的增長.
3 討 論
抑菌實驗中的植物組沒有檢測出亮氨酸氨基肽酶活性,表明該蛋白酶完全由根際微生物分泌,海馬齒根系不分泌該種胞外酶.大部分水環境中的研究表明亮氨酸氨基肽酶活性主要是由異養細菌產生的[19-21].而也有研究者認為蛋白酶可以由植物根系釋放,如Gramms等[22]研究發現,豆科、禾本科和茄科的一些植物種類能夠向根系區域大量釋放氧化酶和水解酶,這些酶包括蛋白酶、過氧化物酶、漆酶、單酚氧化酶、脂肪酶以及酯酶等.另有研究表明,亮氨酸氨基肽酶是植物體內廣泛存在的一種金屬蛋白酶[23],可以有效切割N端的亮氨酸、精氨酸和蛋氨酸殘基[24],可能對蛋白半衰期具有重要的調節作用,關系細胞的生長和凋亡[25].
從整體上看,不抑菌實驗中的植物組和細菌組的酶活基本呈現先升高后降低的趨勢,這一趨勢基本上符合目前比較流行的胞外酶活性調控機制——“抑制-誘導”假說[26-27].該學說認為當海水中存在大量初級代謝產物以及光合作用產物時,這些易于被吸收利用的低分子質量產物(UDOM)容易進入細胞內,以維持細菌的正常生長及新陳代謝.同時這些UDOM(如氨基酸、單糖和有機酸等)限制了胞外酶的活性(產物抑制)并且抑制了酶的合成(代謝抑制).而當海水中的UDOM降低到臨界值,藻類細胞通過裂解釋放出大量的高分子化合物(如蛋白質、多糖、酯類或核酸等)時,胞外酶的抑制被解除,酶活性顯著升高.
在不抑菌實驗中,植物組的酶活性最高值和細菌密度最大值的出現時間均早于細菌組,并且植物組的酶活始終高于細菌組.表明海馬齒根系可以促進根際細菌的生長和亮氨酸氨基肽酶活性的升高.也有其他研究證實植物根系可以增強根際胞外酶活性[28-30],如棉花、小麥、水芹、西紅柿和水葫蘆等植物能向根際環境中提供氨基酸、糖類和維生素等養料,改善根際微生態環境,間接促進胞外酶活性的升高.
由于亮氨酸氨基肽酶完全由根際細菌分泌,根際細菌在蛋白質的根際降解過程中起著主要的作用,但是不抑菌實驗的細菌組的降解率不高且明顯低于植物組,說明根際細菌的高效降解必須以海馬齒根系為依托.海馬齒根系和根際細菌的協同作用使得蛋白質的降解效率明顯提高,其原因一方面可能是植物可以通過根系將氧氣釋放進入水體,保證微生物有充足的氧氣對蛋白質進行降解.另一方面是根系分泌物可以改變根系周圍環境的pH值[31]、可能為微生物提供共代謝的底物[32-33].許多實驗已經證實,向土壤中添加根系分泌物同樣能刺激微生物的活性.Abdel-Nasser[34]證實向土壤中添加人工合成的大麥根系分泌物可以刺激土壤中放線菌、真菌和細菌的數量.向土壤中添加燕麥的根系分泌物可以促進菲的降解[35],而向土壤中添加玉米的根系分泌物可以顯著提高14C-芘的礦化率[36].由此可以看出根系分泌物在有機物根際降解中的重要作用.
4 結 論
1)本研究的不抑菌實驗結果表明海馬齒的根際微生物具有降解蛋白質的能力,其降解效率在有植物根系存在時明顯高于無根系時的降解效率,說明植物根系對于根際微生物降解蛋白質有重要促進作用.
2)抑菌實驗表明氨芐霉素-頭孢噻肟-萬古霉素的抗生素組合能有效抑制海馬齒根際微生物的生長.在有效抑菌的情況下,整個實驗過程中,植物組與細菌組都未檢測到亮氨酸氨基肽酶的活性,說明能高效降解可溶性蛋白質的亮氨酸氨基肽酶完全來源于根際微生物,而非由海馬齒根系所分泌.
3)以上表明,在可溶性蛋白質的根際降解中,根際細菌是降解的執行者,但是根際細菌的高效降解是以植物根系為依托的,植物根系和根際細菌構成互惠共生的關系.這為更好利用海馬齒生態浮床修復富含有機氮的海水環境提供了理論依據.
[1]鄭天凌,王斐,徐美珠,等.臺灣海峽水域的β-葡萄糖苷酶活性 [J].應用與環境生物學報,2001,7(2):175-182.
[2]Hoppe H.Use of fluorogenic model substrates for extracellular enzyme activity(EEA)measurement of bacteria[M]∥ Handbook of methods in aquatic microbial ecology.Boca Raton:Lewis Publishers,1993:423-431.
[3]王斐,鄭天凌,洪華生.細菌胞外酶的生態作用 [J].海洋科學,1999,3(3):33-36.
[4]Sinsabaugh R,Antibus R,Linkins A,et al.Wood decomposition:nitrogen and phosphorus dynamics in relation to extracellular enzyme activity [J].Ecology,1993,74(5):1586-1593.
[5]Wright R R,Hobbie J E.Use of glucose and acetate by bacteria and algae in aquatic ecosystem [J].Ecology,1966,47:447-464.
[6]唐昌林.中國植物志[M].北京:科學出版社,1996:30-32.
[7]張志英,黃凌風,姜丹,等.浮床種植海馬齒對富營養化海水氮、磷移除能力的初步研究 [C]∥中國環境科學學會2009年學術年會論文集.北京:北京航空航天大學出版社,2009:50-55.
[8]陸建良,梁月榮,張凌云.考馬斯亮藍法在茶湯可溶性蛋白含量分析中的應用和改良 [J].茶葉,2002,28(2):89-93.
[9]曲春香,沈頌東,王雪峰,等.用考馬斯亮藍測定植物粗提液中可溶性蛋白質含量方法的研究 [J].蘇州大學學報:自然科學版,2006,22(2):82-85.
[10]袁燕,段玲燕,郭麗紅,等.BCA法測定新的植物毒素——蒜頭果蛋白質量濃度 [J].昆明學院學報,2009,31(3):60-61.
[11]Weinberger F,Hoppe H,Friedlander M.Bacterial induction and inhibition of a fast mecrotic response in Gracilaria conferta (Rhodophyta)[J].Journal of Applied Phycology,1997,9(3):277-285.
[12]de Souza M,Chu D,Zhao M,et al.Rhizosphere bacteria enhance selenium accumulation and volatilization by Indian mustard [J].Plant Physiology,1999,119(2):565-574.
[13]宋福行,焦念志.細菌胞外酶活性的調控因素 [J].海洋科學,2001,25(9):16-18.
[14]Martinez J.Variability in ecohydroytic enzyme activities of pelagic marine bacteria and its significance for substrate processing in the sea[J].Aquatic Microbial Ecology,1996,10(4):223-230.
[15]Hoppe H.Significance of exoenzymatic activities in the ecology of brackish water:measurements by means of methylumbelliferyl-substrates[J].Marine Ecology Pro-gress Series,1983,11(1):299-308.
[16]Ecology A.Coupling between bacterioplankton species composition,population dynamics,and organic matter degradation[J].Aquatic Microbial Ecology,1999,17(1):13-26.
[17]Hoppe H,Kim S,Gocke K.Microbial decomposition in aquatic environments:combined process of extracellular enzyme activity and substrate uptake[J].Applied and Environmental Microbiology,1988,54(3):784-790.
[18]鄭天凌,王斐,徐美珠,等.臺灣海峽海域細菌產量、生物量及其在微食物環中的作用[J].海洋與湖沼,2002,33(4):415-423
[19]Rosso A L,Azam F.Proteolytic activity in coastal oceanic waters:depth distribution and relationship to bacterial populations[J].Marine Ecology Progress Series,1987,41:231-240.
[20]Jacobsen T R,Rai H.Determination of aminopeptidase activity in lakewater by a short term kinetic assay and its application in two lakes of differing eutrophication[J].Archiv Fur Hydrobiologie,1988,113:359-370.
[21]Fabiano M,Danovaro R.Enzymatic activity,bacterial distribution,and organic matter composition in sediments of the Ross Sea (Antarctica)[J].Applied and Environmental Microbiology,1998,64:3838-3848.
[22]Gramss G,Voigt K D,Kirsche B.Oxidoreductase enzymes liberated by plant roots and their effects on soil humic material [J].Chemosphere,1999,38 (7):1481-1494.
[23]Bartling D,Weiler E W.Leucine aminopeptidase from Arabidopsis-thaliana-molecular evidence for a phylogenetically conserved enzyme of protein-turnover in higher-plants [J].European Journal of Biochemistry,1992,205(1):425.
[24]Gu Y Q,Walling L L.Specificity of the wound-induced leucine aminopeptidase (LAP-A)of tomato-activity on dipeptide and tripeptide substrates[J].European Journal of Biochemistry,2000,267(4):1178.
[25]Varshavsky A.The N-end rule:functions,mysteries,uses[J].Proceedings of The National Academy of Sciences of the United States of America,1996,93(22):12142.
[26]Chróst R.Characterization and significance ofα-glucosidase activity in lake water[J].Limnology and Oceanography,1989,34(4):660-672.
[27]Chróst R.Microbial enzyme in aquatic environments[M]∥Aquatic microbial ecology,biochemical and molecular approaches.New York:Springer,1990:47-79.
[28]李傳榮,許景偉,宋海燕,等.黃河三角洲灘地不同造林模式的土壤酶活性 [J].植物生態學報,2006,30(5):802-809.
[29]Garcia C,Roldan A,Hernandez T.Ability of different plant species to promote microbiological processes in semiarid soil[J].Geoderma,2005,124(1/2):193-202.
[30]Siegel B.Plant peroxidases:an organismic perspective[J].Plant Growth Regulation,1993,12(3):303-312.
[31]Dakora F,Phillips D.Root exudates as mediators of mineral acquisition in low-nutrient environments[J].Plant and Soil,2002,245(1):35-47.
[32]Shann J.The role of plants and plant/microbial systems in the reduction of exposure[J].Environmental Health Perspectives,1995,103(Sup.5):13-15.
[33]Walton B,Anderson T.Microbial degradation of trichloroethylene in the rhizosphere:potential application to biological remediation of waste sites [J].Applied and Environmental Microbiology,1990,56(4):1012-1016.
[34]Abdel-Nasser M,Moawad Hassan.Changes in numbers of microorganisms during decomposition of root exudates in soil[J].Zentralbl Bakteriol Parasitenkd Infektionskr Hyg,1975,130(8):738-744.
[35]Miya R,Firestone M.Enhanced phenanthrene biodegradation in soil by slender oat root exudates and root debris[J].Journal of Environmental Quality,2001,30(6):1911-1918.
[36]Yoshitomi K J,Shann J R.Corn(Zea mays L.)root exudates and their impact on14C-pyrene mineralization[J].Soil Biology and Biochemistry,2001,33(12/13):1769-1776.

