伶鼬榧螺(Olivamustelina)的分子鑒定及其形態變異
李海濤,何薇,周鵬,陳凱彪,董燕紅
(1.國家海洋局 南海環境監測中心,廣東 廣州 510300)
伶鼬榧螺(Olivamustelina)的分子鑒定及其形態變異
李海濤1,何薇1,周鵬1,陳凱彪1,董燕紅1
(1.國家海洋局 南海環境監測中心,廣東 廣州 510300)
根據榧螺螺旋部的差異可將其分為兩種不同的形態類型,類型Ⅰ(Morphotype Ⅰ)的個體螺旋部陷入體螺層中,其殼頂與前部數螺層愈合;類型Ⅱ(Morphotype Ⅱ)的個體螺旋部高出殼頂,縫合線明顯,此類型可明確鑒定為伶鼬榧螺(Olivamustelina)。本研究通過線粒體COⅠ和16S rRNA基因序列的分析對兩種形態類型的榧螺進行了分子鑒定。結果表明:兩種形態類型的榧螺含有共享的單倍型;不同形態類型間的遺傳距離和同一形態類型內的遺傳距離重疊;兩者的單系性在系統發育分析中沒有得到顯現,而是共同構成一個單系。因此,兩種形態類型的個體均為伶鼬榧螺。類型Ⅰ可能是伶鼬榧螺中一種少見的有別于典型個體的形態變異類型。
伶鼬榧螺;COⅠ基因;16S rRNA基因;形態變異
1 引言
榧螺屬(Oliva)隸屬于新腹足目(Neogastropoda)榧螺科(Olividae),是一類具有美麗外殼的貝類。榧螺屬的貝殼具有一定的藥用價值,有清燥潤肺、平膽潛陽之功效,主治高血壓、青盲內障、骨蒸勞熱等[1]。近年來,由于野生資源衰竭,部分榧螺種類已處于瀕危狀態[2]。
伶鼬榧螺(Olivamustelina)是南海沿岸水域較為常見的種類,也是榧螺屬最主要的入藥種類。典型的伶鼬榧螺其螺旋部高出殼頂,且縫合線明顯。但在自然界中也存在另外一類個體,其貝殼花紋與伶鼬榧螺相似,但螺旋部陷入體螺層中,殼頂與前部螺層愈合。這兩種形態類型的差別屬于種內還是種間變異,尚未得到其他研究的證實。
由于缺乏肋紋等特征,貝殼的顏色和花紋目前仍是榧螺屬分類最重要的依據。但這類特征也常發生變異,Bert[3]描述了一類殼色發生變異的伶鼬榧螺個體,其淺棕色的貝殼上伴有縱向的青灰色斑紋,明顯有別于正常個體的“之”字形花紋。形態可塑性為分類學家帶來極大困難,使其難以區分種內和種間變異的界限。
分子生物學的發展提供了一種新的物種鑒定手段。線粒體細胞色素c氧化酶亞基Ⅰ(cytochromecoxidase subunit Ⅰ,COⅠ)和16S rRNA基因已在貝類的物種鑒定中得到廣泛應用[4-7]。本研究旨在通過線粒體COⅠ和16S rRNA基因序列的分析來鑒別兩種不同形態的榧螺,以期為資源的開發利用和保護,以及其他學科的研究提供基礎資料。
2 材料和方法
2.1 實驗材料
實驗所用的榧螺標本采自珠江口及汕頭南澳島海域(表1),腹足肌以90%酒精或-80℃冰凍保存。榧螺標本的殼色和花紋相似,但依據螺旋部的差異可分為2種不同的形態類型:類型Ⅰ(Morphotype Ⅰ)個體的螺旋部陷入體螺層(圖1a,1b),螺旋部的所有螺層愈合(圖1c);類型Ⅱ(Morphotype Ⅱ)個體的螺旋部高出殼頂,縫合線明顯(圖1d,1e),螺旋部的螺層不愈合(圖1f),此類型的個體可鑒定為伶鼬榧螺。共選取兩種形態類型的31個個體用于形態學測量分析,其中24個用于分子生物學分析(表1)。

圖1 兩種不同形態類型榧螺的貝殼Fig.1 Shells of two Morphotypes of Oliva species
表1 用于DNA序列分析的榧螺標本信息
Tab.1 Specimen information ofOlivaspecimens used for DNA sequence analyses
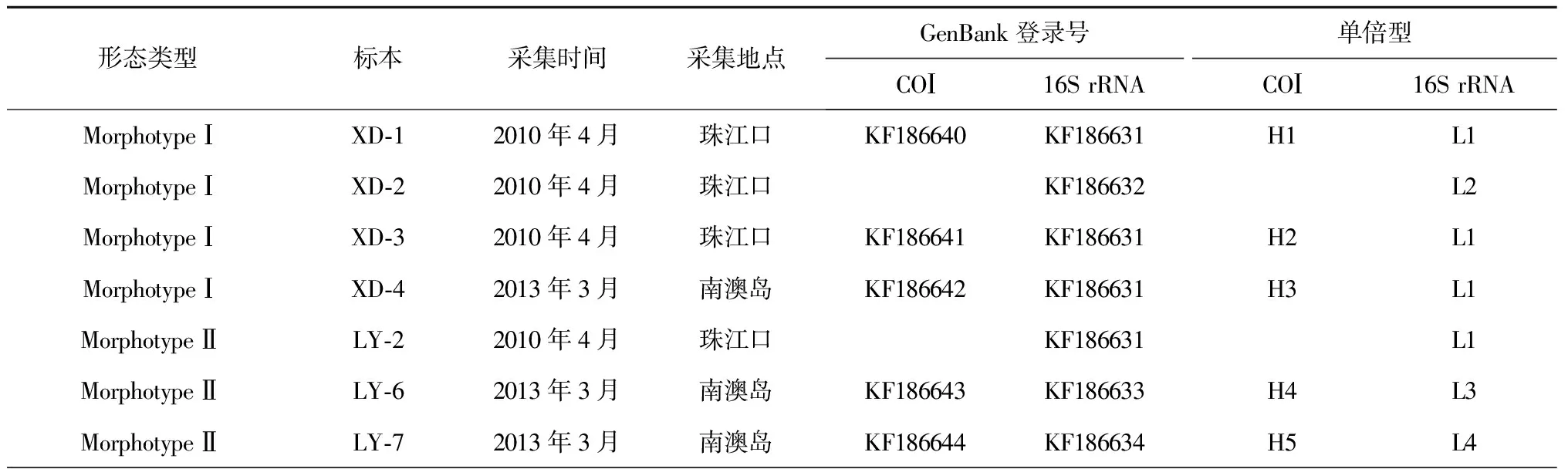
形態類型標本采集時間采集地點GenBank登錄號單倍型COⅠ16SrRNACOⅠ16SrRNAMorphotypeⅠXD?12010年4月珠江口KF186640KF186631H1L1MorphotypeⅠXD?22010年4月珠江口KF186632L2MorphotypeⅠXD?32010年4月珠江口KF186641KF186631H2L1MorphotypeⅠXD?42013年3月南澳島KF186642KF186631H3L1MorphotypeⅡLY?22010年4月珠江口KF186631L1MorphotypeⅡLY?62013年3月南澳島KF186643KF186633H4L3MorphotypeⅡLY?72013年3月南澳島KF186644KF186634H5L4
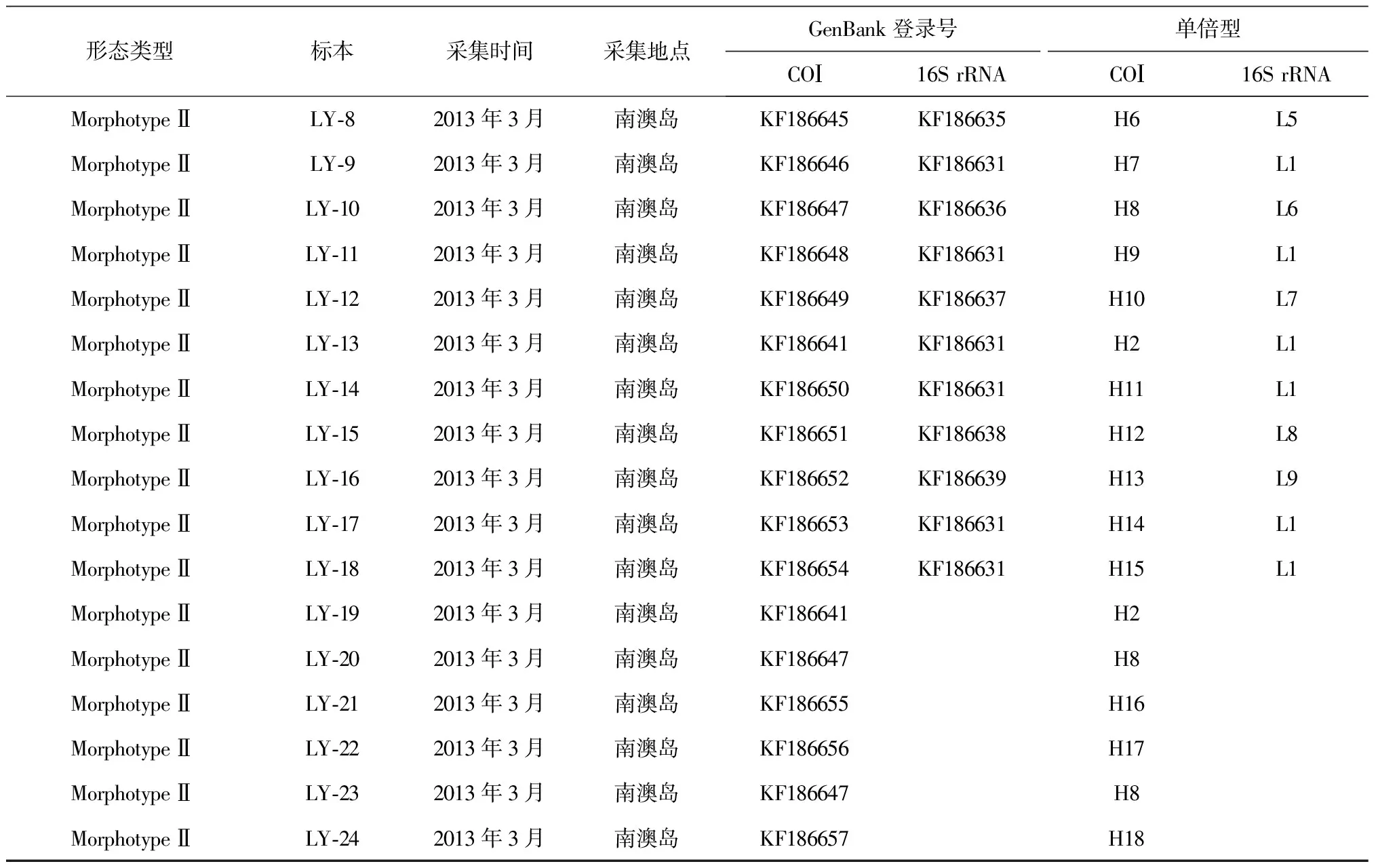
續表1
2.2 貝殼形態數據的測量和分析
用游標卡尺(精度0.2 mm)測量兩種形態類型榧螺標本的殼高(SL)和殼寬(SW),比較兩種形態類型的SL及殼高與殼寬之比(SL/SW)的差異。
2.3 模板DNA制備、PCR擴增和序列分析
采用堿裂解法制備模板DNA,具體步驟為:剪取小塊腹足肌,去離子水清洗兩次,吸干水分后置于180 μL 0.05 mol/L NaOH溶液中,95℃消化15 min,然后加入20 μL 1 mol/L Tris-Cl(pH7.6),混勻后10 000 r/min離心10 min,上清液作為模板使用。用于擴增COⅠ基因的引物為LCO-1490(5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′)和LCO-2198 (5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′)[8];擴增16S rRNA基因的引物為16sar-L(5′-CGC CTG TTT ATC AAA AAC AT-3′)和16sbr-H(5′-CCG GTC TGA ACT CAG ATC ACG T-3′)[9]。PCR反應體系總體積為50 μL,其中:2×buffer 25 μL,dNTP 400 μmol/L,引物各0.3 μmol/L,KOD FX高保真PCR酶(TOYOBO公司)1.0 U,模板DNA1.2 μL,加去離子水補足至50 μL。PCR反應條件為:94℃預變性2 min;98℃變性15 s,52℃退火30 s,68℃延伸45 s,共35個循環;最后68℃延伸10 min。PCR產物經1%瓊脂糖凝膠電泳檢測后回收純化,然后進行雙向測序,測序引物與擴增引物相同。
將所測的序列與GenBank和國際生命條形碼數據庫BOLD Systems中查得的其他榧螺屬序列一起用DAMBE(Version 5.2.65)軟件進行多序列比對。使用MEGA 5.10軟件計算變異位點、堿基組成和基于Kimura 雙參數模型(Kimura 2-parameter,K2P)下各個類群內部及類群之間的遺傳距離,并利用該軟件中的鄰接法(Neighbor-joining,NJ)和非加權配對算術平均法(Unweighted Pair Group Method with Arithmetic mean,UPGMA)構建所有單倍型系統進化樹,可靠性經過1 000次自展(Bootstrap)檢驗。采用DNASP 5.10軟件計算單倍型多樣性指數(Hd)和核苷酸多樣性指數(Pi)。
3 結果
3.1 形態測量數據的分析
從貝殼的SL及SL/SW比值來看,兩種形態類型的榧螺是大范圍重疊的,通過SL/SW比值無法將兩者明確區分(見表2)。

表2 兩種不同形態榧螺貝殼的形態測量數據
3.2 線粒體基因的序列分析
共成功獲得22條COⅠ基因序列,各序列長度均為658 bp(不含引物區),無插入/缺失位點,A、T、G、C的平均含量分別為23.4%、39.1%、20.5%和17.0%,A+T的含量明顯高于G+C的含量。22條COⅠ基因序列定義了18種單倍型(H1~H18,GenBank登錄號KF186640~KF186657)。其中,1個Morphotype Ⅰ個體(XD-3)和2個Morphotype Ⅱ個體(LY-13和LY-19)共享單倍型H2。
在COI基因序列上共檢測到52個變異位點(22個為簡約信息位點),占核苷酸總數的7.9%,其中46個發生在密碼子第三位堿基,6個發生在密碼子第一位堿基。所有核苷酸的變異未導致氨基酸序列的變化。MorphotypeⅠ個體間的遺傳距離為0.019~0.025,Morphotype Ⅱ個體間的遺傳距離為0~0.028,兩種形態類型之間的遺傳距離為0~0.026,同一形態類型內及兩種形態類型間的遺傳距離完全重疊,無明顯的界限。上述兩種形態的個體與其他榧螺,如O.spicata(GenBank登錄號FM999165)、O.sayana(GenBank登錄號U86333)、O.amalda(BOLD序列號NEOGA900-10、NEOGA901-10和NEOGA902-10)、Olivasp.(BOLD序列號NEOGA879-10)、Olivasp.1(BOLD序列號NEOGA877-10)間的遺傳距離較大,在0.129~0.241之間。
共成功獲得18條16S rRNA基因序列,其長度為508~510 bp,存在2個位點的插入/缺失。A、T、G、C的平均含量分別為34.5%、29.5%、19.9%和16.1%,A+T的含量顯著高于G+C的含量。18條16S rRNA基因序列共定義了9種單倍型(L1~L9,GenBank登錄號KF186631~KF186639)。其中,2個Morphotype Ⅰ個體和8個Morphotype Ⅱ個體共享單倍型L1,這一單倍型序列與來自日本Ise bay的一條伶鼬榧螺序列[10](GenBank登錄號AB121038)完全相同。
在16S rRNA基因序列上共檢測到10個變異位點,占核苷酸總數的2.0%,其中4個為簡約信息位點。兩種形態類型內的遺傳距離分別為0~0.008和0~0.012,兩種形態類型間的遺傳距離為0~0.014,形態類型內和形態類型間的遺傳距離完全重疊。而它們與O.spicata(FM999114)間的遺傳距離則高達0.104~0.112。
基于COⅠ基因序列計算的單倍型多樣性指數(Hd)和核苷酸多樣性指數(Pi)分別為Hd=(0.974±0.024)和Pi=(0.015 5±0.001 1);基于16S rRNA基因序列計算的結果分別為Hd=(0.706±0.120)和Pi=(0.003 6±0.001 0)。單倍型多樣性指數高于核苷酸多樣性指數,且COⅠ基因所反映的單倍型多樣性指數和核苷酸多樣性指數均高于16S rRNA基因。
3.3 系統發育關系
基于COⅠ和16S rRNA 基因序列分別構建了NJ樹和UPGMA樹,兩種樹的拓撲結構基本一致。因此本文僅給出NJ樹,其自展支持率(僅大于70%的列出)標注在節點斜線左側,UPGMA樹的自展支持率標注在節點斜線右側。
基于COⅠ基因序列構建的NJ樹(見圖2)顯示,18個單倍型合在一起形成一個單系,支持率為99%,在系統樹中處于較為進化的位置。兩種形態類型榧螺的單系性在系統樹上均沒有得到顯現,相互間無法明確區分。
在基于16S rRNA 基因序列構建的NJ樹(見圖3)中,9個單倍型與GenBank中的一條伶鼬榧螺序列(GenBank登錄號AB121038)共同形成一個單系群,支持率為99%。兩種形態類型的單系性也未在系統樹上得到顯現。
4 討論
貝殼的形態受多種生物和非生物因素的影響,因此僅依據貝殼形態來進行分類學和系統學研究越來越有爭議性[11]。腹足類的個體差異、性別差異和以及發育階段的不同均可能導致貝殼形態特征的變化[7]。
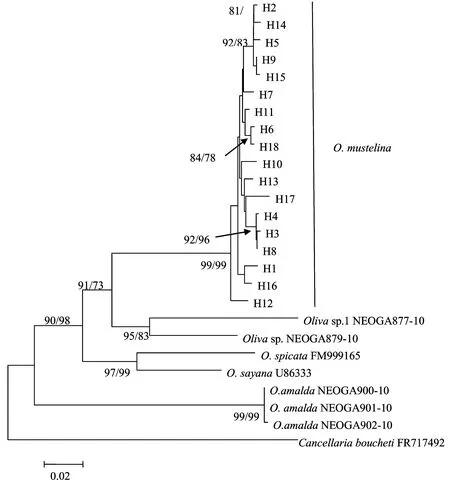
圖2 基于COⅠ基因序列通過鄰接法構建的系統發育樹Fig. 2 Molecular phylogenetic tree based on COⅠ gene sequences using Neighbor-joining method
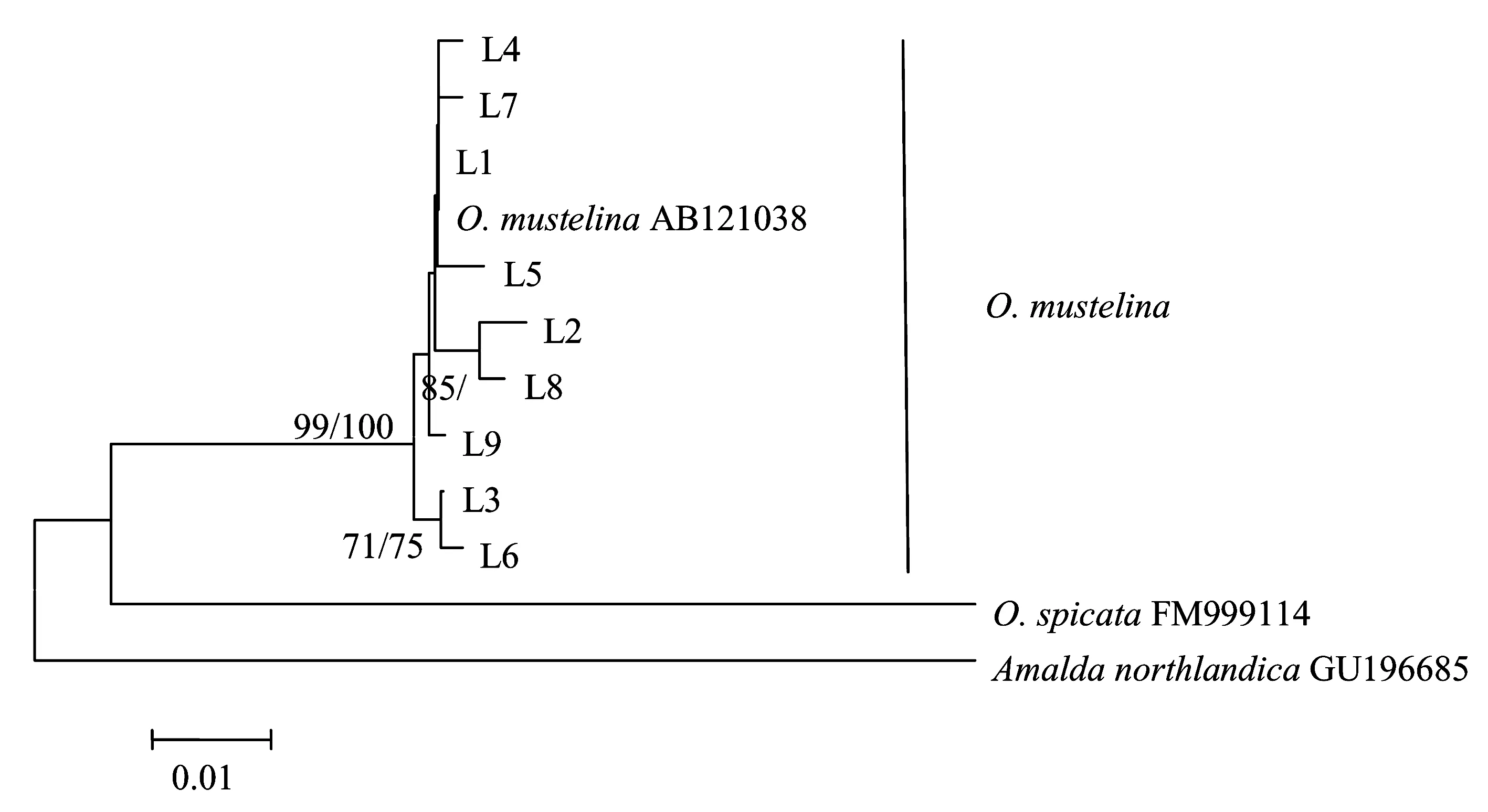
圖3 基于16S rRNA基因序列通過鄰接法構建的系統發育樹Fig.3 Molecular phylogenetic tree based on 16S rRNA gene sequences using Neighbor-joining method
基于線粒體COⅠ和16S rRNA 基因序列的分析結果表明兩種形態類型的榧螺之間沒有明顯的遺傳差異:兩者享有共同的單倍型;遺傳距離完全重疊;各自無法形成單系,而是共同形成一個單系群,且獲得很高的支持率。因此,兩種類型的個體為同一物種,即伶鼬榧螺。COⅠ基因為蛋白質編碼序列,在種內沒有發生堿基的插入/缺失,其變異也未造成氨基酸序列的改變。COⅠ基因的進化速率較16S rRNA基因更快,本文的研究結果與其他研究結論[4,12]是一致的。COⅠ基因所揭示的伶鼬榧螺單倍型多樣性指數和核苷酸多樣性指數均明顯高于16S rRNA基因。
榧螺屬的分類問題由來已久,其現生種的拉丁學名達500多個,遠遠超過實際的種類數,同種異名現象十分嚴重[13]。殼頂是否陷入體螺層是伶鼬榧螺與陷頂榧螺(O.concavospira)的形態區別之一,但伶鼬榧螺中也存在螺旋部陷入體螺層的個體,且其螺旋部的螺層常愈合在一起。因此,這些特征在用于屬下種間的鑒別時需要十分慎重,也不宜作為亞種區分的依據。Bert[3]描述的有別于典型伶鼬榧螺形態的個體除顏色和花紋不同之外,其螺旋部也明顯低矮。本研究中的Morphotype Ⅰ個體也可能是伶鼬榧螺中一種較為少見的形態變異類型,這類個體在采集標本中的數量不多。
[1] 《中國藥用動物志》協作組.中國藥用動物志[M].天津:天津科學技術出版社,1983:37.
Cooperative Group of China Medicinal Animal Fauna. China Medicinal Animal Fauna[M]. Tianjin: Tianjin Science and Technology Press,1983: 37.
[2] 傅秀梅,王長云,邵長倫,等.中國海洋藥用生物瀕危珍稀物種及其保護[J]. 中國海洋大學學報(自然科學版),2009,39(4):719-728.
Fu Xiumei,Wang Changyun,Shao Changlun,et al. Endangered and rare species of marine medicinal organisms and their protection in China[J]. Periodical of Ocean University of China(Natrual Sience),2009,39(4): 719-728.
[3] Bert C. New form ofOlivamustelina Lamarck,1811 form SW Taiwan[J]. Bulletin of Malacology R.O.C.,1984,10: 7-8.
[4] Yu Z N,Kong X Y,Zhang L S,et al. Taxonomic status of fourCrassostreaoysters from China as inferred from mitochondrial DNA sequences[J]. Journal of Shellfish Research,2003,22(1): 31-38.
[5] 張亮,黃艷艷,劉煥章.利用mtDNA 16S rRNA 序列差異鑒定江西青嵐湖的河蚌物種[J].水生生物學報,2004,28(3):294-299.
Zhang Liang,Huang Yanyan,Liu Huanzhang. The species identification of Mussels in Qinglan Lake(Jiangxi Province) based on sequence differences of mitochondrial 16S rRNA gene[J]. Acta Hydrobiologica Sinica,2004,28(3): 294-298.
[6] Duda T F,Bolin M B,Meyer C P,et al. Hidden diversity in a hyperdiverse gastropod genus: discovery of previously unidentified members of aConusspecies complex[J]. Molecular Phylogenetics and Evolution,2008,49: 867-876.
[7] 班紹君,薛東秀,張濤,等.三種殼口顏色脈紅螺(Rapanavenosa)形態學和線粒體16S rRNA與COⅠ基因片段差異比較分析[J].海洋與湖沼,2012,43(6):1209-1217.
Ban Shaojun,Xue Dongxiu,Zhang Tao,et al. Variance analysis of three color morphs ofRapanavenosabased on morphological traits and mt16S rRNA and COI partial sequences[J]. Oceanologia et Limnologia Sinica,2012,43(6): 1209-1217.
[8] Folmer O,Black M,Hoeh W,et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit 1 from diverse metazoan invertebrates[J]. Molecular Marine Biology and Biotechnology,1994,3: 294-299.
[9] Simon C,Frati F,Beckenbach A,et al. Evolution,weighting and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers[J]. Annals Entomological Society of America,1994,87: 651-701.
[10] Hayashi S. The molecular phylogeny of the Buccinidae (Caenogastropoda: Neogastropoda) as inferred from the complete mitochondrial 16S rRNA gene sequences of selected representatives[J]. Molluscan Research,2005,25(2): 85-98.
[11] Wilke T,Falniowski A. The genusAdriohydrobia(Hydrobiidae: Gastropoda): polytypic species or polymorphic populations?[J]. Journal of Zoological Systematics and Evolutionary Research,2001,39: 227-234.
[12] 蘇天鳳,黃建華,吳進鋒,等.2種東風螺線粒體基因序列多態性研究[J].中國水產科學,2007,14(3):369-376.
Su Tianfeng,Huang Jianhua,Wu Jinfeng,et al. Genetic polymorphism of mitochondrial DNA sequences inBabyloniaareolataandBabyloniaformosae[J]. Journal of Fishery Sciences of China,2007,14(3): 369-376.
[13] Tursch B,Germain L. Studies on Olividae. Ⅰ:A morphometric approach to theOlivaproblem[J]. Indo-Malayan Zoology,1985,2: 331-352.
Molecular identification ofOlivamustelinaand its morphological variation
Li Haitao1,He Wei1,Zhou Peng1,Chen Kaibiao1,Dong Yanhong1
(1.SouthChinaSeaEnvironmentalMonitoringCenter,StateOceanicAdministration,Guangzhou510300,China)
Based on the morphologic difference of spire,Olivasnails with similar shell colors and patterns can be divided into two morphotypes. Morphotype Ⅰ is characterized by the sunk spire in its concavity below the shoulder of the body whorl and the whorls of spire fused together. Morphotype Ⅱ shows the identical conchological characteristics of typical specimens ofO.mustelina. The mitochondrial cytochrome c oxidase subunit Ⅰ (COⅠ) and 16S rRNA gene segments of these two morphotypes were sequenced. Genetic analyses clearly showed many common haplotypes and lack of significant genetic differentiation between the two morphotypes. In addition,further phylogenetic analyses showed that all haplotypes were grouped in a monophyletic clade. Thus,the two morphotypes are not genetically diagnosable,and therefore should be assigned to speciesO.mustelina. Morphotype Ⅰ may be a new form ofO.mustelinathat differ from the typical specimens.
Olivamustelina; COⅠ gene; 16S rRNA gene; morphological variation
10.3969/j.issn.0253-4193.2015.04.011
2014-06-04;
2015-01-03。
海洋公益性行業科研專項(201305010,201105003);南海區海洋環境質量綜合評價方法(DOMEP(MEA)-01-03)。
李海濤(1981—),男,湖北省沙洋縣人,主要從事貝類分類學研究。E-mail:haitaoli1981@126.com
Q959.212
A
0253-4193(2015)04-0117-07
李海濤,何薇,周鵬,等. 伶鼬榧螺(Olivamustelina)的分子鑒定及其形態變異[J].海洋學報,2015,37(4):117—123,
Li Haitao,He Wei,Zhou Peng,et al. Molecular identification ofOlivamustelinaand its morphological variation[J]. Haiyang Xuebao,2015,37(4):117—123,doi: 10.3969/j.issn.0253-4193.2015.04.011

