轉導蘆葦總DNA耐鹽變異水稻SSH文庫的構建及部分基因特異表達
肇瑩 肖軍 楊鎮等


摘要:通過花粉管通道轉導蘆葦總DNA到水稻遼星1號中,以獲得的2個耐鹽變異水稻品系H1、H2為材料,對6張葉的水稻幼苗進行0.3%濃度的NaCl溶液誘導處理24 h。提取經NaCl溶液誘導后的葉片mRNA,經反轉錄成 cDNA,經過酶解后加接頭和2次雜交,構建差異表達的抑制消減(SSH)cDNA文庫。以NaCl溶液誘導后的耐鹽變異水稻H1、H2的cDNA為檢測子(tester),以水稻遼星1號cDNA為驅動子(driver)。所構建的文庫容量分別達到了240、281個,表明所構建的文庫質量良好。對文庫中克隆進行測序分析,在NCBI水稻數據庫中進行BLAST比對,獲得了15個沒有同源性的EST序列,其中9個EST序列功能與植物的抗鹽性有關,通過RT-PCR技術進一步驗證了蘆葦總DNA轉導到水稻中,使水稻獲得了抗鹽性。
關鍵詞:水稻;蘆葦;抑制消減雜交;cDNA文庫;基因表達
中圖分類號: S511.01 文獻標志碼: A
文章編號:1002-1302(2015)03-0013-04
我國沿海鹽漬稻區常利用鹽水灌溉,過量施用肥料導致土壤鹽分積累以及水稻產量下降[1]。因此,選育耐鹽的水稻新品種、充分開發利用鹽堿地對于實現農業增收、保障糧食安全具有重要的意義[2-3]。通過生物技術手段可以加速耐鹽水稻品種選育進程,但是耐鹽目的基因的轉入由于存在基因克隆、質粒構建等一系列問題而受到限制。筆者將具有耐鹽性狀的蘆葦總DNA通過花粉管通道法轉導至水稻中,經過幾代的人工海水篩選獲得2個穩定耐鹽的水稻品系。抑制消減雜交(suppression subtra ctive hybridization,SSH)技術是一種篩選、分離差異表達基因的方法[4-5],目前已在小麥[6]、水稻[7-9]、馬鈴薯[10]、紫稈檉柳[11-12]、楊樹[13]等多種植物中得到廣泛應用。本研究以2個耐鹽變異水稻為材料,構建水稻抑制消減雜交文庫,對陽性克隆進行測序,并結合生物信息學對測序結果進行分析,旨在為通過花粉管通道轉導蘆葦總DNA獲得耐鹽變異水稻提供可靠的分子生物學證據。
1 材料與方法
1.1 材料
試驗材料為遼星1號水稻轉導蘆葦總DNA獲得的2個耐鹽變異品系后代H1、H2,用濃度為0.3%的NaCl溶液對其誘導處理24 h。將處理完的水稻倒數第3張葉片經液氮速凍后保存于-80 ℃超低溫冰箱備用。植物總RNA提取試劑盒、Taq DNA 聚合酶、超純dNTP(10 mmol/L)均購自天根生化科技(北京)有限公司;反轉錄酶SuperscriptⅢ購自 Invitrogen 生物技術有限公司;pGEM-T easy克隆試劑盒購自Promega公司;LA Taq DNA 聚合酶購自TaKaRa公司;瓊脂糖購自西班牙;瓊脂糖凝膠回收試劑盒購自Xygen公司;其他生化試劑購自Sigma公司;引物由Invotrigen公司合成。
1.2 方法
1.2.1 葉片總RNA的提取與純化
分別取濃度為0.3%的NaCl溶液誘導處理24 h的耐鹽變異水稻H1、H2和對照遼星1號葉片100 mg,加入液氮充分研磨成粉末,參照天根生化科技(北京)有限公司植物總RNA提取試劑盒操作說明提取純化總RNA。對純化后的RNA進行1.2%瓊脂糖凝膠電泳,觀察提取物質量。分光光度計檢測RNA樣品濃度。RNA樣品濃度計算公式如下:
RNA原液濃度(ng/μL)=D260 nm×稀釋倍數×40。根據D260 nm/D280 nm比值估測RNA質量。
1.2.2 SSH cDNA文庫的構建
采用Invitrogen公司SuperScript Ⅲ Transcriptase反轉錄酶合成cDNA第1鏈。以cDNA第1鏈為模板合成雙鏈cDNA,利用QIAGEN PCR產物純化試劑盒純化雙鏈cDNA。采用Rsa Ⅰ限制性內切酶對雙鏈cDNA進行單酶切,酶切后用QIAGEN PCR產物純化試劑盒進行純化與濃縮,取5 μL酶切產物進行瓊脂糖凝膠電泳檢測。分別以耐鹽變異水稻H1、H2為檢測子(tester),對照遼星1號為驅動子 (driver)進行SSH雜交,構建耐鹽變異水稻特異表達基因的正向cDNA文庫。將SSH-PCR純化產物做TA克隆、藍白斑篩選,挑選白斑單菌落在37 ℃氨芐青霉素抗性條件下培養,提取質粒以通用引物T7(5′-TAATACGACTCACTC TATAGGG-3′)測序,得到EST序列。將測序得到的EST序列在NCBI水稻數據庫中進行核酸BLAST比對。
1.2.3 RT-PCR驗證
對SSHcDNA文庫中篩選的與水稻cDNA文庫查找沒有同源性的EST序列設計引物,用Primer Premier 5軟件設計引物,由Invotrigen公司合成引物,采用 RT-PCR 技術驗證耐鹽變異水稻H1、H2差異表達基因。
2 結果與分析
2.1 耐鹽變異水稻H1、H2葉片總RNA純度、質量檢測
將提取的耐鹽變異水稻H1、H2和對照遼星1號(CK)總RNA用1.2%瓊脂糖凝膠進行電泳檢測。由圖1可知,28 S、18 S條帶清晰。根據條帶亮度可以判斷,水稻葉片總RNA主要由28 S、18 S組成。同時譜帶亮度明顯,表明提取的總RNA未被降解,純度較高。D 260 nm/D 280 nm比值為196,大于1.8。由此可知,提取的耐鹽變異水稻H1、H2和對照葉片總RNA純度較高、質量完整,滿足進一步抑制消減雜交的要求。
2.2 雙鏈cDNA合成與酶切電泳檢測
1.2%瓊脂糖凝膠檢測結果(圖2)顯示,酶切前后條帶大小發生明顯變化,說明酶切效果很好。未經酶切的雙鏈 cDNA 大小在0.4~10.0 kb之間呈現彌散帶型,且在750 bp附近有比較清晰的條帶,酶切后的cDNA大小在0.1~2.0 kb呈現彌散帶型,且在100、300 bp附近有比較清晰的條帶,說明酶切可以充分進行SSH雜交。
2.3 SSH雜交電泳結果
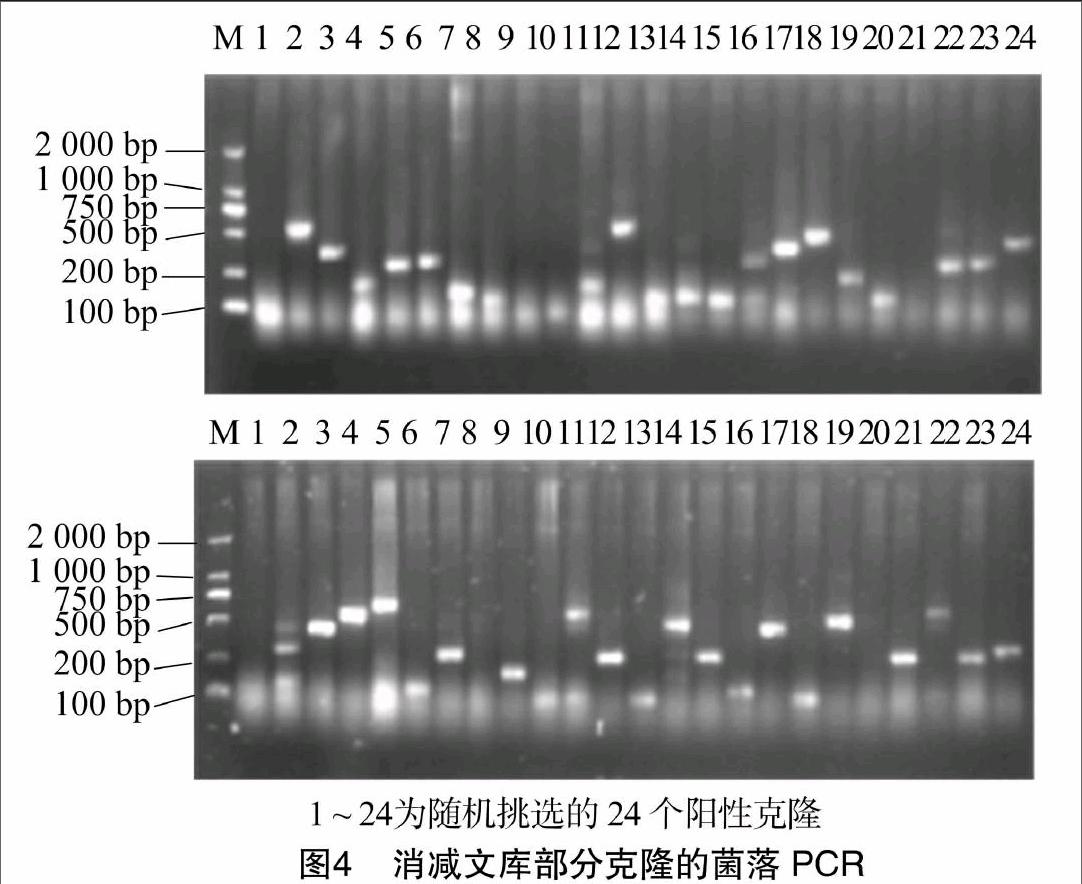
以2次雜交所得產物為模板進行2輪PCR擴增,將PCR產物進行1%瓊脂糖凝膠檢測,耐鹽變異后代H1、H2經第1次消減雜交后,對這些產物進行PCR,結果顯示:泳道1、3出現彌散帶,并且有主帶出現,雖然主帶不明顯,但也說明差異表達基因已經被均衡消減,達到了去除高豐度基因的目的。在第1次雜交混合物中加入新制備的變性CK cDNA,結果顯示,第2輪PCR后在泳道2、4中150、300 bp附近有比較清晰的條帶,說明經過第2次抑制消減雜交后,已經富集誘導表達cDNA目的片段(圖3、圖4)。
2.4 TA克隆及陽性克隆篩選
將第2次PCR擴增產物經QIAGEN PCR產物純化試劑盒純化后與pGEM-Teasy載體連接,轉化大腸桿菌。對所構建的抑制消減雜交文庫進行藍白斑點篩選并計算重組率[重組率=白色菌落數/(藍色菌落數+白色菌落數)×100%],結果顯示,耐鹽變異水稻H1、H2雜交文庫重組率分別為962%、932%。隨機挑選的24個陽性克隆均能擴增出單一清晰條帶。H1、H2雜交文庫插入的片段大小為300~1 000 bp,個別插入片段大于1 000 bp,平均插入片段大小為400 bp。
2.5 SSH文庫測序分析
對挑選的經過菌落PCR檢測過的差異表達克隆進行測序,以T7(ACGACTCACTATAGGGCTTTTT)為引物。H1文庫得到240個高質量的EST序列,H2文庫得到281個高質量的EST序列。將測序質量合格的序列去除序列兩端載體序列、接頭序列后提交到NCBI水稻數據庫中進行核酸BLAST比對,結果表明,H1文庫有234個EST序列在水稻cDNA數據庫中有很高同源性,6個EST序列沒有同源性。H2文庫有272個EST序列在水稻cDNA數據庫中有很高的同源性,9個EST序列沒有同源性。將獲得的15個與水稻沒有同源性的EST序列進行功能比對,其中9個EST序列與植物抗鹽性有關,說明通過轉導蘆葦總DNA獲得的水稻H1、H2、遼星1號在基因水平上存在差異,蘆葦DNA與抗鹽相關的基因片段轉導到水稻DNA中從而導致其耐鹽性發生改變(表1)。
2.6 引物設計結果
選取4個在水稻cDNA數據庫中沒有同源性的EST序列與5個具有與抗鹽性相關的基因EST序列設計引物(表2)。
2.7 RT-PCR驗證
從SSH-cDNA文庫中挑選9個陽性克隆進行RT-PCR驗證。提取水稻耐鹽變異新品種以及對照株的RNA為模板,進行反轉錄合成cDNA,用上述步驟中合成的特異性檢測引物進行PCR擴增,取10 μL PCR擴增產物進行1%瓊脂糖凝膠電泳。RT-PCR擴增后電泳檢測,部分結果如圖5所示,X177、X137檢測引物可從水稻耐鹽變異株中擴增出目的條帶,對照株遼星1號中沒有擴增出特異性條帶,表明遼星1號變異品系成功轉入了蘆葦總DNA。X181、X219、D562檢測引物在耐鹽變異新品種材料中的表達量較高,條帶亮度較為明顯,對照株中的表達量較低,PCR條帶較暗,目的基因在植株體內表達量不是很高。D23沒有擴增出目的條帶,說明陽性克隆特異性弱。X205、D546檢測引物在遼星1號以及耐鹽變異株中均擴增出了目的片段且豐度相同,這是因為消減雜交構建文庫時可能沒有完全消減掉相同的片段,由此說明抑制消減雜交技術還有待改進。X144在對照遼星1號中擴增出了目的條帶而在水稻耐鹽變異株中沒有擴增出目的條帶,表明這個陽性克隆是假陽性。為了驗證片段的正確性,片段進行RT-PCR擴增后的PCR產物再次進行測序,將所得的序列與之前的序列進行比對,發現均屬于相同的片段。
3 結論與討論
通過花粉管通道將外源DNA導入整體植物是一種簡單易行的生物技術,目前已被廣大作物育種工作者用于作物的基因轉移[14]。此技術不但在小麥、棉花、水稻上得以利用,而且在大豆[15]、花生、煙草、白菜、茄子[16]、黃瓜等作物上都進行了供體DNA的轉移。花粉管通道轉基因技術初期階段是以總DNA為外源基因供體,雖能引起后代發生變異,但對外源DNA進入胚囊、卵細胞或受精卵以及核基因組或細胞質基因組的行為,帶有標記基因的質粒或目的基因導入后整合機理,以及引起特定而廣泛變異的機制和轉化后的表達與調節等問題缺乏理論認識,致使國內外一些學者對其可靠性提出了質疑。自從花粉管通道轉基因技術問世,最普遍的疑問是認為變異植株來自種子混雜、異花授粉或自然突變。本研究首次利用SSH技術對水稻通過花粉管通道技術轉導蘆葦總DNA獲得的變異后代H1、H2進行測序研究,最終獲得了許多非來源于水稻的表達序列標簽。未知基因比例高達300%、34.5%,這些未知基因可能來源于供體蘆葦DNA片段與水稻合成相關的新基因。這些EST為最終闡明通過花粉管通道轉導蘆葦總DNA獲得耐鹽變異后代機制提供了直接可靠的分子生物學證據。
3.1 RNA樣本的制備
構建一個高質量的全長cDNA的前提是能提取到結構完整、純度高的總RNA,否則就不能保證所構建的文庫涵蓋90%以上的基因。RNA提取的過程中,應該創造一個無RNaseA的環境,防止RNA降解,并且選擇一種既簡單又快捷的提取RNA的方法。本研究獲得的總RNA質量完整,純度較高,為構建高質量的耐鹽變異后代H1、H2的cDNA文庫奠定了良好的基礎。
3.2 對SSH各個環節進行實時監控
本試驗抑制消減雜交過程中,由反轉錄合成cDNA及 RsaⅠ 酶切的檢測,到加接頭后的接頭連接效率分析直到進行消減cDNA片段的消減效率分析,都要對各個試驗環節進行隨時監控,以保證最終能得到高質量的差異表達cDNA文庫。接頭連接效率同整個試驗效果直接相關,接頭連接后,要進行接頭連接效率檢測。
3.3 陽性克隆的鑒定
菌斑培養的方法是抗生素、藍白斑篩選,一般情況下,使用抗生素篩選則空載體不能成活。藍白斑篩選可以判斷目標片斷是否插入到載體中,白斑或者淺藍斑表示該菌落質粒中已經含有目標片斷。為了能夠更準確地判斷該克隆是否是有水稻誘導后特異表達的基因(正交庫)或者誘導后不表達或者表達量減少的基因(反交庫),應當進一步進行菌落PCR鑒定,對目標基因的特異性進行驗證。
3.4 EST序列的分析
RT-PCR把以RNA當作模板的cDNA合成同PCR結合在一起,是一種快速靈敏的分析基因表達的方法,主要應用于對表達信息進行檢測,除此之外還可以用來檢測基因表達的差異,相比于其他包括RNase保護分析、Northern印跡、原位雜交以及S1核酸酶分析在內的RNA分析技術更加靈敏,更加便于操作。本試驗利用RT-PCR差異片段進行擴增,可以有效避免DNA擴增過程所帶來的內含子的影響,從而可以更高效、快速地驗證差異片段的正確性。
3.5 EST序列的分析
泛肽(ubiquitin)是存在于真核細胞中含有76個氨基酸殘基的高度保守序列的小肽。泛肽途徑是真核細胞選擇性降解蛋白質的1種級聯反應途徑,不但能降解變性的、異常的或短命的蛋白,還能降解植物色素、內膜蛋白、細胞周期蛋白等天然蛋白、轉錄因子,因而在轉錄水平的調節、蛋白的降解、蛋白穩定狀態的調節、程序性細胞死亡、細胞周期的控制等過程中有重要作用。泛素類蛋白被各種脅迫所誘導而表達且該家族成員對脅迫反應是相互獨立的。菜豆泛肽基因能響應高溫、水楊酸、病毒侵染等脅迫,表明菜豆的泛肽水解途徑在清除脅迫條件下累積的損傷蛋白和介導植物的抗逆性具有重要作用。本研究所獲EST序列(登錄號:NM_001156199.1)與玉米泛素結合酶、EST序列(登錄號:XM_002265287.2)的同源性較高。鹽脅迫對光反應的影響主要是對光系統Ⅰ(PSⅠ)、光系統Ⅱ(PSⅡ)以及電子傳遞的影響,通常認為PSⅡ是光抑制的原初位點及主要作用部位。PSⅡ能介導非循環電子傳遞,而且是進行水光解放氧的部位,在光合作用響應環境脅迫中起著極為關鍵的作用。本研究所得到的EST序列(登錄號:XM_002518218.1)與蓖麻PSⅡ葉綠素a P680假定蛋白的同源性較高。有學者認為,逆境脅迫下蛋白水解酶活性上升同清除因脅迫而產生的畸變蛋白(aberrantproteins)有關。研究表明,大麥根系蛋白水解酶活性在鹽脅迫處理下下降,這可能是因為形成了具有蛋白酶抑制劑作用的鹽適應蛋白,在鹽脅迫條件下維持根系中較高的蛋白質含量。本研究所獲EST序列(登錄號:XM_003580628.1)與二穗短柄草的ATP依賴的Clp蛋白水解酶的同源性較高。
參考文獻:
[1]李繼開. 水稻抗鹽育種前途光明[J]. 鹽堿地利用,1992(3):10-12.
[2]李廷軒. 保護地土壤次生鹽漬化的研究進展[J]. 西南農業學報,2001,14(增刊1):103-107.
[3]黃 易,張玉龍. 保護地生產條件下的土壤退化問題及其防治對策[J]. 土壤通報,2004,35(2):212-216.
[4]Diatchenko L,Lau Y F,Campbell A P,et al. Suppression subtractive hybridization:a method for generating differentially regulated or tissue-specific cDNA probes and libraries[J]. Proceedings of the National Academy of Sciences of the United States of America,1996,93(12):6025-6030.
[5]Diatchenko L,Lukyanov S,Lau Y F,et al. Suppression subtractive hybridization:a versatile method for identifying differentially expressed genes[J]. Methods in Enzymology,1999,303:349-380.
[6]王 轉,臧慶偉,郭志愛,等. 小麥幼苗期水分脅迫所誘導基因表達譜的初步分析[J]. 遺傳學報,2004,31(8):842-849.
[7]米麗萍,衛 春,黃俊生,等. 水稻凋亡基因rPDCD5的克隆和表達分析[J]. 遺傳,2004,26(6):893-897.
[8]Xia M,Wang S F,Wang X B,et al. Identification of phosphorus starvation inducible genes in rice by suppression subtractive hybridization[J]. Acta Botanica Sinica,2003,45:736-741.
[9]袁紅雨,祝莉莉,唐 明,等. 利用抑制消減雜交分離受褐飛虱取食下調的水稻基因[J]. 植物生理與分子生物學學報,2004,30(3):305-310.
[10]Tian Z D,Liu J,Xie C H.Isolation of resistance related genes to phytophthora infestans with suppression subtractive hybridization in the R-gene-free potato[J]. Acta Genetica Sinica,2003,30:597-605.
[11]楊傳平,王玉成,劉桂豐,等. 應用SSH技術研究NaHCO3脅迫下檉柳基因的表達[J]. 遺傳學報,2004,31(9):926-933.
[12]劉桂豐,侯英杰,王玉成,等. 干旱脅迫下剛毛檉柳消減文庫的構建及分析[J]. 植物研究,2005,25(1):69-73.
[13]孫洪波,王國英,孫振元,等. 應用抑制差減雜交法分離粗枝大葉黃楊幼苗的冷誘導表達基因[J]. 中國農業科學,2005,38(1):135-139.
[14]Hess D. Attempts to transfor kanamycin resistance of bacterial orgin in Petunia hybrida using pollen as vectors[J]. Biochem Pflanzen,1981,11176:322-328.
[15]雷勃鈞. 導入外源總DNA獲得大豆早熟新品系[J]. 作物學報,1996,22(2):173-177.
[16]林棲鳳,鄧用川,黃 薇,等. 紅樹DNA導入茄子獲得耐鹽性后代的研究[J]. 生物工程進展,2001,21(5):40-44.

