固有無序超嗜熱菌FlgM蛋白在兩種不同溫度下的構象變化特征
朱玉風 曹贊霞 趙立嶺 王吉華(山東師范大學物理與電子科學學院,濟南5004;山東省功能大分子生物物理重點實驗室,山東德州50;德州學院生物物理研究所,山東德州50)
固有無序超嗜熱菌FlgM蛋白在兩種不同溫度下的構象變化特征
朱玉風1,2,3曹贊霞2,3趙立嶺2,3王吉華2,3,*
(1山東師范大學物理與電子科學學院,濟南250014;2山東省功能大分子生物物理重點實驗室,山東德州253023;3德州學院生物物理研究所,山東德州253023)
基于分子動力學模擬方法比較了超嗜熱菌FlgM蛋白在常溫(293 K)和生理溫度(358 K)下的結構特征.基于GROMACS軟件包,采用OPLS-AA分子力場和TIP3P水模型,對超嗜熱菌FlgM蛋白在293和358 K進行了2組獨立的長時間分子動力學模擬,每組體系模擬時間為1500 ns.主要分析了兩種溫度下超嗜熱菌FlgM蛋白的二級結構特征、整體構象變化及半無序化區域和結構化區域的構象特征.研究結果表明:在常溫下,N端具有一定程度的螺旋成分,但在生理溫度下,超嗜熱菌FlgM蛋白的結構變得松散,螺旋結構減少,構象穩定性減弱,H1螺旋散開,FlgM蛋白構象靈活性增強,不穩定程度增加.這些不同溫度的結構變化說明:半無序化區域(N端)在非結合狀態下有一定的螺旋結構,但該段螺旋的穩定性隨溫度升高而降低.超嗜熱菌FlgM蛋白會通過增加結構的無序程度使結構更加靈活,以適應高溫,從而使該類固有無序蛋白更好地行使其功能,如提高同其他成分的結合速率等.
FlgM蛋白;固有無序蛋白;分子動力學模擬;溫度適應性;結構特征
www.whxb.pku.edu.cn
1 引言
自然界億萬年的發展進化使細菌具有極強的適應性.在溫度適應性方面,如嗜熱菌和嗜冷菌這類嗜極細菌能夠在極端的溫度下生存.嗜熱蛋白、嗜常溫蛋白和嗜冷蛋白1的相關研究表明,嗜冷蛋白具有高柔韌性和低耐熱性,而嗜熱蛋白能形成較穩定的結構,這些都表明溫度能夠影響蛋白質結構. FlgM蛋白是一類典型的溫度感應蛋白質,同源FlgM蛋白在不同生理溫度的細菌中廣泛存在,如在生理溫度273 K(0°C)的Leptothrix cholodnii和生理溫度358 K(85°C)的Aquifex aeolicus.FlgM蛋白作為RNA轉錄因子抑制劑,是鞭毛綜合體運動性和趨藥性基因的負調節器,在革蘭氏陰性和革蘭氏陽性細菌中大量存在.2-7FlgM蛋白的基因功能在物種間高度保守,但同源FlgM蛋白在序列結構上差別很大.8
我們的前期預測研究8表明:39個物種的同源FlgM蛋白的生理溫度與其結構無序性具有相關性.其生理溫度的變化范圍為0°C(273 K)到85°C(358 K),不同蛋白具有不同程度的序列長度、序列相似性、二級結構含量及無序程度等等.39個同源FlgM蛋白均為固有無序蛋白,而且隨著生理溫度的升高,蛋白長度變短,無序區的無序程度減小.這39個同源FlgM蛋白在無序程度上相差很大,分為有序態、半無序態和全無序態.其中序列較短的蛋白(長度<97個氨基酸殘基)具有高溫適應性(溫度>45°C 或318 K),其高溫適應性在結構上表現為N端為半無序態且具有較高螺旋含量.8而非高溫最適生長溫度的FlgM蛋白的N端含有極少二級結構,呈全無序態.這些結果表明:不同物種的FlgM蛋白通過由含有極少二級結構的全無序態轉變為具有較高螺旋含量的半無序態來適應高溫,全無序到半無序的轉變可能是自然界生物適應高溫的策略.
超嗜熱菌(the thermophile Aquifex aeolicus,A. aeolicus)可適應85-95°C的高溫.其FlgM(簡寫A.a. FlgM)蛋白是固有無序蛋白質,9同時具有半無序特征.10固有無序蛋白的無序特征有利于其同其他成分發生相互作用.11,12圓二色譜(CD)實驗研究發現,13A.a.FlgM蛋白在室溫20°C(293 K)下稀溶液和粘性溶液中都表現出明顯的α螺旋結構(A.a.FlgM蛋白在室溫下具有活性),形成α螺旋的百分含量高達40.5%;隨著溫度升高直至生理溫度85°C(358 K)時,α螺旋特性逐漸減弱.CD實驗證明A.a.FlgM蛋白柔性結構與溫度明顯相關.
但是,這個半無序蛋白在原子水平的構象如何隨著溫度發生變化?該蛋白半無序區的構象集合特征如何?半無序區和結構區的變化有何不同?這些變化與其高溫適應性調節有何關聯?這一系列問題都有待解決.到目前為止,已經有針對A.a.FlgM蛋白的實驗方面的結構研究,13,14但并沒有揭示溫度是如何影響A.a.FlgM蛋白的結構特征的.分子動力學模擬15-18作為一種有效工具,能將蛋白結構及其結構變化可視化和量化,能夠清楚地得出蛋白結構及其變化.
本文使用分子動力學模擬的方法研究A.a. FlgM蛋白在不同溫度下的結構特征,比較不同溫度對半無序蛋白A.a.FlgM構象變化的影響.為了與實驗信息相比較,分子動力學模擬選擇293 K(20°C)和358 K(85°C)兩個溫度.分析了兩個溫度下的主鏈二面角、二級結構、代表性構象、回旋半徑、均方根漲落(RMSF)等參數.
2 材料和方法
2.1超嗜熱菌FlgM(A.a.FlgM)蛋白的結構
A.a.FlgM蛋白的序列14為:1MVNRIELSRLI GLLLETEKRKNEQKESGTNKIEDKVTLSKIAQEL SKNDVEEKDLEKKVKELKEKIEKGEYEVSDEKV VKGLIEFFT88,共88個殘基.2004年14首次測定了A.a.FlgM蛋白的晶體結構,使用X射線晶體衍射(分辨率:0.325 nm和0.23 nm)得到超嗜熱菌σ28/FlgM復合物的晶體結構(PDB ID:1SC5),該結構中沒有1-2號和18-31號殘基信息.使用Discovery Studio建模軟件,在晶體結構的基礎上將18-31號缺失殘基補齊,殘基序列為:3NRIELSRLIGLLLETEKRKNTEQ KESGTNKIEDKVTLSKIAQELSKNDVEEKDLEKK VKELKEKIEKGEYEVSDEKVVKGLIEFFT88,共86個殘基.我們以此結構(圖1)作為本文模擬研究的初始結構.該結構有四段螺旋:2-14、35-46、56-67及74-83片段,依次為H1、H2、H3、H4螺旋(86個殘基,序號從1開始).
2.2分子動力學模擬和體系準備
基于GROMACS軟件(版本4.5.3),19采用OPLSAA力場20及TIP3P水模型,21研究A.a.FlgM蛋白.初始構象見2.1節,采用NPT系綜,將蛋白溶解在周期性截斷八面體盒子里,蛋白到盒子的最小距離設定為1.0 nm.帶電基團選擇在中性pH下的質子化狀態.添加的水分子為13495個,加入了2個Na+離子替換2個水分子使pH保持電中性.首先使用最陡下降法方法進行第一次能量優化,然后使用共軛梯度法進行第二次能量優化.本模擬采用particle-mesh Ewald(PME)方法22,23處理長程靜電相互作用,采用cut-off方法處理范德華相互作用,采用長度為0.12 nm的網格,溫度耦合采用V-rescale算法,24壓強耦合采用Berendsen算法,25每10步更新一次鄰居列表.模擬溫度設定為293 K(20°C)和358 K(85°C)兩個溫度,對得到的體系進行約束分子動力學模擬.對293 K的模擬,采用六步升溫,設定為50、100、150、200、250、293 K.對358 K的模擬,采用七步升溫,設定為50、100、150、200、250、293、358 K.隨后在兩個不同溫度下分別進行1500 ns的自由動力學模擬.最后,取各自最后200 ns的坐標軌跡進行后續分析.通過計算二級結構、殘基漲落、聚類分析、快照分析、回旋半徑等等,分析了溫度對半無序蛋白二級結構、結構松散程度、構象變化的影響.
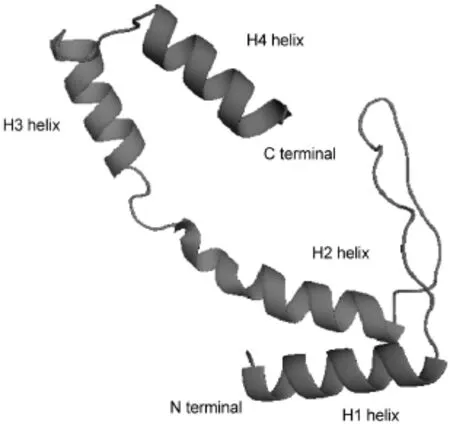
圖1 A.a.FlgM蛋白分子動力學模擬的初始結構Fig.1 Initiating structure ofA.a.FlgM protein on molecular dynamics simulations
3 結果與討論
3.1A.a.FlgM蛋白具有鮮明的固有半無序態蛋白特征
我們的早期研究8基于SPINE-D10,26和SPINEX27,28對A.a.FlgM蛋白進行無序程度和二級結構預測.SPINE-D是基于序列集成神經網絡無序預測程序,預測結果以無序程度P取值從0到1呈現(0和1分別對應完全有序態和完全無序態),能夠很好地計算和區分全無序區與半無序區.在該預測結果中,A.a.FlgM蛋白有兩段半無序區:2-64及78-88片段,一段有序區:65-77片段;有四段螺旋結構:3-15、33-47、58-65及76-87片段.預測結果表明A.a.FlgM蛋白屬于固有半無序蛋白,其N端部分屬于半無序區,而C端部分屬于結構化區域.
3.2不同溫度下A.a.FlgM蛋白的總體構象變化情況
為了探討溫度對A.a.FlgM蛋白總體構象的影響,我們分析了構象snapshot和殘基漲落.Snapshot可以從一系列蛋白質構象中得到一組具有代表性的構象,縮減了構象數據,方便進行總體構象分析.在1500 ns的軌跡中,每100 ns軌跡聚類得到一個代表性構象(聚類是基于構象相互之間的Cα原子的均方根偏差進行分析的,cutoff設為0.1),共聚類15次,得到15個代表性構象,如圖2所示.為了更加明顯地呈現結構變化,另外使用了STRIDE程序29,30計算了二級結構隨時間的變化來分析蛋白質在不同溫度下的二級結構變化情況,31,32如圖3所示.初始構象含有四段螺旋結構,隨著模擬時間的增長,C端的兩個螺旋H3和H4在兩個溫度下都解開.在293 K下,H1和H2螺旋基本保持穩定,與在復合物中的結構基本相同;但在358 K下,H2螺旋基本保持不變,H1螺旋慢慢解開,并且有向折疊片轉變的趨勢.
另一方面,我們分析了構象總體的穩定性,計算了兩個不同溫度下最后200 ns即1301-1500 ns的殘基漲落情況,如圖4所示.殘基的均方根漲落反映了構象系綜中原子的漲落情況,RMSF越大表示分子動力學模擬過程中殘基偏離參考構象的程度越大.在圖4中,1-20號殘基、25-32號殘基、49-86號殘基漲落明顯增加,即H1、H3和H4螺旋穩定性較差. 在RMSF概率分布中,變化最明顯的是0-0.2和0.2-0.6區間內,溫度升高,RMSF在0-0.2之間的概率明顯減少,RMSF在0.2-0.6之間的概率明顯增加.溫度升高之后殘基漲落整體增加,結構穩定性變差,但是358 K左右是超嗜熱菌的生理溫度,說明FlgM蛋白功能的實現需要一定的無序程度.
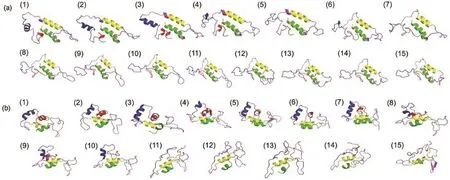
圖2 293和358 K溫度下在0-1500 ns內從snapshot取出的連續構象Fig.2 Sequential conformations from snapshot during 0-1500 ns at temperatures of 293 and 358 K(a)293 K,(b)358 K.(1)-(15)indicate the most clustered structure of selected group.Conformations are represented as cartoon in the same proportion.The four helixes colored with green,yellow,blue,and red,respectively.
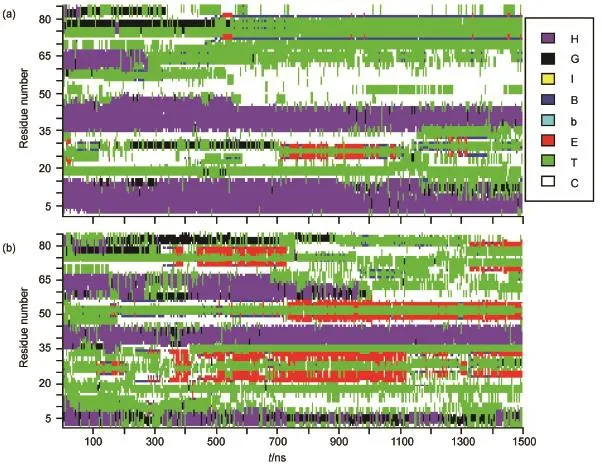
圖3 293和358 K溫度下在0-1500 ns內各構象二級結構隨時間變化Fig.3 The second structure content plotted as a function of time of each conformation during 0-1500 ns at temperatures of 293 and 358 K(a)293 K;(b)358 K.The structures of H,G,I,B,b,E,T,C are colored of blueviolet,black,yellow,blue,cyan,red,green,and white,respectively. H:Alpha helix;G:3-10 helix;I:PI-helix;E:extended conformation;B or b:Isolated bridge;T:Turn;C:Coil(none of the above)
3.3在不同溫度下A.a.FlgM蛋白的聚類分析
前面分析發現蛋白在358 K下穩定性減弱.根據預測結果,A.a.FlgM蛋白的N端部分屬于半無序化區域,而C端部分屬于結構化區域.為了分析溫度對半無序化區域和結構化區域是否存在不同影響,我們選取了N端的1-38號殘基片段和C端的47-84號殘基片段各38個殘基作為研究對象,進行聚類分析.聚類使用g_cluster程序,基于構象相互之間的Cα原子的均方根偏差進行分析,cutoff設為0.1,結果如表1所示.從表1可以看出,無論是半無序化區域還是結構化區域,358 K下聚類數都要比293 K下多,表明358 K下的構象類型較多,構象采樣更豐富;同時說明相同時間內,采樣構象數多,即構象變化范圍更廣闊.在生理溫度下,A.a.FlgM蛋白的靈活性增大,有利于它同其它成分相互作用,進而行使其生物功能.
3.4在不同溫度下A.a.FlgM蛋白的二級結構變化
前面是基于整體分析溫度對構象的影響.下面基于二面角分布和二級結構形成情況,分析溫度對二級結構的影響.
首先,使用g_rama程序計算1301-1500 ns內殘基主鏈二面角進而得到其分布,使用平均力勢(PMF)分布來表征.二面角平均力勢PMF定義為:
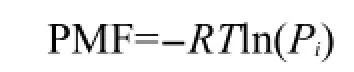
其中R為理想氣體常數,T為溫度,Pi為殘基主鏈二面角落入區域i的幾率.區域i為-180°≤ψ≤180° 且-180°≤φ≤180°范圍內的小區間.兩模擬體系的殘基主鏈二面角平均力勢分布如圖5所示.平均力勢越低,相應構象被采樣的幾率就越大,圖中紅色區域為平均力勢極小區域,相鄰力勢曲線間隔為2 kJ·mol-1.由圖5可知,在293 K下半無序區域的主鏈二面角主要分布在α螺旋區域,其他區域較少;結構化區域的主鏈二面角主要分布在β折疊區域.在358 K下,半無序區域的主鏈二面角平均分布在β折疊區域和α螺旋區域;結構化區域的主鏈二面角主要分布在β折疊區域.能量低點說明相應二級結構形成的可能性較大,但不一定形成.表2給出了不同溫度下不同片段落在α螺旋區域和β折疊區域的平均力勢極小值的相對深度及坐標.結果表明,對半無序化片段,溫度升高后其在α螺旋區域的采樣明顯減少,β折疊區域的采樣明顯增加;而對結構化區域,溫度升高后其在α螺旋區域的采樣和β折疊區域的采樣無明顯變化.有實驗13發現,在0 mol·L-1葡萄糖濃度下,293 K下形成31%的α螺旋結構和11%的β折疊結構,358 K下形成24%的α螺旋結構和21% 的β折疊結構,這就意味著溫度升高后α螺旋結構成分減少,β折疊結構成分增加,我們的模擬結果與實驗結果是相符合的.
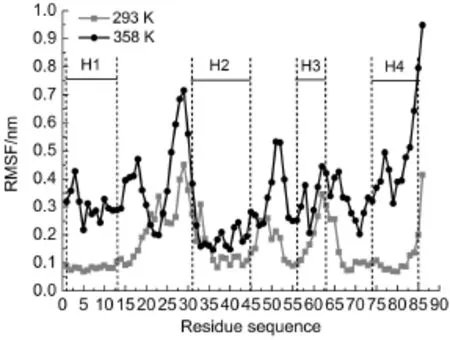
圖4 293和358 K溫度下在1301-1500 ns內各殘基的均方根漲落分布(RMSF)Fig.4 Root mean square fluctuation(RMSF)of each residue during 1301-1500 ns at temperatures of 293 and 358 K
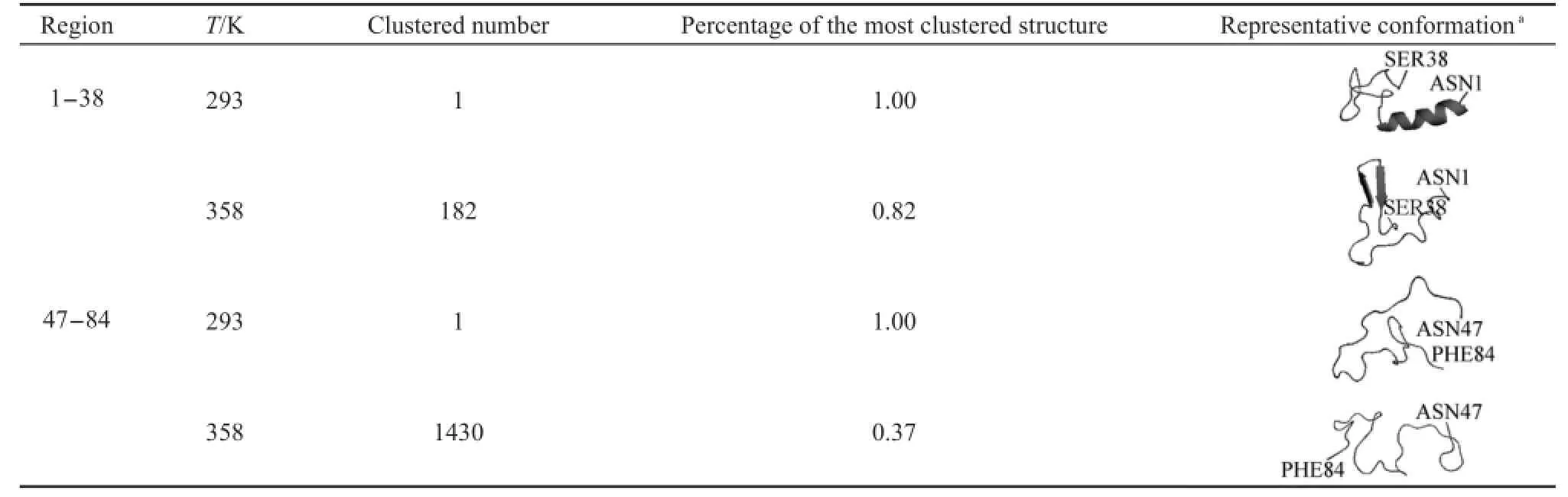
表1 293和358 K下1301-1500 ns構象聚類分布Table 1 Clustered conformation distribution during 1301-1500 ns at temperatures of 293 and 358 K
同時,使用STRIDE軟件29,30分別計算1301-1500 ns時間內的殘基形成二級結構的變化情況.結果表明,293 K下,螺旋(包括α螺旋和3-10螺旋)結構所占的比例為18.8%,β折疊片結構所占的比例為0.8%,Turn結構所占的比例為45.7%,無規卷曲(Coil)結構所占的比例為31.2%.358 K下,螺旋(包括α螺旋和3-10螺旋)結構所占的比例為12.9%,β折疊片結構所占的比例為13.8%,Turn結構所占的比例為48.0%,Coil結構所占的比例為24.4%.說明隨著溫度增加α螺旋含量減少,而β折疊結構增加.
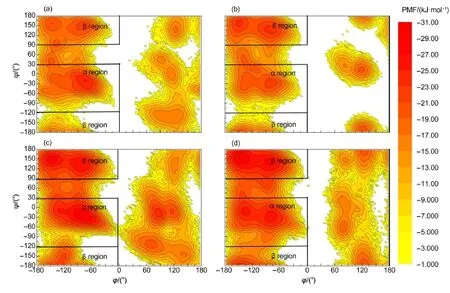
圖5 293和358 K溫度下半無序化區域和結構化區域中殘基主鏈Ramachandran二面角(φ,ψ)對應的平均力勢(PMF)分布Fig.5 Distributions of potentials of mean force(PMF)plotted as a function of the Ramachandran angles(φ,ψ)of residue backbones for semi-disordered region and structured region respectively at the temperatures of 293 and 358 K(a)semi-disordred region at the temperature of 293 K;(b)structured region at the temperature of 293 K;(c)semi-disordered region at the temperature of 358 K;(d)structured region at the temperature of 358 K.Neighboring contour lines are separated by 2 kJ·mol-1.

表2 A.a.FlgM蛋白殘基主鏈二面角(φ,ψ)PMF極小值分布及相應坐標Table 2 Distributions of the PMF minima of Ramachandran angle(φ,ψ)of residue backbones ofA.a.FlgM protein and corresponding coordinates
為進一步分析比較293和358 K下形成螺旋結構的差異,計算了各殘基形成螺旋結構或折疊結構的概率,如圖6所示.在293 K下,形成兩段螺旋:2-14段殘基,形成概率介于17.6%-99.0%;35-45段殘基,形成概率介于17.6%-99.0%.在358 K下,同樣形成兩段螺旋:2-8段殘基,形成概率介于14.5%-66.0%;37-45段殘基,形成概率介于63.9%-99.4%.在兩個不同溫度下,第二段螺旋沒有明顯變化,第一段螺旋形成概率明顯減小.初始結構具有4段螺旋,在兩種溫度下,H3和H4都已經消失,與圖2結果一致,表明在這部分區域即C端區域只有與核苷酸聚合酶σ因子結合時才形成穩定的結構,而在單體情況下呈無序狀,與SPINE-D預測結果一致.螺旋H2在兩種溫度下變化不大,表明該部分區域穩定性較強;H1在生理溫度(358 K)時穩定性較差.上述分析表明,A.a.FlgM蛋白為適應溫度變化,可能主要通過N端無序化區域的螺旋形成概率或者無序程度的變化來進行調節的.其他同源FlgM蛋白是否也具有類似的溫度適應性規律,有待于進一步研究.
3.5在不同溫度下A.a.FlgM蛋白的三級結構分析
以上分析已經證明了溫度對A.a.FlgM蛋白二級結構形成具有影響,為了更準確地分析構象穩定性的變化,使用參數Contact進行了蛋白質三級結構的分析.Contact是三維蛋白質分子中所有氨基酸殘基Cα原子之間距離的反映.定義距離閾值為0.8 nm,33即若兩殘基Cα原子之間距離小于0.8 nm,則認為它們之間存在Contact.計算兩溫度下1301-1500 ns內的Contact分布,如圖7所示.Percent為殘基間形成Contact的概率.圖中顏色越深,表示形成Contact的概率越大.圖中293 K下,在1-40號殘基片段之間有“U”型結構,說明在20號殘基附近形成彎曲;在65-80號殘基片段附近也有部分彎曲.在358 K下,在20-30、45-60、65-85號殘基片段附近有部分“U”型結構.20-30、45-60號殘基片段附近有明顯彎曲形成,結構變化較大.在358 K還有一部分Contact形成,零散分布,結合作用較弱.由此可以得出溫度升高使得A.a.FlgM蛋白的構象更加卷曲,構象穩定性減弱.
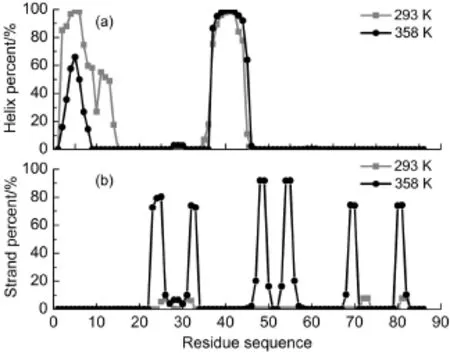
圖6 293和358 K下A.a.FlgM蛋白二級結構的螺旋結構(a)和折疊結構(b)分布Fig.6 Residue-specific second structure helix(a)and strand(b)percentages ofA.a.FlgM protein respectively at the temperatures of 293 and 358 K
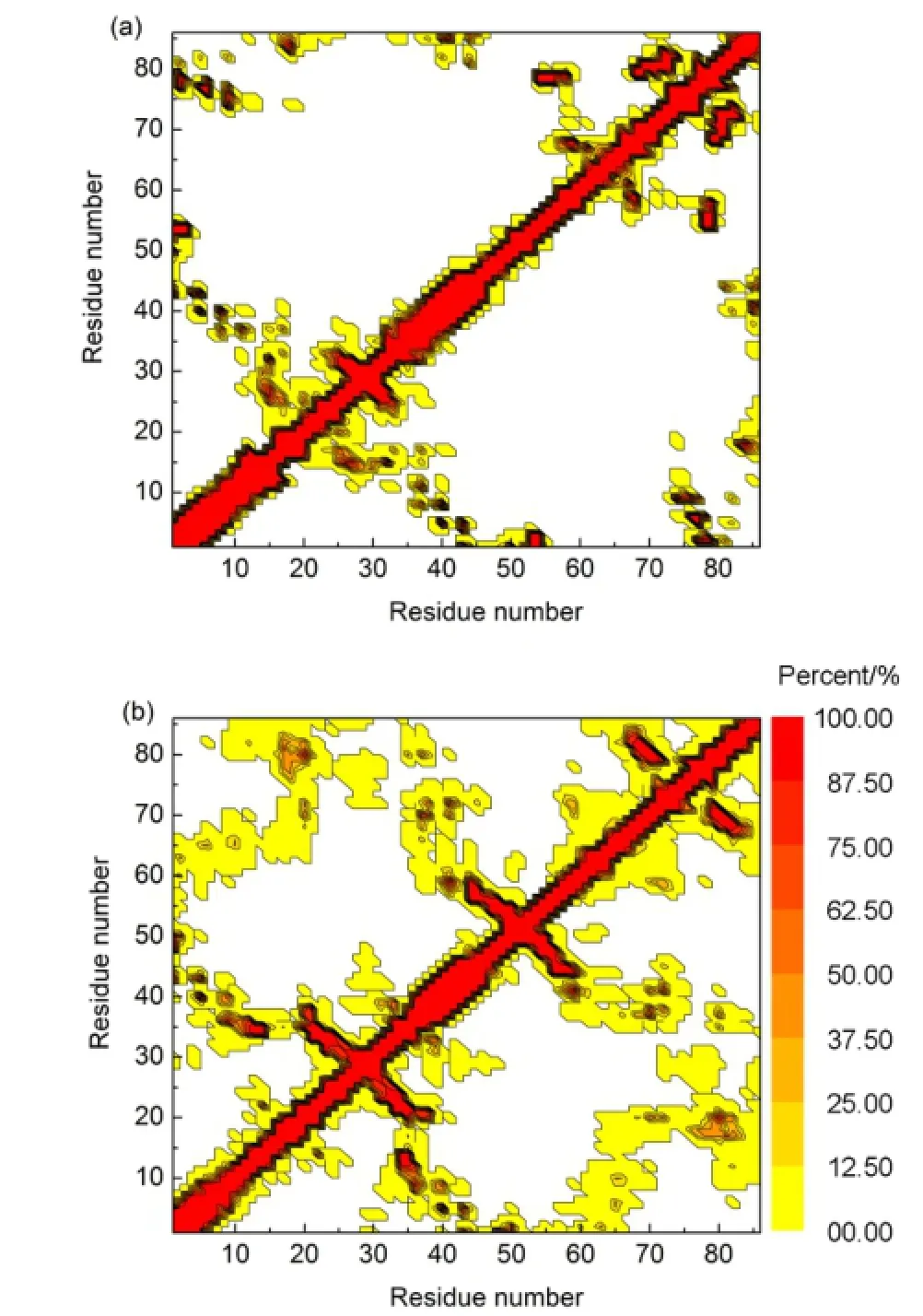
圖7 293和358 K下1301-1500 ns模擬內各殘基間contact形成情況Fig.7 Formation of contacts between residues at the temperatures of 293 and 358 K during the 1301-1500 ns simulation(a)293 K;(b)358 K.Percent is representing the formation rates of contact between residues.
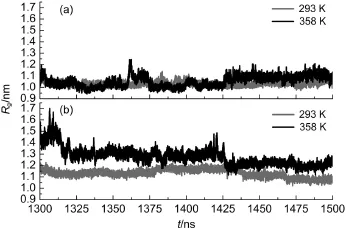
圖8 293和358 K溫度下A.a.FlgM蛋白半無序化區域和結構化區域在1301-1500 ns模擬內回旋半徑(Rg)分布Fig.8 Distributions of radius of gyration(Rg)plotted as a function of time at the semi-disordered region and structured region fromA.a.FlgM protein at the temperatures of 293 and 358 K during the 1301-1500 ns simulation(a)semi-disordered region,(b)structured region
3.6在不同溫度下A.a.FlgM蛋白半無序化區域和結構化區域的結構特征
蛋白回旋半徑反映了蛋白的松散程度.我們選取了N端的1-38號殘基片段和C端的47-84號殘基片段各38個殘基作為研究對象,分析不同無序程度區域的回旋半徑受溫度的影響(見圖8).對于N端半無序化區域而言,其回旋半徑受溫度影響較小,358 K下略大于293 K下的變化.而對于C端結構化區域,其回旋半徑受溫度影響比較大.同時,對N端半無序化區域和C端結構化區域進行比較發現,C端結構區域的伸展范圍在生理溫度下更廣,有利于它同其他成分相互作用.由此可以看出,半無序蛋白A.a.FlgM蛋白的N端半無序化區域的伸展程度隨溫度變化較小,而且高溫的伸展程度與低溫的不相上下,C端的結構化區域的伸展程度隨溫度的變化較大,且高溫時的伸展程度明顯大于低溫的情況.這給我們以啟迪,是否可以通過溫度影響伸展效應來區分有序化區域和無序化區域?值得進一步研究.
4 結論
A.a.FlgM蛋白是超嗜熱菌鞭毛組裝的負反饋階段的關鍵蛋白,而高溫影響A.a.FlgM蛋白結構這一關鍵對其功能的實現至關重要,實驗表明溫度升高會導致A.a.FlgM蛋白構象擴展.為了深入探索溫度對A.a.FlgM蛋白構象影響的微觀過程,本文模擬研究了A.a.FlgM蛋白在293和358 K下的動力學特征.采用OPLS-AA力場以及TIP3P水模型,完成了兩個1500 ns的獨立長時間分子動力學模擬,總模擬時間長達3000 ns.針對模擬軌跡,計算了殘基主鏈二面角、二級結構、回旋半徑、RMSF等參數,分析了半無序化區域及結構化區域的結構特征等.分析結果表明,在生理溫度下,H1螺旋散開,A.a.FlgM蛋白構象靈活性增加,不穩定程度增加,結構松散性增加;半無序區在非結合狀態下有一定的螺旋結構,并且該段螺旋的穩定性隨溫度升高而降低,同時溫度升高會使得蛋白C端結構區的伸展范圍更廣.處于結構化區域的C端是A.a.FlgM蛋白的功能化區域,溫度升高提高了A.a.FlgM蛋白的靈活性,有利于它同其他成分或蛋白發生相互作用.這些研究,有助于我們認識固有無序蛋白質的溫度效應,為揭示有序和無序的區別提供了新的啟迪.
References
(1)Feller,G.J.Phys.:Condes.Matter 2010,22(32),323101.doi:10.1088/0953-8984/22/32/323101
(2)Ohnishi,K.;Kutsukake,K.;Suzuki,H.;Iino,T.Mol.Genet. Metab.1990,221(2),139.
(3)Gillen,K.L.;Hughes,K.T.J.Bacteriol.1991,173(20),6453.
(4)Ohnishi,K.;Kutsukake,K.;Suzuki,H.;Lino,T.Mol. Microbiol.1992,6(21),3149.doi:10.1111/mmi.1992.6.issue-21
(5)Gillen,K.L.;Hughes,K.T.J.Bacteriol.1993,175(21),7006.
(6)Hughes,K.T.;Gillen,K.L.;Semon,M.J.;Karlinsey,J.E. Science 1993,262(5137),1277.doi:10.1126/science.8235660
(7)Kutsukake,K.Mol.Genet.Metab.1994,243(6),605.
(8)Wang,J.H.;Yang,Y.D.;Cao,Z.X.;Li,Z.X.;Zhao,H.L.;Zhou,Y.Q.Biophys.J.2013,105(11),1.
(9)Dunker,A.K.;Brown,C.J.;Lawson,J.D.;Iakoucheva,L.M.;Obradovic,Z.Biochemistry 2002,41(21),6573.doi:10.1021/ bi012159+
(10)Zhang,T.;Faraggi,E.;Li,Z.X.;Zhou,Y.Q.Cell Biochem. Biophys.2013,76(3),1193.
(11)Huang,Y.Q.;Liu,Z.R.Acta Phys.-Chim.Sin.2010,26,2061.[黃永棋,劉志榮.物理化學學報,2010,26,2061.]doi:10.3866/ PKU.WHXB20100644
(12)Liu,Z.R.;Huang,Y.Q.Protein Sci.2014,23(5),539.doi:10.1002/pro.2443
(13)Molloy,R.G.;Ma,W.K.;Allen,A.C.;Greenwood,K.;Bryan,L.;Sacora,R.;Williams,L.;Gage,M.J.Biochim.Biophys.Acta 2010,1804(7),1457.doi:10.1016/j.bbapap.2010.03.002
(14)Sorenson,M.K.;Ray,S.S.;Darst,S.A.Mol.Cell 2004,14(1),127.doi:10.1016/S1097-2765(04)00150-9
(15)Xu,Z.Y.;Zhao,L.L.;Cao,Z.X.;Wang,J.H.Acta Phys.-Chim.Sin.2012,28,1665.[許朝瑩,趙立嶺,曹贊霞,王吉華.物理化學學報,2012,28,1665.]doi:10.3866/PKU. WHXB201204182
(16)Luo,F.;Gao,J.;Cheng,Y.H.;Cui,W.;Ji,M.J.Acta Phys.-Chim.Sin.2012,28,2191.[羅芳,高劍,成元華,崔巍,計明娟.物理化學學報,2012,28,2191.]doi:10.3866/ PKU.WHXB201207063
(17)Dong,X.Y.;Du,W.J.;Liu,F.F.Acta Phys.-Chim.Sin.2012,28,2735.[董曉燕,都文婕,劉夫鋒.物理化學學報,2012,28,2735.]doi:10.3866/PKU.WHXB201207162
(18)Cao,J.;Cao,Z.X.;Zhao,L.L.;Wang,J.H.Acta Phys.-Chim. Sin.2012,28,479.[曹劍,曹贊霞,趙立嶺,王吉華.物理化學學報,2012,28,479.]doi:10.3866/PKU.WHXB201111231
(19)Van der Spoel,D.;Lindahl,E.;Hess,B.;Groenhof,G.;Mark,A. E.;Berendsen,H.J.J.Comput.Chem.2005,26(16),1701.
(20)Jorgensen,W.L.;Tirado-Rives,J.Proc.Natl.Acad.Sci.U.S.A. 2005,102(19),6665.doi:10.1073/pnas.0408037102
(21)Jorgensen,W.L.;Chandrasekhar,J.;Madura,J.D.;Impey,R. W.;Klein,M.L.J.Chem.Phys.1983,79(2),926.doi:10.1063/ 1.445869
(22)Essmann,U.;Perera,L.;Berkowitz,M.L.;Darden,T.;Lee,H.;Pedersen,L.G.J.Chem.Phys.1995,103(19),8577.doi:10.1063/1.470117
(23)Darden,T.;York,D.;Pedersen,L.J.Chem.Phys.1993,98(12),10089.doi:10.1063/1.464397
(24)Bussi,G.;Donadio,D.;Parrinello,M.J.Chem.Phys.2007,126(1),014101.doi:10.1063/1.2408420
(25)Berendsen,H.J.;Postma,J.P.M.;Van Gunsteren,W.F.;DiNola,A.;Haak,J.J.Chem.Phys.1984,81(8),3684.doi:10.1063/1.448118
(26)Zhang,T.;Faraggi,E.;Xue,B.;Dunker,A.K.;Uversky,V.N.;Zhou,Y.Q.J.Biomol.Struct.Dyn.2012,29(4),799.doi:10.1080/073911012010525022
(27)Faraggi,E.;Yang,Y.D.;Zhang,S.S.;Zhou,Y.Q.Structure 2009,17(11),1515.doi:10.1016/j.str.2009.09.006
(28)Faraggi,E.;Zhang,T.;Yang,Y.D.;Kurgan,L.;Zhou,Y.Q. J.Comput.Chem.2012,33(3),259.doi:10.1002/jcc.v33.3
(29)Frishman,D.;Argos,P.Proteins:Struct.Funct.Bioinf.1995,23 (4),566.
(30)Heinig,M.;Frishman,D.Nucleic Acids Res.2004,32(suppl 2),W500.
(31)Liu,F.F.;Ji,L.;Dong,X.Y.;Sun,Y.J.Phys.Chem.B 2009,113(32),11320.doi:10.1021/jp905580j
(32)Liu,F.F.;Dong,X.Y.;He,L.Z.;Middelberg,A.P.J.;Sun,Y. J.Phys.Chem.B 2001,115(41),11879.
(33)Tegge,A.N.;Wang,Z.;Eickholt,J.;Cheng,J.L.Nucleic Acids Res.2009,37(suppl 2),W515.
Conformational Change Characteristics in the lntrinsically Disordered FlgM Protein from a Hyperthermophile at Two Different Temperatures
ZHU Yu-Feng1,2,3CAO Zan-Xia2,3ZHAO Li-Ling2,3WANG Ji-Hua2,3,*
(1Department of Physics and Electronic Science,Shandong Normal University,Jinan 250014,P.R.China;2Shandong Province Key Laboratory of Biophysics for Functional Macromolecules,Dezhou 253023,Shandong Province,P.R.China;3Institute of Biophysics,Dezhou University,Dezhou 253023,Shandong Province,P.R.China)
The aim of this work was to compare the structural characteristics of the FlgM protein from the thermophile aquifex aeolicus at room temperature(293 K)and at the physiological temperature(358 K)using molecular dynamics simulations.Two independent long-time molecular dynamics simulations were performed using the GROMACS software package at 293 and 358 K,respectively.The OPLS-AAforce field and the TIP3P water model were used.Each simulation was run for 1500 ns.We mainly analyzed the secondary structural characteristics,the overall conformation variation,the conformational characteristics of a semi-disordered region and the structured region of the FlgM protein at two different temperatures.The results indicate that the helix structure of the N terminal increased at room temperature.The FlgM protein had the following characteristics at the physiological temperature:the structure loosed,the helix structure reduced in size,the conformational stability weakened,the H1 helix spread,the conformational flexibility increased,and the degree of instability increased.In summary,the semi-disordered region(N terminal)formed a helical structure in the unbound state and its stability decreased with an increase in temperature.The FlgM protein adapts to temperature by increasing the degree of disorder,creating a more flexible structure by improving the binding rate.
September 15,2014;Revised:December 16,2014;Published on Web:December 16,2014.?
.Email:jhw25336@126.com;Tel:+86-534-8985933.
FlgM protein;Intrinsically disordered protein;Molecular dynamics simulation;Temperature adapting;Structure characteristic
?Editorial office ofActa Physico-Chimica Sinica
O643;O641
10.3866/PKU.WHXB201412161
The project was supported by the National Natural Science Foundation of China(61271378,31000324).
國家自然科學基金(61271378,31000324)資助項目

