植物凝集素結構多樣性與藥物設計
趙蘊杰,劉志超1222,劉琴,陳高,陳禪友,曾辰
1.喬治華盛頓大學物理系,美國華盛頓哥倫比亞特區20052 2.江漢大學生命科學學院,武漢430056
*論著——生物信息技術*
植物凝集素結構多樣性與藥物設計
趙蘊杰1,2,劉志超12221,2,劉琴,陳高,陳禪友,曾辰
1.喬治華盛頓大學物理系,美國華盛頓哥倫比亞特區20052 2.江漢大學生命科學學院,武漢430056
植物凝集素蛋白最早發現于19世紀,可特異性結合碳水化合物與糖分子。植物凝集素蛋白結合碳水化合物的高度特異性使我們可以將其作為生物工具應用到不同領域。例如,植物凝集素的重要特征之一就是可以特異性結合昆蟲消化道表面的糖分子,從而起到抗蟲的作用。植物凝集素也可以特異性結合癌細胞膜表面的糖分子,從而導致細胞凋亡或抑制腫瘤的生長。研究表明,僅豆科植物的凝集素分子表面就有大約40萬個潛在的結合位點可以特異性結合不同的糖分子。因此,研究植物凝集素的結構特征信息對更好地理解植物凝集素的結構功能關系與應用有重要的意義。近年來,計算生物學的飛速發展使我們可以在分子層次上研究植物凝集素。本文主要從計算生物學角度綜述植物凝集素的結構,序列保守性與動力學等研究現狀,并以菜豆凝集素為例討論植物凝集素的藥物設計和醫學應用。
植物凝集素;三級結構;進化;藥物設計;醫學應用
植物凝集素最早發現于19世紀80年代,至今已有一百多年的歷史,因其凝集紅細胞與特異性結合碳水化合物的能力,具有非常廣泛的用途[1-4]。例如,昆蟲和高等動物的消化道表面有很多細胞膜糖蛋白,植物凝集素與其特異性結合會引起昆蟲和高等動物的不適,從而起到保護植物的作用。植物凝集素也會與器官暴露在上皮細胞的糖分子特異性結合,利用植物凝集素特異性結合碳水化合物的特性,可以將其作為生物粘附劑輸送藥物到特定靶標[5-7]。另外,植物凝集素還可以刺激細胞有絲分裂。研究表明,僅豆科植物的凝集素分子表面就有大約40萬個潛在的結合位點,植物凝集素具有非常廣泛的應用前景[8,9]。因此,研究植物凝集素的結構和功能關系,如何利用植物凝集素的結構和生物學特性逐漸成為了熱點研究問題。
2013年諾貝爾化學獎授予了Martin Kaplus, Michael Levitt和AriehWarshel以表彰他們在發展多尺度復雜化學系統模型方面所做的貢獻[10,11]。近年來,計算生物學領域的飛速發展給我們提供了一種可以理解和捕捉生物實驗中涉及幾千甚至幾百萬原子協同發生化學反應的工具,尤其是分子力場和分子動力學模擬的不斷發展讓我們可以捕捉生物分子在原子層次上的動態行為[12-17]。計算生物學在植物凝集素領域也有非常廣泛的應用,例如Surolia等用分子動力學模擬研究香蕉凝集素二聚體在不同溫度下的結構穩定性情況[18,19]。香蕉凝集素是一種同源二聚體的非糖基化蛋白,高溫動力學模擬結果表明,香蕉凝集素二聚體接觸面的三級結構相互作用對二聚體結構的穩定至關重要。在400~500K的高溫動力學模擬中,該二聚體結構仍然十分穩定,僅僅在無規卷曲有較大的柔性變化。Surolia等進一步模擬了糖基化勁直刺桐凝集素,模擬結果表明糖基化勁直刺桐凝集素二聚體接觸面上三級相互作用的動力學特征和靜態結構中觀察到的三級結構相互作用特征有較大不同[20]。因此,綜合考慮植物凝集素的靜態與動力學特征對深刻理解植物凝集素的結構與生物功能關系,設計生物粘附劑輸送藥物分子和設計抑制劑降低植物凝集素毒性等應用均有非常大的幫助。
本文主要從計算生物學的角度綜述植物凝集素的現狀及其應用,主要內容有:(1)總結蛋白質結構數據庫(Protein Data Bank,簡稱PDB)中植物凝集素的三級結構特征;(2)以菜豆凝集素為例分析植物凝集素的序列保守性,靜態結構與動力學特征;(3)以菜豆凝集素為例討論如何設計抑制劑降低植物凝集素的凝血毒性。
1 植物凝集素三級結構特征
植物凝集素需要折疊成特定的三級結構才能實現其生物學功能,因此充分了解植物凝集素的三級結構特征對理解其生物學功能至關重要。蛋白質三級結構數據庫是目前最主要的收集生物大分子三級結構的數據庫,通過X射線晶體衍射、核磁共振、電子衍射和冷凍電鏡等實驗手段測定蛋白質、核酸等生物大分子的三級結構[21]。目前,蛋白質三級結構數據庫共收集了106710個生物分子結構數據。本章節我們將結合蛋白質三級結構數據庫中測定的植物凝集素實驗結構討論植物凝集素的三級結構特征。
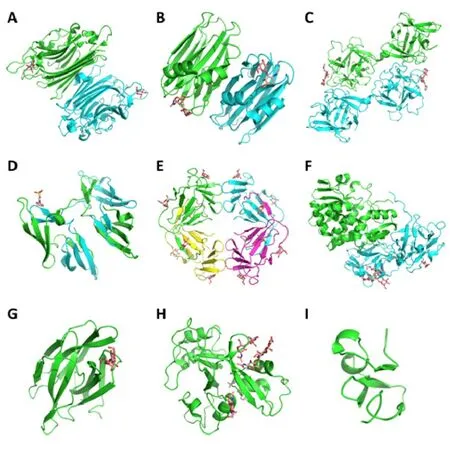
圖1 植物凝集素蛋白質三級結構示意圖不同顏色分別表示二聚體或多聚體中不同的蛋白質鏈或糖分子Fig.1 Cartoon representations of lectin proteins from different fold families. Dimer structures are colored in blue and green. Sugars are colored in orange. (A) Legume lectin (PDB code: 3WCR). (B) Agaricusbisporus agglutinin homologs (PDB code: 1Y2V). (C) Amaranthins (PDB code: 1JLX). (D) Cyanovirin family (PDB code: 1L5B). (E) Galanthusnivalis agglutinin (PDB code: 1MSA). (F) Ricin-B (PDB code: 1HWM). (G) Jacalin (PDB code: 1C3M). (H) LysM (PDB code: 4EBY). (I) Hevein (PDB code: 1HEV).
研究表明,植物凝集素可根據序列和糖分子結合位點結構分為12類結構[22]。豆科植物凝集素(Legume lectin)廣泛分布在如菜豆,刀豆,黃豆,豌豆,扁豆,花生等植物中,可特異性識別半乳糖,甘露糖,葡萄糖等糖分子。圖1A為菜豆凝集素的結構,該單體結構為一個7條β折疊片形成的β折疊層和6條β折疊片形成的β折疊層通過β轉角連接組成,單體長度一般為250個氨基酸左右,并進一步形成穩定的二聚體結構(PDB code:3WCR)[23]。ABA(Agaricusbisporus agglutinin homologs)植物凝集素單體一般由140個氨基酸左右的序列組成,可特異性識別T antigen和N-glycans等分子。圖1B為ABA植物凝集素結構(PDB code: 1Y2V)[24],該凝集素單體為一個4條β折疊片形成的β折疊層和6條β折疊片形成的β折疊層通過螺旋-環-螺旋的模體結構連接組成,并進一步形成二聚體結構,T antigen結合在螺旋-環-螺旋模體結構區域。Amaranthins植物凝集素主要分布在莧屬植物中,可特異性結合T antigen和GalNAc等分子。圖1C為Amaranthins植物凝集素結構,該單體結構由兩個150個氨基酸左右的β-trefoil通過α螺旋串聯組成,并進一步通過頭-尾相互作用形成穩定的二聚體結構(PDB code: 1JLX)[25]。CVN(Cyanovirin family)植物凝集素最早在藍藻中發現,因其與人類免疫缺陷病毒(HIV)表面的糖分子有較高親和性,是潛在的抗病毒分子工具。圖1D為CV-N凝集素結構,該單體結構主要由4個β折疊層連接組成(101個氨基酸左右),并進一步通過頭-尾相互作用形成穩定的二聚體結構(PDB code:1L5B)[26]。GNA(Galanthusnivalis agglutinin)植物凝集素最早從雪蓮花中發現,可特異性結合甘露糖分子。實驗研究表明,GNA凝集素的空間拓撲結構非常保守,主要由3個β折疊層(每個β折疊層均有一個甘露糖結合位點)形成β-prism結構,凝集素單體一端插入另外一個單體形成β折疊層進而形成二聚體,并進一步形成穩定的四聚體結構(圖1E,PDB code:1MSA)[27]。Ricin-B植物凝集素最早在蓖麻中發現,為最早發現的植物凝集素,該凝集素可以特異性結合GalNAc等分子。晶體結構研究表明,典型的Ricin-B凝集素單體為兩個串聯的重復結構域組成,每個結構域為三對稱的β-trefoil結構(圖1F,PDB code:1HWM)[28]。Jacalin植物凝集素可特異性結合半乳糖和甘露糖分子,圖1G為菊科植物的Jacalin凝集素結構(實驗僅解析出單體結構,PDB code:1C3M)[29],該結構域由12個β折疊片形成3個重復的β折疊結構,并進一步形成二聚體和四聚體結構。LysM植物凝集素可以特異性結合甘露糖和氨基葡萄糖等分子,空間拓撲結構不十分保守,典型的LysM單體結構為40個氨基酸組成的β-α-α-β三對稱結構,并形成多聚體結構(圖1H,實驗僅解析出單體結構,PDB code:4EBY)[30]。Hevein是1975年在橡膠樹中發現的植物凝集素類型,其序列長度為43個氨基酸左右,空間拓撲結構并不十分保守。圖1I中橡膠樹Hevein凝集素N端為2個β折疊片段組成的β折疊結構,C端為一個β折疊片段,兩個β折疊結構間由兩個較短的螺旋結構連接組成,并進一步形成四聚體結構(PDB code:1HEV)[31]。CRA(Class V chitinase homologs)植物凝集素最早由刺槐中提取[32],單體長度為337個氨基酸左右。CRA植物凝集素目前還沒有解析出晶體結構,同源建模研究表明該結構主要由TIM‐barrel組成,并進一步形成二聚體結構。Nic(Nictaba family)和EEA(Euonymus europaeuslectin)植物凝集素可以特異性結合甘露糖和聚糖等分子,并形成二聚體結構,目前沒有實驗結構。
植物凝集素多為二聚體或多聚體結構,形成二聚體主要有3種方式:(1)通過單體β折疊結構之間的相互作用形成二聚體結構;(2)通過單體頭-尾相互作用形成二聚體結構;(3)通過單體一端插入另一單體形成二聚體結構。植物凝集素二聚體結構的穩定性對其發揮生物學功能十分關鍵。
2 菜豆凝集素保守性與動力學分析
菜豆是我們日常生活中的常用食材,我們以菜豆凝集素(PDB code: 3WCR)為例對凝集素二聚體結構的序列保守性和動力學特征進行分析(比對方法為CSBLAST,序列庫為UNIREF90)[33-35]。圖2A為序列保守性投影到其三級結構的結果。序列保守性共分為9個等級,1-3為不保守氨基酸序列(偏藍),7-9為保守氨基酸序列(偏紅)。總體來說,45.4%的氨基酸序列較為保守(7-9),25.8%的氨基酸序列不保守(1-3),大部分β片結構均非常保守,其中4個β片結構(LEU67-TYR72,ASP107-PRO115,HIS160-VAL165和TRP225-THR234)組成了菜豆凝集素三級結構的拓撲骨架,6個β片結構(SER23-PHE29,SER88-ASN96,ALA184-ASP191,LEU196-TYR203,THR208-THR215和ASP245-LEU255)與另一單體的相應結構部分形成三級相互作用,從而形成穩定的二聚體結構。糖結合位點區域的兩個β片結構(THR44-VAL45和LEU51-ARG52)的氨基酸序列不保守,有較多突變發生。菜豆凝集素要形成穩定的二聚體結構才能發揮生物學功能,因此菜豆凝集素三級結構拓撲骨架上的氨基酸序列和二聚體接觸面上的氨基酸序列均非常保守。不同的凝集素蛋白可以特異性結合不同的糖分子,因此糖結合位點附近的氨基酸序列不保守,有較多突變。
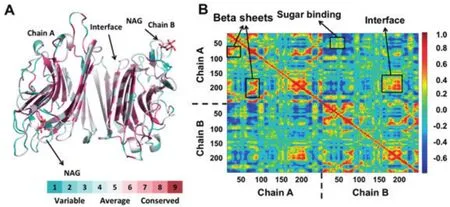
圖2 菜豆凝集素序列保守性和動力學關聯系數分析Fig.2 Lectin sequence conservation and dynamical correlation analysis. (A) Cartoon representation of the lectin dimer structure colored by sequence conservation scores. (B) Dynamical correlation analysis of the motions of lectin dimer obtained from MD simulations.
生物分子靜態結構分析只能反映該分子某一時刻的結構特征,其動力學特征對全面了解結構功能關系有非常重要的幫助。為了更加全面地描述菜豆凝集素分子的生物學特性,我們對菜豆凝集素的二聚體結構進行了分子動力學模擬分析(模擬軟件為GROMACS,力場為Amber03,水模型為TIP3P,模擬時間為50ns,初始結構為菜豆凝集素二聚體結構PDB code:3WCR)[36-38]。動力學網絡中的關聯分析(數值區間為-1~1)可以描述分子在動力學模擬過程中不同結構域或氨基酸殘基之間的協同運動情況,動力學關聯分析為正值表示兩個結構域(或氨基酸殘基)之間有較為緊密的協同運動關系(紅色區域),動力學關聯分析為負值表示兩個結構域之間的運動特征相反(藍色區域),動力學關聯分析為0則表示兩個結構域之間沒有關系(淺綠色區域)。圖2B為動力學關聯分析的結果,數值大于0.7表明氨基酸殘基之間有非常高的運動協同性,主要為同一個二級結構單元中的氨基酸殘基。數值在0.5-0.6之間表明氨基酸殘基之間有較高的運動協同性,例如菜豆凝集素三級結構拓撲骨架的β片結構(LEU37-ARG41,LEU67-TYR72,SER88-ASN96,LEU196-TYR203和THR208-THR215)之間有較高的運動協同性(圖2B),二聚體接觸面上的β片結構(SER23-PHE29,SER88-ASN96,ALA184-ASP191,LEU196-TYR203,THR208-THR215和ASP245-LEU255)之間也有較高的運動協同性(圖2B)。菜豆凝集素需要形成穩定的二聚體結構才能發揮生物學功能,因此動力學模擬中接觸面氨基酸殘基之間的三級相互作用使兩個凝集素單體緊密結合,并協同運動。
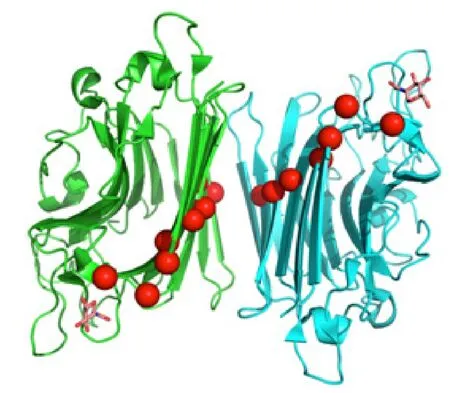
圖3 菜豆凝集素糖分子結合位點間的主要動力學通路Fig.3 Optimal pathway that enforces the correlation between the two sugar binding sitesis shown as the red spheres. These spheres represent the CA atoms of amino acids along the optimal pathway. The two monomers are colored in green and blue. The sugar molecules are colored in orange.
特異性結合糖分子是菜豆凝集素的重要生物學特征,因此我們也著重分析了菜豆凝集素二聚體兩個單體中糖結合位點之間的運動協同性。結果表明,不同單體的糖分子結合位點之間也有較高的運動協同性(圖2B)。我們進一步利用動力學網絡模型分析了不同單體糖分子結合位點之間的動力學通信路徑,兩個結合位點間通過15個氨基酸連接(A:ASN33, A:ARG31, A:PHE29, A:TRP249, A:SER92, A:LEU187, A:ALA199, A:ILE211, B:SER200, B:LEU187, B:SER92, B:TRP249, B:PHE29, B:ARG31, B:ASN33)(圖3)。糖結合位點附近的氨基酸殘基保守性較低,如ASN33(1)和ARG31(4)。動力學通信路徑上的其它氨基酸殘基均較為保守,如A:PHE29(7),A:TRP249(9),A:ALA199(8),B:TRP249(9), B:PHE29(7)等。菜豆凝集素不同單體的糖結合位點氨基酸殘基通過保守的氨基酸通路來保持協同作用,從而發揮相應的生物學功能。
3 植物凝集素藥物設計與醫學應用
植物凝集素可以特異性結合不同的碳水化合物分子,在生物醫學中有廣泛的應用。然而部分含有植物凝集素的食物被食用后會結合器官表皮上的糖分子,從而導致胃腸或肝臟無法正常地代謝蛋白質,并制造有毒副產品。如果食用過多與血型不兼容的植物凝集素,體內毒素會急劇增加。因此,如何設計抑制劑來降低植物凝集素的毒性,進而緩解或者消除植物凝集素引發的癥狀也是近年來研究食品安全等問題的熱點之一。在此,我們也以日常食用的菜豆凝集素為例討論如何設計菜豆凝集素抑制劑來緩解菜豆凝集素產生的毒性癥狀。方法的主要思路為:(1)在目標蛋白質結構中尋找并分析潛在的藥物靶點;(2)結合潛在藥物靶點的結構信息(藥物靶點的空腔體積,表面積,關鍵氨基酸等)篩選或設計藥物。針對菜豆凝集素,我們首先計算分析了單體結構上所有的潛在藥物靶點。圖4中球形結構區域為菜豆凝集素單體中空腔體積最大的三個潛在藥物靶點分布情況,其中藍色區域的空腔體積最大(體積為263.87 ?3),表面積為345.51 ?2;紫色區域的空腔體積次大(體積為230.21?3),表面積為369.47?2;黃色區域的空腔體積為194.62?3,表面積為449.84?2。抑制劑分子若結合在藍色或者紫色區域會影響該區域的局部結構,可能進一步改變菜豆凝集素單體結構,從而降低其二聚體的結合能力。黃色靶點區域在菜豆凝集素二聚體的結合面,抑制劑如果結合在黃色靶點區域,將直接和菜豆凝集素單體發生競爭,從而直接降低菜豆凝集素二聚體的形成效率,降低菜豆凝集素的凝血毒性。目前,我們已根據該黃色區域的結構特征篩選并設計了短肽來抑制菜豆凝集素形成二聚體結構,并已進行了相應的凝血毒性實驗,有較好效果。
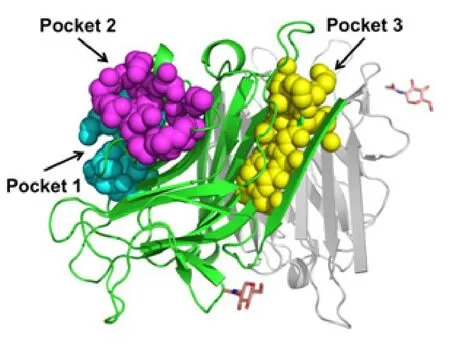
圖4 菜豆凝集素潛在藥物靶點預測結果Fig.4 Sphere representation of the top three pockets of lectin monomer (PDB: 3WCR). The volumes of pocket 1 (colored in blue), pocket 2 (colored in purple), and pocket 3 (colored in yellow) are 263.87 ?3,230.21?3, and 194.62 ?3, respectively. The surface areas of the three pockets are 345.51 ?2, 369.47 ?2, 449.84 ?2, respectively.
4 結 論
本文以計算生物學的視角并結合菜豆凝集素實例,從植物凝集素的三級結構、序列保守性、動力學特征和抑制劑設計等方面討論了植物凝集素的研究進展及藥物設計的可能性。
1)目前蛋白質結構數據庫中的植物凝集素主要可分為12個較大的結構類型,大多數植物凝集素都形成二聚體或者多聚體結構。
2)菜豆凝集素序列保守性分析表明二聚體接觸面上的氨基酸較為保守(形成穩定的二聚體結構),糖結合位點的氨基酸差異性較大(特異性結合糖分子)。動力學關聯分析表明二聚體接觸面氨基酸,不同糖分子結合位點的氨基酸均有較好的運動協同性。
3)以菜豆凝集素為例設計短肽抑制劑來降低菜豆凝集素的凝血毒性,討論了植物凝集素藥物設計的可行性與可操作性。
隨著計算生物學的不斷發展,利用植物凝集素的生物學特性并結合計算生物學的優勢,將極大促進植物凝集素抗蟲,抗凝血,抗病等的應用推廣,為植物凝集素應用研究開辟新的思路。
(References)
[1] BanerjeeN, Sengupta S, Roy A, et al.Functional alteration of a dimeric insecticidal lectin to a monomeric antifungal protein correlated to its oligomericstatus[J]. PLoS One, 2011, 6(4): e18593.
[2]MalikA, Lee J, Lee J. Community-based network study of protein-carbohydrate interactions in plant lectins using glycan array data[J]. PLoS One, 2014, 9(4): p. e95480.
[3]Elgavish S, ShaananB. Chemical characteristics of dimer interfaces in the legume lectinfamily[J]. Protein Sci, 2001, 10(4): 753-761.
[4]Loris R, Hamelryck T, BouckaertJ ,et al.Legume lectin structure[J]. BiochimBiophysActa, 1998, 1383(1): 9-36.
[5]Plattner VE, Wagner M, Ratzinger G,et al. Targeted drug delivery: binding and uptake of plant lectins using human 5637 bladder cancer cells[J]. Eur J Pharm Biopharm, 2008, 70(2):572-576.
[6]Wirth M, Gerhardt K, Wurm C,et al. Lectin-mediated drug delivery: influence of mucin on cytoadhesion of plant lectins in vitro[J]. J Control Release, 2002, 79(1-3):183-191.
[7]Wang XY, Koller R, Wirth M,et al. Lectin-Grafted PLGA Microcarriers Loaded with Fluorescent Model Drugs: Characteristics, Release Profi les, and CytoadhesionStudies[J]. Sci Pharm, 2014, 82(1): 193-205.
[8] Badari N AR, Sivaramakrishna A, Marimuthu KM, et al.A comparative study of phytohaemagglutinin and extract of Phaseolus vulgaris seeds by characterization and cytogenetics[J]. SpectrochimActa A MolBiomolSpectrosc, 2015, 134: 143-137.
[9]JafféWG. Hemagglutinins, in Toxic Constituents of Plant Foodstuffs[M]. Academic Press, 1969: 69-101.
[10]Jorgensen, WL. Foundations of biomolecularmodeling[J]. Cell, 2013, 155(6): 1199-1202.
[11] CuiQ, NussinovR. Making biomolecular simulations accessible in the post-Nobel Prize era[J]. PLoSComputBiol, 2014. 10(8):e1003786.
[12]ZhaoY, GongZ, Xiao Y.Improvements of the hierarchical approach for predicting RNA tertiary structure[J]. J BiomolStructDyn, 2011, 28(5): 815-826.
[13]Zhao Y, Huang Y, Gong Z, et al. Automated and fast building of three-dimensional RNA structures[J]. Sci Rep, 2012, 2: 734.
[14]Zhao Y, Zeng C, Tarasova NI, et al. A new role for STAT3as a regulator of chromatintopology[J]. Transcription, 2013, 4(5): 227-231.
[15]Chen H, Zhao Y, Li H,et al. Break CDK2/Cyclin E1 interface allosterically with small peptides[J]. PLoS One,2014, 9(10): e109154.
[16]Du C, Chen H, Zhao Y, et al.How Far and How Fast Can One Move on Neutral Network?[J]. Journal of Theoretical & Computational Chemistry, 2013, 12(8): 1341010.
[17]Wang J, Zhao Y, Zhu C, et al.3dRNAscore: a distance and torsion angle dependent evaluation function of 3D RNA structures[J]. Nucleic Acids Res, 2015, 2: gkv141.
[18] GuptaGS. Vishveshwara, SuroliaA. Stability of dimeric interface in banana lectin: Insight from molecular dynamics simulations[J]. IUBMB Life, 2009, 61(3): 252-260.
[19] GuptaGS. Sinha, SuroliaA. Unfolding energetics and stability of banana lectin[J]. Proteins, 2008, 72(2): 754-760.
[20]KaushikS, Mohanty D, Surolia A.Role of glycosylation in structure and stability of Erythrinacorallodendronlectin (EcorL): a molecular dynamics study[J]. Protein Sci, 2011, 20(3): 465-481.
[21]Berman HM, Westbrook J, FengZ et al., The Protein Data Bank[J]. Nucleic Acids Res, 2000. 28(1): 235-42.
[22] Van DEJ. History of plant lectin research, in Lectins[M]. Springer, 2014:3-13.
[23] Nagae M, Soga K, Morita-Matsumoto K, et al., Phytohemagglutinin from Phaseolus vulgaris (PHA-E) displays a novel glycan recognition mode using a common legume lectinfold[J]. Glycobiology, 2014. 24(4): 368-78.
[24] Carrizo ME, Capaldi S, Perduca M, et al., The Antineoplastic Lectin of the Common Edible Mushroom (Agaricusbisporus) Has Two Binding Sites, Each Specific for a Different Configuration at a Single EpimericHydroxyl[J]. Journal of Biological Chemistry, 2005, 280(11): 10614-10623.
[25]Transue TR, Smith AK, Mo H, et al., Structure of benzyl T-antigen disaccharide bound to Amaranthuscaudatusaggl utinin[J]. Nat StructBiol, 1997. 4(10): 779-83.
[26] Barrientos LG, Louis JM, Botos I, et al., The Domain-Swapped Dimer of Cyanovirin-N Is in a Metastable Folded State[J]. Structure. 10(5): 673-686.
[27] Hester G, Kaku H, Goldstein JI, et al., Structure of mannose-specific snowdrop (Galanthusnivalis) lectin is representative of a new plant lectinfamily[J]. Nature Structural Biology, 1995.2: 472-479.
[28] Pascal JM, Day PJ, Monzingo AF, et al., 2.8-A crystal structure of a nontoxic type-II ribosome-inactivating protein, ebulinl[J]. Proteins, 2001. 43(3): 319-26.
[29] Bourne Y, Zamboni V, Barre A, et al., Helianthus tuberosuslectin reveals a widespread scaffold for mannosebinding lectins[J]. Structure, 1999. 7(12): 1473-82.
[30] Liu T, Liu Z, Song C, et al., Chitin-induced dimerization activates a plant immune receptor[J]. Science, 2012. 336(6085): 1160-4.
[31]Andersen NH, Cao B, Rodriguez-Romero A, et al., Hevein: NMR assignment and assessment of solution-state folding for the agglutinin-toxin motif[J]. Biochemistry, 1993. 32(6): 1407-22.
[32] Van DEJ, Culerrier R, Barre A, et al., A Novel Family of Lectins Evolutionarily Related to Class V Chitinases: An Example of Neofunctionalization in Legumes[J]. Plant Physiology, 2007. 144(2): 662-672.
[33] UniProt, C., The Universal Protein Resource (UniProt) in 2010[J]. Nucleic Acids Res, 2010. 38(Database issue): D142-8.
[34]Angermuller, C, A. Biegert, and J. Soding, Discriminative modelling of context-specific amino acid substitution probabilities[J]. Bioinformatics, 2012. 28(24): 3240-7.
[35]Biegert, A. and J. Soding, Sequence context-specific profiles for homology searching[J]. ProcNatlAcadSci U S A, 2009. 106(10): 3770-5.
[36] Duan Y, Wu C, Chowdhury S, et al., A point-charge force field for molecular mechanics simulations of proteins based on condensed-phase quantum mechanical calculations[J]. J ComputChem, 2003. 24(16): 1999-2012.
[37]Mahoney, M.W. and W.L. Jorgensen, A five-site model for liquid water and the reproduction of the density anomaly by rigid, nonpolarizable potential functions[J]. Journal of Chemical Physics, 2000. 112(20): 8910-8922.
[38] Pronk S, Pall S, Schulz R, et al., GROMACS 4.5: a highthroughput and highly parallel open source molecular simulation toolkit[J]. Bioinformatics, 2013. 29(7): p. 845-54.
Plant lectin protein structural classifi cation and drug design
ZHAO Yunjie1,2, LIU Zhichao1, LIU Qin2, CHEN Gao2, CHEN Chanyou2, ZENG Chen1,2
1 Department of Physics, The George Washington University, Washington DC, 20052, USA 2 School of Life Sciences, Jianghan University, Wuhan 430056, China
Plant lectinis a specifi c carbohydrate-bindingor sugar-binding protein which wasfi rst discoveredin the 19th century.Itshigh specifi city in carbohydratebindingmakes it a useful molecular tool for different biological applications. For example,plant lectin’s ability to target sugar or carbohydrate in the gastrointestinaltract of insects provides a defense mechanism against pests. Lectin protein can also bind to the membrane of cancer cellsinducing apoptosis or inhibiting growth.Previous studies showed that there are over 400,000 estimated binding sites for kidney bean lectins alone. Structural information of plant lectinsisthus essentialfor understanding their biological functions and applications.Here we report some of the most prevalenttertiary structures of plant lectins, and further probed the dynamical properties of kidney bean lectin via molecular dynamics simulations. Thestructural and dynamical information may shed some insights in lectin protein engineering and drug design for industrial and medical applications.
Plant lectin; tertiary structure; evolution; drug design; medical application
R730.58
A doi 10.11966/j.issn.2095-994X.2015.01.01.12
2015-02-24;
2015-03-21
武漢市黃鶴英才項目
趙蘊杰,博士后研究員,研究方向為生物物理學,電子信箱:yjzhao.wh@gmail.com;陳禪友(通信作者),教授,研究方向為植物遺傳改良和利用,電子郵箱:ccy@jhun.edu.cn;曾辰(通信作者),教授,研究方向為生物物理學,電子信箱:chenz@gwu.edu
引用格式:趙蘊杰,劉志超,劉琴,等.大數據癌癥風險預測系統[J].世界復合醫學, 2015 , 1(1): 68-73.