我國葡萄酒中布魯塞爾酒香酵母的檢測和鑒定
曹培鑫,馬 濤,楊凱迪,劉延琳,2,*
(1.西北農林科技大學葡萄酒學院,陜西 楊凌 712100;2.陜西省葡萄與葡萄酒工程技術研究中心,陜西 楊凌 712100)
我國葡萄酒中布魯塞爾酒香酵母的檢測和鑒定
曹培鑫1,馬濤1,楊凱迪1,劉延琳1,2,*
(1.西北農林科技大學葡萄酒學院,陜西 楊凌712100;2.陜西省葡萄與葡萄酒工程技術研究中心,陜西 楊凌712100)
為快速且準確地檢測葡萄酒中布魯塞爾酒香酵母的存在情況,本研究比較了酒香酵母鑒別培養基(Dekkera bruxellensis differential medium,DBDM)和WLN(wallersteins laboratory nurtrient)培養基分離檢測布魯塞爾酒香酵母的效果,并利用特異聚合酶鏈式反應(polymerase chain reaction,PCR)擴增法對上述兩種培養基的分離檢測效果作了進一步驗證。結果表明,上述兩種培養基均能對葡萄酒中的布魯塞爾酒香酵母進行分離鑒別,而DBDM培養基較少出現假陽性菌落,具有更好的選擇性,結合特異PCR擴增可準確鑒定布魯塞爾酒香酵母。此外,利用DBDM培養基結合特異PCR擴增法對國內5 個主要葡萄酒產區的酒樣進行布魯塞爾酒香酵母感染情況的調查發現,河北、山東和吉林的葡萄酒中存在布魯塞爾酒香酵母感染的情況,尤其是在橡木桶中陳釀的葡萄酒。因此,國內葡萄酒行業應對酒香酵母感染問題足夠重視。
葡萄酒;布魯塞爾酒香酵母;鑒別培養基;檢測鑒定
酒香酵母屬(Brettanomyces/Dekkera)于1904年被首次提出,最先是在啤酒中發現的,后來不斷在葡萄酒中發現。目前公認的分類中,酒香酵母屬有以下5 個種:Brettanomyces custersianus、Brettanomyces naardenensis、Brettanomyces nanus、Dekkera anomalus以及Dekkera bruxellensis。關于酒香酵母屬的名稱,經常會有Brettanomyces和Dekkera兩種方法表示,最新的分類命名法對Dekkera anomalu和Dekkera b ruxellensis兩種能夠產生孢子的酒香酵母用Dekkera表示,其他3 種不能產生孢子的用Brettanomyces表示[1]。由于其分類命名的變更,很多報道中依然采用了Brettanomyces的形式。本實驗全部采用最新的命名方式,即以Dekkera bruxellensis來表示布魯塞爾酒香酵母。
Dekkera bruxellensis是葡萄酒中最常發現 的酒香酵母,存在于葡萄和葡萄酒、釀酒設備以及橡木桶中。其會利用葡萄酒中固有的對羥基肉桂酸,如阿魏酸和對香豆酸等,在羥基肉桂酸脫羧酶作用下產生4-乙烯基苯酚和4-乙烯基愈創木酚,進一步在乙烯基還原酶的作用下轉化為4-乙基苯酚和4-乙基愈創木酚等對葡萄酒風味影響極大的代謝產物[2-3]。目前在世界各地的葡萄酒產區中都分離檢測到Dekkera bruxellensis的存在[4-10]。酒香酵母在葡萄酒中存在時,合適的 條件下會給葡萄酒帶來煙熏、香料以及動物香 等香氣,增加葡萄酒香氣的復雜性,然而如果任由其在葡萄酒中存在不加處理,其產生的過高濃度的4-乙基苯酚和4-乙基愈創木酚會帶來創可貼、鼠尿、馬汗等不良風味,使葡萄酒的質量受到影響,引起消費者的抗拒,從而造成酒廠的經濟損失[11-12]。酒香酵母相對于釀酒酵母對SO2以及酒精的耐受性更強,通常的葡萄酒處理手段很難完全將其除去,因此,對葡萄酒中酒香酵母的檢測極為重要。目前對葡萄酒中Dekkera bruxellensis的檢測,傳統方法是利用鑒別培養基分離檢測,常用的鑒別培養基是添加放線菌酮的WLN(wallersteins laboratory nurtrient)培養基[7]和DBDM(Dekkera bruxellensis differential medium)培養基[13]。Dekkera bruxellensis在葡萄酒中有時會呈現一種存活但不可培養的狀態(viable but not culturable,VBNC)[14-16],在此狀態下其生理代謝仍會進行,但處于一種不生長繁殖的狀態,直接涂布平板其不能生長,出現假陰性的檢測結果,因此,在涂布平板檢測時需要對酒樣進行預處理,使其脫離VBNC狀態。形態觀察并不能夠準確鑒定,需要結合其他手段進行鑒定。因此,對酒香酵母的分子鑒定和檢測技術也有越來越多的研究,包括早期的26S rRNA基因D1~D2區測序[17],對5.8S-ITS-18S區的聚合酶鏈式反應-限制性片段長度多態性(polymerase chain reactionrestriction fragment length polymorphism,PCR-RFLP)分析[18],這兩種方法需要對擴增片段進行測序或者酶切分析,操作相對復雜且用時較長,而針對Dekkera bruxellensis設計的特異性PCR擴增鑒定[4,19-20]可以較快地完成鑒定。另外還有熒光原位交技術[21]、巢式PCR[10]以及實時定量PCR[5,7,22]等技術也逐漸用于對Dekkera bruxellensis的檢測和鑒定,但對設備條件要求較高。
相比于國際葡萄酒行業對Dekkera bruxellensis在葡萄酒發酵和貯存過程中發揮的作用和可能帶來的風險的關注,國內葡萄酒行業對其還沒有足夠的重視,對于葡萄酒中Dekkera bruxellensis快速檢測方法的開發以及其防范和治理等,都需要深入的研究。針對這一現狀,本研究比較WLN和DBDM培養基分離檢測Dekkera bruxellensis的效果,并利用特異PCR擴增法對上述兩種培養基的分離檢測效果作了進一步驗證。并選用分離鑒別Dekkera bruxellensis效果較好的DBDM培養基結合特異PCR擴增法對我國不同產區的酒樣進行Dekkera bruxellensis的分離檢測,調查我國葡萄酒中該酵母的感染情況。本研究為Dekkera bruxellensis的分離鑒定以及檢測提供技術支撐,并為我國葡萄酒行業酒香酵母的存在情況提供理論依據,對國內Dekkera bruxellensis的進一步研究將發揮重要作用。
1 材料與方法
1.1材料
1.1.1酒樣來源
從國內主要的葡萄酒產區采集葡萄酒樣品,取樣為酒精發酵剛結束的發酵罐中的葡萄酒和正在儲酒池、儲酒罐以及橡木桶中陳釀的葡萄酒,陳釀的葡萄酒選擇不同年份、品種以及不同儲酒容器進行取樣,盡量從容器底部取樣,每個葡萄酒樣品取樣量為500 mL,其他信息見表1。
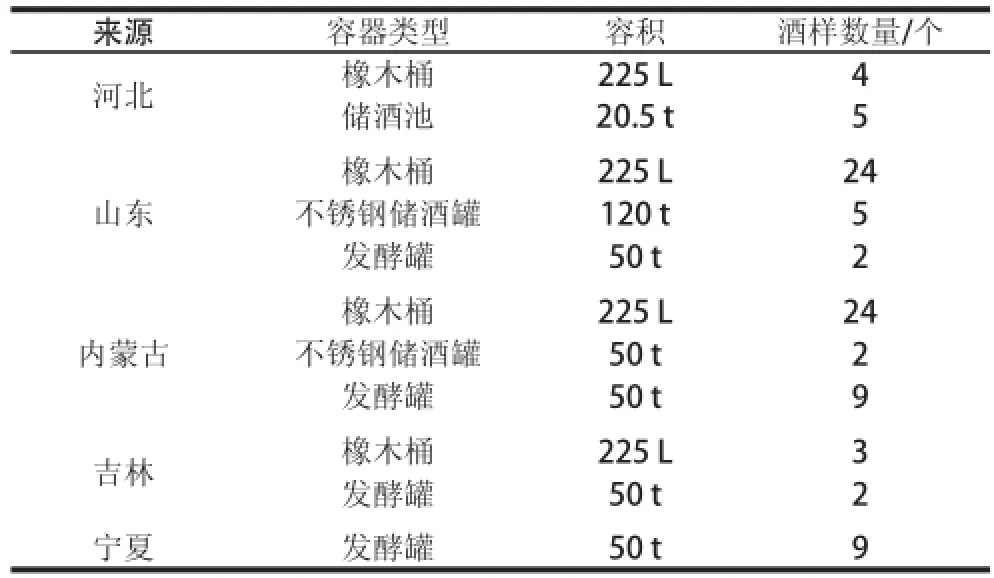
表1 本研究所用葡萄酒樣品來源Table 1 Wine samples from different areas in China used in this study
1.1.2對照菌株
實驗中所用菌株如表2所示,UCD系列菌株為加利福尼亞大學戴維斯分校的菌株保藏中心(Culture Collection,University of California,Davis)提供的Dekkera bruxellensis標準菌株。其余為本實驗室分離篩選鑒定并保藏的菌株。

表2 不同種屬酵母菌株Table 2 Yeast strains belonging to different genera
1.1.3培養基和試劑
YPD液體培養基:酵母浸粉10 g/L、蛋白胨20 g/L、葡萄糖20 g/L,121 ℃滅菌20 min。
DBDM培養基[13]:無氨基酵母氮源(yeast nitrogen base without amino acids,YNB)6.7 g/L、乙醇體積分數6%、氯霉素10 mg/L、放線菌酮10 mg/L、對香豆酸10 mg/L、溴甲酚綠22 mg/L、瓊脂20 mg/L,山梨酸調pH值至5.4,瓊脂高溫滅菌,其他過濾除菌。
WLN培養基[23]:葡萄糖50 g/L、蛋白胨5 g/L、酵母浸粉4 g/L、KH2PO40.55 g/L、KCl 0.425 g/L、CaCl20.125 g/L、MgSO40.125 g/L、FeCl30.025 g/L、MnSO40.025 g/L、溴甲酚綠22 mg/L、瓊脂20 g/L,調pH值至6.2。另外添加50 mg/L放線菌酮。
PCR試劑:10×PCR buffer,2.5 mmol/L dNTP,25 mmol/L MgCl2,5 U/μL Taq Polymerase,均購自寶生物公司。引物濃度為10 μmol/L,由生工生物工程(上海)股份有限公司合成。
1.2方法
1.2.1酒樣預處理
由于SO2以及酒精的脅迫,Dekkera bruxellensis在葡萄酒中可能會以存活但不可培養的狀態存在,直接涂布不能在培養基上生長,因此先對酒樣進行預處理,將1 mL酒樣接在5 mL YPD液體培養基(含10 mg/L放線菌酮,以抑制其他酵母的生長)中,28 ℃條件下靜置活化24~48 h。
1.2.2兩種鑒別培養基比較
以河北的9 個酒樣(酒樣編號為CC1~CC9)涂布兩種鑒別培養基,比較其分離檢測效果。取100 μL原酒樣品和經預處理活化后的酒樣,分別涂布WLN和DBDM培養基,28 ℃條件下培養7~20 d,觀察菌落形成情況。
1.2.3DNA的提取
采用石英砂破壁提取法[24]。YEPD平板培養基上培養2~3 d;1.5 mL離心管,用滅過菌的牙簽挑取0.3 g菌體,加入3/10總體系體積的石英砂,加200 μL酵母裂解液,在勻漿機振蕩,每2 min取下搖30 s,共循環4 次;65 ℃水浴10 min。之后加入等體積(200 μL)5 mol/L CH3COOK,混勻,冰浴8 min;13 300 r/min離心5 min,吸350 μL上清液至新的離心管中;加入0.1 倍體積(35 μL)3 mol/L CH3COONa,加入0.6 倍體積異丙醇(200 μL),混勻,后冰浴8 min,13 300 r/min離心5 min后收集沉淀;200 μL TE溶解,加入RNase 3 μL,65 ℃水浴10 min;加入200 μL氯仿-異戊醇(24∶1,V/V),抽提一次,12 000 r/min離心10 min;小心取上清液150 μL至新的離心管中,加入0.1 倍體積(20 μL)3 mol/L CH3COONa及2.5 倍體積(375 μL)無水乙醇,混勻后13 300 r/min離心8 min,收集沉淀DNA;500 μL 70%乙醇洗滌沉淀,13 300 r/min離心8 min收集沉淀,吹干后加50 μL TE溶解,-20 ℃保藏(長期保存)。
1.2.4特異引物PCR擴增鑒定
利用Cecchini等[4]以Dekkera bruxellensis ITS1~ITS2區為靶序列設計的特異引物對,DekITS(5'-GACACGTGGAATAAGCAAGG-3')和BruxITR(5'-ATTATCCCCTCACTCCCCTC-3'),擴增片段長度為308 bp。PCR反應體系:2.5 μL 10×PCR buffer,1.0 μL dNTP,引物各0.5 μL,1.5 μL MgCl2,0.25 μL Taq Polymerase,1 μL DNA模板,17.95 μL ddH2O,總體積25 μL。反應條件:95 ℃預變性4 min;95 ℃變性30 s,60 ℃退火30 s,70 ℃延伸30 s,循環36 次;70 ℃保溫10 min。擴增產物經10 g/L瓊脂糖凝膠電泳檢測(100 V,1 h),后置于凝膠成像儀內拍照觀察。
1.2.526S rRNA基因D1~D2區測序驗證
對特異PCR擴增為陽性的菌落進一步進行26S rRNA基因D1~D2區測序,驗證其為Dekkera bruxellensis。使用通用引物NL1(5'-GCATATCAATAAGCGGAGGAAAAG-3')和NL4(5'-GGTCCGTGTTTCAAGACGG-3')進行26S rRNA基因D1/D2區的擴增[17],PCR反應體系:5.0 μL 10×PCR buffer,1.6 μL dNTP,引物各1 μL,3.0 μL MgCl2,0.5 μL Taq Polymerase,1 μL DNA模板,36.9 μL ddH2O,總體積50 μL。PCR擴增程序為:95 ℃預變性5 min;94 ℃變性1 min,52 ℃退火1 min,72 ℃延伸1 min 20 s,循環36 次;72 ℃保溫8 min。擴增產物經10 g/L瓊脂糖凝膠檢測后,送生工生物工程(上海)股份有限公司進行純化和測序分析。
1.2.6不同產區葡萄酒中酒香酵母的分離檢測
采用分離檢測效果較好的鑒別培養基結合特異PCR擴增對其他地區的酒樣進行Dekkera bruxellensis分離檢測。
2 結果與分析
2.1兩種鑒別培養基的比較

表3 鑒別培養基上菌落形成情況Table 3 Colony formation on differential media
9 個酒樣活化前后分別在WLN培養基和DBDM培養基上進行涂布檢測,菌落形成情況見表3。在WLN培養基上,未經活化的酒樣有6 個樣品出現菌落,經YPD活化后,原本沒有菌落形成的CC6酒樣也有菌落形成,共7 個酒樣檢測到菌落形成。在WLN培養基上,共出現了4 種不同形態的菌落:ⅠW,奶油狀,白色圓形突起,開始白色,后來稍帶藍綠色;ⅡW,奶油狀,深綠色,扁平圓形菌落;ⅢW,不規則較大菌落,扁平,表面有褶皺,藍綠色;ⅣW,油狀,稍突起,黃綠色圓形菌落(圖1)。未經活化的酒樣有5 個樣品在DBDM培養基上形成菌落,活化后,原本沒有菌落形成的CC6酒樣有菌落形成。DBDM培養基上有兩種形態的菌落,ⅠD,奶油狀,純白色圓形突起;ⅡD,奶油狀,黃綠色圓形菌落。與Dekkera bruxellensis標準菌株形態對比,初步斷定ⅠW和ⅠD菌株為Dekkera bruxellensis。

圖1 鑒別培養基上的菌落形態Fig.1 Colony types on differential media
Benito等[25]比較鑒別培養基不同選擇因子的作用時,發現放線菌酮是最強的選擇因子,但并不能完全抑制其他酵母的生長,需要結合山梨酸、酒精等次級選擇因子才能更具選擇性。Morneau等[23]比較15 種不同酵母菌在WLN和DBDM培養基上的生長情況,發現在WLN培養基中放線菌酮達到100 mg/L時,除了Dekkera bruxellensis還有3 種酵母能夠生長,而在DBDM培養基上則只有Dekkera bruxellensis能夠生長。實驗各酒樣菌落分布如表4所示,結果與之前的這些結論基本一致。WLN培養基成分營養豐富,雖然其添加了更高質量濃度的放線菌酮以抑制其他酵母的生長,但由于選擇因素單一,部分其他酵母對放線菌酮存在抗性,出現多種形態的菌落,對分離檢測結果的觀察造成影響;而DBDM培養基除了有相對較低質量濃度的放線菌酮作為主要選擇因子,還有山梨酸和酒精作為次級選擇因子,并且不含有葡萄糖等常規性碳源,其選擇能力較高,實驗中出現其他菌落較少。相比于WLN培養基,DBDM培養基是更為理想的分離檢測Dekkera bruxellensis的鑒別培養基。
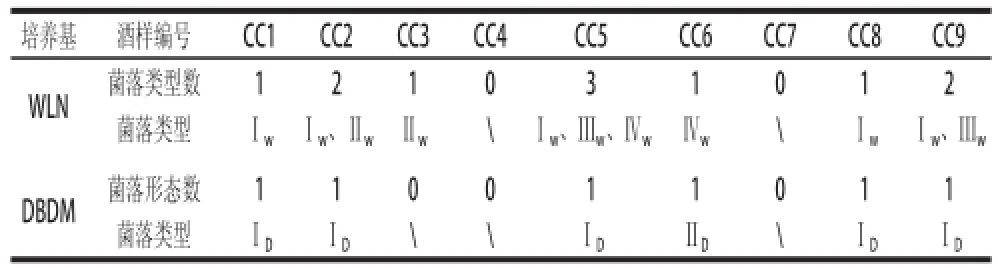
表4 每個酒樣在鑒別培養基上的菌落形態Table 4 Strain types from each wine sample on differential media
2.2PCR特異性擴增和26S rRNA基因D1~D2區測序鑒定
選擇培養基雖然能夠在形態上對菌落進行初步判定,但由于培養條件、菌株差異等因素,會使菌落在不同培養時期甚至同一時期內菌種內不同菌株的形態表現不同,因此,只根據菌落形態難以準確對Dekkera bruxellensis進行鑒定和檢測,需要結合其他方法進一步鑒定。
2.2.1PCR特異性擴增鑒定
Cecchini等[4]利用17 種酵母菌和6 種細菌驗證了該引物的特異性,實驗中進一步用對實驗室之前研究發現的葡萄和葡萄酒中最為常見的8 種酵母以及3 株Dekkera bruxellensis標準菌株驗證其特異性,結果如圖2所示,只有Dekkera bruxellensis的3 株菌有目的片段,其余酵母均未擴增出條帶,結合實驗和Cecchini等[4]報道可以確定該引物對特異性良好。
對WLN培養基上4 種形態的菌落和DBDM培養基上2 種形態的菌落進行特異引物PCR擴增鑒定,其結果如圖2所示,只有符合Dekkera bruxellensis形態特征的ⅠW和ⅠD型菌落擴增出與目的片段大小相符的條帶,其他形態的菌落均未有條帶,因此可以判斷ⅠW和ⅠD形態的菌落為Dekkera bruxellensis。為進一步驗證,分別從CC1、CC2、CC5、CC8、CC9挑選一株該形態菌株進行擴增,如圖3所示,均有目的擴增片段。表明DBMD培養基和WLN培養基能夠較為準確地對葡萄酒中的Dekkera bruxellensis進行檢測;這5 個酒樣中均存在Dekkera bruxellensis。

圖2 不同酵母PCR擴增結果Fig.2 PCR amplification of different yeast species

圖3 實驗菌株PCR擴增結果Fig.3 PCR amplification of strains isolated from wine samples
2.2.226S rRNA基因D1~D2區測序
為進一步驗證,分別從5 個酒樣的WLN和DBDM培養基上選取一株特異PCR擴增陽性的菌株,進行26S rRNA基因D1~D2區進行測序分析,測得的序列在國家生物技術信息中心(National Center for Biotechnology Information,NCBI)上進行對比,結果見表5,表明其確實為Dekkera bruxellensis。
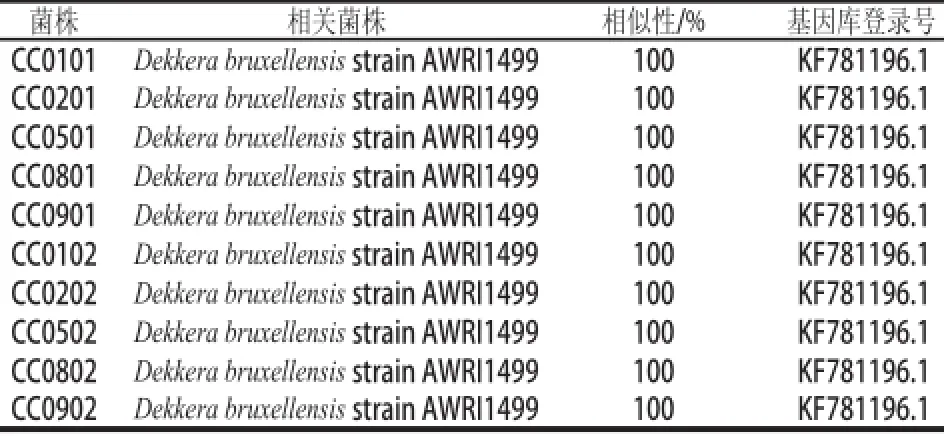
表5 被測菌株26S rRNA基因D1~D2區與相關菌株序列相似性Table 5 The identity of 26S rRNA gene D1/D2 fragment between the sequenced strains and related strains
26S rRNA基因D1~D2區測序結果表明,鑒別培養基結合PCR特異性擴增可以準確地對Dekkera bruxellensis進行鑒定和檢測,可以替代操作相對復雜、用時較長的26S rRNA基因D1~D2測序和5.8S-ITS-18S rRNA基因的PCR-RFLP方法。
2.3不同產區葡萄酒中酒香酵母的分離檢測
利用上述分離檢測效果較好的DBDM培養基,對另外4 個地區的酒樣進行分離檢測,并對篩選得到的菌落進一步進行PCR特異性擴增鑒定。綜合檢測結果見表6,在研究的5 個地區中,山東、河北和吉林的酒樣中存在酒香酵母,分別有6、5、3 個酒樣被感染,山東與吉林9 個酒樣的PCR特異擴增結果見圖4。內蒙古的35 個酒樣中均未檢測到Dekkera bruxellensis的存在,酒樣采集相對豐富,可以確認沒有感染;而寧夏的酒樣雖然此實驗中沒有分離到Dekkera bruxellensis,但由于樣品量少,且酒樣都是來自于較少出現Dekkera bruxellensis的發酵罐,難以說明其不存在感染。實驗發現存在Dekkera bruxellensis的酒樣均為陳釀期間的葡萄酒,尤其是橡木桶,這與之前報道的酒香酵母大多存在于陳釀期間結果一致,原因是酒香酵母生長相對較慢,抗逆性強,在陳釀期間沒有其他酵母的競爭以及其可以利用橡木桶的纖維二糖作為碳源,在此時期內會慢慢繁殖[26];部分研究人員也在葡萄和發酵結束的葡萄酒中分離到Dekkera bruxellensis[1],但此實驗中在剛發酵完的葡萄酒中沒有分離檢測到其存在,可能并不存在或者是由于其數量較少沒有分離到。

Table 6 Contamination of Dekkera bruxellensisnsis in wines from different areas表6 不同地區葡萄酒中Dekkera bruxellensis感染情況

圖4 山東和吉林酒樣分離菌株PCR擴增結果Fig.4 PCR amplification of strains isolated from wine samples produced in Shandong and Jilin
3 結 論
WLN培養基和DBDM培養基均能對葡萄酒中的布魯塞爾酒香酵母進行初步分離和鑒定,根據葡萄酒樣品在兩種培養基上形成的菌落形態,可以初步判斷葡萄酒中是否存在布魯塞爾酒香酵母。與WLN培養基相比,DBDM培養基有更好的選擇性,較少出現假陽性菌落,分離檢測效果相對較好,結合特異引物PCR能夠準確地對Dekkera bruxellensis進行鑒定。
在調查的5 個國內主要葡萄酒產區的葡萄酒樣品中,河北、山東和吉林產區的葡萄酒均檢測到Dekkera bruxellensis的存在,尤其是陳釀在橡木桶中的葡萄酒,調查中發現感染Dekkera bruxellensis的酒樣大部分為在橡木桶中陳釀的葡萄酒。國內葡萄酒行業應對酒香酵母感染問題足夠重視,以避免其可能帶來的風險。
[1]OELOFSE A, PRETORIUS I, du TOIT M. Significance of Brettanomyces and Dekkera during winemaking: a synoptic review[J]. South African Journal for Enology and Viticulture, 2008, 19(2): 128-144.
[2]CHATONNET P, DUBOURDIE D, BOIDRON J N, et al. The origin of ethylphenols in wines[J]. Journal of the Science of Food and Agriculture, 1992, 60(2): 165-178.
[3]CHATONNET P, VIALA C, DUBOURDIEU D. Influence of polyphenolic components of red wines on the microbial synthesis of volatile phenols[J]. American Journal of Enology and Viticulture,1997, 48(4): 443-448.
[4]CECCHINI F, IACUMIN L, FONTANOT M, et al. Dot blot and PCR for Brettanomyces bruxellensis detection in red wine[J]. Food Control,2013, 34(1): 40-46.
[5]TOFALO R, SCHIRONE M, CORSETTI A, et al. Detection of Brettanomyces spp. in red wines using real-time PCR[J]. Journal of Food Science, 2012, 77(9): M545- M549.
[6]PUIG A, BERTRAN E, FRANQUET R, et al. Brettanomyces bruxellensis prevalence in wines produced and marketed in Spain[J]. Annals of Microbiology, 2011, 61(1): 145-151.
[7]TESSONNIERE H, VIDAL S, BARNAVON L, et al. Design and performance testing of a real-time PCR assay for sensitive and reliable direct quantification of Brettanomyces in wine[J]. International Journal of Food Microbiology, 2009, 129(3): 237-243.
[8]AGNOLUCCI M, VIGENTINI I, CAPURSO G, et al. Genetic diversity and physiological traits of Brettanomyces bruxellensis strains isolated from Tuscan Sangiovese wines[J]. International Journal of Food Microbiology, 2009, 130(3): 238-244.
[9]RODER C, KONIG H, FROHLICH J. Species-specific identification of Dekkera/Brettanomyces yeasts by fluorescently labeled DNA probes targeting the 26S rRNA[J]. FEMS Yeast Research, 2007, 7(6): 1013-1026.
[10] IBEAS J I, LOZANO I, PERDIGONES F, et al. Detection of Dekkera-Brettanomyces strains in sherry by a nested PCR method[J]. Applied and Environmental Microbiology, 1996, 62(3): 998-1003.
[11] SU.REZ R, SU.REZ-LEPE J A, MORATA A, et al. The production of ethylphenols in wine by yeasts of the genera Brettanomyces and Dekkera: a review[J]. Food Chemistry, 2007, 102(1): 10-21.
[12] 游雪燕, 莊海寧, 馮濤. 葡萄酒中Brettanomyces酒香酵母屬不良風味的研究進展[J]. 中國釀造, 2012, 31(12): 9-12.
[13] RODRIGUES F, GONCALVES G, PEREIRA-da-SILVA SMALFEITO-FERREIRA M, et al. Development and use of a new medium to detect yeasts of the genera Dekkera/Brettanomyces[J]. Journal of Applied Microbiology, 2001, 90(4): 588-599.
[14] AGNOLUCCI M, REA F, SBRANA C, et al. Sulphur dioxide affects culturability and volatile phenol production by Brettanomyces/Dekkera bruxellensis[J]. International Journal of Food Microbiology, 2010,143(1/2): 76-80.
[15] SERPAGGI V, REMIZE F, RECORBET G, et al. Characterization of the “viable but nonculturable” (VBNC) state in the wine spoilage yeast Brettanomyces[J]. Food Microbiology, 2012, 30(2): 438-447.
[16] ZUEHLKE J M, EDWARDS C G. Impact of sulfur dioxide and temperature on culturability and viability of Brettanomyces bruxellensis in wine[J]. Journal of Food Protection, 2013, 76(12):2024-2030.
[17] KURTZMAN C P, ROBNETT C J. Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit (26S)ribosomal DNA partial sequences[J]. Antonie van Leeuwenhoek,1998, 73(4): 331-371.
[18] ESTEVE-ZARZOSO B, BELLOCH C, URUBURU F, et al. Identification of yeasts by RFLP analysis of the 5.8S rRNA gene and the two ribosomal internal transcribed spacers[J]. International Journal of Systematic Bacteriology, 1999, 49(1): 329-337.
[19] COCOLIN L, RANTSIOU K, IACUMIN L, et al. Molecular detection and identification of Brettanomyces/Dekkera bruxellensis and Brettanomyces/Dekkera anomalus in spoiled wines[J]. Applied and Environmental Microbiology, 2004, 70(3): 1347-1355.
[20] CONTRERAS A, SALINAS F, GANGA A, et al. Polymerase chain reaction confirmatory method for microbiological detection of Brettanomyces bruxellensis in wines[J]. Journal of Rapid Methods & Automation in Microbiology, 2008, 16(4): 308-319.
[21] STENDER H, KURTZMAN C, HYLDIG-NIELSEN J J, et al. Identification of Dekkera bruxellensis (Brettanomyces) from wine by fluorescence in situ hybridization using peptide nucleic acid probes[J]. Applied and Environmental Microbiology, 2001, 67(2): 938-941.
[22] ANDORRA I, ESTEVE-ZARZOSO B, GUILLAMON J M, et al. Determination of viable wine yeast using DNA binding dyes and quantitative PCR[J]. International Journal of Food Microbiology,2010, 144(2): 257-262.
[23] MORNEAU A D, ZUEHLKE J M, EDWARDS C G. Comparison of media formulations used to selectively cultivate Dekkera/ Brettanomyces[J]. Letters in Applied Microbiology, 2011, 53(4): 460-465.
[24] 周小玲, 沈微, 饒志明, 等. 一種快速提取真菌染色體 DNA 的方法[J].微生物學通報, 2004, 31(4): 89-92.
[25] BENITO S, PALOMERO F, MORATA A, et al. Identifying yeats belonging to the Brettanomyces/Dekkera genera through the use of selective-differential media[J]. African Journal of Microbiology Research, 2012, 34(6): 6348-6357.
[26] WEDRAL D, SHEWFELT R, FRANK J. The challenge of Brettanomyces in wine[J]. LWT-Food Science and Technology, 2010,43(10): 1474-1479.
Detection and Identification of Dekkera bruxellensis in Chinese Wines
CAO Peixin1, MA Tao1, YANG Kaidi1, LIU Yanlin1,2,*
(1. College of Enology, Northwest A&F University, Yangling712100, China;2. Shaanxi Engineering Research Center for Wine and Viticulture, Yangling712100, China)
Dekkera bruxellensis is one of the major spoilage microorganisms in wines. In order to detect Dekkera bruxellensis in wines rapidly and accurately, the separation and detection efficiency of Dekkera bruxellensis using differential medium (DBDM) and Wallerstein nutrient (WLN) medium were compared. A specific polymerase chain reaction (PCR)assay was also used to further testify the results. The results showed that both DBDM and WLN could be useful to detect Dekkera bruxellensis in wines, while DBDM was more selective to identify Dekkera bruxellensis accurately when combined with the specific PCR. The application of DBDM combined with the specific PCR to investigate the infection of Dekkera bruxellensis in wines from five major producing regions in China showed that among the investigated regions, Dekkera bruxellensis spoilage was found in wines from three regions including Hebei, Shandong and Jilin, especially in the wines aged in oak barrels. Therefore, the Chinese wine industry should pay much attention to the issue of Dekkera bruxellensis spoilage.
wine; Dekkera bruxellensis; differential medium; detection and identification
TS261.1
A
1002-6630(2015)23-0172-06
10.7506/spkx1002-6630-201523032
2015-01-19
中央高校基本科研業務費專項資金項目(重點項目)(Z109021201);國家現代農業(葡萄)產業技術體系建設專項(CARS-30-jg-3)
曹培鑫(1990—),男,碩士研究生,研究方向為葡萄酒微生物。E-mail:736008703@qq.com
劉延琳(1966—),女,教授,博士,研究方向為葡萄酒及釀酒微生物。E-mail:yanlinliu@nwsuaf.edu.cn

