色譜與質譜聯用技術在蛋白質翻譯后修飾研究中的進展及應用
林琳等
摘 要 蛋白質翻譯后修飾是調控各種細胞信號通路和細胞命運最為關鍵的分子機制之一。在分子水平上,蛋白質翻譯后修飾的“寫入”、“擦除”和“識別”是實現其動態調控功能的核心過程,均屬于生物分析化學的研究范疇。基于液相色譜與生物質譜聯用的蛋白質組學技術經歷了近十年的高速發展,已成為系統水平上表征各種蛋白質翻譯后修飾的標準方法。本文綜述了蛋白質翻譯后修飾分析相關的關鍵技術和方法進展,主要包括:各種翻譯后修飾蛋白質和多肽的富集方法、基于高效液相色譜的多維分離方法和各種生物質譜鑒定方法,并重點闡述了近年來的新的發展方向。基于相關技術的快速發展和大規模鑒定數據的產生,本文重點介紹了5種具有重要生物學功能的蛋白質翻譯后修飾,包括:磷酸化、糖基化、泛素化、乙酰化和甲基化。生物分析化學領域的方法和技術創新必將進一步促進對蛋白質翻譯后修飾的系統水平研究的發展。
關鍵詞 蛋白質翻譯后修飾; 蛋白質組學; 質譜; 綜述
1 引 言
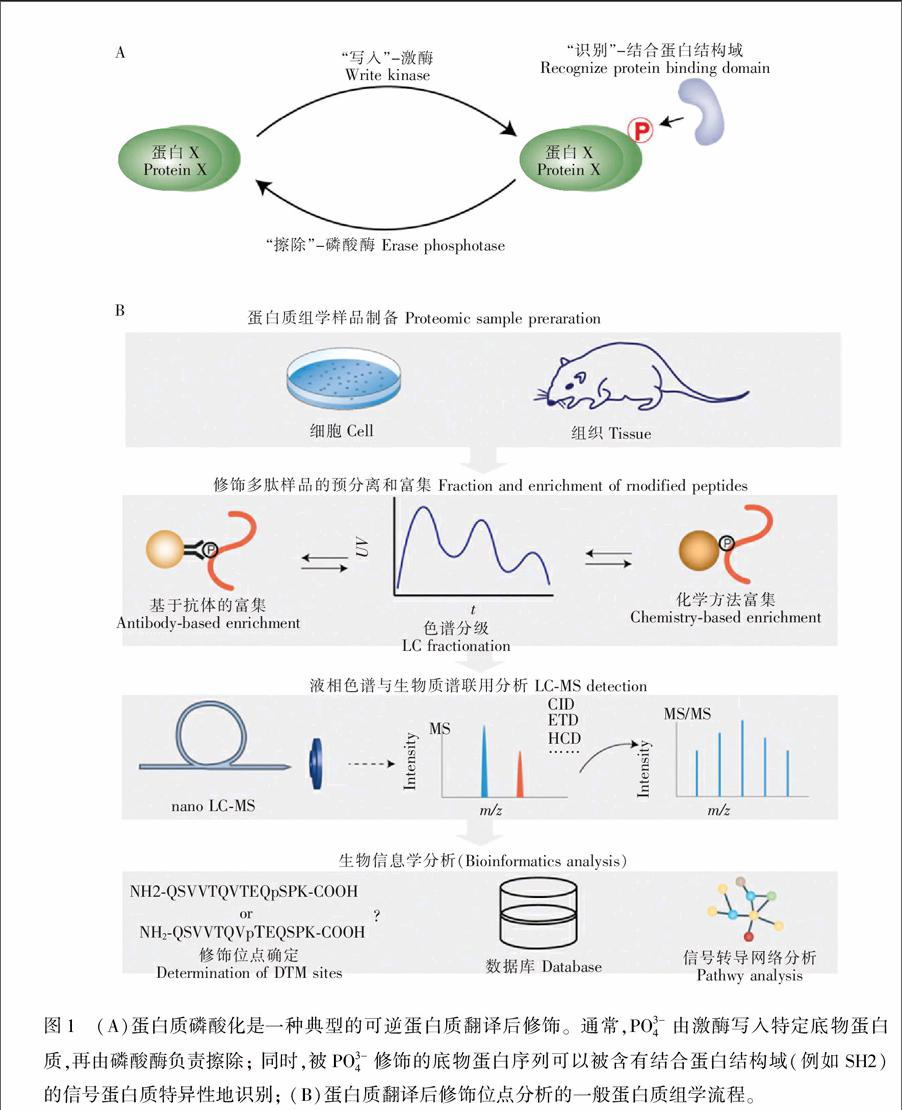
蛋白質翻譯后修飾是蛋白質水平發生的一種重要的生物化學過程,可為蛋白質的特定氨基酸位點引入各種化學基團,例如磷酸根、糖基、甲基、乙酰基和泛素鏈等。在哺乳動物表達的所有蛋白質中, 超過50%的蛋白質可以在特定時間和亞細胞空間發生各種各樣的翻譯后修飾,并且調控著許多重要的生物學功能[1]。目前,已陸續發現近300種蛋白質翻譯后修飾,其中很多重要的翻譯后修飾都是以可逆方式被相關酶調控的[2]。這些可逆的蛋白質翻譯后修飾是動態調控蛋白質功能的重要方式,同時也為生物體系提供了更高層次的復雜度。以具有重要生物學功能以及研究最為廣泛的蛋白質磷酸化為例,使底物蛋白質發生磷酸化修飾的酶是激酶,而使磷酸化蛋白質發生去磷酸化的酶是磷酸酶。在人類基因組中,分別有518種激酶和約137種磷酸酶。激酶和磷酸酶常有多種底物蛋白質,而每種底物蛋白質往往有多個磷酸化位點。這些被磷酸化的氨基酸序列常會特異性地被蛋白結構域所識別(例如SH2、PTB、BRCT結構域等),從而形成基于磷酸化的蛋白質間相互作用[3,4]。如圖1A所示,正是由于細胞內存在著數目龐大的可以“寫入”、“擦除”和“識別”磷酸化位點的蛋白質,形成了一個極為復雜的由磷酸化介導的信號轉導網絡。從分子水平上,上述細胞內的生物化學過程主要涉及各種修飾基團的“寫入”和“擦除”,以及蛋白質間基于特定修飾基團的“識別”。這類分子機理正是生物分析化學研究的核心生物學問題之一。
傳統的蛋白質翻譯后修飾研究主要依賴于基于特異性抗體的免疫檢測技術或放射性標記技術。這些方法對研究由單一位點翻譯后修飾介導的細胞信號轉導過程起著不可替代的作用。然而,由于上述技術存在操作要求高、特異性抗體制備周期長等缺點,很難實現蛋白質翻譯后修飾的大規模檢測。近年來,基于液相色譜與生物質譜聯用技術(LC-MS)的蛋白質組學策略發展迅速,為系統水平上的蛋白質翻譯后修飾研究提供了強有力的研究工具[5,6]。應用于蛋白質翻譯后修飾研究的研究策略與鑒定一般多肽樣品的LC-MS流程是基本一致的。然而,蛋白質翻譯后修飾研究常具有極大的技術挑戰,這主要是因為:(1)被修飾蛋白質通常僅占總體蛋白質表達量的很小一部分; (2)翻譯后修飾基團常具有化學穩定性差、在樣品制備和LC-MS分析過程中容易丟失的缺點; (3)蛋白質翻譯后修飾常是動態調節過程,這為樣品制備和LC-MS分析提出了更高的要求。
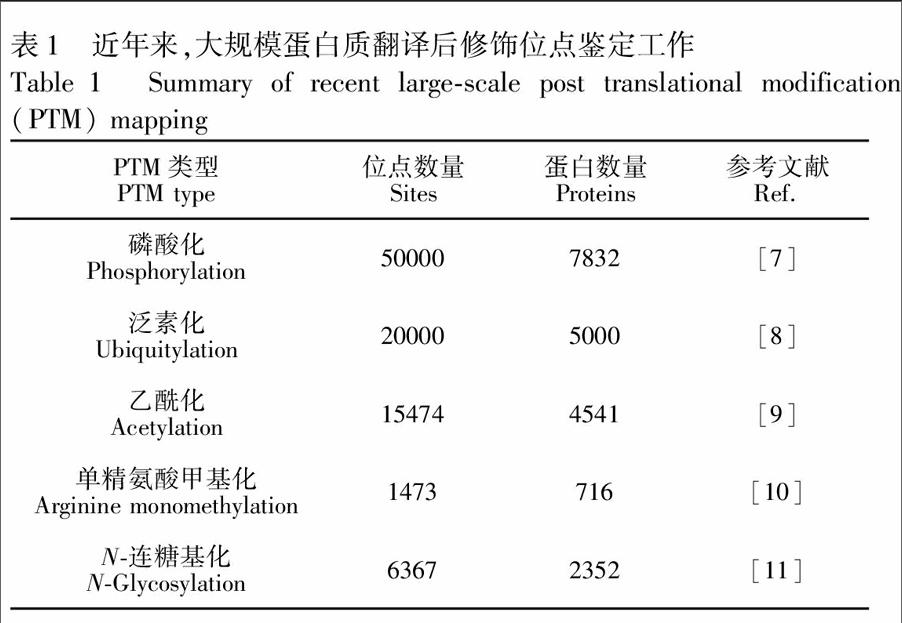
如圖1B所示,蛋白質翻譯后修飾位點分析的一般流程主要分為以下4個步驟:蛋白質組學樣品制備、修飾多肽樣品的預分離和富集、LC-MS分析和生物信息學分析。作為一種通用型分析技術策略,蛋白質組學技術可以實現對包括細胞、組織和體液在內的所有生物樣品的全面LC-MS分析。近十年來,隨著質譜硬件技術的快速發展,生物質譜的靈敏度和掃描速度都有了顯著地提升; 與此同時,各種在線和離線色譜分離技術和針對多種重要翻譯后修飾的選擇性富集方法也層出不窮,為蛋白質翻譯后修飾研究奠定了堅實的技術基礎。目前,針對磷酸化等幾種重要蛋白質翻譯后修飾的蛋白質組學研究均可實現在幾千到上萬個位點的定性和定量表征(表1)。本文主要針對翻譯后修飾蛋白及相應的酶解多肽的色譜富集技術、色譜分離技術及質譜檢測技術進行
了綜述。基于其生物學重要性和相關分析技術快速發展,本文重點綜述蛋白質磷酸化、糖基化、泛素化、乙酰化和甲基化相關的研究進展,并對未來發展方向提出若干展望。
2 翻譯后修飾蛋白及多肽的
富集方法
2.1 磷酸化修飾
磷酸化修飾是細胞信號轉導和功能調控過程中最重要的翻譯后修飾類型之一。由于磷酸化翻譯后修飾的豐度極低,僅約占所在蛋白質總量的1%,因此,必須發展特異性的富集方法,以實現質譜的有效分析。目前,幾種親和富集方法發展較為完備,可以高效地從細胞酶解液中將磷酸化修飾的肽段富集出來,包括金屬氧化物親和色譜法(Metal oxide affinity chromatography,MOAC)、固定化金屬離子親和色譜法(Immobilized metal ion affinity chromatography,IMAC)和基于特異性抗體的富集方法。
2.1.1 金屬氧化物親和色譜法(MOAC) 金屬氧化物親和色譜是一類最常用和最為有效的磷酸化肽富集方法。基于金屬氧化物對磷酸基團的高親和力,可以實現對磷酸化多肽的高選擇性富集(一般可以達90%以上)。多種金屬氧化物已經被用于富集磷酸化肽,包括二氧化鈦(TiO2)[12]、二氧化鋯(ZrO2)[13],五氧化二鈮(Nb2O5)[14]等,其中TiO2以其優異的富集效率和富集選擇性被廣泛使用。TiO2富集的一般流程包括以下步驟:首先將樣品酸化(如0.1%(V/V)三氟乙酸),這個過程是為了將樣品中的非磷酸化酸性肽段質子化,防止它們非特異性地吸附在TiO2顆粒上。然后將樣品與TiO2顆粒充分接觸,以實現對磷酸化肽的特異性吸附。經過洗滌步驟之后,磷酸化多肽在堿性條件下(如5%(V/V)氨水)從TiO2顆粒上洗脫下來。含有酸性氨基酸的多肽(如谷氨酸和天冬氨酸)常會競爭磷酸化肽的結合位點,從而降低富集方法的選擇性。因此,上樣緩沖液中常要加入2,5二羥苯甲酸[15]、鄰苯二甲酸[16]或谷氨酸[17]等,有效抑制酸性多肽的吸附,從而提高富集選擇性。
2.1.2 固定化金屬離子親和色譜法(IMAC) IMAC是另一種被廣泛應用的磷酸化多肽富集方法。通常,正電性的金屬離子可以被含有亞胺乙酸(IDA)或氨三乙酸(NTA)金屬螯合基團的載體固定化,可以基于靜電相互作用力, 可選擇性地富集負電性的磷酸化多肽。各種各樣的金屬離子可以被固定化,并用于高效富集磷酸化多肽,例如Fe3+[18],Ti4+[19],Zr4+[20]等。IMAC的操作流程與MOAC類似:首先,多肽樣品在酸性條件下上樣,然后清洗非特異性吸附多肽,最后用高pH值的洗脫液或者磷酸鹽緩沖液洗脫被富集的磷酸化多肽。
通常,IMAC富集法對多磷酸化肽的富集效果強于對單磷酸化肽的富集。因此,通過結合MOAC富集法,可以很好地實現對磷酸化多肽的全面富集。在Sequential elution from IMAC(SIMAC)方法中[21],基于Fe3+-IMAC的富集首先可以將多磷酸化肽很好地富集,然后再串聯使用TiO2富集,以實現對大多數單磷酸化肽的全面富集。Zhou等[19,22]發展了一種新的高效磷酸化多肽富集方法Ti4+-IMAC。通過設計并合成多種含有固定化磷酸根的微球,可以實現對Ti4+等金屬離子的高效螯合和磷酸化肽的高選擇性富集。與其它4種常用磷酸化多肽富集方法(Fe3+-IMAC, Zr4+-IMAC, TiO2和ZrO2)相比,Ti4+-IMAC方法展示出更高的富集選擇性和富集效率。該方法的高富集選擇性主要是源于聚合物微球表面所含有的具有靈活性的間隔臂,以及Ti3+與PO34之間的高選擇性作用力。
2.1.3 基于抗體的富集法 酪氨酸磷酸化發生率非常低,一般只約占人體全部磷酸化修飾的1%[23]。因此,發展可以區分絲氨酸和蘇氨酸磷酸化肽的高選擇性酪氨酸磷酸化肽富集方法就顯得尤為必要。基于固定化抗體的富集方法是目前針對酪氨酸磷酸化肽最有效的富集方法。目前, 最有效的富集抗體包括4G10抗體和PY100抗體。以PY100抗體為例,Rikova等[24]從41種非小細胞肺癌細胞和150種非小細胞肺癌腫瘤中共富集并鑒定到2700多個蛋白和4551個酪氨酸磷酸化位點,是目前最大規模的酪氨酸磷酸化蛋白質組學分析。基于固定化抗體的富集方法的富集選擇性一般較差,通過結合MOAC或IMAC富集方法可以大大提高富集選擇性[7,25]。然而,引入額外的富集步驟也會大大增加微量樣品的損失。在本研究組的前期工作中,通過系統優化抗體富集的各步驟,實現了一步抗體富集鑒定超過800個酪氨酸磷酸化位點[26]。
2.2 糖基化修飾
蛋白質糖基化修飾參與著很多重要的細胞功能,例如細胞粘附,受體膜蛋白激活,細胞免疫反應等。大多數位于細胞膜,內質網和細胞外的蛋白質均會發生糖基化修飾。在眾多蛋白質糖基化修飾中,發生在天冬酰胺上的N-連糖基化修飾和發生在絲氨酸和蘇氨酸上的O-連糖基化修飾是最常見和研究最廣泛的糖基化修飾。應用于富集糖基化蛋白質或多肽的方法主要有固定化凝集素和酰肼化學兩種。
2.2.1 固定化凝集素富集 基于凝集素對多糖的選擇性吸附作用,將凝集素固定到合適的基質上可以實現對糖蛋白或者糖肽的特異性富集[27]。伴刀豆凝集素(Con A)、麥胚凝集素(WGA)、花生凝集素(PNA)和橙黃網孢盤菌凝集素(AAA)是針對N-連糖基化蛋白富集廣泛使用的幾種凝集素。由于不同凝集素識別不同的糖型,通過結合使用凝集素ConA、WGA、N-乙酰葡糖胺和凝集素RCA120, Zielinska等[11]發展了一種膜輔助樣品處理方法用于糖基化蛋白的富集和糖基化位點鑒定,共從4種小鼠組織和血漿中鑒定5753個糖基化位點,是目前最大的N-連糖基化數據集。
2.2.2 基于酰肼化學的富集
另一種廣泛使用的糖基化蛋白和糖肽富集方法是由Zhang等[28]發展的基于酰肼化學的N-連糖基化富集方法。其主要步驟是:首先將糖基化蛋白用高碘酸鈉氧化; 被氧化糖鏈產生的醛基可以與含有酰肼基團的樹脂發生共價鍵反應,從而實現對糖基化蛋白的高選擇性富集; 所固定化的糖基化蛋白可以被糖苷水解酶酶解釋放,可以用于后續的質譜分析。這種方法最大的優點是反應效率高和特異性好。最近,Zhang等[29]報道了一種基于氨氧基樹脂的氧化糖基化蛋白富集方法。與傳統的酰肼化學法相比,該方法具有更高的反應活性,從而大大降低了樣品處理的周期。
2.3 泛素化修飾
泛素是含有76個氨基酸的多肽序列。一個或多個該多肽序列可以通過其C末端的兩個甘氨酸殘基選擇性地修飾到多個底物蛋白質的賴氨酸位點上。蛋白質泛素化修飾是激活蛋白質降解機制的關鍵性信號,在DNA修復和細胞凋亡等重要生物學過程中都起著至關重要的作用。傳統的泛素化蛋白質的富集主要通過在其泛素鏈末端引入親和富集標簽實現。以酵母細胞為模型和6×His tag標簽對泛素化進行標記,Peng等[30]首次實現了對泛素化蛋白及其泛素化位點的蛋白質組學鑒定。然而,該方法的顯著缺陷是缺乏對泛素化位點的選擇性富集。基于胰蛋白酶酶解產生的泛素化多肽會殘留兩個甘氨酸殘基的特征,目前最為有效的泛素化多肽富集方法是基于上述雙甘氨酸殘基的抗體富集法[31]。通過結合各種離線色譜分離策略,該抗體富集法已經被廣泛應用于鑒定成千上萬個泛素化位點[32]。
2.4 乙酰化修飾
蛋白質乙酰化修飾主要發生在蛋白質賴氨酸位點上,是組蛋白和轉錄因子等關鍵功能性蛋白質的重要調控機制。大規模乙酰化位點鑒定主要得益于特異性抗體和先進的質譜儀器的發展。芝加哥大學趙英明團隊首先發展了一種針對乙酰化蛋白的抗體,并首次實現對蛋白質乙酰化位點的系統水平分析,共鑒定了388個乙酰化位點[33]。最近,Mann等[34]使用相同抗體并結合先進的軌道阱質譜儀(Orbitrap)實現了對乙酰化位點最大規模的鑒定,共從人類急性髓系白血病細胞器中鑒定到約3600個賴氨酸乙酰化位點。這充分說明了特異性抗體對低豐度修飾多肽富集的高效率和先進質譜技術對大規模蛋白質組學分析的重要性。
2.5 甲基化修飾
蛋白質甲基化修飾主要發生在蛋白質的賴氨酸和精氨酸位點上。與乙酰化修飾類似,蛋白質甲基化修飾也是DNA損傷修復、基因轉錄和信號轉導的重要調控途徑。然而,與其它類型翻譯后修飾相比,蛋白質甲基化修飾的大規模蛋白質組學研究一直比較落后。這主要是因為甲基化修飾基團較小,很難發展針對其化學結構的有效富集方法。Boisvert等[35]首先使用4種特異性精氨酸甲基化抗體(ASYM24、ASYM25、SYM10和SYM11)對甲基化蛋白質進行了蛋白質組學分析。最近,Cell Signaling公司發展了多種針對不同類型甲基化的特異性抗體,實現了對超過1000多個精氨酸甲基化位點和約160個賴氨酸甲基化位點的大規模鑒定[10]。
正如引言中所述,大部分重要的蛋白質翻譯后修飾都有相應的特異性蛋白質結合結構域[36]。例如,人蛋白質組中有120個可以特異性識別酪氨酸磷酸化的SH2蛋白質結合結構域。該結構域已經被廣泛地應用于高選擇性地檢測和富集酪氨酸磷酸化蛋白質[37]。在人蛋白質組中也存在著5種甲基化蛋白質結合結構域: Tudor, Chromo, MBT, PWWP和Agenet[38]。最近, Moore等[39]篩選獲得一種MBT結構域,可以實現對賴氨酸甲基化蛋白質的廣譜性富集和大規模蛋白質組學分析。可以預期基于蛋白質結合結構域的親和富集方法將在蛋白質翻譯后修飾研究中起到越來越重要的作用。
2.6 逐步富集多種修飾蛋白
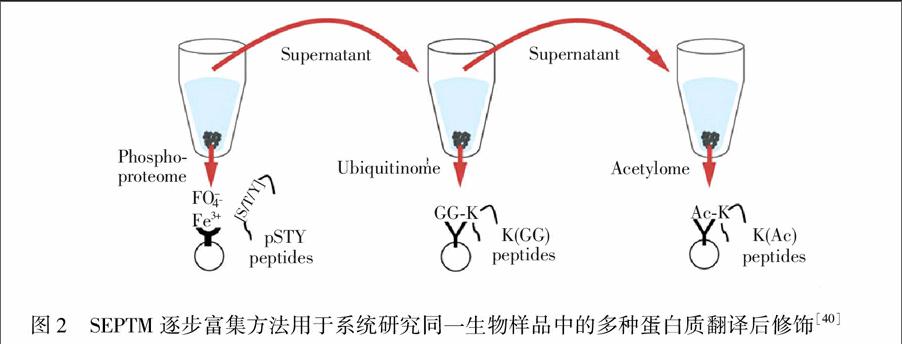
多種蛋白質翻譯后修飾的協同調控是調控各種生物學過程的重要分子機制。實現對這種協同調控機制的系統水平表征,將為生物學研究提供更高維度的分子機理。最近Mertins等[40]發展了一種針對同一生物學樣品中蛋白質磷酸化、乙酰化和泛素化修飾多肽的逐步富集方法(SEPTM),從而實現了對特定樣品中多種蛋白質翻譯后修飾的全面分析。該方法依次對同一生物樣品使用IMAC親和富集、泛素化特異性抗體富集和乙酰化特異性抗體富集,共檢測到20000多個磷酸化修飾位點、15000多個泛素化修飾位點和3000多個乙酰化修飾位點(圖2)。在這些被修飾的多肽中有0.3%的多肽存在多種修飾類型同時發生的現象。Swaney等[41]針對蛋白質磷酸化和泛素化修飾多肽進行了類似的串聯富集和大規模質譜鑒定,最終發現在466個蛋白中同時存在2100個磷酸化修飾位點和2189個泛素化修飾位點。
圖2 SEPTM逐步富集方法用于系統研究同一生物樣品中的多種蛋白質翻譯后修飾[40]
Fig.2 Serial enrichment of different post-translational modification (SEPTM) enables sequential enrichment of different PTMs from the same biological sample[40]
3 多維液相色譜分離
蛋白質組學樣品均具有極高的復雜度,因此需要對其進行高效的分離以提高質譜鑒定效率。在蛋白質翻譯后修飾研究中,在選擇性富集修飾多肽前或者富集后常需要對酶解多肽進行有效的預分離。高效液相色譜是目前蛋白質組學策略中的標準化分離手段,這主要是因為其具有極高的酶解多肽分離能力,并且能很好地與質譜儀進行在線聯用[42]。為了顯著提高液相色譜的分離效率,降低多肽的復雜度,多維液相色譜是目前最為廣泛使用的色譜分離模式[43]。基于在線低pH反相液相色譜與質譜儀之間的良好相容性,其它色譜分離模式與在線低pH反相液相色譜之間的的分離正交性是多維液相色譜分離中最重要的考慮因素[44]。以應用磷酸化蛋白質組學為例,介紹目前最為廣泛使用的幾種多維液相色譜分離模式。
3.1 離子交換色譜分離
離子交換色譜用于多肽分離主要是基于磷酸化多肽與固定相表面的強陽離子交換基團(SCX)或強陰離子交換基團(SAX)間的靜電相互作用實現分離。另外,在高乙腈濃度流動相中,眾多離子交換色譜填料可以展示出很好的親水相互作用色譜(HILIC)性質,從而可以基于親水相互作用分離酶解多肽[45]。在磷酸化蛋白質組學中,SCX是被最為廣泛使用的一種色譜分離模式[18]。SCX分離磷酸化多肽的主要原理是:在酸性條件(如pH 2.7)下,磷酸化多肽的N端氨基和C端的賴氨酸或精氨酸殘基都被質子化,磷酸基團的存在會降低多肽的價態。因此,磷酸化肽在SCX色譜填料上的保留較常規多肽更弱,從而實現了磷酸化多肽的有效富集和分離。通過與IMAC富集方法結合,Hottlin等[46]從9種小鼠組織中共鑒定到36000多個磷酸化位點,很好地展示了SCX與反相液相色譜的高正交性和其應用于磷酸化蛋白質組學研究的強大分離能力。與SCX相比,SAX對磷酸化肽具有更有效的保留,因此也被廣泛地應用于磷酸化蛋白質組學研究中[47,48]。其主要優勢是可以對多磷酸化肽、單磷酸化肽和非磷酸化肽實現更有效的分離。然而,離子交換色譜模式進行多肽分離一般都需要使用高濃度鹽進行梯度洗脫,從而引入了額外的柱后除鹽步驟,導致樣品損失。
3.2 基于高pH反相液相色譜的分離
多肽在反相色譜中的分離是基于其與固定相上的C18基團之間的疏水相互作用實現的。Gilar等[50]首次將高pH反相色譜和低pH反向色譜結合使用,進行二維液相色譜分離,獲得了優秀的正交性。該二維分離模式正交性的主要原因可能是肽段在不同的pH條件下電荷分布不同。中科院大連化物所鄒漢法研究組對該方法在磷酸化多肽的分離方面進行了系統優化,并創造性地引入了等間隔先流出組分和后流出組分樣品混合法(圖3)[49]。該方法的優勢是:(1)去除了額外的除鹽步驟,從而大大降低了微量多肽樣品的損失; (2)通過合并樣品可以大大減少質譜分析時間,但同時又不降低二維色譜分離的正交性。正是由于上述顯著的樣品處理優勢,該分離策略成為目前蛋白質組學領域中最為廣泛使用的多維色譜分離模式[32,51]。基于上述介紹的多維色譜模式, Ficarro等[52]進一步發展了一種基于高pH反相色譜,SAX色譜和低pH反相色譜的三維色譜分離系統; 通過與質譜進行在線聯用,最大化地實現了復雜磷酸化多肽樣品的分離和高靈敏度質譜檢測。作者從1×106~3×106個細胞中(約5~10 μg)共鑒定了約2500個非冗余的磷酸化多肽,是目前應用于蛋白質翻譯后修飾研究最靈敏的LC-MS技術。
4 生物質譜鑒定
應用于蛋白質翻譯后修飾的生物質譜技術主要需要完成以下任務:(1)鑒定發生修飾的蛋白質; (2)鑒定發生修飾的氨基酸位點及修飾基團的化學結構; (3)定量分析修飾蛋白占總體蛋白表達量的比例。存在于蛋白質上的修飾基團常具有化學穩定性差和豐度低的特點,因此對于質譜檢測提出了更高的要求。近年來,多種溫和的碎裂技術相繼誕生,大大促進了修飾多肽的質譜檢測效果。以研究最為廣泛的磷酸化修飾為例,本文闡述應用于修飾多肽檢測的重要質譜碎裂技術,主要包括:碰撞誘導解離(Collision-induced dissociation, CID)、高能碰撞解離技術(High-energy collisional dissociation,HCD)、基于電子誘導的電子捕獲解離(Electron capture dissociation,ECD)和電子轉移解離技術(Electron transfer dissociation,ETD)。
4.1 碰撞誘導解離(CID)
CID是串聯質譜法中最常用的碰撞解離方法。它利用惰性氣體分子撞擊肽段母離子使其在酰胺鍵處斷裂,產生具有豐富序列信息的b-和y-型離子。由于磷酸基團在CID過程中易發生中性丟失,影響了它對磷酸化多肽序列的有效鑒定。中性丟失的程度與很多因素有關,如電荷態、化學結構、質子化試劑、碎裂能量等。酪氨酸磷酸化易丟失HPO3(80 Da)的中性碎片,而絲氨酸和蘇氨酸磷酸化更傾向于丟失H3PO4 (98 Da)的中性碎片[53]。這主要是由于酪氨酸磷酸化的CO鍵鍵能更大,不易碎裂。
為了解決磷酸化肽易發生中性丟失的問題,在離子阱質譜中常進一步采用第三級質譜MS3碎裂,以獲得更多的碎片離子信息[54]。多級活化解離,又稱虛擬的MS3,將發生中性丟失的產物離子和母離子同時活化,并記錄所有的碎片離子[55]。MSA方法常用于低分辨的離子阱質譜中,而在高分辨、高質量精度類質譜(如Orbitrap)中的應用不多[54]。這主要是因為這類質譜可以高通量地獲得母離子的精確質量數,因此常規的MS2碎裂已經可以滿足檢測需求,額外的MS3和多級活化解離反而會消耗本可以用于測序新肽段的質譜時間。
4.2 高能碰撞誘導解離(HCD)
HCD通過在高能碰撞室中提供比 CID更高的碰撞能量和更短的活化時間來解離離子[56,57]。與基于離子阱的CID相比,HCD所產生的中性丟失峰較少,主要產生序列特異性的y-型離子和某些b-型離子(圖4)。在HCD中,b-型離子傾向于進一步解離成a-型離子,因此b-型離子的數目較y-型離子少。同時,由于HCD的能量更大,它常產生更小的碎片離子,如磷酸化酪氨酸特異性的Immonium離子。HCD碎裂克服了常規離子阱存在的1/3低質量范圍限制,同時提供了比CID更高的分辨率,是Orbitrap質譜儀的標配解離模式,被廣泛用于蛋白質翻譯后修飾的研究。
4.3 基于電子誘導的解離(ECD/ETD)
與CID相比,基于電子誘導的解離方法,電子捕獲解離(ECD)和電子轉移解離(ETD),是一種溫和的軟解離方式,能夠產生肽骨架碎裂信息并保留修飾基團。在ECD過程中,母離子被近熱電子活化,酰胺鍵上的氧原子從鄰近的氨基酸殘基上轉移一個質子,此過程放熱使N-Cα鍵發生斷裂產生c-型和z-型離子[58]。在ETD過程中,熒蒽自由基陰離子攜帶電子與肽段陽離子發生相互作用,此過程使多肽電荷數降低并誘導肽骨架的碎裂,產生一系列互補的c-型和z-型離子[59]。與CID相比,ETD更適合高電荷態和包含堿性殘基肽段的測序。CID和ETD的互補使用有利于增加蛋白的鑒定數目和提高序列覆蓋度,對大規模鑒定磷酸化肽十分有利[60]。另外,ECD和ETD方法是目前應用于糖基化多肽糖鏈結構解析最為有效的裂解方法[61]。基于上述的解離方法,Heck研究小組發展了一種集成化的解離技術,名為EThcD。顧名思義,該方法將適合磷酸化位點分析的ETD和HCD碎裂技術結合起來[62]。ETD不能斷裂N-端連接脯氨酸殘基的NCα鍵,這限制了它對脯氨酸含量高的磷酸化肽的修飾位點的確認。而EThcD方法很好地克服了這方面的缺陷,能夠產生豐富的b/y和c/z型離子。
5 結論與展望
蛋白質翻譯后修飾多達300多種,它們具有化學穩定性差、豐度低和動態變化等特點,在各種生物學過程中發揮著重要的調控作用。基于LC-MS的蛋白質組學技術已經實現了對成千上萬種修飾位點的大規模定性和定量分析,這其中主要包括磷酸化、糖基化、泛素化、乙酰化和甲基化修飾。如本文所述,對這些重要翻譯后修飾的大規模蛋白質組學分析的實現主要歸功于眾多方法學的巨大進步,主要包括高選擇性和高效富集方法的發展、高容量和高正交性多維色譜技術的發展以及高靈敏度和高掃描速度生物質譜技術的發展。隨著通用型多維色譜技術和生物質譜技術的不斷完善,新的蛋白質翻譯后修飾的大規模蛋白質組學研究更多局限于高選擇性和高效富集方法的發展。如表1所示,在目前主要研究的蛋白質翻譯后修飾中,甲基化修飾的大規模質譜鑒定仍處于較低的水平。這主要是因為各種甲基化修飾基團的分子尺寸較小,很難發展高特異性的富集方法。在傳統抗體技術無法很好地解決這一難題的情況下,基于MBT甲基化蛋白質結合結構域的富集方法為甲基化蛋白質組研究提供了新穎的高效富集方法[39]。由于幾乎所有的重要蛋白質翻譯后修飾都有眾多與之對應的蛋白質結合結構域,可以預期該類富集方法將會成為未來蛋白質翻譯后修飾富集方法發展的重要方向。
此外,隨著多種重要蛋白質翻譯后修飾大規模蛋白質組學分析方法的逐步成熟,越來越多的研究者開始關注在相關生物體系中的應用研究,尤其是對多種翻譯后修飾在同一蛋白質上的協同作用研究。目前的相關研究主要針對大規模質譜鑒定效果最好的翻譯后修飾,即磷酸化、泛素化和乙酰化。可以預期,隨著其它相關蛋白質翻譯后修飾富集方法的不斷發展和完善,針對特定生物學體系的多種翻譯后修飾的協同作用研究將會得到進一步發展,并在相關生物學研究中起到關鍵作用。總之,色譜與質譜聯用的技術已經為蛋白質翻譯后修飾研究提供了革命性的系統水平研究工具,傳統生物學領域的“一個基因,一個蛋白質,一種生物學功能”的理念已經被完全打破。基于色譜與質譜聯用技術的蛋白質組學策略所產生的海量質譜數據, 已經推動蛋白質翻譯后修飾及相關生物學領域進入了全新的系統生物學研究時代。
References
1 Scott J D, Pawson T. Science, 2009, 326: 1220-1224
2 Witze E S, Old W M, Resing K A, Ahn N G. Nat.Methods, 2007, 4: 798-806
3 Choudhary C, Mann M. Nat. Rev. Mol. Cell. Biol., 2010, 11: 427-439
4 Altelaar A F, Munoz J, Heck A J. Nat. Rev. Genet., 2013, 14: 35-48
5 Nilsson C L. Anal. Chem., 2012, 84: 735-746
6 Wang F, Song C, Cheng K, Jiang X, Ye M, Zou H. Anal. Chem., 2011, 83: 8078-8085
7 Sharma K, D′Souza R C, Tyanova S, Schaab C, Wisniewski J R, Cox J, Mann M. Cell Reports, 2014, 8: 1583-1594
8 Udeshi N D, Svinkina T, Mertins P, Kuhn E, Mani D R, Qiao J W, Carr S A. Mol. Cell. Proteomics, 2013, 12: 825-831
9 Lundby A, Lage K, Weinert B T, Bekker-Jensen D B, Secher A, Skovgaard T, Kelstrup C D, Dmytriyev A, Choudhary C, Lundby C, Olsen J V. Cell Reports, 2012, 2: 419-431
10 Guo A, Gu H, Zhou J, Mulhern D, Wang Y, Lee K A, Yang V, Aguiar M, Kornhauser J, Jia X, Ren J, Beausolei S A, Silva J C, Vemulapalli V, Bedford M T, Comb M J. Mol. Cell. Proteomics, 2014, 13: 372-387
11 Zielinska D F, Gnad F, Wisniewski J R, Mann M. Cell, 2010, 141: 897-907
12 Olsen J V, Blagoev B, Gnad F, Macek B, Kumar C, Mortensen P, Mann M. Cell, 2006, 127: 635-648
13 Sugiyama N, Masuda T, Shinoda K, Nakamura A, Tomita M, Ishihama Y. Mol. Cell. Proteomics, 2007, 6: 1103-1109
14 Ficarro S B, Parikh J R, Blank N C, Marto J A. Anal.Chem., 2008, 80: 4606-4613
15 Larsen M R, Thingholm T E, Jensen O N, Roepstorff P, Jrgensen T J D. Mol. Cell. Proteomics, 2005, 4: 873-886
16 Bodenmiller B, Mueller L N, Mueller M, Domon B, Aebersold R. Nat. Methods, 2007, 4: 231-237
17 Wu J, Shakey Q, Liu W, Schuller A, Follettie M T. J. Proteome Res., 2007, 6: 4684-4689
18 Villen J, Gygi S P. Nat. Protocols, 2008, 3: 1630-1638
19 Zhou H, Ye M, Dong J, Corradini E, Cristobal A, Heck A J, Zou H F, Mohammed S. Nat. Protocols, 2013, 8: 461-480
20 Feng S, Ye M L, Zhou H J, Jiang X G, Jiang X N, Zou H F,Gong B. Mol. Cell. Proteomics, 2007, 6: 1656-1665
21 Thingholm T E, Jensen O N, Robinson P J, Larsen M R. Mol. Cell. Proteomics, 2008, 7: 661-671
22 Zhou H, Ye M, Dong J, Han G, Jiang X, Wu R, Zou H F. J.Proteome Res., 2008, 7: 3957-3967
23 Hunter T, Sefton B M. Proc. Natl. Acad. Sci. U. S. A., 1980, 77: 1311-1315
24 Rikova K, Guo A, Zeng Q, Possemato A, Yu J, Haack H. Cell, 2007, 131: 1190-1203
25 Jorgensen C, Sherman A, Chen G I, Pasculescu A, Poliakov A, Hsiung M, Larsen, B, Wilkinson D G, Linding R, Pawson T. Science, 2009, 326: 1502-1509
26 Tian R, Wang H, Gish G D, Petsalaki E, Pasculescu A, Shi Y, Mollenauerd M, Bagshawa R D, Yoseff N, Huntere T, Gingrasa A C,Weissd A,Pawsona T. Proc. Natl. Acad. Sci. U. S. A., 2015, 112: E1594-E1603
27 Kaji H, Saito H, Yamauchi Y, Shinkawa T, Taoka M, Hirabayashi J, Kasai K, Takahashi N, Isobel T. Nat. Biotechnol., 2003, 21: 667-672
28 Zhang H, Li X J, Martin D B, Aebersold R. Nat. Biotechnol., 2003, 21: 660-666
29 Zhang Y, Yu M, Zhang C, Ma W, Zhang Y, Wang C, Lu H. Anal.Chem., 2014, 86(15): 7920-7924
30 Peng J, Schwartz D, Elias J E, Thoreen C C, Cheng D, Marsischky G, Roelofs J, Finley D, Gygi S P. Nat. Biotech., 2003, 21: 921-926
31 Kim W, Bennett E J, Huttlin E L, Guo A, Li J, Possemato A, Sowa M, Rad R, Rush J, Comb M J, Harper J W, Gygi S P. Mol. Cell, 2011, 44: 325-340
32 Udeshi N D, Mertins P, Svinkina T, Carr SA. Nat.Protocols, 2013, 8: 1950-1960
33 Kim S C, Sprung R, Chen Y, Xu Y, Ball H, Pei J, Cheng T, Kho Y, Xiao Hao, Lin X, Grishin N V,White M, Yang X J, Zhao Y. Mol.Cell, 2006, 23: 607-618
34 Choudhary C, Kumar C, Gnad F, Nielsen M L, Rehman M, Walther T C,Olsen J V, Mann M. Science, 2009, 325: 834-840
35 Boisvert F M, Cté J, Boulanger M C, Richard S. Mol. Cell. Proteomics, 2003, 2: 1319-1330
36 Seet B T, Dikic I, Zhou M M, Pawson T. Nature Reviews Mol. Cell.Biol., 2006, 7: 473-483
37 Machida K, Thompson C M, Dierck K, Jablonowski K, Karkkainen S, Liu B, Zhang H, Nash P D, Newman D K, Nollau P, Pawson T, Renkema G H, Saksela K, Schiller M R, Shin D G, Mayer B J. Mol.Cell, 2007, 26: 899-915
38 Chen C, Nott T J, Jin J, Pawson T. Nat. Rev. Mol.Cell.Biol., 2011, 12: 629-642
39 Moore K E, Carlson S M, Camp N D, Cheung P, James R G, Chua K F,Wolf-Yadlin A, Gozani O. Mol. Cell, 2013, 50: 444-456
40 Mertins P, Qiao J W, Patel J, Udeshi N D, Clauser K R, Mani D R, Burgess M W, Gillette M A, JaffeJ D, Carr S A. Nat. Methods, 2013, 10: 634-637
41 Swaney D L, Beltrao P, Starita L, Guo A, Rush J, Fields S, Krogan N J, Villén J. Nat. Methods, 2013, 10: 676-682
42 Link A J, Eng J, Schieltz D M, Carmack E, Mize G J, Morris D R, Garvik B M, Yates J R. Nat. Biotechnol., 1999, 17: 676-682
43 Motoyama A, Yates J R, 3rd. Anal. Chem., 2008, 80: 7187-7193
44 Gilar M, Olivova P, Daly A E, Gebler J C. Anal. Chem., 2005, 77: 6426-6434
45 Boersema P J, Mohammed S, Heck A J. Anal. Bioanal. Chem., 2008, 391: 151-159
46 Huttlin E L, Jedrychowski M P, Elias J E, Goswami T, Rad R, Beausoleil S A, Villén J, Haas W, Sowa M E, Gygi S P. Cell, 2010, 143: 1174-1189
47 Han G, Ye M, Zhou H, Jiang X, Feng S, Jiang X, Tian R, Wan D, Zou H, Gu J. Proteomics, 2008, 8: 1346-1361
48 Wisniewski J R, Nagaraj N, Zougman A, Gnad F, Mann M. J. Proteome Res., 2010, 9: 3280-3289
49 Song C X, Ye M L, Han G H, Jiang X N, Wang F J, Yu Z Y, Zou H. Anal. Chem., 2010, 82(1): 53-56
50 Gilar M, Olivova P, Daly A E, Gebler J C. J. Sep. Sci., 2005, 28: 1694-1703
51 Kim M S, Pinto S M, Getnet D, Nirujogi R S, Manda S S, Chaerkady R. Nature, 2014, 509: 575-581
52 Ficarro S B, Zhang Y, Carrasco-Alfonso M J, Garg B, Adelmant G, Webber J T. Mol. Cell. Proteomics, 2011, 10: O111 011064
53 Boersema P J, Mohammed S, Heck A J R. J. Mass Spec., 2009, 44: 861-878
54 Villen J, Beausoleil S A, Gygi S P. Proteomics, 2008, 8: 4444-4452
55 Schroeder M J, Shabanowitz J, Schwartz J C, Hunt D F, Coon J J. Anal. Chem., 2004, 76(13): 3590-3598
56 Olsen J V, Macek B, Lange O, Makarov A, Horning S, Mann M. Nat. Methods, 2007, 4: 709-712
57 Jedrychowski M P, Huttlin E L, Haas W, Sowa M E, Rad R, Gygi S P. Mol. Cell. Proteomics, 2011, 10: M111 009910
58 Syrstad E A, Turecek F. J. Am. Soc. Mass Spec., 2005, 16: 208-224
59 Chi A, Huttenhower C, Geer L Y, Coon J J, Syka J E P, Bai D L. Proc. Natl. Acad. Sci. U. S. A., 2007, 104: 2193-2198
60 Phanstiel D H, Brumbaugh J, Wenger C D, Tian S, Probasco M D, Bailey D J. Nat. Methods, 2011, 8: 821-827
61 Zhu Z, Su X, Clark D F, Go E P, Desaire H. Anal. Chem., 2013, 85: 8403-8411
62 Frese C K, Zhou H, Taus T, Altelaar A F M, Mechter K, Heck A J R. J.Proteome Res., 2013, 12: 1520-1525

