稻瘟病菌重金屬抗性蛋白的生物信息學分析
方淑梅, 王 偉, 杜沖曉, 趙東磊, 李 超, 梁喜龍
(黑龍江八一農墾大學, 大慶 163319)
?
稻瘟病菌重金屬抗性蛋白的生物信息學分析
方淑梅#, 王 偉#, 杜沖曉, 趙東磊, 李 超, 梁喜龍*
(黑龍江八一農墾大學, 大慶 163319)
稻瘟病菌重金屬抗性蛋白是稻瘟病菌在進化過程中產生的分泌型抗性蛋白,其可能對該病原菌在高濃度重金屬環境中的生長、繁殖及生活史完成具有重要作用。為了明確稻瘟病菌重金屬抗性蛋白的生物信息學特性,本研究利用多種生物信息學網站與軟件對獲得的3種重金屬抗性蛋白的理化性質、結構域、三級結構、亞細胞定位等進行分析,并構建了系統進化樹,同時預測了蛋白的組織表達特異性。該研究結果可為從遺傳學方面深入了解重金屬抗性蛋白在稻瘟病菌生長發育、繁殖和致病過程中的真正功能奠定基礎。
生物信息學; 稻瘟病菌; 重金屬抗性蛋白; 結構分析; 理化特性
稻瘟病菌(Magnaportheoryzae)屬于半知菌亞門,灰梨孢屬,可引起水稻稻瘟病,每年給全球水稻產量造成的損失超過1億t,給世界糧食安全生產帶來嚴重威脅[1-2]。因稻瘟病菌具有許多植物病原真菌生命循環的重要特點,且其侵染過程、致病機制和相伴隨的基因表達在絲狀真菌中具有典型性,該菌現已成為植物病原真菌致病機理和分子生物學研究的一種重要模式真菌[3]。
重金屬對生物機體的作用具有雙重性,Cu、Zn、Mn等微量元素可作為多數酶的輔助因子維持細胞的正常代謝,但濃度過高則對生物體有害。其他重金屬,如Cd、Pb等并非生命活動所需,含量超標同樣會對生物產生毒害。隨著工業化的迅猛發展,農藥、化肥的大量使用及嚴重的環境污染,土壤中的重金屬含量大量增加,已嚴重威脅植物和土壤微生物的生存。研究發現,自然界中的大多數原核和真核微生物,暴露在一定量的重金屬環境中都能利用自身的代謝機制盡量減少毒害或受毒后迅速恢復生長,繁殖后代,同時在遺傳上顯示出某些特定的生物反應[4-5]。這一事實主要與微生物在長期的進化過程中產生的抗性蛋白以及形成的多種適應性有關,例如分泌重金屬抗性蛋白。目前,有關芽胞桿菌屬[6]、葡萄球菌屬[7]、短桿菌屬[8]和根瘤菌屬[9]部分細菌,以及黑曲霉[10]等對重金屬(如Cd,Cr,Ni)的抗性機制已有報道。隨著稻瘟病菌基因組測序的完成,眾多學者對稻瘟病菌各方面的研究已較為深入,但對稻瘟病菌重金屬抗性蛋白的研究仍然較少。為此,本文利用生物信息學方法分析了稻瘟病菌基因組中可能的重金屬抗性基因及蛋白序列,探討了稻瘟病菌的重金屬抗性機制,旨在為從反向遺傳探究它們在稻瘟病菌生長發育和侵染致病過程中所起的作用奠定理論基礎。
1 研究對象與分析方法
1.1 稻瘟病菌重金屬抗性蛋白基因的獲取與確定
利用稻瘟病菌數據庫網站(http:∥www.broadinstitute.org/annotation/genome/magnaporthe grisea /MultiHome.html),輸入檢索詞heavy metal tolerance protein,獲取所有重金屬抗性蛋白基因序列及其相關信息。利用稻瘟病菌在線網站(http:∥fungi.ensembl.org/Magnaporthe_oryzae/Info/Index),通過基因座位號檢索確定重金屬抗性蛋白基因的外顯子數、編碼外顯子數、內含子數和基因在染色體上的位置,并利用MapChart 2.2軟件,參照基因和染色體的長度按比例進行繪制。
1.2 稻瘟病菌重金屬抗性蛋白的系統進化分析
將稻瘟病菌重金屬抗性蛋白序列提交到NCBI(http:∥www.ncbi.nlm.nih.gov/),利用同源性分別獲取相應的同源蛋白,然后利用ClustalX 2.0軟件進行氨基酸序列多重匹配分析,參數設為默認值,所生成的 .aln文檔用MEGA 4.0軟件轉化為.meg文檔后,進一步用Neighbor-Joining法繪制系統進化樹,其中Test of Phylogeny選擇Bootstrap,Replications=500,Random seed=64238,Model選擇poisson correction。
1.3 稻瘟病菌重金屬抗性蛋白的理化特性分析與亞細胞定位
利用ExPASy(http:∥web.expasy.org/prot-param/)在線軟件,分析稻瘟病菌重金屬抗性蛋白的分子量、等電點、親水性及穩定性;利用SignalP 4.1在線軟件(http:∥www.cbs.dtu.dk/services/SignalP/)進行信號肽預測,其中organism group選真核生物,其余參數均設為默認值;利用亞細胞定位在線軟件ProtcComp v.9.0(http:∥linux1.softberry.com/berry.phtml?topic=protcompan&group=programs&subgroup=proloc)確定各蛋白在細胞中的具體位置。
1.4 稻瘟病菌重金屬抗性蛋白的結構域分析
首先利用Smart(http:∥smart.embl-heidelberg.de/)在線網站中的所有鏈接數據庫對各重金屬抗性蛋白進行結構域預測分析,然后利用在線軟件DomainDraw(http:∥domaindraw.imb.uq.edu.au/),根據蛋白質分子總長度及結構域起始與結束位置按比例繪制結構域示意圖。
1.5 稻瘟病菌重金屬抗性蛋白的三級結構分析
利用蛋白質三級結構在線預測軟件(http:∥zhanglab.ccmb.med.umich.edu/)中的I-TASSER預測重金屬抗性蛋白的三級結構,獲取三級結構3D模型以及相關信息[11]。
1.6 稻瘟病菌重金屬抗性蛋白的EST表達分析
首先利用NCBI中的BLAST蛋白在線比對軟件,比對方式選擇tblastn,選擇Expressed sequence tags數據庫,導入FASTA格式蛋白序列,獲取相應mRNA序列的version號,然后利用NCBI中的UniGene程序分析預測稻瘟病菌重金屬抗性蛋白的表達情況。
2 結果與分析
2.1 重金屬抗性蛋白及基因的獲取與確定
本研究從稻瘟病菌數據庫中共獲取3個重金屬抗性蛋白及相應基因,其中蛋白ID分別為XP_003712732、XP_003709867和XP_003713406,基因座位分別為MGG_05190、MGG_11754和MGG_14069(表1)。3個重金屬抗性蛋白在大小及外顯子與內含子數方面均存在一定差異。從表1可知,XP_003712732蛋白最大,但其外顯子數與實際的編碼外顯子數并不相同,因此該蛋白是一個提前終止的蛋白,而XP_003713406蛋白所對應基因的外顯子數和內含子數最多,因此MGG_14069基因在成熟轉錄體的形成上可能較為復雜。
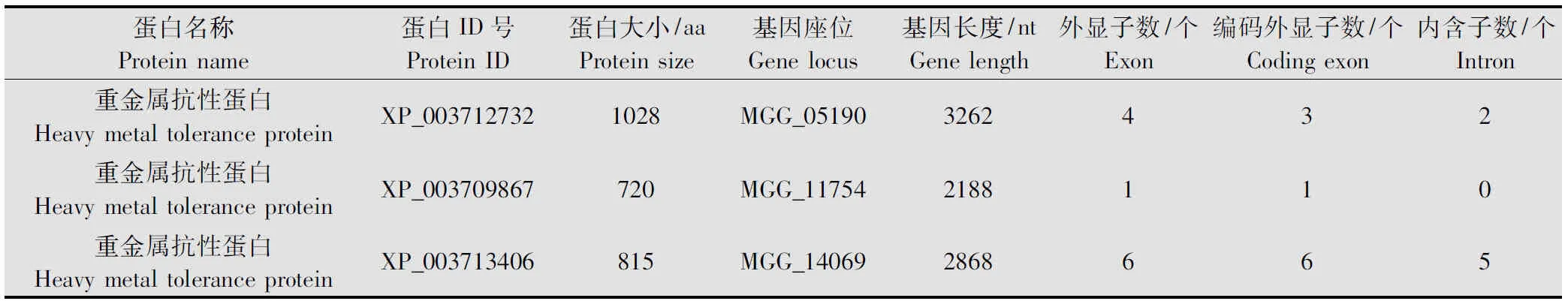
表1 稻瘟病菌重金屬抗性蛋白及基因的相關信息
2.2 重金屬抗性蛋白的系統進化分析及相應基因的染色體定位
為了明確稻瘟病菌重金屬抗性蛋白的進化關系,本文利用MEGA 4.0軟件,采用Neighbor-Joining法繪制了系統進化樹。圖1顯示,稻瘟病菌的3種重金屬抗性蛋白沒有聚在一起,遺傳距離較遠,因此進化關系較復雜。其中XP_003712732與全蝕病菌中的EJT81035具有相同結點,遺傳距離相等,表明兩者同源性最高;而與XP_003713406遺傳距離相等的是粗糙脈孢菌的NCU03228,表明兩者同源性最高,親緣關系也最近;而XP_003709867單獨成一分支,與XP_003712732和XP_003713406的遺傳距離均較遠,表明其在進化上經歷了不同的進化途徑。經染色體定位分析表明,3種稻瘟病菌重金屬抗性蛋白分別位于不同的染色體上,結果如圖2所示:其中XP_003709867位于第1號染色體上,XP_003713406位于第2號染色體上,XP_003712732位于第3號染色體上,并且均分布在染色體的兩端位置,這與3種蛋白進化途徑的差異有一定的對應性。

圖1 稻瘟病菌重金屬抗性蛋白的系統進化樹Fig.1 Phylogenetic tree of heavy metal tolerance proteins in Magnaporthe oryzae
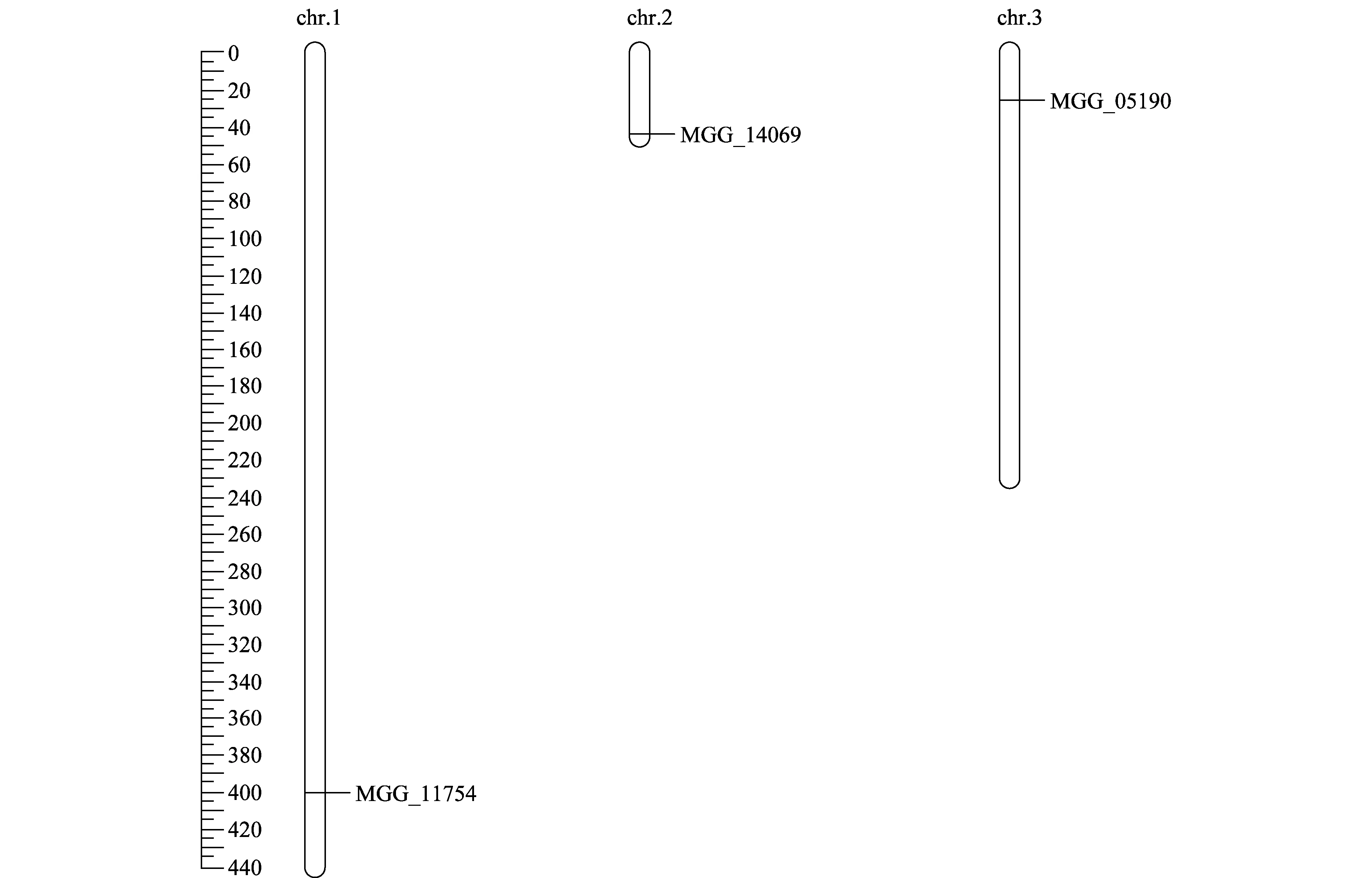
圖2 稻瘟病菌重金屬抗性蛋白的染色體定位Fig.2 Chromosomal localization of heavy metal tolerance proteins in Magnaporthe oryzae
2.3 重金屬抗性蛋白的理化特性與亞細胞定位
從表2可以看出,3種蛋白質的分子量存在一定的差異,其中XP_003712732分子量最大,為114.64 ku,XP_003709867分子量最小,為79.59 ku;3種蛋白均不含信號肽,屬于非分泌型的親水蛋白;在等電點方面,XP_003712732和XP_003709867偏堿性,XP_003713406偏酸性;在穩定性方面, XP_003709867和XP_003713406為穩定性蛋白,而XP_003712732屬于不穩定蛋白,為進一步確定重金屬抗性蛋白的特征,筆者利用在線軟件ProtcComp v.9.0進行亞細胞定位分析,結果顯示,3種蛋白質皆存在于線粒體中。
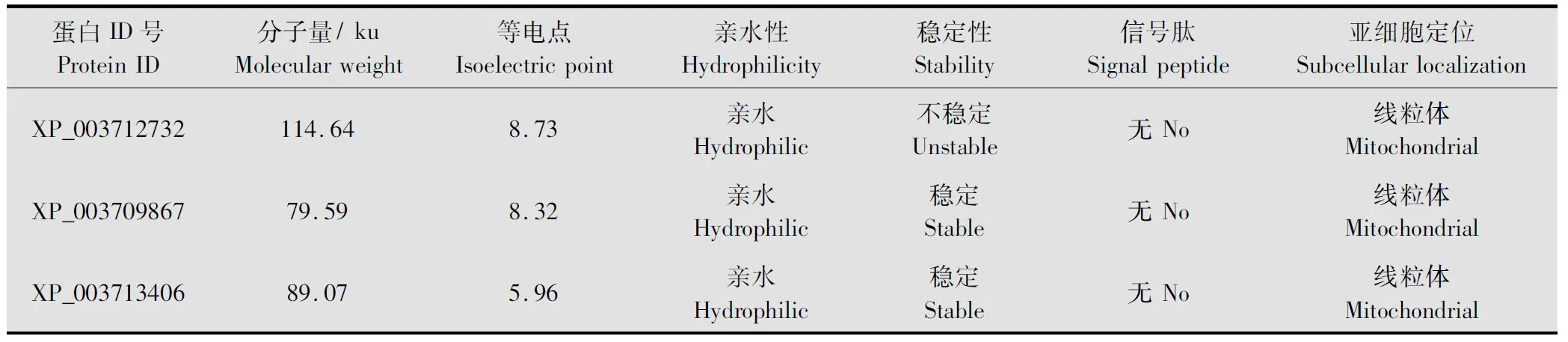
表2 稻瘟病菌重金屬抗性蛋白的理化性質及亞細胞定位
2.4 重金屬抗性蛋白的結構域分析
利用NCBI網站和Smart在線軟件對稻瘟病菌重金屬抗性蛋白的結構域進行了分析預測,結果如圖3所示,在蛋白質的N端均具有transmembrane region結構域,但XP_003712732含有的數量較多,而在XP_003713406中只含有一個transmembrane region結構域。在蛋白質的中間區域和C端部分, 3種蛋白有較大的不同,其中,XP_003709867不含ABC_membrane結構域,而其余2種蛋白均含有該結構域,同時只有XP_003712732的C端含有low complexity結構域,AAA結構域在3種蛋白中均含有。因此中間區域和C端部分可能是稻瘟病菌重金屬抗性蛋白分類的根本特征,更是形成功能多樣性的決定部分。
2.5 重金屬抗性蛋白的三級結構預測
蛋白質的功能與其三維結構密切相關,筆者利用蛋白質三級結構在線預測軟件中的I-TASSER成功預測出了3個重金屬抗性蛋白的三維結構,結果
見表3和圖4。3種蛋白質三級結構預測的標準化Z值(Norm.Z-score)值均大于1,因此三級結構的預測準確性高。同時通過與模板的比對參數來看,XP_003713406同一性值最低,分別為0.20和0.17;結合三維結構圖進行比較,XP_003712732和XP_003709867兩種蛋白質的結構較相似,兩者與XP_003713406的結構差異較大。
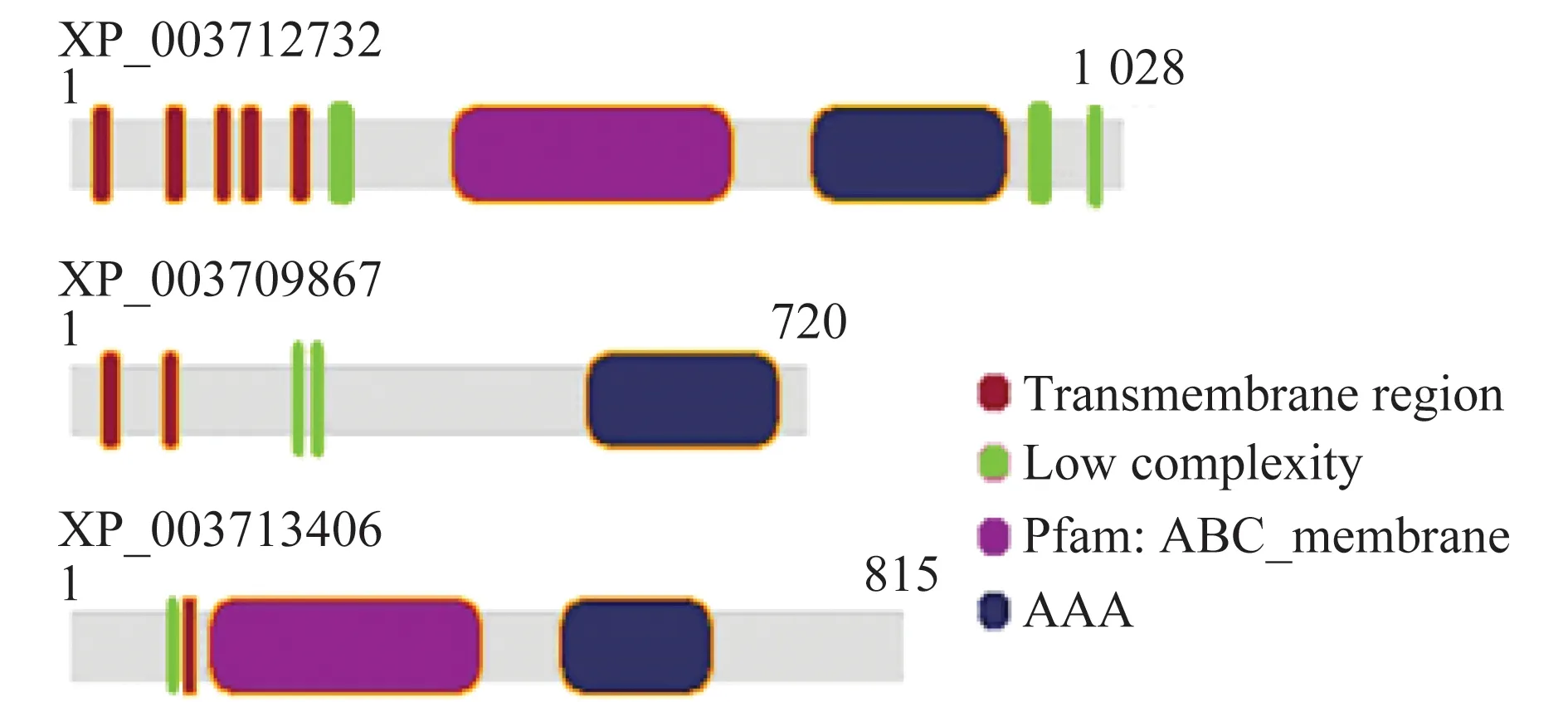
圖3 稻瘟病菌重金屬抗性蛋白的功能結構域示意圖Fig.3 Functional domains of heavy metal tolerance proteins in Magnaporthe oryzae

蛋白ID號ProteinID一致性1Identity1一致性2Identity2覆蓋率Coveragerate標準化Z值Norm.Z-scoreXP_0037127320.360.230.573.51XP_0037098670.270.270.813.59XP_0037134060.200.170.704.18
1) 一致性1為線性對齊區域模板與本研究蛋白序列的相似性百分比;一致性2為整個模板鏈與本研究蛋白序列的相似性百分比。
Identity 1 is the percentage sequence identity of the templates in the threading aligned region with the query sequence; Identity 2 is the percentage sequence identity of the whole template chains with the query sequence.
2.6 重金屬抗性蛋白基因的EST表達分析
對稻瘟病菌重金屬抗性蛋白基因進行EST表達分析,結果如表4所示。MGG_14069結果未知,MGG_05190在分生孢子中表達,MGG_11754在菌絲體中表達。由此可見,重金屬抗性蛋白基因可能在維持稻瘟病菌正常生長發育及繁殖等過程中扮演著重要角色,同時這種表達部位上的差異性也許是稻瘟病菌的營養體與繁殖體適應重金屬逆境的一種策略。
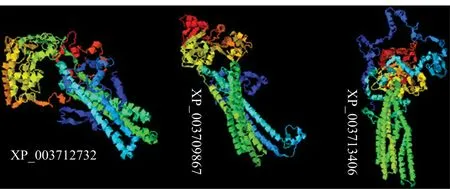
圖4 XP_003712732、XP_003709867和XP_003713406的三級結構模型Fig.4 The tertiary structure models of XP_003712732,XP_003709867 and XP_003713406

蛋白IDProteinID基因座位Genelocus分生孢子Conidium菌絲體Mycelium未知UnknownXP_003712732MGG_05190●XP_003709867MGG_11754●XP_003713406MGG_14069●
1) ●代表表達部位。
● indicates expression site.
3 結論與討論
目前,對植物細胞內重金屬抗性基因的研究較為普遍,多種重金屬抗性蛋白與重金屬離子之間的聯系已被揭示,并且已分離鑒定了諸多相關蛋白家族[12],但對稻瘟病菌重金屬抗性蛋白的研究卻較少。本研究通過生物信息學方法共獲得3個稻瘟病菌重金屬抗性蛋白及相應基因序列,同時進行了相應分析。
蛋白質的理化性質和結構特征對其功能具有決定作用[13]。研究發現,3個重金屬抗性蛋白不含信號肽,具有親水性,在等電點、分子量、氨基酸數目等方面存在一定的差異,這與進化分析中存在差異性的結果相一致。在對重金屬抗性蛋白進行亞細胞定位時發現,3個重金屬抗性蛋白均位于線粒體中,這與前人對水稻的研究結果基本一致[14]。XP_003712732的穩定性較差,推測其可能在發揮重金屬抗性作用時效果不顯著。
本研究發現3個重金屬抗性蛋白各具有1個AAA保守結構域。XP_003712732和XP_003713406還各具有1個ABC_membrane非保守結構域。ABC_membrane具有跨膜轉運功能,這一結構域對于稻瘟病菌在處于高濃度或有毒重金屬環境下,將有毒或過量的重金屬轉運出細胞,保證真菌正常的生長、繁殖尤為重要。通過表達部位分析發現,MGG_05190和MGG_11754兩個基因分別在分生孢子和菌絲體中表達,而MGG_14069基因未獲得確切的表達部位。蛋白XP_003712732穩定性較XP_003713406差,XP_003713406與XP_003709867相比呈略酸性,分子量相對較大,推測蛋白XP_003713406可能在稻瘟病菌的重金屬抗性方面地位更為主要,值得在今后的正反向遺傳學研究中進行重要關注。
[1] Bednarek P, Osbourn A. Plant-microbe interactions:chemical diversity in plant defense [J]. Science, 2009, 324(5928):746-748.
[2] Talbot N J. On the trail of a cereal killer:Exploring the biology ofMagnaporthegrisea[J]. Annual Review of Microbiology, 2003, 57:177-202.
[3] 林雄杰, 周潔, 王宗華,等. 稻瘟病菌幾丁質結合蛋白的生物信息學分析[J]. 中國農學通報, 2011, 27(21):239-245.
[4] Peters J M, Wiley L M, Zidenberg-Cherr S, et al. Periconceptional zinc deficiency affects uterine3H-estradiol binding in mice [J]. Teratogenesis, Carcinogenesis, and Mutagenesis, 1995, 15(1):23-31.
[5] 池振明.現代微生物生態學[M].第2版.北京:科學出版社,2010.
[6] Pal A, Dutta S, Paul A K. Reduction of hexavalent chromium by cell-free extract ofBacillussphaericusAND 303 isolated from serpentine soil [J]. Current Microbiology, 2005, 51(5):327-330.
[7] Endo G, Silver S. CadC, the transcriptional regulatory protein of the cadmium resistance system ofStaphylococcusaureusplasmid pI258 [J].Journal of Bacteriology,1995,177(15):4437-4441.
[8] 田美娟.深海重金屬抗性菌的分離、鑒定以及菌株BrevibacteriumsanguinisNCD-5鉻(Ⅵ)還原機制的初探[D].廈門:廈門大學,2006.[9] Xie Pin, Hao Xiuli, Mohamad O A, et al. Comparative study of chromium biosorption byMesorhizobiumamorphaestrain CCNWGS0123 in single and binary mixtures [J]. Applied Biochemistry and Biotechnology, 2013, 169(2):570-587.
[10]Magyarosy A, Laidlaw R D, Kilaas R, et al. Nickel accumulation and nickel oxalate precipitation byAspergillusniger[J]. Applied Microbiology and Biotechnology, 2002, 59(2/3):382-388.
[11]Zhang Yang. I-TASSER server for protein 3D structure prediction [J]. BMC Bioinformatics, 2008, 9:40.
[12]金楓, 王翠, 林海建,等. 植物重金屬轉運蛋白研究進展[J]. 應用生態學報, 2010, 21(7):1875-1882.
[13]Itoh H, Ueguchi-Tanaka M, Sato Y, et al. The gibberellin signaling pathway is regulated by the appearance and disappearance of SLENDER RICE1 in nuclei [J]. Plant Cell, 2002, 14(1):57-70.
[14]Sahoo R K, Tuteja N. OsSUV3 functions in cadmium and zinc stress tolerance in rice (OryzasativaL.cv IR64)[J]. Plant Signaling & Behavior, 2014, 9(1):e27389.
(責任編輯:楊明麗)
Bioinformatics analysis of heavy metal tolerance proteins inMagnaportheoryzae
Fang Shumei, Wang Wei, Du Chongxiao, Zhao Donglei, Li Chao, Liang Xilong
(Heilongjiang Bayi Agricultural University, Daqing 163319, China)
Heavy metal tolerance proteins are secretory protein produced byMagnaportheoryzaeduring its evolution. They may play an important role in its growth, reproduction and life cycle in the environment polluted with high concentrations of heavy metals. In this study, three heavy metal tolerance proteins have been obtained inM.oryzaedatabase. To identify their characteristics, some websites and softwares were used to analyze the physiochemical property, domains, tertiary structure, subcellular localization and tissue-specific expression. Moreover, the phylogenetic relationship was analyzed by constructing phylogenetic tree. These results may provide a guideline for further study on the function of heavy metal tolerance proteins in the development, reproduction and pathogenesis ofM.oryzae.
bioinformatics;Magnaportheoryzae; heavy metal tolerance protein; structural analysis; physicochemical property
2014-08-16
2014-09-30
國家級大學生創新創業訓練計劃項目(201310223001);國家自然科學基金(31201163);黑龍江省教育廳科學技術研究(12521381)
Q811.4
A
10.3969/j.issn.0529-1542.2015.04.016
* 通信作者 E-mail:790007483@qq.com
# 共同第一作者

