基于分子描述符和機(jī)器學(xué)習(xí)方法預(yù)測(cè)和虛擬篩選乳腺癌靶向蛋白HEC1抑制劑
何 冰 羅 勇 李秉軻 薛 英,3 余洛汀,*邱小龍 楊登貴
(1四川大學(xué)華西醫(yī)院生物治療國(guó)家重點(diǎn)實(shí)驗(yàn)室, 腫瘤中心及生物治療協(xié)同創(chuàng)新中心, 成都 610041;2成都師范學(xué)院化學(xué)與生命科學(xué)學(xué)院, 成都 611130; 3四川大學(xué)化學(xué)學(xué)院, 成都 610064;4江蘇兆邦生物醫(yī)藥研究院有限公司, 江蘇 南通 226000; 5江蘇海門(mén)慧聚藥業(yè)有限公司, 江蘇 海門(mén) 226123)
基于分子描述符和機(jī)器學(xué)習(xí)方法預(yù)測(cè)和虛擬篩選乳腺癌靶向蛋白HEC1抑制劑
何 冰1,2羅 勇1李秉軻2薛 英1,3余洛汀1,*邱小龍4,5楊登貴4
(1四川大學(xué)華西醫(yī)院生物治療國(guó)家重點(diǎn)實(shí)驗(yàn)室, 腫瘤中心及生物治療協(xié)同創(chuàng)新中心, 成都 610041;2成都師范學(xué)院化學(xué)與生命科學(xué)學(xué)院, 成都 611130;3四川大學(xué)化學(xué)學(xué)院, 成都 610064;4江蘇兆邦生物醫(yī)藥研究院有限公司, 江蘇 南通 226000;5江蘇海門(mén)慧聚藥業(yè)有限公司, 江蘇 海門(mén) 226123)
HEC1(癌癥高表達(dá)蛋白)是紡錘體檢查點(diǎn)控制、著絲粒功能、細(xì)胞存活的關(guān)鍵的有絲分裂調(diào)節(jié)器, 與原發(fā)性乳腺癌的不良預(yù)后有關(guān). 篩選具有高親和力的HEC1新型抑制劑對(duì)探索乳腺癌的靶向治療具有重要意義.本文從結(jié)構(gòu)多樣性的化合物庫(kù)中篩選HEC1抑制劑. 通過(guò)對(duì)分子描述符的特征篩選, 采用支持向量機(jī)(SVM)和隨機(jī)森林(RF)方法分別對(duì)HEC1抑制劑和非抑制劑建立了分類模型. 經(jīng)對(duì)比, RF模型顯示了更好的預(yù)測(cè)精度.我們采用RF模型對(duì)HEC1抑制劑進(jìn)行了虛擬篩選, 從“in-house”實(shí)體庫(kù)篩選得到2個(gè)潛在的HEC1抑制劑分子.隨后對(duì)篩出的化合物進(jìn)行了體外活性實(shí)驗(yàn), 發(fā)現(xiàn)對(duì)乳腺癌細(xì)胞株MDA-MB-468和MDA-MB-231均有一定程度的抗腫瘤活性. 研究結(jié)果表明, 機(jī)器學(xué)習(xí)方法對(duì)于設(shè)計(jì)和虛擬篩選HEC1抑制劑有良好的效果.
HEC1; 選擇性抑制劑; 機(jī)器學(xué)習(xí)方法; 支持向量機(jī); 隨機(jī)森林; 虛擬篩選
1 引 言
乳腺癌是女性癌癥高發(fā)性惡性腫瘤, 全球范圍內(nèi)位于女性惡性腫瘤首位, 嚴(yán)重威脅女性身心健康.乳腺癌靶向治療是在分子水平對(duì)其通路靶點(diǎn)設(shè)計(jì)藥物, 通過(guò)藥物與受體或調(diào)節(jié)分子結(jié)合, 下調(diào)受體表達(dá)或者活化下游基因, 使得腫瘤細(xì)胞凋亡或者抑制其生長(zhǎng). 目前乳腺癌常見(jiàn)靶向藥物有人表皮生長(zhǎng)因子受體(HER)靶向藥物曲妥珠單抗(rastuzumab)、帕妥珠單抗(pertuzumab)、西妥昔單抗(cetuximab);還有小分子酪氨酸激酶抑制劑吉非替尼(gefitinib)、厄洛替尼(erlotinib)、拉帕替尼(lapatinib); 哺乳動(dòng)物雷帕霉素靶蛋白靶向藥物依維莫司(everolimus); 血管內(nèi)皮生長(zhǎng)因子(VEGF)靶向藥物貝伐單抗(bevacizumab); 多聚二磷酸腺苷核糖聚合酶靶向藥物抑制劑Olaparib已進(jìn)入臨床II期.1
HEC1是癌癥高表達(dá)蛋白,2相對(duì)于正常細(xì)胞, 它在癌癥細(xì)胞系中的表達(dá)量更高. HEC1也是一類周期蛋白, 主要在G2/M期表達(dá), 參與有絲分裂、動(dòng)粒組裝、有絲分裂檢驗(yàn)點(diǎn)以及染色體穩(wěn)定性的維持等, 在腫瘤的發(fā)生發(fā)展中占有重要作用. 利用小分子抑制劑來(lái)研究HEC1在癌癥中高表達(dá)的作用對(duì)于臨床具有極大的意義.3
HEC1抑制劑相關(guān)機(jī)理已有不少研究.4–6Wu等7通過(guò)酵母雙雜交篩選得到靶向Hec1/Nek2復(fù)合物的小分子抑制劑INH1, 該抑制劑通過(guò)抑制Nek2對(duì)Hec1的磷酸化從而破壞Nek2-Hec1通路, 另一方面在降低Nek2表達(dá)量同時(shí)并不降低Hec1表達(dá)量的前提下, 導(dǎo)致中期染色體排列錯(cuò)誤, 細(xì)胞不能進(jìn)行正常的有絲分裂, 最終導(dǎo)致細(xì)胞的死亡. 這在腫瘤治療中可能具有重要意義. Qiu等8運(yùn)用反向酵母雙雜交系統(tǒng)篩選得到特異性阻斷Hec1與Nek2蛋白–蛋白相互作用的小分子化合物, 該小分子能夠特異性降低Hec1的磷酸水平, 能夠?qū)е氯旧w不穩(wěn)定現(xiàn)象的出現(xiàn). 目前報(bào)道的Hec1/Nek2的抑制劑主要是加州大學(xué)歐文分校研究的INH系列化合物.9–12
篩選得到對(duì)HEC1蛋白高親和力、高選擇性的小分子抑制劑, 特異性阻斷Nek2對(duì)HEC1的磷酸化作用, 對(duì)于乳腺癌靶向治療有重要意義. 機(jī)器學(xué)習(xí)方法在預(yù)測(cè)化合物的藥效動(dòng)力學(xué)、藥代動(dòng)力學(xué)和毒性等方面有非常好的效果.13–15本研究采用支持向量機(jī)(SVM)和隨機(jī)森林(RF)兩種機(jī)器學(xué)習(xí)方法, 通過(guò)內(nèi)部五重交叉驗(yàn)證和袋外數(shù)據(jù)(OOB)估計(jì)對(duì)訓(xùn)練模型進(jìn)行優(yōu)化, 以期獲得良好的預(yù)測(cè)結(jié)果. 隨后, 我們從這兩種模型中選取出與HEC1抑制劑相關(guān)的物理化學(xué)特征, 用于HEC1潛在抑制劑的虛擬篩選, 并對(duì)篩選出的已有化合物進(jìn)行了體外活性測(cè)試.
2 材料與方法
2.1 分子描述符
分子描述符是對(duì)分子的結(jié)構(gòu)和物理化學(xué)屬性的定量描述符號(hào), 廣泛應(yīng)用于定量結(jié)構(gòu)活性關(guān)系(QSAR)和其他機(jī)器學(xué)習(xí)模型中, 用于預(yù)測(cè)各種藥物及小分子的毒理學(xué)與藥理學(xué)特征.16–22我們采用課題組編寫(xiě)的分子描述符計(jì)算程序, 采用AM1半經(jīng)驗(yàn)方法對(duì)化合物三維(3D)結(jié)構(gòu)進(jìn)行優(yōu)化, 然后分別計(jì)算了化合物189個(gè)描述符相對(duì)應(yīng)的取值. 隨后, 我們對(duì)SVM模型采用遞歸特征消除(RFE)方法, 對(duì)RF模型使用程序自帶的隨機(jī)選擇方法, 剔除掉這189個(gè)描述符中與體系不相干的或多余的描述符, 僅保留和體系最相關(guān)的關(guān)鍵描述符, 用于進(jìn)一步虛擬篩選或其它研究. 這兩種特征選擇方法將在后文中詳細(xì)介紹. 本研究涉及的189個(gè)分子描述符組參見(jiàn)表S1(Supporting Information), 這189個(gè)描述符的詳細(xì)說(shuō)明請(qǐng)參見(jiàn)我們之前的文章.16–19,23
2.2 數(shù)據(jù)集
本研究所用的數(shù)據(jù)集為122個(gè)HEC1抑制劑和122個(gè)HEC1非抑制劑. HEC1抑制劑均從文獻(xiàn)收集得到.8,24為保證所選出的HEC1抑制劑具備一定程度的高效性, 我們?nèi)コ薍EC1抑制劑中的IC50值大于10 μmolL–1的分子, 確定HEC1抑制劑的IC50值范圍為7.5 到9431 nmolL–1. 通過(guò)對(duì)以上范圍抑制劑分子描述符的計(jì)算, 我們將具有重復(fù)結(jié)構(gòu)的分子舍棄,最終確定了122個(gè)HEC1抑制劑加入到數(shù)據(jù)集中.
為充分表示HEC1非抑制劑的廣泛性并作為對(duì)比, 我們根據(jù)k均值聚類25的方法, 從MDDR (MDL Drug Data Report)數(shù)據(jù)庫(kù)中, 挑選出假定存在且對(duì)HEC1沒(méi)有抑制作用的非抑制劑. 我們把MDDR中的未被報(bào)道具有任何HEC1抑制活性的分子(去除掉有無(wú)效的結(jié)構(gòu)或分子描述符的分子, 剩下超過(guò)15萬(wàn)個(gè)分子), 根據(jù)計(jì)算得到它們的189個(gè)分子描述符的取值, 并分成122個(gè)聚類, 然后挑選出每個(gè)聚類中距離相應(yīng)聚類的質(zhì)心最近的分子, 即得到122個(gè)HEC1非抑制劑化合物.
對(duì)選定的244個(gè)分子, 均通過(guò)ChemBioDraw軟件繪制得到其二維(2D)結(jié)構(gòu), 并用Corina軟件將2D結(jié)構(gòu)轉(zhuǎn)換為3D結(jié)構(gòu), 再用ChemBio3D軟件對(duì)3D結(jié)構(gòu)進(jìn)行優(yōu)化.26,27根據(jù)這些分子在化學(xué)空間中的相似性和分布的廣泛性, 我們將122個(gè)抑制劑和122個(gè)非抑制劑其分成HEC1的訓(xùn)練集和測(cè)試集. 訓(xùn)練集和測(cè)試集中化合物的結(jié)構(gòu)、類別和來(lái)源參見(jiàn)表S2和S3 (Supporting Information).
2.3 機(jī)器學(xué)習(xí)方法
本研究采用了兩種最新的機(jī)器學(xué)習(xí)方法——SVM和RF, 通過(guò)SVM和RF方法執(zhí)行的預(yù)測(cè)可以歸結(jié)為一個(gè)二元分類問(wèn)題(即分為HEC1抑制劑或非抑制劑). 對(duì)于SVM和RF理論的深入描述可以很容易從一些經(jīng)典著作和優(yōu)秀論文中獲得, 因此這里僅對(duì)SVM和RF分類的主要思想做一個(gè)簡(jiǎn)述.
SVM是一種基于統(tǒng)計(jì)學(xué)習(xí)理論中的結(jié)構(gòu)風(fēng)險(xiǎn)最小化(SRM)原則的方法, 而統(tǒng)計(jì)學(xué)習(xí)理論是一種著名的與核函數(shù)相關(guān)的機(jī)器學(xué)習(xí)方法.28,29SVM方法通過(guò)使用核函數(shù)(本研究使用高斯徑向基函數(shù)(RBF), 把輸入變量投射到高維特征空間中, 然后從輸入向量中選擇一個(gè)所謂支持向量的小的子集, 在變換之后的空間中, 通過(guò)最大間隔的原則構(gòu)建一個(gè)最優(yōu)化的分類超平面, 從而把這些輸入向量分成了兩種不同的類別.30
RF方法則是一種決策樹(shù)自然生長(zhǎng)且很多個(gè)決策樹(shù)預(yù)測(cè)器組合在一起的分類方法. 每棵決策樹(shù)依賴于對(duì)輸入向量進(jìn)行隨機(jī)獨(dú)立抽樣所獲得的數(shù)值,且森林中的所有決策樹(shù)都具有相同的分布.31,32每棵樹(shù)都不受干涉地自然生長(zhǎng)到最大的規(guī)模, 然后對(duì)于一個(gè)新的數(shù)據(jù)點(diǎn)給出自己的預(yù)測(cè). 也就是說(shuō), 這顆樹(shù)投票決定這一新數(shù)據(jù)點(diǎn)的類別. 當(dāng)大量的決策樹(shù)生成以后, 整個(gè)森林就選擇最多數(shù)的投票結(jié)果作為對(duì)這個(gè)數(shù)據(jù)點(diǎn)類別的判定. 在對(duì)訓(xùn)練集的每次引導(dǎo)過(guò)程中, 大約三分之一的實(shí)例在訓(xùn)練過(guò)程遺漏而組成袋外數(shù)據(jù)估計(jì)(OOB)樣本. 作為評(píng)估總預(yù)測(cè)精度指標(biāo)的一部分, OOB在內(nèi)部評(píng)估了RF的性能, 顯示了使用相同大小的測(cè)試集具有和訓(xùn)練集同樣的精確度, 是一種和經(jīng)典的交叉驗(yàn)證相類似的新的評(píng)估方法.33,34此外, RF可以根據(jù)對(duì)變量值隨機(jī)排序所引起的系統(tǒng)性能降低的程度, 選擇出對(duì)模型預(yù)測(cè)能力相對(duì)重要的描述符.18盡管分子的某種特定描述符與其活性之間的關(guān)系被隱藏在一個(gè)“黑箱”中, RF模型仍然可以在訓(xùn)練過(guò)程中衡量每個(gè)描述符對(duì)于預(yù)測(cè)精度的貢獻(xiàn)程度.35
2.4 特征選擇方法和模型建立
引入RFE方法可以提高機(jī)器學(xué)習(xí)方法的分類性能, 并能篩選出一些最相關(guān)的描述符, 這些描述符可以把數(shù)據(jù)集區(qū)分為陰性和陽(yáng)性兩類. RFE在生物化學(xué)很多領(lǐng)域, 例如藥代動(dòng)力學(xué)相關(guān)的藥物活性以及毒理學(xué)屬性等,14,15,16,20顯示出了高效性分類和預(yù)測(cè)能力, 并且發(fā)現(xiàn)很多有價(jià)值的特征, 獲得了令人滿意的推廣. 因此, RFE和SVM相結(jié)合的方法(SVM + RFE)一開(kāi)始就被應(yīng)用于本研究中. 我們通過(guò)五重交叉驗(yàn)證方法, 從訓(xùn)練集的預(yù)測(cè)結(jié)果中分別選擇了與HEC1抑制劑密切相關(guān)的一組描述符, 并使用RFE方法, 從整個(gè)訓(xùn)練集中產(chǎn)生一個(gè)有RFE特征的SVM模型.
對(duì)RF模型的每棵樹(shù), 均由隨機(jī)選擇的mtry個(gè)描述符和與訓(xùn)練集不同的自引導(dǎo)樣本共同建立. RF模型的性能通過(guò)OOB估計(jì)從內(nèi)部進(jìn)行評(píng)估, OOB測(cè)試所選用的數(shù)據(jù)與構(gòu)建森林所用到的數(shù)據(jù)互相獨(dú)立.在RF的訓(xùn)練過(guò)程中, 描述符的相對(duì)重要性亦可通過(guò)其自帶的特征選擇程序計(jì)算得到.
2.5 性能評(píng)估
機(jī)器學(xué)習(xí)方法的預(yù)測(cè)效果可以通過(guò)真陽(yáng)性(TP), 真陰性(TN), 假陽(yáng)性(FP)和假陰性(FN)這四個(gè)指標(biāo)來(lái)進(jìn)行衡量. TP是預(yù)測(cè)正確的HEC1抑制劑的數(shù)量, TN是預(yù)測(cè)正確的HEC1非抑制劑的數(shù)量, FP是把HEC1的非抑制劑錯(cuò)誤地預(yù)測(cè)成抑制劑的數(shù)量, FN是把HEC1的抑制劑錯(cuò)誤地預(yù)測(cè)成非抑制劑的數(shù)量.
一些精確性函數(shù)可以用來(lái)衡量模型的預(yù)測(cè)效果, 例如靈敏度(SE, 即HEC1抑制劑的預(yù)測(cè)精度), 特異性(SP, 即HEC1非抑制劑的預(yù)測(cè)精度), 錯(cuò)誤率(ER), 總預(yù)測(cè)精度Q, 馬氏相關(guān)系數(shù)C等. 以上這些變量和函數(shù)之間的相互作用關(guān)系如下所示:

3 結(jié)果與討論
3.1 模型建立與RFE方法的效果
SVM模型根據(jù)我們課題組發(fā)展的程序13–15建立,而RF模型則由 Breiman和Cutler36提出的Fortran代碼生成. 在SVM模型中, 聯(lián)合五重交叉驗(yàn)證方法, RFE方法的參與顯著提高了訓(xùn)練集的預(yù)測(cè)精度, 詳細(xì)結(jié)果參見(jiàn)表1.
在預(yù)測(cè)HEC1抑制劑和非抑制劑的過(guò)程中, 當(dāng)SVM的參數(shù)σ被確定為5的時(shí)候, 模型表現(xiàn)出了最好的預(yù)測(cè)效果. 對(duì)于HEC1抑制劑和非抑制劑而言, 未使用RFE方法的SVM模型(記為SVM)的平均預(yù)測(cè)精度是95.83%和98.67%; 與此相比, 使用了RFE方法的SVM模型(記為SVM + RFE)的以上數(shù)值分別為100%和100%. "SVM"模型的Q和C的平均預(yù)測(cè)值分別為97.41%和0.9484, 而"SVM + RFE"模型的Q和C值則分別是100%和1. 從以上結(jié)果可以看出, 對(duì)于HEC1抑制劑和非抑制劑, Q和C這四項(xiàng)指標(biāo)的平均預(yù)測(cè)精度而言, 結(jié)合了RFE方法的SVM模型比起單純的SVM模型顯示出了較明顯的改善. 此外, 使用RFE方法還能有效選出一組與化合物的HEC1抑制活性最相關(guān)的分子描述符.
特征選擇是通過(guò)隨機(jī)地減少森林中樹(shù)與樹(shù)之間的相關(guān)度來(lái)提高RF模型的預(yù)測(cè)性能, 因此RF模型的每個(gè)節(jié)點(diǎn)在構(gòu)建決策樹(shù)的過(guò)程中使用了特征選擇. 樹(shù)的數(shù)量ntree經(jīng)優(yōu)化取值為500, 用于生成穩(wěn)定的袋外數(shù)據(jù)估計(jì)(OBB)預(yù)測(cè)率.31參數(shù)mtry的取值范圍是從1到描述符的總個(gè)數(shù), 對(duì)于本研究1 ≤ mtry≤ 189, mtry的默認(rèn)值一般設(shè)置為描述符總個(gè)數(shù)的平方根, 即13. 本研究對(duì)每個(gè)mtry值對(duì)應(yīng)的模型都進(jìn)行了預(yù)測(cè),通過(guò)對(duì)相應(yīng)的OOB預(yù)測(cè)錯(cuò)誤率的比較, 選出使OOB預(yù)測(cè)錯(cuò)誤率最小時(shí)對(duì)應(yīng)的mtry值為最優(yōu)值, 即mtry= 9的時(shí)候.
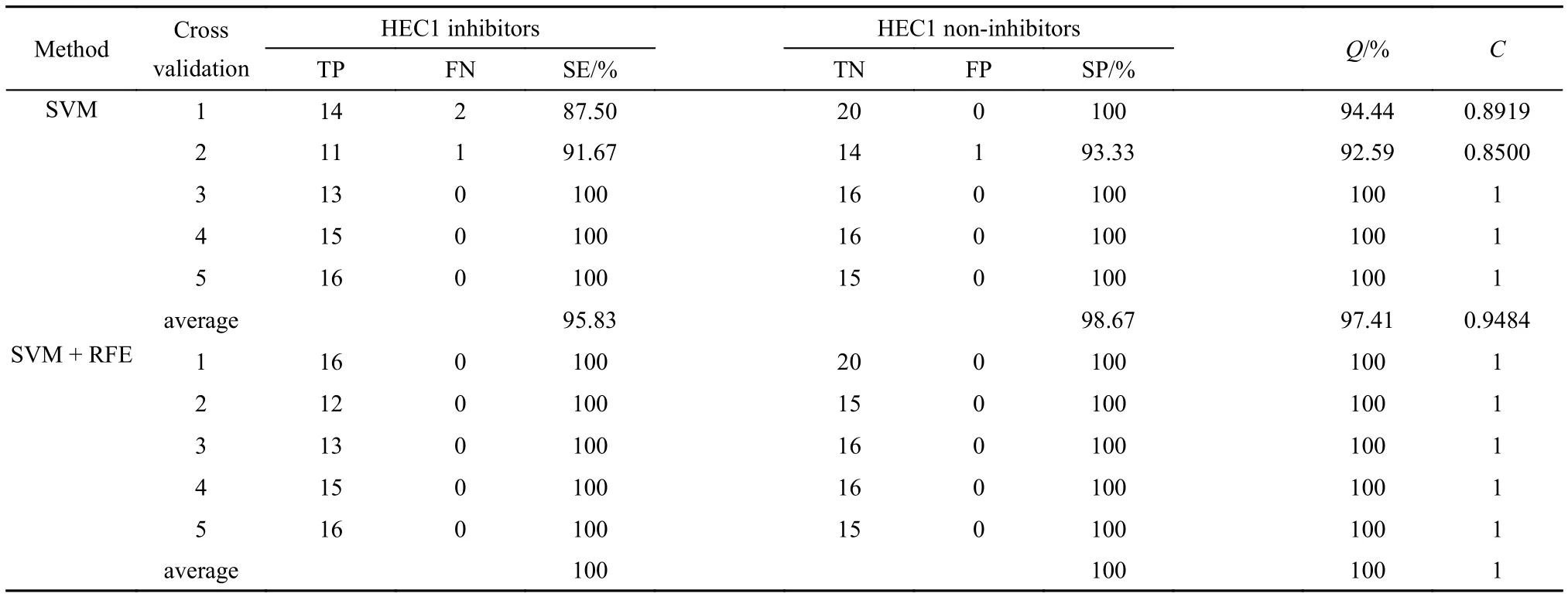
表1 SVM五重交叉驗(yàn)證預(yù)測(cè)結(jié)果Table1 Prediction performance of SVM by 5-fold cross-validation
我們通過(guò)測(cè)試集進(jìn)一步評(píng)估所生成模型的效能和可靠性. 表2給出了HEC1體系中用RFE選出的描述符所生成的SVM模型對(duì)測(cè)試集的抑制劑和非抑制劑的預(yù)測(cè)精度. 作為對(duì)比, 使用全部189個(gè)描述符所生成的SVM模型相應(yīng)的預(yù)測(cè)精度也列在表中.
在該體系中, “SVM”模型和“SVM + RFE”模型對(duì)非抑制劑的預(yù)測(cè)精度相同, 均為100%, 但是對(duì)于抑制劑而言, 使用RFE之后預(yù)測(cè)精度略微有所降低,對(duì)“SVM + RFE”模型為98%, 對(duì)“SVM”模型為100%.
根據(jù)表2數(shù)據(jù)顯示, 這些模型中所采用的分子描述符數(shù)量減少, 模型的多余度和分子描述符的不相干性得到了極大程度的降低, 體系中的描述符從189個(gè)降到了12個(gè). 表明RFE方法在選擇最相關(guān)的特征和剔除掉多余分子描述符方面是高度有效的, 大大提高了HEC1體系的抑制劑和非抑制劑分類研究的效率.
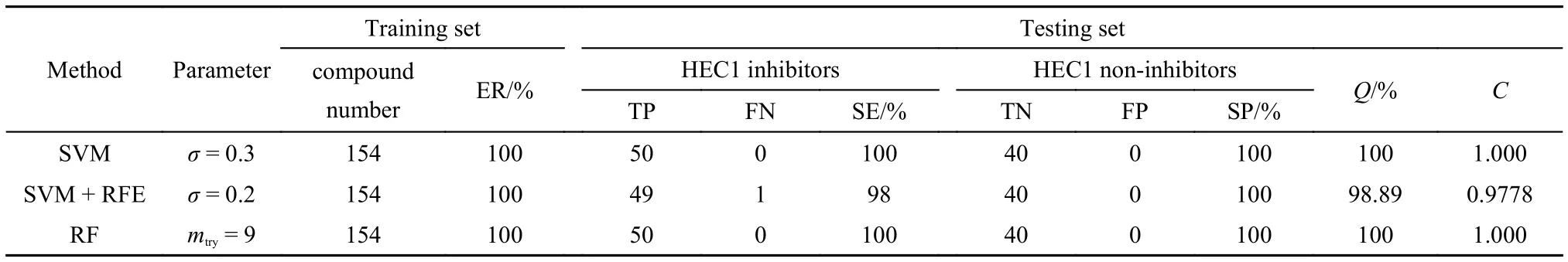
表2 預(yù)測(cè)精度的比較Table2 Comparison of the prediction accuracies
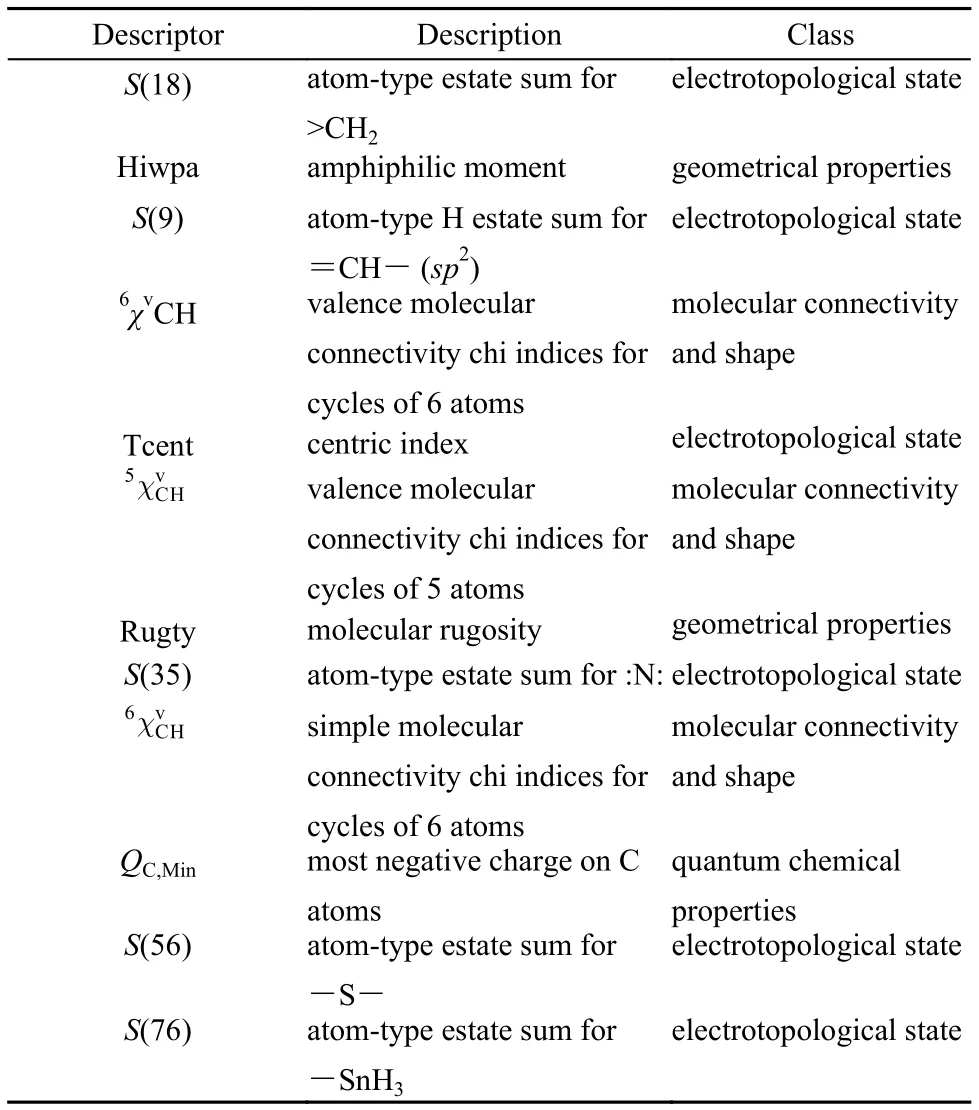
表3 SVM中SVM + RFE模型選擇出的12個(gè)最相關(guān)的描述符Table3 12 descriptors selected by (SVM + RFE) model in the SVM
3.2 模型驗(yàn)證與機(jī)器學(xué)習(xí)方法的性能
表2給出了HEC1體系中SVM和RF方法對(duì)抑制劑和非抑制劑預(yù)測(cè)精度的詳細(xì)比較, 特別是采用SVM方法所建立模型的參數(shù)值也在表中得到體現(xiàn).對(duì)于“SVM + RFE”模型的參數(shù)σ = 0.2, 而對(duì)于“SVM”模型則為0.3. 相應(yīng)的模型在這兩個(gè)參數(shù)下都給出了最好的預(yù)測(cè)結(jié)果.
如表3所示, 對(duì)于HEC1體系, SVM中“SVM + RFE”模型對(duì)抑制劑的預(yù)測(cè)精度為98%, 對(duì)非抑制劑的則為100%, 而“SVM”模型和RF方法對(duì)抑制劑和非抑制劑的預(yù)測(cè)精度均為100%. 相應(yīng)的, “SVM + RFE”模型的Q和C值分別為98.89%和0.9778, 而“SVM”模型的Q和C值和RF方法的相同, 分別是100%和1.
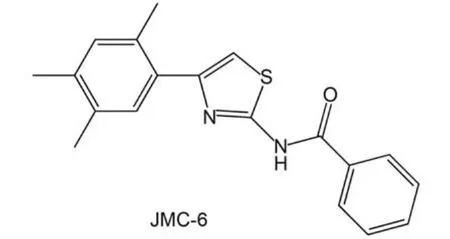
圖1 SVM模型對(duì)測(cè)試集預(yù)測(cè)錯(cuò)誤的HEC1抑制劑的結(jié)構(gòu)Fig.1 Structures of the misclassified HEC1 inhibitors by using testing set in SVM model
由于完全正確的預(yù)測(cè)準(zhǔn)確率, 使得RF模型和“SVM”模型中沒(méi)有預(yù)測(cè)錯(cuò)誤的抑制劑和非抑制劑分子. 而對(duì)于“SVM + RFE”模型, SVM的測(cè)試集中有1個(gè)抑制劑被預(yù)測(cè)錯(cuò)誤, 該抑制劑的結(jié)構(gòu)信息可以參見(jiàn)圖1. 預(yù)測(cè)錯(cuò)誤的原因可能和RFE方法的描述符較少(12個(gè))造成的預(yù)測(cè)誤差有關(guān).
基于以上數(shù)據(jù)來(lái)看, 對(duì)于HEC1體系, 盡管“SVM”模型與RF模型具有同樣優(yōu)越的預(yù)測(cè)性能, 但是“SVM”模型擁有全部189個(gè)描述符, 相較于RF模型用自帶的特征選擇方法挑出的25個(gè)最相關(guān)描述符而言過(guò)于復(fù)雜和冗余. 此外, RF方法的計(jì)算速度要比SVM方法快的多, 拋開(kāi)性能不論, 僅從時(shí)間和計(jì)算成本上考慮, 顯然RF模型更利于后續(xù)的虛擬篩選工作.
3.3 最相關(guān)描述符與對(duì)模型的解釋
在HEC1體系中, 通過(guò)特征選擇, SVM和RF預(yù)測(cè)模型提取了與抑制劑和非抑制劑活性最相關(guān)的分子描述符. 部分描述符可為特定類別化合物相關(guān)的結(jié)構(gòu)和物理化學(xué)屬性提供更深層次的了解. 通過(guò)RFE方法的SVM模型, 體系分子描述符從189個(gè)選出12個(gè)(表3).
RF模型選定了25個(gè)(表4)與預(yù)測(cè)性能高度相關(guān)的分子描述符, 并按照這些描述符對(duì)預(yù)測(cè)性能的貢獻(xiàn)值的高低而對(duì)它們進(jìn)行排序(圖2). 體系的RF模型所排出的描述符序列的前三位分別為S(56)(-S-原子類型電拓?fù)鋺B(tài)之和),5χCH(五元環(huán)的簡(jiǎn)單分子連通性Chi指數(shù))和(五元環(huán)的價(jià)分子連通性Chi指數(shù)), 這三個(gè)描述符中的兩個(gè)S(56)和也同時(shí)被體系的SVM模型的RFE方法所選出.

表4 腺苷受體模型PROCHECK評(píng)價(jià)結(jié)果Table4 Evaluation results of PROCHECK of modeled adenosine receptor
在HEC1的母核結(jié)構(gòu)中(圖3),24存在含有S原子的五元環(huán), 和所選出的貢獻(xiàn)最大的三個(gè)描述符表述一致. RF選出的nsulph描述符反映了五元環(huán)中的S原子; S(35)、S(34)反映了母核結(jié)構(gòu)中的N; S(9)、S(20)、S(25)反映了母核結(jié)構(gòu)的碳碳雙鍵; nhyd反映了母核結(jié)構(gòu)中的氫鍵. 表明所選出的描述符能夠很好地代表HEC1抑制劑的結(jié)構(gòu)特征. 除此之外, SVM模型和RF模型中還有以下重合的描述符: S(18)(>CH2原子類型電拓?fù)鋺B(tài)之和), S(9)(=CH-[sp2]原子類型H電拓?fù)鋺B(tài)之和), Tcent(中心指數(shù)), Rugty(分子粗糙度), S(35)(:N:原子類型電拓?fù)鋺B(tài)之和), QC,Min(C原子上最大的負(fù)電荷). 以上結(jié)果顯示兩種方法提取出的描述符有所交叉, 但各有自己獨(dú)特的描述符, 說(shuō)明學(xué)習(xí)過(guò)程是不同的.
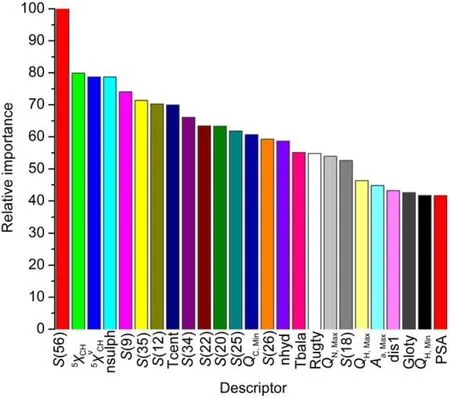
圖2 RF模型中得到的25個(gè)對(duì)HEC1抑制劑和非抑制劑預(yù)測(cè)最相關(guān)的描述符的相對(duì)重要性排名Fig.2 Relative importance of the 25 highest ranked descriptors in the RF model for the prediction of HEC1 inhibitors and non-inhibitors
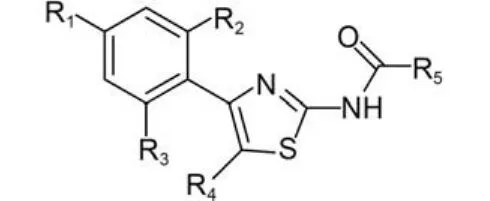
圖3 HEC1抑制劑母核結(jié)構(gòu)Fig.3 Mother nuclear structure of HEC1 inhibitors
3.4 虛擬篩選in-house數(shù)據(jù)庫(kù)
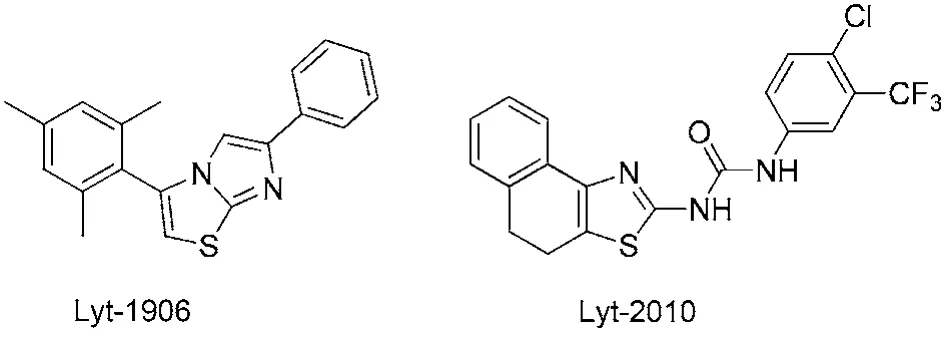
測(cè)試集用來(lái)驗(yàn)證所建立模型的性能, 預(yù)測(cè)精度越高, 說(shuō)明模型的性能就越好. 根據(jù)這一原則, 我們采用了效果更優(yōu)的RF模型來(lái)對(duì)HEC1的抑制劑進(jìn)行虛擬篩選. 我們對(duì)實(shí)驗(yàn)室“in-house”數(shù)據(jù)庫(kù)(2100個(gè)實(shí)體化合物和片段)進(jìn)行篩選, 最終得到了2個(gè)HEC1的潛在選擇性抑制劑(圖4).
我們虛擬篩選得到的兩個(gè)分子, 在結(jié)構(gòu)上和數(shù)據(jù)集的分子相比, 均有噻唑環(huán)結(jié)構(gòu)、苯環(huán)結(jié)構(gòu), 以及和噻唑2位相連接的N原子; 且和表4中貢獻(xiàn)值最大的前六位描述符S(35)相吻合. 表明篩選結(jié)果極大可能具有生物活性.

表5 化合物L(fēng)yt-1906和Lyt-2010體外增殖抑制活性Table5 Anti-proliferative activity of compounds Lyt-1906 and Lyt-2010 in vitro
3.5 活性化合物體外抗腫瘤測(cè)試
為驗(yàn)證篩選結(jié)果, 我們對(duì)篩選出的化合物L(fēng)yt-1906和Lyt-2010, 進(jìn)行了體外抗腫瘤活性測(cè)試(實(shí)驗(yàn)方法見(jiàn)Supporting Information實(shí)驗(yàn)1), 選用乳腺癌細(xì)胞株為MDA-MB-468和MDA-MB-231. 如表5所示,化合物L(fēng)yt-1906和Lyt-2010展示了較好的體外增殖抑制活性, IC50值在5.6–58.3 μmolL–1, 結(jié)果驗(yàn)證了虛擬篩選方法的可靠性, 證實(shí)RF模型對(duì)于篩選HEC1抑制劑的可靠性.
4 結(jié) 論
機(jī)器學(xué)習(xí)方法(RF和SVM)對(duì)于HEC1抑制劑與非抑制劑分類模型的建立和虛擬篩選, 具有很好的效果. 尤其對(duì)于像HEC1這樣還沒(méi)有X射線晶體結(jié)構(gòu)的靶點(diǎn)蛋白, 不能通過(guò)小分子和靶點(diǎn)蛋白對(duì)接的方法來(lái)進(jìn)行抑制劑的快速篩選, 采用機(jī)器學(xué)習(xí)方法(RF和SVM)進(jìn)行高通量虛擬篩選, 就具有很重要的現(xiàn)實(shí)意義. 我們通過(guò)對(duì)分子描述符的特征篩選, 采用SVM和RF方法分別對(duì)HEC1抑制劑和非抑制劑建立了分類模型. 在RF模型中使用RF方法得到25個(gè)分子描述符, 而SVM模型中使用RFE方法選擇出12個(gè)最相關(guān)描述符, 大幅降低了分子描述符的冗余程度.我們對(duì)兩種模型的預(yù)測(cè)效果做了對(duì)比, 采用具有更好預(yù)測(cè)性能的RF模型對(duì)HEC1抑制劑做了虛擬篩選, 得到了2個(gè)潛在的HEC1抑制化合物. 對(duì)篩選得到的化合物所進(jìn)行的體外抗腫瘤活性測(cè)試, 均顯示出一定活性. 在此基礎(chǔ)上, 后期可以通過(guò)進(jìn)一步修飾改構(gòu)和生物學(xué)實(shí)驗(yàn), 以發(fā)現(xiàn)HEC1抑制劑, 并對(duì)乳腺癌的治療發(fā)揮良好作用.
Supporting Information: The 189 molecular descriptors of this study are listed in Table S1, and the structure, category and source of compounds in the training set and test set are provided in Tables S2 and S3. Test method of antitumor activity in vitro was in experiment 1. This information isavailable free of charge via the internet at http://www.whxb.pk u.edu.cn.
(1)Gan, S. J.; Wang, Q.; Zhu, L. M.; Xie, H.; Ding, X. F. Basic & Clin. Med. 2015, 35 (1), 134. [甘紹舉, 王 青, 朱麗敏,謝 浩, 丁先鋒. 基礎(chǔ)醫(yī)學(xué)與臨床, 2015, 35 (1), 134.]
(2)Chen, Y.; Riley, D. J.; Chen, P. L.; Lee, W. H. Mol. Cell Biol. 1997, 17 (10), 6049.
(3)Du, X. L.; Wang, M. R. Acta Acad. Med. Sin. 2007, 29 (1), 137. [杜小莉, 王明榮. 中國(guó)醫(yī)學(xué)科學(xué)院學(xué)報(bào), 2007, 29 (1), 137.]
(4)Hu, C. M.; Zhu, J.; Guo, X. E.; Chen, W.; Qiu, X. L.; Ngo, B.; Chien, R.; Wang, Y. V.; Tsai, C. Y.; Wu, G.; Kim, Y.; Lopez, R.; Chamberlin, A. R.; Lee, E. H.; Lee, W. H. Oncogene 2015, 34, 1220. doi: 10.1038/onc.2014.67
(5)Huang, L. Y.; Chang, C. C.; Lee, Y. S.; Chang, J. M.; Huang, J. J.; Chuang, S. H.; Kao, K. J.; Lau, G. M.; Tsai, P. Y.; Liu, C. W.; Lin, H. S.; Lau, J. Y. Mol. Cancer Ther. 2014, 13 (6), 1419.
(6)Lee, Y. S.; Chuang, S. H.; Huang, L. Y.; Lai, C. L.; Lin, Y. H.; Yang, J. Y.; Liu, C. W.; Yang, S. C.; Lin, H. S.; Chang, C. C.; Lai, J. Y.; Jian, P. S.; Lam, K.; Chang, J. M.; Lau, J. Y.; Huang, J. J. J. Med. Chem. 2014, 57 (10), 4098. doi: 10.1021/jm401990s
(7)Wu, G.; Qiu, X. L.; Zhou, L.; Zhu, J.; Chamberlin, R.; Lau, J.; Chen, P. L.; Lee, W. H. Cancer Res. 2008, 68 (20), 8393. doi: 10.1158/0008-5472.CAN-08-1915
(8)Qiu, X. L.; Li, G.; Wu, G.; Zhu, J.; Zhou, L.; Chen, P. L.; Chamberlin, A. R.; Lee, W. H. J. Med. Chem. 2009, 52 (6), 1757. doi: 10.1021/jm8015969
(9)Chen, Y.; Riley, D. J.; Zheng, L.; Chen, P. L.; Lee, W. H. J. Biol. Chem. 2002, 277 (51), 49408. doi: 10.1074/jbc.M207069200
(10)Diaz-Rodríguez, E.; Sotillo, R.; Schvartzman, J. M.; Benezra, R. Proc. Natl. Acad. Sci. U. S. A. 2008, 105 (43), 16719. doi: 10.1073/pnas.0803504105
(11)Ferretti, C.; Totta, P.; Fiore, M.; Mattiuzzo, M.; Schillaci, T.; Ricordye, R.; Di Leonardo, A.; Degrassi, F. Cell Cycle 2010, 9 (20), 4174. doi: 10.4161/cc.9.20.13457
(12)Wei, R.; Ngo, B.; Wu, G.; Lee, W. H. Mol. Biol. Cell 2011, 22 (19), 3584. doi: 10.1091/mbc.E11-01-0012
(13)Xue, Y.; Li, H.; Ung, C.; Yap, C.; Chen, Y. Chem. Res. Toxicol. 2006, 19, 1030. doi: 10.1021/tx0600550
(14)Xue, Y.; Yap, C. W.; Sun, L. Z.; Cao, Z. W.; Wang, J.; Chen, Y. Z. J. Chem. Inf. Comput. Sci. 2004, 44, 1497. doi: 10.1021/ci049971e
(15)Xue, Y.; Li, Z.; Yap, C. W.; Sun, L.; Chen, X.; Chen, Y. Z. J. Chem. Inf. Comput. Sci. 2004, 44, 1630. doi: 10.1021/ci049869h
(16)Yang, X. G.; Chen, D.; Wang, M.; Xue, Y.; Chen, Y. Z. J. Comput. Chem. 2009, 30, 1202. doi: 10.1002/jcc.v30:8
(17)Yang, X. G.; Lv, W.; Chen, Y. Z.; Xue, Y. J. Comput. Chem. 2010, 31, 1249.
(18)Lv, W.; Xue, Y. Eur. J. Med. Chem. 2010, 45, 1167. doi: 10.1016/j.ejmech.2009.12.038
(19)Cong, Y.; Yang, X.; Lv, W.; Xue, Y. J. Mol. Graph. Model. 2009, 28, 236. doi: 10.1016/j.jmgm.2009.08.001
(20)Luan, F.; Liu, H.; Ma, W.; Fan, B. Eur. Med. Chem. 2008, 43, 43. doi: 10.1016/j.ejmech.2007.03.002
(21)Ung, C. Y.; Li, H.; Yap, C. W.; Chen, Y. Z. Mol. Pharmacol. 2007, 71, 158.
(22)Li, H.; Ung, C.; Yap, C.; Xue, Y.; Li, Z.; Cao, Z.; Chen, Y. Chem. Res. Toxicol. 2005, 18, 1071. doi: 10.1021/tx049652h
(23)Li, B. K.; Cong, Y.; Tian, Z. Y.; Xue, Y. Acta Phys. -Chim. Sin. 2014, 30 (1), 171. [李秉軻, 叢 湧, 田之悅, 薛 英. 物理化學(xué)學(xué)報(bào), 2014, 30 (1), 171.] doi: 10.3866/PKU.WHXB201311041
(24)Huang, J. J.; Lau, J. Improved Modulators of HEC1 Activity and Methods. CN Patent 103038231.A, 2013-04-10. [Huang, J. J., Lau, J. HEC1活性調(diào)節(jié)劑及其方法: 中國(guó), CN103038231.A[P]. 2013-04-10.]
(25)Duda, R. O.; Hart, P. E. Pattern Classification and Scene Analysis; John Wiley & Sons: Hoboken, New Jersey, USA, 1973.
(26)ChemDraw 7.0.1 ed.; CambridgeSoft Corporation, Cambridge: Massachusetts, USA, 2007.
(27)Corina 3.4 edn.; Molecular Networks GmbH Computerchemie: Erlangen, Germany, 2006.
(28)Burges, C. J. Data Min. Knowl. Disc. 1998, 2, 121.
(29)Vapnik, V. N. The Nature of Statistical Learning Theory; Springer: Berlin & Heidelberg, Germany, 1995.
(30)Doucet, J. P.; Barbault, F.; Xia, H.; Panaye, A.; Fan, B. Curr. Comput-Aid. Drug. 2007, 3, 263. doi: 10.2174/157340907782799372
(31)Svetnik, V.; Liaw, A.; Tong, C.; Culberson, J. C.; Sheridan, R. P.; Feuston, B. P. J. Chem. Inf. Comput. Sci. 2003, 43, 1947. doi: 10.1021/ci034160g
(32)Breiman, L. Mach. Learn. 2001, 45, 5. doi: 10.1023/A: 1010933404324
(33)Khandelwal, A.; Krasowski, M. D.; Reschly, E. J.; Sinz, M. W.; Swaan, P. W.; Ekins, S. Chem. Res. Toxicol. 2008, 21, 1457. doi: 10.1021/tx800102e
(34)Breiman, L. Out-of-bag Estimation, 1996, http://citeseerx.ist.psu.edu.sci-hub.org/viewdoc/download? doi=10.1.1.45.3712&rep=rep1&type=pdf (accessed Mar 15, 2015).
(35)Breiman, L. Wald Lecture II, Looking inside the Black Box, 2005. http://www.stat.berkeley.edu/users/breiman (accessed Mar 15, 2015).
(36)Breiman, L.; Cutler, A. Random Forests, Version 5.1, 2004. http://www.stat.berkeley.edu/~breiman/RandomForests/ cc_home.htm (accessed Mar 15, 2015).
Predicting and Virtually Screening Breast Cancer Targeting Protein HEC1 Inhibitors by Molecular Descriptors and Machine Learning Methods
HE Bing1,2LUO Yong1LI Bing-Ke2XUE Ying1,3YU Luo-Ting1,*QIU Xiao-Long4,5YANG Teng-Kuei4
(1State Key Laboratory of Biotherapy and Cancer Center, West China Hospital, Sichuan University, and Collaborative Innovation Center for Biotherapy, Chengdu 610041, P. R. China;2College of Chemistry and Life Science, Chengdu Normal University, Chengdu 611130, P. R. China;3College of Chemistry, Sichuan University, Chengdu 610064, P. R. China;4Zhaobang Bio-Med. Institute Co., Ltd., Nantong 226000, Jiangsu Province, P. R. China;5Wisdom Pharmaceutical Co., Ltd., Haimen 226123, Jiangsu Province, P. R. China)
Highly expressed in cancer 1 (HEC1) is a conserved mitotic regulator that is critical for spindle checkpoint control, kinetochore functionality, and cell survival. Overexpression of HEC1 has been detected in a variety of human cancers, and it is linked to poor prognosis of primary breast cancers. Thus, it is important to screen novel inhibitors with high affinity for HEC1. Machine learning (ML) methods were exhibiting good predicting capability in several aspects of the diverse compounds, such as pharmacokinetics,pharmacodynamics, and toxicity. In this work, two ML methods, support vector machines (SVMs) and random forests (RFs), were used to develop a classification method for searching inhibitors and non-inhibitors of HEC1 from the chemical library of structural diversity by screening characteristics of molecular descriptors. Both ML methods achieved promising prediction accuracies, and the RF model showed better performance. We performed virtual screening of HEC1 inhibitors by the RF model from an in-house database to screen potential HEC1 inhibitors. Two novel potential candidates were found. In vitro experiments of the two compounds showed that both had a certain degree of antitumor activity for the MDA-MB-468 and MDA-MB-231 breast cancer cell lines. Our study shows that ML methods are promising to design and virtually screen inhibitors of HEC1.
HEC1; Selective inhibitor; Machine learning method; Support vector machine; Random forest; Virtual screening
O641
10.3866/PKU.WHXB201507301
Received: April 2, 2015; Revised: July 30, 2015; Published on Web: July 30, 2015.
*Corresponding author. Email: luodyu@163.com.
The project was supported by “the Category 1.1 New Drug SKLB1312 of Antitumor (Breast Cancer), which is the Cooperation Project between West China Hospital of Sichuan University and Jiangsu Zhaobang Biological and Medical Research Institute Co., Ltd.”.
四川大學(xué)華西醫(yī)院與江蘇兆邦生物醫(yī)藥研究院有限公司合作項(xiàng)目“抗腫瘤(乳腺癌)一類新藥SKLB1312”資助
? Editorial office of Acta Physico-Chimica Sinica

