蛋白質組學在水產品品質與安全研究中的應用
李學鵬,陳 楊,蔡路昀,儀淑敏,徐永霞,勵建榮
(渤海大學食品科學研究院,遼寧省食品安全重點實驗室,遼寧 錦州 121013)
蛋白質組學在水產品品質與安全研究中的應用
李學鵬,陳 楊,蔡路昀,儀淑敏,徐永霞,勵建榮*>
(渤海大學食品科學研究院,遼寧省食品安全重點實驗室,遼寧 錦州 121013)
蛋白質組學技術在水產品品質與安全研究中已有較多應用。本文介紹了蛋白質組學的研究途徑,綜述了蛋白質組學在水產品物種鑒定、過敏原檢測、致病性和腐敗性微生物的檢測與鑒定、貯藏加工過程中品質變化機制研究等方面的應用進展,并對其發展方向和應用前景進行了展望。
蛋白質組學;質譜分析法;物種鑒定;過敏原檢測;品質與安全
蛋白質組(proteome)是指一個細胞在特定時間和特定環境條件下所有蛋白質的表達,是由澳大利亞Macquarie大學的Wilkins和Williams在1994年首先提出的[1]。如今,蛋白質組學技術已經廣泛應用于基礎生物學和臨床醫學等科學領域,為應用研究的高速發展和發病機制的研究奠定了基礎。在食品科學領域中,蛋白質組學的研究與應用雖然還處于起步階段,但在各個方面已經廣泛開展。
魚類等水產品是人體膳食營養素的主要來源之一,在發展中國家,人均年消耗量在9.0~15.1 kg之間[2],且攝入量還在逐年增加,這與魚類中富含的不飽和脂肪酸有關,這些不飽和脂肪酸有助于預防和治療心血管疾病、神經系統和炎癥疾病[3];同樣,魚類是優質動物性蛋白質來源之一,也能提供豐富微量元素,包括各種維生素(A、D、E、K、B1、B6、B12)和礦物質(Fe、I、P、Ca、K)等。
如今,商業全球化使得我們可以在一個國家捕獲魚,在另一個國家加工,并在其他國家銷售,在創造大量商業機會的同時也伴隨著各種風險。常見現象就是物種摻假,將低值魚或非法捕獲的魚替代高經濟價值的魚,欺騙消費者,并對人類健康造成了潛在危害。因為人們一旦食用了含有過敏原的魚類等水產品,常會引起過敏反應,加重敏感病人的癥狀或引發并發癥。此外,水產品在捕撈、運輸、加工、貯藏過程中,組成成分發生改變,進而影響感官品質和安全性。因此,需要一種準確、靈敏、快速的檢測方法,以便直接鑒定和監測水產食品中潛在的威脅。在過去的20年,為了達到這個目的,研究者發現了一些DNA和蛋白質分子標記[4-5],尤其是蛋白質組學技術取得的成功使這個目標得以實現,蛋白質組學在水產品物種鑒定、過敏原分析、致病性和腐敗性微生物鑒定、貯藏加工過程中品質變化等研究領域發揮著越來越重要的作用。
1 蛋白質組學
蛋白質組學(proteomics)作為一門學科,它是以蛋白質組為研究對象,應用相關技術,從整體水平上來研究蛋白質的存在形式、表達水平、翻譯后的修飾、功能及相互作用等[6]。因此,蛋白質組學研究不僅包括蛋白質結構和功能,還包括蛋白質修飾、蛋白質間的交互作用及分析它們在細胞內的位置和豐度的變化。
蛋白質組學始于20世紀70年代雙向凝膠電泳(twodimensional gel electrophoresis,2-DE)的發展[7]。2-DE技術是在單一凝膠上分離數以千計蛋白質的第一個方法。而質譜分析法是蛋白質組學研究中不可或缺的一項工具,主要包括基質輔助激光解析離子化飛行時間質譜(matrix-assisted laser desorption/ionizationtime of flight,MALDI-TOF)、電噴霧電離-離子阱質譜(electrospray-ion trap,ESI-IT)和串聯質譜(mass spectrometry/mass spectrometry,MS/MS)[8]。如今,利用生物信息學的方法處理數據,擴大了蛋白質組學的應用范圍,為蛋白質和肽的高通量識別及量化提供了一個強大的途徑。蛋白質組學研究方法取得的巨大成功,使它在食品科學領域的應用成為現實。目前,科研院所、行業機構和監管機構共同努力下,利用蛋白質組學研究已經獲得了一些有關食品成分、食品質量和安全方面的研究成果,蛋白質組學技術被提議作為最有希望的戰略食品認證方案[9-10]。
2 蛋白質組學研究流程
根據蛋白質分離、質譜分析方式的不同,蛋白質組學可分為:自下而上蛋白質組學(bottom-up proteomics)和自上而下(top-down proteomics)蛋白質組學兩種。Bottom-up proteomics是經典的蛋白質組學研究方法,它的主要步驟包括:分離出感興趣的蛋白,使用蛋白酶(如胰蛋白酶)進行消化,進行質譜分析[8]。Top-down proteomics也稱鳥槍法蛋白質組學,其基本過程是蛋白質混合物經過簡單分離或不經過分離就被酶解為肽段混合物,肽段混合物經液相色譜分離和離子化后,經串聯質譜(MS/MS)進行分析[11]。該方法具有高靈敏度和重現性,可在短時間內獲得大量鑒定結果,因此在蛋白質組研究中被廣泛采用。然而也存在許多缺點,比如易丟失蛋白質亞型信息、難以識別和表征蛋白質等,尤其是對于水產品這類缺乏基因組測序物種的蛋白質。兩種蛋白質組學的研究流程如圖1所示[12]。
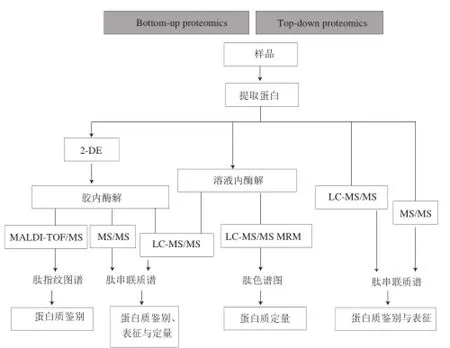
圖1 食品科學研究中常用的蛋白質組學工作流程[12]Fig.1 Proteomic workflow commonly used for food research[12]
Carrera等[13-16]提出了用于魚類物種認證的蛋白質組學方法,并根據研究目的和研究方法的差異,將蛋白質組學研究分為兩個連續階段:發現階段(discovery phase)和目標驅動階段(target-driven phase),如圖2所示。
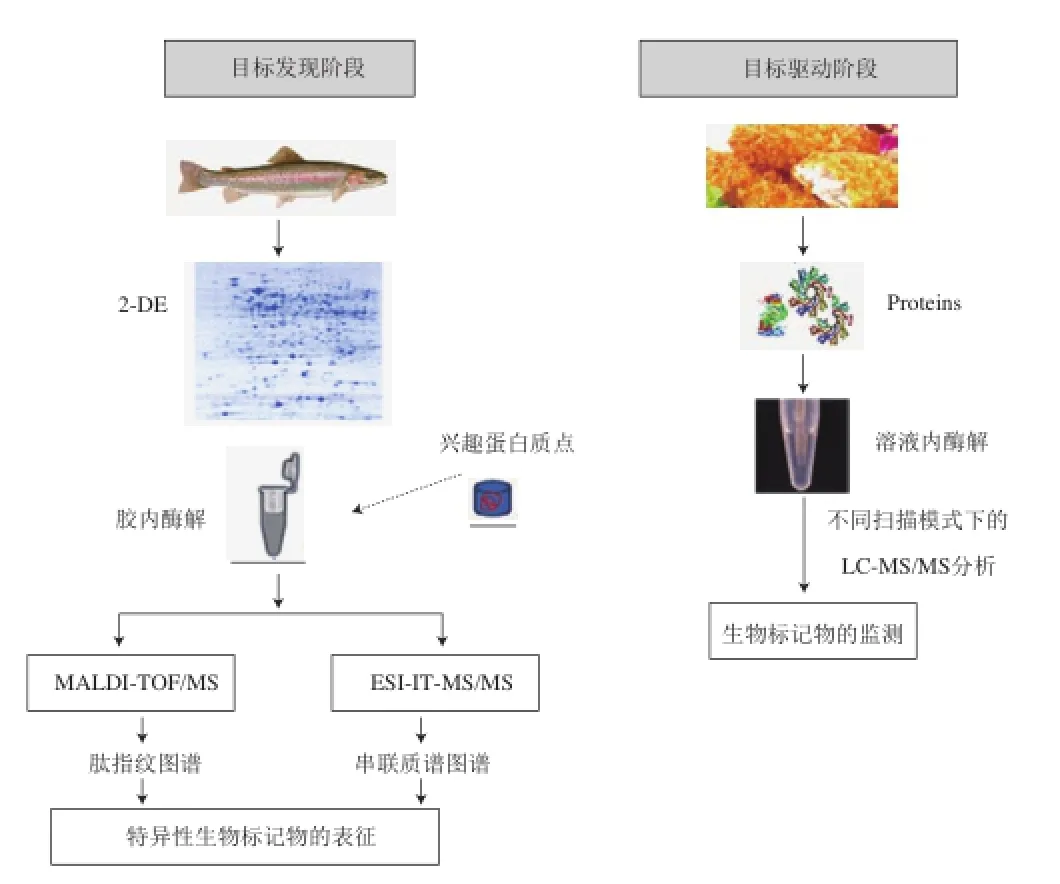
圖2 基于生物標記物鑒定與監測的蛋白質組學研究途徑[16]Fig.2 Proteomics pipeline used for identification and detection of species-specific peptide biomarkers[16]
2.1 發現階段(discovery phase)
發現階段的目標是全面探索特定的蛋白質組,以篩選潛在的蛋白質生物標記物,通常采用Bottom-up proteomics。蛋白質的分離純化可采用雙向凝膠電泳(2-DE)和液相色譜法。其中,2-DE由于其高分辨率,是區分復雜生物樣品蛋白質的最有效方法,也是蛋白質組分析中最廣泛應用的蛋白質分離方法。對于未完全測序的生物(如魚類),2-DE是最好的選擇,因為這類生物蛋白質是基于特定蛋白質以及與來自其他物種的同源蛋白質之間肽片段的比較來識別的。當然,2-DE也有局限性,如對疏水性和難溶性蛋白質的分離效果不佳等。
質譜是表征和鑒定蛋白質的首選方法,利用生物質譜鑒定蛋白質的過程分為質譜數據的獲得和質譜解析兩步驟。純化后的蛋白質被酶切為肽段混合物,離子化后經一級質譜產生肽質量指紋圖譜(peptide mass fingerprint,PMF)[17],或經串聯質譜產生肽碎片指紋圖譜(peptide fragmentation fingerprint,PFF)[18]。現有的解析圖譜方法包括序列庫搜索、圖譜庫搜索、從頭測序(de novo sequencing)以及從頭測序結合容錯性搜索的方法。其中,蛋白質序列庫搜索法是常用的解析圖譜方法。一些經典的搜庫軟件已得到廣泛應用,其中包括MASCOT、SEQUEST、X!Tandem等[6]。因此,相應的蛋白質序列數據庫在蛋白質組學研究中是很重要的,如果分離出來的蛋白不存在于數據庫中,但是有高度同源性的蛋白存在,那么最佳匹配的蛋白質是最接近的同源,通常是目標蛋白質的相關種類。如果數據庫蛋白的序列相似性太低,必須采用從頭測序法,并通過手動或借助計算機輔助工具來解釋MS/MS的圖譜[18]。
2.2 目標驅動階段(target-driven phase)
目標驅動階段的蛋白質組學方法主要用于監測已被鑒定出的物種特異性肽標記物。如前所述,對于沒有完成基因組測序的物種,物種特異性肽的篩選和鑒定需要采用繁瑣的從頭測序法。一旦獲得了這種物種特異性肽,質譜分析儀集中分析由選擇反應監測(selectedreaction monitoring,SRM)、多反應監測(multiple reaction monitoring,MRM)和選擇離子監測(selected MS/MS ion monitoring,SMIM)等模式得到的幾種肽化合物[19-21]。監測離子對(母離子對和碎片離子對)是鑒定肽標記物的常用方法。目標驅動蛋白質組學方法的優勢在于它的高特異性和靈敏性,能夠對質量濃度低于ng/mL級的復雜樣品中的蛋白質進行定量分析[20]。
3 蛋白質組學在水產品質量與安全評價中的應用
3.1 物種鑒定
魚類物種的鑒定是水產品質量與安全研究中的一個重要領域,已引起政府、水產品加工企業和消費者們的極大關注[16]。傳統的魚類認證主要是通過解剖學和生態學特征進行鑒定,然而,對于相似的魚種類別,特別是經過加工的產品來說,傳統鑒定方法很難實現。因此,需要一個快速、可信的分子識別技術,滿足政府和水產加工企業所需的物種標識和可追溯性的要求,從而確保產品質量,保護消費者健康。
目前已經存在的魚類物種鑒定技術主要有以DNA為基礎的物種鑒定等技術,但DNA技術存在一定的局限性。在魚制品加工過程中,細胞完整性被破壞,釋放出水解酶,加上加工過程中的熱處理和酸性條件,導致DNA的完整性被破壞,減少了片段長度擴增以及提高具有非特異性識別的機會。蛋白質電泳和免疫學技術已廣泛應用于魚類物種的鑒定[4-5]。等電聚焦(isoelectric focusing,IEF)在1990年就被美國化學會(Association of Official Analytical Chemists,AOAC)作為魚種鑒定的官方認證方法。與基因組不同,蛋白質組不是一個靜態的實體,蛋白質會隨著組織和環境條件的變化而變化,蛋白質組學比基因組學的方法產生更多的信息,并可表征除了物種鑒定以外的有關品質的更多信息。因此,蛋白質組學方法已成為水產品品種鑒偽和可追溯的一種更快、更敏感和高通量的方法。
2-DE可以通過檢測潛在的物種特異性蛋白質組或蛋白質點來區分相近物種的魚類,如鱈魚[11]、河豚魚[22]、鱸魚[23]、金槍魚[24]等。2-DE法檢測潛在的物種特異性蛋白質,通常采用經典的自下向上蛋白質組學方法(bottom-up proteomics),采用胰蛋白酶消化目標蛋白點,通過質譜得到肽指紋圖譜。利用肽質量指紋圖譜技術(peptide mass fingerprinting,PMF)可以分析無須鱈魚科的10 個近似商業品種[11]。肌漿蛋白中的小清蛋白PRVB具有較高的熱穩定性,被選為魚類物種鑒定的目標蛋白質,其MALDI-TOF質量指紋圖譜被視為魚類物種鑒定的分子標記物[12]。通過監測小清蛋白的肽質量可以區分新鮮的、加工的和煮過的水產品。Mazzeo等[25]利用MALDI-TOF/MS指紋技術直接對整個肌漿蛋白進行分析(樣品不經2-DE分離),通過分析特定PRVB的質量圖譜,成功實現了25 種(包括鱸形目、鱈形目和蝶形目)魚類的區分與鑒定。
3.2 過敏原檢測
食物過敏是人類常見的一種過敏性疾病, 絕大部分是由食物蛋白質引起,免疫球蛋白E(IgE)介導的Ⅰ型超敏反應。近年來,食物過敏現象越來越引起人們的重視。據聯合國糧食及農業組織(The Food and Agriculture Organization of the United States,FAO)統計,世界范圍內約有2%的成人,8%的兒童對食物過敏,其中兒童主要以牛奶和雞蛋過敏為主,而成人主要以海鮮類食物過敏為主[26]。在FAO公布的八大類過敏食物中,水產品占了兩大類。為了確保消費者的安全,歐盟規定生產者在水產品及其制品的標簽中必須標明14 種過敏原[27]。然而,由于生產過程可能存在交叉污染,市場上流通的產品中很可能含有痕量的過敏原,而極少量的過敏原蛋白即可引起嚴重的過敏反應。因此,可靠、精確、快捷的過敏原檢測方法是非常必要的。
β-PRVBs是主要的魚類過敏原,大量出現在魚類白色肌肉中[28]。Carrera等[29]建立了一種可以快速、直接檢測任何食物中的魚源β-PRVBs的方法。該方法采用目標驅動蛋白質組學方法,通過熱處理使β-PRVBs快速分離,利用高強度聚焦超聲法加速樣品的胰蛋白酶消化,采用選擇離子監測模式(SMIM)進行質譜分析,實現了對19 種常見的β-PRVBs肽標記物的檢測。由于PRVBs對熱穩定,因此該方法能夠快速直接檢測存在于任何產品包括經過加工的及預煮的產品中的β-PRVBs。
Yu等[30]采用蛋白質組學技術篩選出了斑節對蝦(Penaeus monodon)中的過敏原,結果表明,該過敏原是一種與精氨酸激酶具有高度相似性的蛋白質,具有精氨酸激酶的活性,并可與對蝦過敏人群血清中IgE發生反應,導致皮膚過敏反應。
3.3 致病性和腐敗性微生物的檢測與鑒定
微生物是導致大部分水產品腐敗變質的主要原因,但只有極少種類的特定腐敗菌參與腐敗過程產生不可接受的異味,使其喪失商品價值。另外,水產品加工過程中致病微生物的污染事件引起消費者食物中毒,同樣給水產加工企業帶來重大的經濟損失。水產品食物中毒大都是由其所攜帶的致病菌(包括大腸桿菌、沙門氏菌、金黃色葡萄球菌和副溶血性弧菌等)產生的毒素引起的。因此,對水產品中的主要腐敗菌和致病菌進行鑒定、檢測及有效控制是延長水產品貨架期、提高質量和安全性、保障行業健康發展的關鍵技術需求。
目前食品中微生物的鑒定主要是通過傳統方法、表型實驗及16S rRNA基因序列鑒定等方法實現的,存在效率偏低、精確度不高等缺點,而蛋白質組學技術在腐敗菌和致病菌的檢測、鑒定中顯示出強大優勢。Mazzeo等[31]通過對細菌細胞膜全蛋白的MALDI-TOF/MS分析實現了對24 種食源性致病菌和腐敗菌的檢測與鑒別,其中包括埃希氏桿菌屬、耶爾森氏菌屬、變形菌屬、沙門氏菌屬、葡萄球菌屬、微球菌、乳球菌屬、假單胞菌屬、明串珠菌和李斯特菌屬等。細菌細胞膜中低分子質量蛋白質的MALDI-TOF/MS分析已成功用于新鮮和加工水產品的安全性評估,主要的腐敗和致病性革蘭氏陰性細菌,包括嗜水氣單胞菌、鮑氏不動桿菌、假單胞菌屬和腸桿菌屬等被鑒定出來[32-33]。B?hme等[34]報道了利用MALDITOF/MS鑒定出水產品中革蘭氏陽性菌的研究,包括芽孢桿菌、李斯特菌屬、梭狀芽孢桿菌屬和葡萄球菌屬等。Fernández-No等[35-37]利用MALDI-TOF/MS鑒定出水產品中生物胺的產生菌和引起大菱鲆等養殖魚類腐敗和傳染性疾病的副乳房鏈球菌(Streptococcus parauberis)。
如今已經有多個商業數據庫用于細菌的MALDITOF/MS鑒定,如AXIMMA@SARAMISA(日本島津公司),MicrobeLynx細菌鑒定系統(英國沃特斯公司),NALDI Biotyper(美國布魯克·道爾頓公司)。盡管這些數據庫主要是針對引起人類傳染性疾病的病原菌,但其中有些細菌種類在水產品的腐敗和安全中也發揮重要作用。最近,西班牙Santiago de Compostela University的一個研究組開發了一個新的公共數據庫——Spectrabank,該數據庫中包括水產品中主要的腐敗和致病菌的質量指紋圖譜,并涵蓋了食品中存在的120 種細菌[16]。
3.4 水產品貯藏加工過程中品質變化機制
影響水產品新鮮度和品質的主要因素包括肌肉蛋白質水解和細菌性腐敗,都會引起肌原纖維蛋白和細胞骨架蛋白的變化[38-39]。魚類等水產品在捕撈、宰殺、貯藏、加工等過程中肌肉蛋白質的變化極具復雜性,蛋白質組學分析可為其變化機制的闡釋提供一定的幫助。
宰殺前狀態(包括魚的活力、魚肉密度、環境壓力等)、宰殺過程中糖原損耗和pH值下降等都會影響魚類死后肌肉的完整性和肌肉品質,這些變化可用蛋白質組學技術(主要是2-DE)來表征[40]。Morzel等[41]采用2-DE技術研究了宰殺前生理活性和應激反應對養殖鮭魚肌肉品質的影響,發現了能量產生酶和結構蛋白在表達水平的差異。Veiseth-Kent等[42]采用2-DE技術研究了擁擠應激對三文魚肌肉和血液中蛋白質組的影響,發現了二級、三級應激反應引起的蛋白質表達水平的變化。王彥波等[43]研究了宰殺方式對鯽魚肌肉質構和蛋白質組的影響,發現與直接打頭致死組比較,氮氣致死組2-DE圖譜中小分子質量蛋白質點增多,相同蛋白質的相對含量下降,這些變化與肌肉硬度等品質相關。
利用蛋白質組學技術可以表征貯藏過程中魚類肌肉蛋白質的變化與其品質性狀之間的相關性等信息,從而為貯藏過程中水產品品質變化機制的解析和水產品新鮮度評價提供參考和依據。Kj?rsg?rd等[44]采用2-DE技術研究了大西洋鱈魚貯藏過程中的肌球蛋白、α-輔肌動蛋白、3-磷酸丙糖異構酶等蛋白質的變化并提出了魚類死后肌肉蛋白質降解的一般途徑。Terova等[45]采用2-DE和MS技術比較了不同冷凍貯藏條件的鱈魚肌肉蛋白質圖譜并分析了與魚肉質地變化的相關性。李學鵬[46]、李婷婷[47]等采用2-DE和MS技術分別研究了中國對蝦和大黃魚肌肉蛋白質在冷藏過程中的變化規律,篩選出與新鮮度密切相關的指示蛋白質。
貯藏加工過程中,蛋白質氧化修飾如羰基化反應、硫醇氧化、芳香族羥基化和美拉德反應等,會引起蛋白質的交聯、側鏈修飾和肽鍵斷裂等變化,改變蛋白質的結構性質(如疏水性、凝膠性、水合性等),進而影響水產品的感官和營養品質[48]。采用2-DE、串聯質譜等蛋白質組學技術可以監測檢測蛋白質羰基化反應,跟蹤貯藏過程中蛋白質的氧化水平,并對參與氧化的蛋白質進行鑒別[49-50]。Kj?rsg?rd等[44]利用LC-MS/MS技術鑒別出冷凍虹鱒魚魚片貯藏過程中發生氧化的蛋白質,包括核苷二磷酸激酶、腺苷酸激酶、丙酮酸激酶、肌動蛋白、肌酸激酶、原肌球蛋白、肌球蛋白輕鏈1和2和肌球蛋白重鏈。Pazos等[51]采用2-DE、MALDI-TOF/TOF、LC-ESIIT-MS/MS研究了馬鮫魚肌原纖維蛋白質氧化情況,發現α-肌動蛋白、糖原磷酸化酶、丙酮酸激酶、亞型肌酸激酶、醛縮酶A和亞型甘油醛-3-磷酸脫氫酶等蛋白質均發生了氧化。
4 結 語
蛋白質組學能夠提供參與決定食品種屬、品質、功能與安全性的各種生理機制過程中的蛋白質結構和功能等信息,已廣泛用于食品科學研究領域,極大地拓展了食品科學的研究范圍。近年來,利用蛋白質組學研究水產品品質與安全的研究著作的數量快速增長,這些研究表明,蛋白質組學技術在水產品物種鑒別、過敏原檢測、水產品腐敗與安全性、水產品品質變化機制等研究方面是一個強大的工具。水產品品質與安全性是由多種因素間復雜的相互作用決定的,蛋白質組學只是用于分析的有力工具而不是解決品質與安全問題的最終方法,以蛋白質組學技術為基礎,篩選、建立可靠的生物標記物(如水產品新鮮度指示蛋白等)并將其運用于水產品貯藏加工過程中品質與安全性的評價與控制,是水產品蛋白質組學研究中的一個重要方向。此外,新的研究平臺(如微流體設備、蛋白質芯片)的開發與應用也為現代食品科學研究提供更為廣闊的發展空間,將有助于利用蛋白質組學研究結果實現水產品品質的常規診斷、監測與控制,為水產食品科學研究提供了新的思路和方法。
[1] WASINGER V C, CORDWELL S J, CERPA-POLJAK A,et al. Progress with gene-product mapping of the mollicutes: Mycoplasmagen italium[J]. Electtrophoresis, 1995, 16(1): 1090-1094.
[2] FAO. The state of world fisheries and aquaculture[R]. Rome: Food and Agriculture Organization of the United Nations, 2014.
[3] HOOPER L, THOMPSON R L, HARRISON R A, et al. Risks and benefits of omega 3 fats for mortality, cardiovascular disease, and cancer: systematic review[J]. British Medical Journal, 2006, 332: 752-760.
[4] BOZIARIS I S. Fish and seafood authenticity: species identification[M]//LAGO F C, ALONSO M, VIEITES J M, et al. Seafood processing: technology, quality and safety. Hoboken: John Wiley & Sons Inc., 2013.
[5] TEPEDINO V, BERRINI A, BORROMEO V, et al. Identification of commercial fish species belonging to the orders pleuronectiformes and gadiformes: library of isoelectric focusing patterns[J]. Journal of AOAC International, 2001, 84(5): 1600-1607.
[6] 李學鵬, 勵建榮, 于平, 等. 蛋白質組學及其在食品科學研究中的應用[J]. 中國糧油學報, 2010, 25(2): 141-149.
[7] AEBERSOLD R, MANN M. Mass spectrometry-based proteomics[J]. Nature, 2003, 422: 198-207.
[8] ZHANG Y, FONSLOW B R, SHAN B, et al. Protein analysis by shotgun/bottom-up proteomics[J]. Chemical Review, 2013, 113(4): 2343-2394.
[9] RODRIGUES P M, SILVA T S, DIAS J, et al. Proteomics in aquaculture: applications and trends[J]. Journal of Proteomics, 2012,75(14): 4325-4345.
[10] D’ALESSANDRO A, ZOLLA L. We are what we eat: food safety and proteomics[J]. Journal of Proteome Research, 2011, 11(1): 26-36.
[11] MOTOYAMA A, YATES J R, Ⅲ. Multidimensional LC separations in shotgun proteomics[J]. Analytical Chemistry, 2008, 80(19): 7187-7193.
[12] GALLARDO J M, ORTEA I, CARRERA M. Proteomics and its applications for food authentication and food-technology research[J]. TrAC Trends in Analytical Chemistry, 2013, 52: 135-141.
[13] CARRERA M, CA?AS B, PI?EIRO C, et al. Identification of commercial hake and grenadier species by proteomic analysis of the parvalbumin fraction[J]. Proteomics, 2006, 6(19): 5278-5287.
[14] CARRERA M, CA?AS B, L?PEZ-FERRER D, et al. Fast monitoring of species-specific peptide biomarkers using high-intensity-focusedultrasound-assisted tryptic digestion and selected MS/MS ion monitoring[J]. Analytical Chemistry, 2011, 83(14): 5688-5695.
[15] CARRERA M, CA?AS B, V?ZQUEZ J, et al. Extensive de novo sequencing of new parvalbumin isoforms using a novel combination of bottom-up proteomics, accurate molecular mass measurement by FTICR-MS, and selected MS/MS ion monitoring[J]. Journal of Proteome Research, 2010, 9(9): 4393-4406.
[16] CARRERA M, CA?AS B, GALLARDO J M. Proteomics for the assessment of quality and safety of fishery products[J]. Food Research International, 2013, 54: 972-979.
[17] PAPPIN D J C, HOJRUP P, BLEASBY A. Rapid identification of proteins by peptide-mass fingerprinting[J]. Current Biology, 1993,3(6): 327-332.
[18] ENG J K, MCCORMACK A L, YATES J R. An approach to correlate tandem mass spectral data of peptides with amino acid sequences in a protein database[J]. Journal of the American Society for Mass Spectrometry, 1994, 5(11): 976-989.
[19] GALLIEN S, DURIEZ E, DOMON B. Selected reaction monitoring applied to proteomics[J]. Journal of Mass Spectrometry, 2011, 46(3): 298-312.
[20] JORGE I, CASAS E M, VILLAR M, et al. High-sensitivity analysis of specific peptides in complex samples by selected MS/MS ion monitoring and linear ion trap mass spectrometry: application to biological studies[J]. Journal of Mass Spectrometry, 2007, 42(11): 1391-1403.
[21] UNWIN R D, GRIFFITHS J R, WHETTON A D. A sensitive mass spectrometric method for hypothesis-driven detection of peptide posttranslational modifications: multiple reaction monitoring-initiated detection and sequencing (MIDAS)[J]. Nature Protocols, 2009, 4(6): 870-877.
[22] CHEN R, CHANG L, CHUNG Y, et al. Quantification of cow milk adulteration in goat milk using high-performance liquid chromatography with electrospray ionization mass spectrometry[J]. Rapid Communications in Mass Spectrometry, 2004, 18(10): 1167-1171.
[23] BERRINI A, TEPEDINO V, BORROMEO V, et al. Identification of freshwater fish commercially labelled ‘perch’ by isoelectric focusing and two-dimensional electrophoresis[J]. Food Chemistry, 2006, 96(1): 163-168.
[24] PEPE T, CERUSO M, CARPENTIERI A, et al. Proteomics analysis for the identification of three species of Thunnus[J]. Veterinary Research Communications, 2010, 34(1): 153-155.
[25] MAZZEO M F, GIULIO B D, GUERRIERO G, et al. Fish authentication by MALDI-TOF mass spectrometry[J]. Journal of Agricultural and Food Chemistry, 2008, 56(23): 11071-11076.
[26] SICHERER S H. Epidemiology of food allergy[J]. Journal of Allergy and Clinical Immunology, 2011, 127(3): 594-602.
[27] JEEBHAY M F, ROBINS T G, LEHRER S B, et al. Occupational seafood allergy: a review[J]. Occupational and Environmental Medicine, 2001, 58(9): 553-562.
[28] 劉光明, 曹敏杰, 蔡秋鳳, 等. 水產品過敏原的研究現狀和展望[J].中國食品學報, 2012, 12(5): 1-9.
[29] CARRERA M, CA?AS B, GALLARDO J M. Rapid direct detection of the major fish allergen, parvalbumin, by selected MS/MS ion monitoring mass spectrometry[J]. Journal of Proteomics, 2012, 75(11): 3211-3220.
[30] YU C J, LIN Y F, CHIANG B L, et al. Proteomics and immunological analysis of a novel shrimp allergen, Pen m 2[J]. The Journal of Immunology, 2003, 170(1): 445-453.
[31] MAZZEO M F, SORRENTINO A, GAITA M, et al. Matrix-assisted laser desorption ionization-time of flight mass spectrometry for the discrimination of food-borne microorganisms[J]. Applied and Environmental Microbiology, 2006, 72(2): 1180-1189.
[32] B?HME K, FERN?NDEZ-NO I C, BARROS-VEL?ZQUEZ J, et al. Rapid species identification of seafood spoilage and pathogenic Gram-positive bacteria by MALDI-TOF mass fingerprinting[J]. Electrophoresis, 2011, 32(21): 2951-2965.
[33] B?HME K, FERN?NDEZ-NO I C, BARROS-VEL?ZQUEZ J, et al. Species differentiation of seafood spoilage and pathogenic Gramnegative bacteria by MALDI-TOF mass fingerprinting[J]. Journal of Proteome Research, 2010, 9(6): 3169-3183.
[34] B?HME K, FERN?NDEZ-NO I, GALLARDO J M, et al. Safety assessment of fresh and processed seafood products by MALDI-TOF mass fingerprinting[J]. Food and Bioprocess and Technology, 2011,4(6): 907-918.
[35] FERN?NDEZ-NO I C, B?HME K, GALLARDO J M, et al. Differential characterization of biogenic amine-producing bacteria involved in food poisoning using MALDI-TOF mass fingerprinting[J]. Electrophoresis, 2010, 31(6): 1116-1127.
[36] FERN?NDEZ-NO I C, B?HME K, CALO-MATA P, et al. Isolation and characterization of Streptococcus parauberis from vacuumpackaging refrigerated seafood products[J]. Food Microbiology, 2012,30(1): 91-97.
[37] B?HME K, FERN?NDEZ-NO I C, BARROS-VEL?ZQUEZ J, et al. SpectraBank: an open access tool for rapid microbial identification by MALDI-TOF MS fingerprinting[J]. Electrophoresis, 2012, 33(14): 2138-2142.
[38] DU Xueli, DU Cuihong, LIU Guangming, et al. Effect of a myofibrilbound serine proteinase on the deg of giant protein titin and nebulin[J]. Journal of Food Biochemistry, 2010, 34(3): 581-594.
[39] WU Guoping, CHEN Suhua, LIU Guangming, et al. Purification and characterization of a collagenolytic serine proteinase from the skeletal muscle of red sea bream (Pagrus major)[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2010,155(3): 281-287.
[40] ADDIS M F, PISANU S, PREZIOSA E, et al. 2D DIGE/MS to investigate the impact of slaughtering techniques on postmortem integrity of fish filet proteins[J]. Journal of Proteomics, 2012, 75(12): 3654-3664.
[41] MORZEL M, CHAMBON C, LEFEVRE F, et al. Modifications of trout (Oncorhynchus mykiss) muscle proteins by preslaughter activity[J]. Journal of Agricultural and Food Chemistry, 2006, 54(8): 2997-3001.
[42] VEISETH-KENT E, GROVE H, F?RGESTAD E M, et al. Changes in muscle and blood plasma proteomes of Atlantic salmon (Salmo salar) induced by crowding[J]. Aquaculture, 2010, 309(1): 272-279.
[43] 王彥波, 沈曉琴, 李學鵬, 等. 不同宰殺方式對鯽魚肌肉質構和蛋白質組的影響[J]. 中國食品學報, 2010, 10(6): 145-149.
[44] KJ?RSG?RD I V H, N?RRELYKKE M R, JESSEN F. Changes in cod muscle proteins during frozen storage revealed by proteome analysis and multivariate data analysis[J]. Proteomics, 2006, 6(5): 1606-1618.
[45] TEROVA G, ADDIS M F, PREZIOSA E, et al. Effects of postmortem storage temperature on sea bass (Dicentrarchus labrax) muscle protein degradation: analysis by 2-D DIGE and MS[J]. Proteomics, 2011,11(14): 2901-2910.
[46] 李學鵬. 中國對蝦冷藏過程中品質評價及新鮮度指示蛋白研究[D].杭州: 浙江工商大學, 2011.
[47] 李婷婷. 大黃魚生物保鮮技術及新鮮度指示蛋白研究[D]. 杭州: 浙江工商大學, 2012.
[48] LUND M N, HEINONEN M, BARON C P, et al. Protein oxidation in muscle foods: a review[J]. Molecular Nutrition & Food Research,2011, 55(1): 83-95.
[49] KINOSHITA Y, SATO T, NAITOU H, et al. Proteomic studies on protein oxidation in bonito (Katsuwonus pelamis) muscle[J]. Food Science and Technology Research, 2007, 13(2): 133-138.
[50] KJ?RSG?RD I V H, JESSEN F. Two-dimensional gel electrophoresis detection of protein oxidation in fresh and tainted rainbow trout muscle[J]. Journal of Agricultural and Food Chemistry, 2004, 52(23): 7101-7107.
[51] PAZOS M, MAESTRE R, GALLARDO J M, et al. Proteomic evaluation of myofibrillar carbonylation in chilled fish mince and its inhibition by catechin[J]. Food Chemistry, 2013, 136(1): 64-72.
Application of Proteomics in Studies of Seafood Quality and Safety
LI Xuepeng, CHEN Yang, CAI Luyun, YI Shumin, XU Yongxia, LI Jianrong*
(Food Safety Key Laboratory of Liaoning Province, Food Science Research Institute of Bohai Univ ersity, Jinzhou 121013, China)
Proteomics has been widely applied in the field of seafood quality and safety. The pipeline of proteomics is described in this review. The application of proteomics in fish authentication, allergen detection, detection and identification of pathogenic and spoilage microorganisms, and quality change mechanisms are summarized. Finally, future directions and new potential perspectives are also discussed.
proteomics; mass spectrometry; fish authentication; allergen diction; quality and safety
TS254.4
A
1002-6630(2015)09-0209-06
10.7506/spkx1002-6630-201509039
2014-07-22
國家自然科學基金青年科學基金項目(31301569;31301418);國家自然科學基金面上項目(31471639);
“十二五”國家科技支撐計劃項目(2012BAD29B06);遼寧省博士啟動基金項目(20131003)
李學鵬(1982—),男,副教授,博士,研究方向為水產品貯藏加工與水產食品化學。E-mail:xuepengli8234@163.com
*通信作者:勵建榮(1964—),男,教授,博士,研究方向為水產品貯藏加工與食品安全。E-mail:lijr6491@163.com

