紅曲黃酒傳統釀造過程中的細菌菌群結構及其動態變化
賈瑞博,潘雨陽,胡榮康,周文斌,蔣雅君,郭偉靈,朱風風,吳林秀,劉 斌,饒平凡,倪 莉,呂旭聰,*
(1.福建農林大學國家菌草工程技術研究中心,福建 福州 350002; 2.福建農林大學食品科學學院,福建 福州 350002; 3.福州大學生物科學與工程學院食品科技研究所,福建 福州 350108)
紅曲黃酒傳統釀造過程中的細菌菌群結構及其動態變化
賈瑞博1,2,潘雨陽1,2,胡榮康1,2,周文斌1,2,蔣雅君1,2,郭偉靈1,2,朱風風1,2,吳林秀1,2,劉 斌1,2,饒平凡3,倪 莉3,呂旭聰1,2,3*
(1.福建農林大學國家菌草工程技術研究中心,福建 福州 350002; 2.福建農林大學食品科學學院,福建 福州 350002; 3.福州大學生物科學與工程學院食品科技研究所,福建 福州 350108)
采用變性梯度凝膠電泳(DGGE)技術、克隆文庫技術和限制酶切片段長度多態性(RFLP)等相結合的方法,探究了紅曲黃酒傳統釀造過程中的細菌菌群結構及其動態變化。變性梯度凝膠電泳(DGGE)試驗結果表明,在傳統釀造過程中的初期階段的優勢菌株包括植物乳桿菌Lactobacillusplantarum、戊糖片球菌Pediococcuspentosaceus和短乳桿菌Lactobacillusbrevis,隨著釀造時間的增加,植物乳桿菌將逐漸占據絕對優勢;利用限制酶切片段長度多態性(RFLP)技術對16S rDNA文庫中的克隆子進行酶切分型,共得到19種RFLP圖譜類型,測序鑒定結果與變性梯度凝膠電泳(DGGE)結果相似,但16S rDNA克隆文庫分析檢測到了戊糖片球菌Pediococcuspentosaceus,該菌未見于變性梯度凝膠電泳(DGGE)結果中。變性梯度凝膠電泳(DGGE)技術、克隆文庫技術和限制酶切片段長度多態性(RFLP)等多種方法相結合,有助于更為全面、客觀地研究紅曲黃酒傳統釀造體系中的細菌群落結構及多樣性。
紅曲黃酒;傳統釀造;細菌菌群
黃酒在世界釀造史上獨樹一幟,與啤酒、葡萄酒并稱為世界三大古酒,是中華民族的文化瑰寶,有著5 000多年的悠久歷史,享有“國酒”之美譽[1-2]。黃酒是以谷物為原料,用曲釀造而成的,其營養豐富、酒精度低且具有獨特的風味和功效而深受廣大消費者的喜愛,加上近年來國家大力扶持黃酒產業的發展,其市場需求量逐年穩步上升。紅曲黃酒是以糯米為主要原料,添加紅曲和藥白曲作為糖化發酵劑,經多種微生物釀造而成的一種低度黃酒,以色紅、味醇、香濃而著稱,不僅具有豐富營養成分,還因添加了紅曲進行釀造而具備多種與眾不同的生理功效,是中國黃酒中十分有特色的一類黃酒[3-4]。
紅曲黃酒傳統釀造用曲中含有豐富的霉菌、酵母以及細菌[5], 在紅曲黃酒“雙邊發酵”過程中,微生物參與各種生物化學反應,對紅曲黃酒的產量和品質起到至關重要的作用[6-7]。紅曲黃酒傳統釀造是“群微共酵”的過程,其產量和品質與整個釀造過程中獨特的微生物群落結構變化有著緊密的聯系。然而,目前有關紅曲黃酒中微生物菌群結構及其功能研究較少,且長期以來研究手段單一,未能進一步系統地研究微生物菌群結構與其功能的關系,尤其是釀造過程中菌群結構的動態變化趨勢。不能充分分析釀造系統微生物區系的結構和功能,更不用說充分利用微生物菌種資源。
紅曲黃酒傳統釀造體系是一個復雜的生態系統[8],許多功能微生物可能處于“存活但不能培養”(VBNC)的狀態,無法通過傳統純培養技術從菌群中分離獲得,使得紅曲黃酒獨特風味組分的形成途徑變成“暗箱”。以分子生物學為基礎的免培養微生物分子生態學克服了傳統微生物純培養方法的不足,直接從基因水平上研究復雜體系中微生物菌群結構與功能。為此,本研究采用變性梯度凝膠電泳(DGGE)技術、克隆文庫技術和限制酶切片段長度多態性(RFLP)等相結合的免培養微生物分子生態學方法,跟蹤紅曲黃酒傳統釀造過程中細菌菌群結構動態變化,研究結果不僅可以更為深刻地認識紅曲黃酒釀造機理,也有利于快速篩選和鑒定出適用于紅曲黃酒釀造的細菌菌株。
1 材料與方法
1.1 試驗材料
以糯米為原料,以烏衣紅曲和藥白曲為發酵曲,在釀造工藝實驗室按照福州紅曲黃酒傳統釀造工藝進行釀造[6,9],在3個酒壇中同時進行釀造作為平行試驗,在傳統釀造的第1、2、3、5、7、10、20、46 d分別從3個酒壇中取酒醪樣品20 g進行混合均勻,提取細菌總DNA進行PCR-DGGE分析,取第1、20、46 d的樣品進行細菌16S rDNA克隆文庫分析。
酵母提取物為OXOID產品; CTAB、N,N′-甲叉丙烯酰胺、TEMED、過硫酸銨、Tris、瓊脂糖、氨芐青霉素購自上海生工有限公司;蛋白酶 K、dNTP、IPTG、X-gal、Loading Buffer、膠回收試劑盒為TaKaRa產品;DNA marker為MBI產品;限制性內切酶(Rsa、Msp)為Fermentas公司;用于轉化的菌株選用E.coliDH5α為福建農林大學國家菌草工程技術研究中心保存;pMD19-T vector購自TaKaRa公司;引物均由上海生工合成;其余試劑均為國產分析純試劑。
1.2 細菌DNA提取
采用溶菌酶-蛋白酶K-CTAB法[10]進行樣品中細菌總DNA的提取,保存于-20℃備用。
1.3 細菌16S rDNA基因的擴增
細菌基因組16S rDNA的擴增[11]:以提取的細菌基因組DNA為模板,采用細菌16S rDNA通用引物[7]1492R(5′-GGC TAC CTT GTT ACG ACT T-3′)和細菌特異引物27F(5′-AGA GTT TGA TCC TGG CTC AG-3′)進行PCR擴增,PCR產物經質量分數為1.5%的瓊脂糖凝膠電泳檢測,EB染色,凝膠成像系統觀察和拍照。
1.4 細菌16S rDNA-V3區基因的擴增
16S rDNA-V3區基因的擴增:將純化后的16S rDNA為模板,采用細菌通用引物[8]338f+(5′-CGC CCG CCG CGC GCG GCG GGC GGG GCG GGG GCA CGG GGG GAC TCC TAC-3′)和518r(5′-ATT ACC GCG GCT GCT GG-3′)進行巢式PCR擴增,PCR產物經質量分數為1.5%的瓊脂糖凝膠電泳檢測,EB染色,凝膠成像系統觀察和拍照。
1.5 變性梯度凝膠電泳(DGGE)分析
對PCR產物進行DGGE分析,DGGE變性膠濃度為8%(w/v),變性梯度為40%~60%電泳條件:1×TAE電泳緩沖液,60℃恒溫,65 V恒壓電泳16 h;EB染色,并用圖形分析軟件(Quantity One)對凝膠圖像進行分析。用Coverage C、Evenness指數(E)、Shannon-Wierner指數(H′)、Simpson指數(D),Margalef指數(d)等指標(表1)來分析細菌多樣性。

表1 多樣性指標分析說明Table 1 Description on diversity index analysis
1.6 DGGE條帶的序列測定
對細菌DGGE圖譜較為明顯的條帶進行割膠后放入EP管中,去離子水重復沖洗3次后加入50 μL去離子水碾碎過夜保存,取上清液作為PCR模板擴增,將純化后的PCR產物與pMD19-T 載體連接,轉化至E.coliDH5α細胞中,通過藍白篩選,隨機挑選白斑進行菌落PCR驗證。將經驗證的陽性克隆子交由上海生工生物技術公司進行測序;測序結果用DNAMAN軟件將16S rDNA-V3 高變區克隆子序列去除載體后,通過NCBI查找比對(BLAST分析),選取克隆子序列在GenBank中找尋最相似收錄序列進行比對。
1.7 細菌16S rDNA基因的限制性內切酶酶切(RFLP)
瓊脂糖電泳驗證成功的PCR樣品,分別用限制性內切酶Rsa和Msp進行酶切,在37℃的恒溫水浴鍋中放置過夜進行反應,酶切產物在2%瓊脂糖凝膠上進行電泳;分析克隆子的RFLP譜型,計算每個譜型的出現頻率。將具有不同圖譜類型的克隆挑出,液體擴培后進行測序,序列在NCBI數據庫上進行比對鑒定。
2 結果與分析
2.1 基于DGGE指紋圖譜技術的細菌菌群多樣性分析
對細菌16S rDNA-V3區PCR產物進行DGGE分析,結果如圖1所示。
從克隆鑒定結果(表2)中可以發現,所有序列均可以在GenBank中找到與其序列相似性很高(>98%)的菌株。結合細菌16S rDNA-V3區DGGE圖譜中發現,條帶1、2和3相較其他條帶較亮,可認為是釀造過程中的關鍵菌株,占有較大比例。經鑒定其分別為植物乳桿菌Lactobacillusplantarum、短乳桿菌Lactobacillusbrevis和戊糖片球菌Pediococcuspentosaceus。在釀造初期(第1~10 d),優勢菌株的結構沒有發生很大變化,到了釀造中期(第20 d),戊糖片球菌基本檢測不到,而植物乳桿菌在釀造體系中的比例升高,到了釀造后期(第46 d),基本上只檢測到植物乳桿菌Lactobacillusplantarum。
2.2 基于16S rDNA克隆文庫技術的細菌菌群多樣性分析
2.2.1 16S rDNA克隆文庫的限制性片段長度多態性(RFLP)分析 PCR-RFLP分析采用限制性內切酶MspI(C′CGG)和RsaI(GT’AC)。一共分析了262個克隆子(B1文庫,77個;B20文庫,115個;B46文庫,70個)。B1、B20和B46文庫分別獲得了15、12和4種RFLP帶型,這表明細菌的種類隨著釀造時間的推移而在不斷減少。B1文庫中最豐富的RFLP帶型有16個克隆子(占總克隆子數的20.8%) (圖2-A),B20文庫中最豐富的帶型有42個克隆子(占總克隆子數的36.5%) (圖2-B),而B46文庫中最豐富的帶型有47個克隆子(占總克隆子數的67.1%) (圖2-C)。可以發現,隨著釀造時間的增加,細菌種類不斷減少;在每一個釀造時期,釀造體系中都有優勢細菌,說明在釀造的不同時期,細菌的分布是不均勻的。

表2 DGGE譜圖中相關電泳條帶的測序鑒定結果Table 2 DNA sequencing results associated electrophoretic bands of DGGE spectrum
2.2.2 細菌16S rDNA克隆文庫種群多樣性分析 根據其計算公式,若Coverage C得到的值很大或達到1,則說明庫容已經足夠。由表3中可知,3個文庫的Coverage C都很接近1,說明3個文庫的庫容均滿足要求。從多樣性和均勻度指數來看,細菌的多樣性水平和均勻度隨著釀造的進行呈現不斷下降的趨勢,釀造初期的細菌種類明顯高于釀造后期。這可能是由于在釀造后期,釀造體系中的酒精度不斷攀升和營養物質不斷減少而造成細菌的多樣性不斷下降。
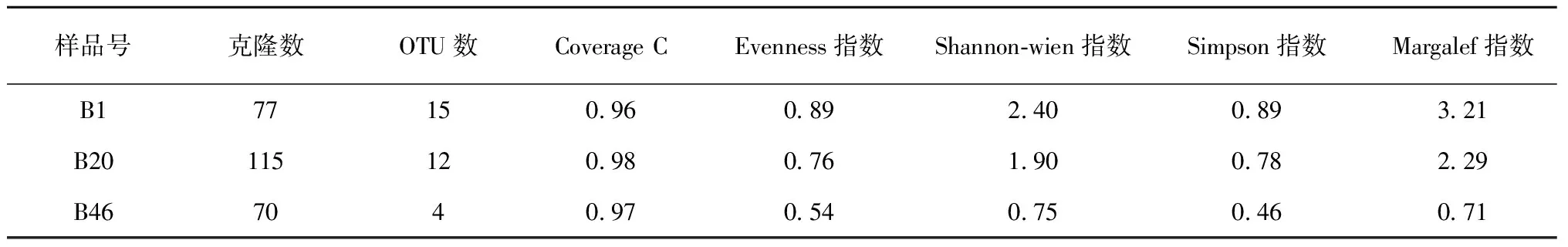
表3 16S rDNA克隆文庫種群多樣性分析Table 3 Species diversity analysis on 16S rDNA clone libraries
2.2.3 序列比對分析 將3個文庫中19種RFLP帶型,均挑選1個代表菌株進行了測序,將鑒定結果結合柱狀圖整理出菌株名及其數量關系,匯總為16S rDNA克隆文庫菌株變化趨勢表(表4)。
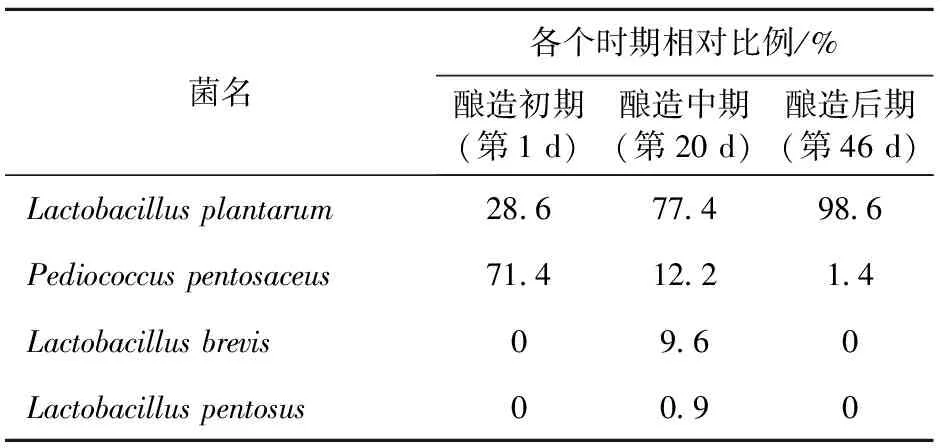
表4 16S rDNA克隆文庫中不同細菌的變化趨勢Table 4 Composition and dynamic changes of bacteria community as revealed by analysis of 16 s rDNA clone library
從表4中可以看出,植物乳桿菌Lactobacillusplantarum在體系中的相對比例從釀造初期的28.6%上升到釀造后期的98.6%,戊糖片球菌Pediococcuspentosaceus在體系中的相對比例從釀造初期的71.4%下降到1.4%,這與細菌DGGE圖譜中的趨勢是吻合的。但16S rDNA克隆文庫在釀造中期檢測到短乳桿菌Lactobacillusbrevis,其相對比例占9.6%,而細菌DGGE圖譜中發現在釀造第1 d就檢測到了短乳桿菌Lactobacillusbrevis,并占有一定比例;16S rDNA克隆文庫在釀造中期還檢測到了戊糖乳桿菌Lactobacilluspentosus,這是細菌DGGE圖譜中所沒有檢測到的。造成兩種方法結果不同的原因可能是由于:(1)用于細菌DGGE所擴增的16S rDNA-V3區的分辨率不夠;(2)使用RFLP方法分析細菌克隆文庫,所選用的限制性內切酶在分析相似性較高的序列時,可能無法根據酶切的帶型清晰地分辨出序列之間的差異,使得該方法低估了研究體系中的微生物多樣性。從這樣的結果可以看出,單一的分子生態學方法有一定的局限性,DGGE技術和克隆文庫技術的結合使用可以得到紅曲黃酒釀造過程中微生物結構更完整的結果。
3 討論與結論
3.1變性梯度凝膠電泳(DGGE)結果顯示,在傳統釀造過程中的初期階段的優勢菌株包括植物乳桿菌、戊糖片球菌和短乳桿菌;但隨著釀造的進行,植物乳桿菌將逐漸占據絕對優勢。利用限制酶切片段長度多態性(RFLP)技術對16S rDNA文庫中的克隆子進行酶切分型,共得到19種RFLP圖譜類型,通過測序鑒定結果顯示16S rDNA克隆文庫中包括植物乳桿菌、戊糖片球菌、短乳桿菌和戊糖乳桿菌。其中,戊糖片球菌的數量從釀造第1 d的71.4%下降至第46 d的1.4%,短乳桿菌在第1 d沒有檢測到,但在第20 d其數量達到13%,在釀造后期同樣沒有檢測到,植物乳桿菌隨著釀造過程的進行,其數量從28.6%上升至98.6%,在釀造過程中,乳酸菌除產生乳酸外,還可能以乳酸為底物產生乙酸、琥珀酸、丙酸和丁酸等其他有機酸,這些有機酸在酯化酶的催化作用通過酯化反應生成乳酸乙酯,乙酸乙酯等香氣組分,豐富了紅曲黃酒的風味成分。陳寶良等[13]對傳統香雪酒(紹興酒中的一種高檔品種)發酵過程中的微生物變化規律及作用進行了研究,發現傳統香雪酒的發酵過程是曲(酒藥和麥曲)的糖化和以多品種、高密度的細菌與酵母協同糖化發酵并行的過程,到中后期是真正意義上的細菌為主的發酵,而酵母菌作用小。據此,在紅曲黃酒傳統釀造的過程中,可以推測細菌對酒的風味有著很大的貢獻,在解析紅曲黃酒傳統釀造過程細菌菌群結構的基礎上,后續研究將進一步探明這些乳酸菌的產香特性,以及乳酸菌與酵母菌之間的相互作用及其對紅曲黃酒特征風味物質形成的影響,最終解決導致紅曲黃酒傳統釀造品質不穩定的瓶頸問題,改進工藝、提升品質。
3.216S rDNA克隆文庫分析得到的結果與變性梯度凝膠電泳(DGGE)得到的結果相似,但16S rDNA克隆文庫分析檢測到了戊糖片球菌,該菌未見于變性梯度凝膠電泳(DGGE)結果中。研究結果說明采用免培養微生物分子生態學方法研究細菌多樣性,雖然較傳統分離培養方法更為快速和準確,但各個技術也存在的一定局限性和不足。因此,本研究采用DGGE結合文庫分析和RFLP等分子生物學方法對樣品進行分析,使其相互補充,有利于獲得關于紅曲黃酒釀造過程中微生物生態更為客觀和準確的信息。
[1]陳玉香, 葉汶坤, 何冬萍, 等. 酵母菌對紅曲黃酒風味物質形成的影響[J].中國食品學報, 2015, 15(7): 218-223.
[2]陳平. 紅曲代謝產物的研究現狀和前景[J]. 廣西輕工業, 2009,(9): 4-5.
[3]Lü X C, WENG X, ZHANG W, et al. Microbial diversity of traditional fermentation starters for Hong Qu glutinous rice wine as determined by PCR-mediated DGGE[J]. Food Control, 2012, 28(2):426-434.
[4]呂旭聰, 翁星, 黃若蘭,等. 紅曲黃酒釀造用曲及傳統釀造過程中酵母菌的多樣性研究[J]. 中國食品學報, 2012, 12(1):182-190.
[5]李艷, 董振玲, 李佳, 等. PCR-DGGE技術檢測羊羔美酒大曲中細菌多樣性[J]. 食品科學, 2015,(12): 142-147.
[6]謝廣發. 黃酒釀造技術[M].北京:中國輕工業出版社,2010.
[7]汪建國, 沈玉根, 陸偉杰, 等. 我國黃酒研究現狀與發展趨勢[J]. 中國釀造, 2012, 31(11): 15-20.
[8]毛青鐘. 黃酒生產特點的探討[J]. 釀酒科技, 2005,(1):65-67.
[9]傅金泉. 黃酒生產技術[M].北京:化學工業出版社,2005.
[10]TAE-WOONKIM, JUN-HWALEE, SUNG-EONKIM. Analysis of microbial communities in doenjang, a Korean fermented soybean paste, nested PCR-denaturing gradient gel electrophoresis[J]. International Journal of Food Microbiology,2009, 131: 265-271.
[11]王海舟,張小波. 分子生物學在微生物生態學中的應用[J]. 萍鄉高等專科學校學報, 2009, 26(3): 72-74.
[12]劉紹雄, 王明月, 王娟, 等. 基于PCR-DGGE技術的劍湖濕地湖濱帶土壤微生物群落結構多樣性分析[J]. 農業環境科學學報, 2013,(7): 1405-1412.
[13]陳寶良, 毛青鐘. 傳統香雪酒發酵過程微生物變化和作用研究[J]. 釀酒, 2010, 37(6): 59-62.
(責任編輯:黃愛萍)
Composition and Dynamic Changes of Bacterial Community During Traditional Hongqu Glutinous Rice Winemaking
JIA Rui-bo1,2,PAN Yu-yang1,2,HU Rong-kang1,2,ZHOU Wen-bin1,2,JIANG Ya-jun1,2,GUO Wei-ling1,2, ZHU Feng-feng1,2,WU Lin-xiu1,2, LIU Bin1,2,RAO Ping-fan3, NI Li3, Lü Xu-cong1,2,3*
(1.NationalEngineeringResearchCenterofJuncaoTechnology,FujianAgricultureandForestryUniversity,Fuzhou,Fujian350002,China; 2.CollegeofFoodScience,FujianAgricultureandForestryUniversity,Fuzhou,Fujian350002,China; 3.InstituteofFoodScienceandTechnology,CollegeofBiologicalScienceandTechnology,FuzchouUniversity,Fuzhou,Fujian350108)
Composition and dynamic changes of the bacterial community during the traditional Hong Qu glutinous rice winemaking were studied using PCR-denaturing gradient gel electropherisis (DGGE), clone library analysis and restriction fragment length polymorphisms (RFLP). Sequences of 9 bacteria strains were obtained by DGGE spectrogram. Through cloning and sequencing, it was found thatLactobacillusplantarum,PediococcuspentosaceuandLactobacillusbreviswere the dominant bacteria during the initial fermentation stage. The RFLP fingerprints showed that 16S rDNA clone libraries had 19 band patterns. Sequencing of different OTUs revealed thatL.plantarum,P.pentosaceus,L.brevisandLactobacilluspentosuspresented in the wine during the entire process, butL.plantarumprevailed at the end. By using various molecular biology methods simultaneously to examine the microbial community in winemaking, a comprehensive and objective profile was obtained.
hongqu glutinous rice wine; traditional brewing; bacterial community
2016-05-09初稿;2016-09-07修改稿
賈瑞博(1991-),男,碩士生,研究方向:食品生物技術(E-mail:13044599915@163.com) *通訊作者:呂旭聰(1984-),男,助理研究員,博士;研究方向:食品生物技術(E-mail:xucong1154@qq.com)
國家自然科學基金項目(31601466);中國博士后科學基金項目(2016T90591、2015M570549);福建農林大學“校杰出青年科研人才”項目(XJQ201607);福建省自然科學基金項目(2016J01095)
TS 262.4
:A
:1008-0384(2016)11-1238-06
賈瑞博,潘雨陽,胡榮康,等.紅曲黃酒傳統釀造過程中的細菌菌群結構及其動態變化[J].福建農業學報,2016,31(11):1238-1243.
JIA R-B,PAN Y-Y,HU R-K,et al.Composition and Dynamic Changes of Bacterial Community During Traditional Hongqu Glutinous Rice Winemaking[J].FujianJournalofAgriculturalSciences,2016,31(11):1238-1243.

