基于16S rRNA和rpoB基因的分子系統發育分析在銅綠假單胞菌鑒定中的應用
何艷霞,向詩非,李澆,何金蕾,張俊榮,袁冬梅,秦翰霄,陳達麗*,陳建平*
(1.四川大學華西基礎醫學與法醫學院寄生蟲學教研室,四川成都610041; 2.湖北民族學院醫學院寄生蟲學教研室,湖北恩施445000; 3.湖北民族學院附屬民大醫院風濕免疫科,湖北恩施445000)
基于16S rRNA和rpoB基因的分子系統發育分析在銅綠假單胞菌鑒定中的應用
何艷霞1,2,向詩非3,李澆1,何金蕾1,張俊榮1,袁冬梅1,秦翰霄1,陳達麗1*,陳建平1*
(1.四川大學華西基礎醫學與法醫學院寄生蟲學教研室,四川成都610041; 2.湖北民族學院醫學院寄生蟲學教研室,湖北恩施445000; 3.湖北民族學院附屬民大醫院風濕免疫科,湖北恩施445000)
為對比16S rRNA和rpoB基因分子系統發育分析與傳統表型分類法對銅綠假單胞菌的鑒定,評估16S rRNA和rpoB基因序列分析在銅綠假單胞菌鑒定中的應用,用表型分類方法對臨床自動微生物鑒定系統鑒定為銅綠假單胞菌的23株分離株進行再鑒定,PCR擴增23株分離株16S rRNA和rpoB基因片段,并測序進行系統發育分析。結果表明,表型再鑒定結果與自動微生物鑒定系統鑒定結果一致。基于兩個基因的系統發育分析均顯示分離株p22與不動桿菌屬序列聚為一枝,其余22株分離株與銅綠假單胞菌序列聚為一枝。因此p22應鑒定為不動桿菌,16S rRNA和rpoB基因序列分析均能準確鑒定銅綠假單胞菌并能較好建立假單胞菌屬內種間關系。
銅綠假單胞菌;16S rRNA;rpoB
銅綠假單胞菌(Pseudomonas aeruginosa)廣泛分布于自然界,是一種條件致病菌,可感染人體的任何部位和組織,出現局部化膿性炎癥。銅綠假單胞菌也廣泛分布于醫院環境,在醫院感染病例中,由銅綠假單胞菌引起的醫院感染占據首位[1]。銅綠假單胞菌耐藥性強,耐藥譜廣,隨著多藥耐藥菌株的出現,尤其是對7種以上或所有抗生素全部耐藥的泛耐藥株的出現,銅綠假單胞菌常使臨床醫生陷入無藥可用的境地。假單胞菌屬自1894年建立以來,其菌種數目在不斷增加,并且隨分類方法的改善,其分類體系不斷被修正,目前,假單胞菌屬已成為革蘭陰性菌中菌種數量最多的屬。不同的菌種作用于不同種類的生物,具有不同的致病特性,準確而快速地鑒定出對人類有嚴重危害的銅綠假單胞菌,對盡早控制銅綠假單胞菌感染及預防銅綠假單胞菌耐藥株的擴散具有非常重要的意義。醫院常采用瓊脂平板培養法對細菌進行分離培養,用全自動微生物鑒定系統進行菌種鑒定和藥物敏感性試驗,其鑒定系統是基于生化試驗做出的鑒定,結果并不十分可靠[2-3]。近30年來,分子生物學技術被越來越多地應用于重要病原菌的鑒定。16S rDNA是細菌染色體上編碼16S rRNA的基因序列,存在于所有細菌染色體基因組中,由于其在進化過程中高度保守,又具有相對的獨特性,已被廣泛應用于細菌的鑒定[4-5]。rpoB基因編碼DNA依賴的RNA聚合酶β亞單位,是一個高度保守的管家基因,也常被用作細菌鑒定的標示基因和系統發育分析的位點,在假單胞菌的鑒定中經常被用到[6-8]。本研究對23株銅綠假單胞菌臨床分離株進行16S rRNA及rpoB基因序列系統發育分析,對比臨床常用的基于生化試驗的分類方法,評估這兩個基因對鑒定銅綠假單胞菌的意義,并為臨床鑒定提供指導意見。
1 材料與方法
1.1 材料
23株銅綠假單胞菌分離自2014年3月至6月間湖北省恩施土家族苗族自治州中心醫院和湖北民族學院附屬民大醫院病人痰液、膿液、支氣管肺泡灌洗液等標本。菌株在醫院檢驗科經分離培養,并經全自動微生物鑒定系統VITEK 32(BioMerieus,Inc.,MarayI’Etoil,France)鑒定為銅綠假單胞菌(可能概率>99.0%),所有菌株均具有必需的生化試驗特征(結果見表1)。綠膿桿菌生化鑒定盒購自廣東環凱公司,細菌基因組提取試劑盒和膠回收試劑盒購自北京天根公司,2×Taq Master Mix購自上海欣百諾公司。PCR引物合成和測序均由成都擎科公司完成。
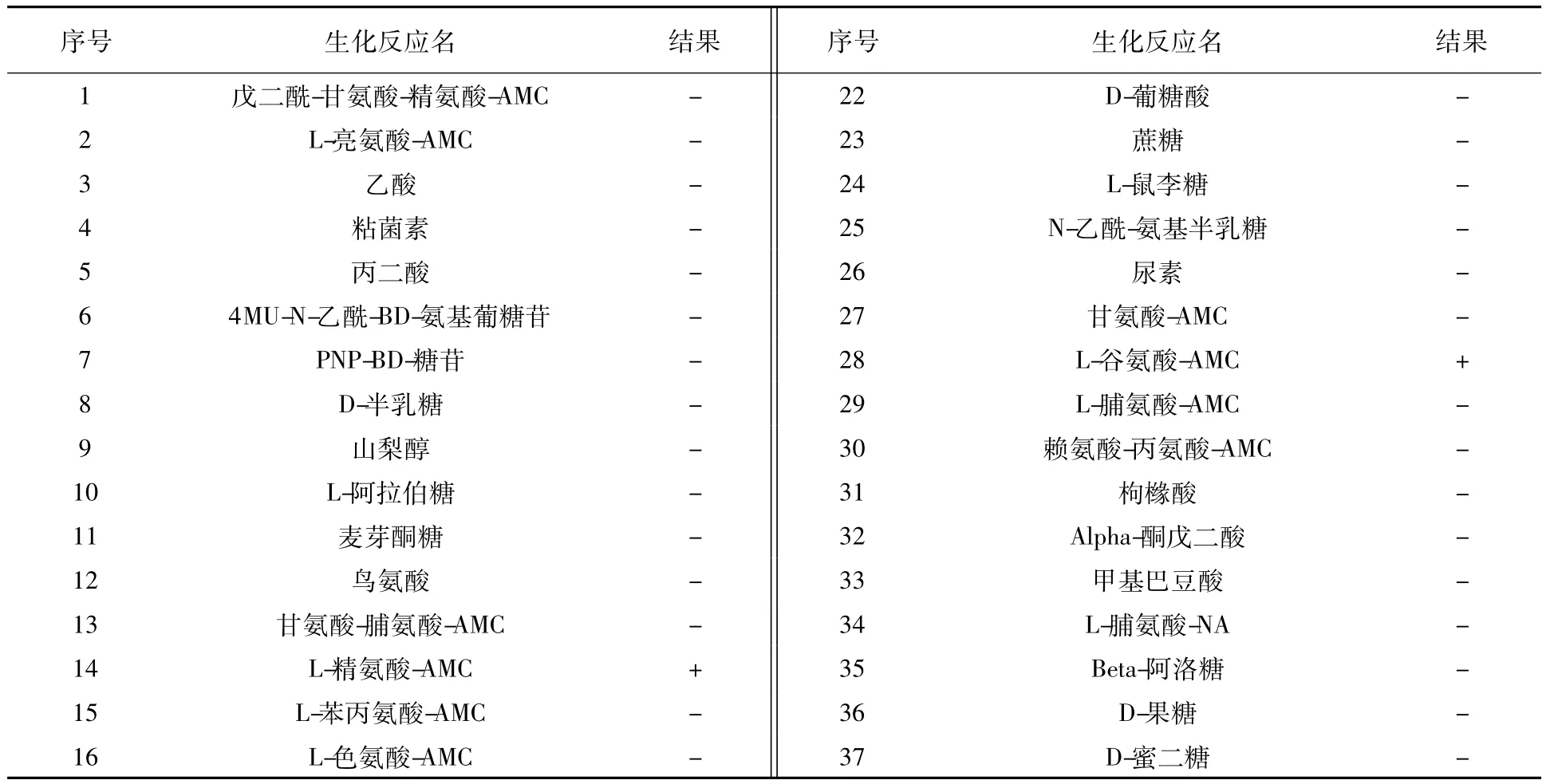
表1 所有臨床分離株生化反應結果列表Table 1The list of biochemical characteristics for all clinical isolates
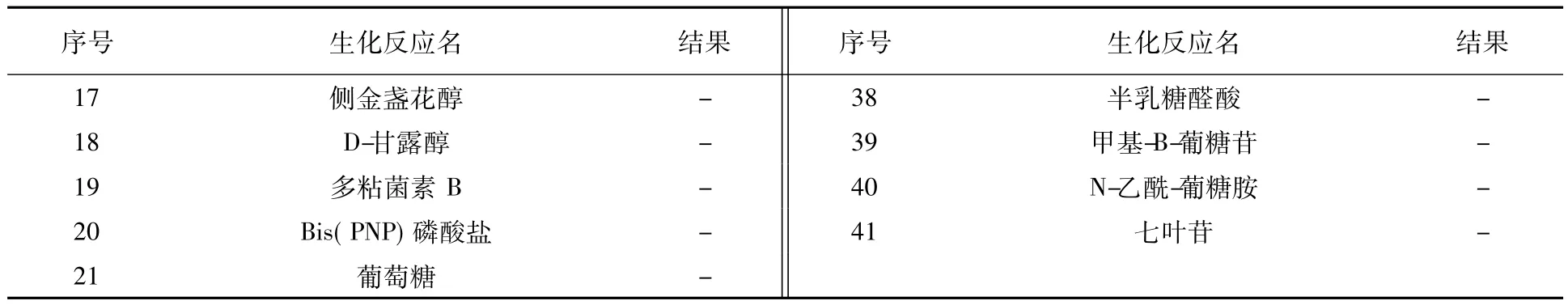
續表1
1.2 方法
1.2.1 細菌培養、再鑒定及DNA提取23個臨床分離株均分別接種于LB平板、麥康凱平板、血平板,37℃培養18~24 h,觀察并記錄在不同培養基上的生長特征。挑取單個菌落進行革蘭染色,并用綠膿桿菌生化鑒定盒進行補充生化鑒定。DNA提取按照試劑盒操作流程進行。
1.2.2 PCR擴增對23個分離株進行16S rRNA基因和rpoB基因PCR擴增。16S rRNA基因擴增選用通用引物27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R(5'-TACGGYTACCTTGTTACGACTT-3')[9]。PCR反應體系為50 μL,包括上、下游引物各0.4 μmol/L,模板2 μL,2×Taq Master Mix 25 μL。反應條件為94℃預變性5 min;94℃變性30 s,58℃退火40 s,72℃延長2 min,共30個循環;72℃終末延長5 min。rpoB基因擴增選用上游引物5'-GGCGAAATGGCWGAGAACCA-3',下游引物5'-GAGTCTTCGAAGTTGTAACC-3'[10]。PCR反應體系同前,反應條件為94℃預變性5 min;94℃變性45 s,50℃退火30 s,72℃延長1 min,共30個循環;72℃終末延長5 min。所有PCR產物均通過1.5%瓊脂糖凝膠電泳鑒定。
1.2.3 基因測序及分子系統發育分析按照產品說明書推薦操作,對23株臨床分離株的16S rRNA基因和rpoB基因PCR產物進行凝膠回收,送成都擎科公司測序,測序結果經校正后上傳GenBank。將新獲得序列在GenBank中進行Blast比對,了解在GenBank中與其同源性較高的序列情況。從GenBank下載假單胞菌屬及其他相關屬16S rRNA基因和rpoB基因參考序列各43條,在表3中列出,Pragiafontium為外類群,與新獲序列進行比對研究。采用軟件CLUSTAL X分別對16S rRNA基因和rpoB基因序列進行多序列匹配排列,采用軟件MEGA 4.0對排列矩陣進行微調,用MrBayes v3.2進行貝葉斯分析,矩陣最優數據模型通過Modeltest 3.7選擇獲得,貝葉斯分析結果使用軟件FigTree v1.4.2查看。
2 結果與分析
2.1 細菌再鑒定
2.1.1 培養特征該23株分離株在LB培養基上均生長良好,菌落扁平、濕潤,培養基呈亮綠色。在麥康凱平板上的表現有4種:形成典型的灰綠色具金屬光澤的菌落;圓形凸起的灰白色半透明似大腸埃希菌菌落;光滑凸起成粘液狀的粘液性菌落;中央凸起、邊緣扁平、表面粗糙的粗糙性菌落。在血平板上可見透明溶血環。
2.1.2 革蘭染色及形態特征經革蘭染色,在光學顯微鏡下觀察,23株銅綠假單胞菌均為革蘭染色陰性,桿菌,長短不一,單獨、成雙或短鏈狀排列,無芽胞,有莢膜。
2.1.3 生化反應在本實驗室再鑒定中,23株臨床分離株具有相同生化反應結果,見表2。該結果支持醫院做出的銅綠假單胞菌的鑒定。

表2 所有臨床分離株補充生化反應結果列表Table 2The list of supplementarybiochemical characteristics for all clinical isolates
2.2 分子系統發育分析
2.2.1 基于16S rRNA基因的分子系統發育分析23株分離株均擴增出約1 500 bp大小的16SrRNA基因片段,測序獲得的序列存儲于Gen-Bank,其編號為KU372980~KU373002(見表3)。p22的16S rRNA序列比對與不動桿菌未定種(Acinetobacter sp.)ADP1基因組序列同源性最高(99%),其次是鮑曼不動桿菌(Acinetobacter baumannii)AC29(98%),其余22株序列(p1、p2、p3、p4、p5、p6、p7、p8、p9、p10、p11、p12、p13、p15、p16、p18、p20、p23、p24、p25、p26、p27,以下簡稱22株)與銅綠假單胞菌(Pseudomonas aeruginosa) F22031基因組序列同源性最高(99%或100%)。Modeltest獲得的最優數據模型為GTR+I+G。對16S rRNA數據矩陣進行貝葉斯分析,構建的進化樹中(圖1)可見所有假單胞菌屬序列以及2個固氮菌屬序列聚成一枝Clade I,其后驗概率(posterior probability)為1.0。22株臨床分離株與銅綠假單胞菌聚成Clade I中的一個亞枝A,其后驗概率為0.95。另一個分離株p22則與不動桿菌屬序列聚成一枝Clade II,后驗概率為1.0。
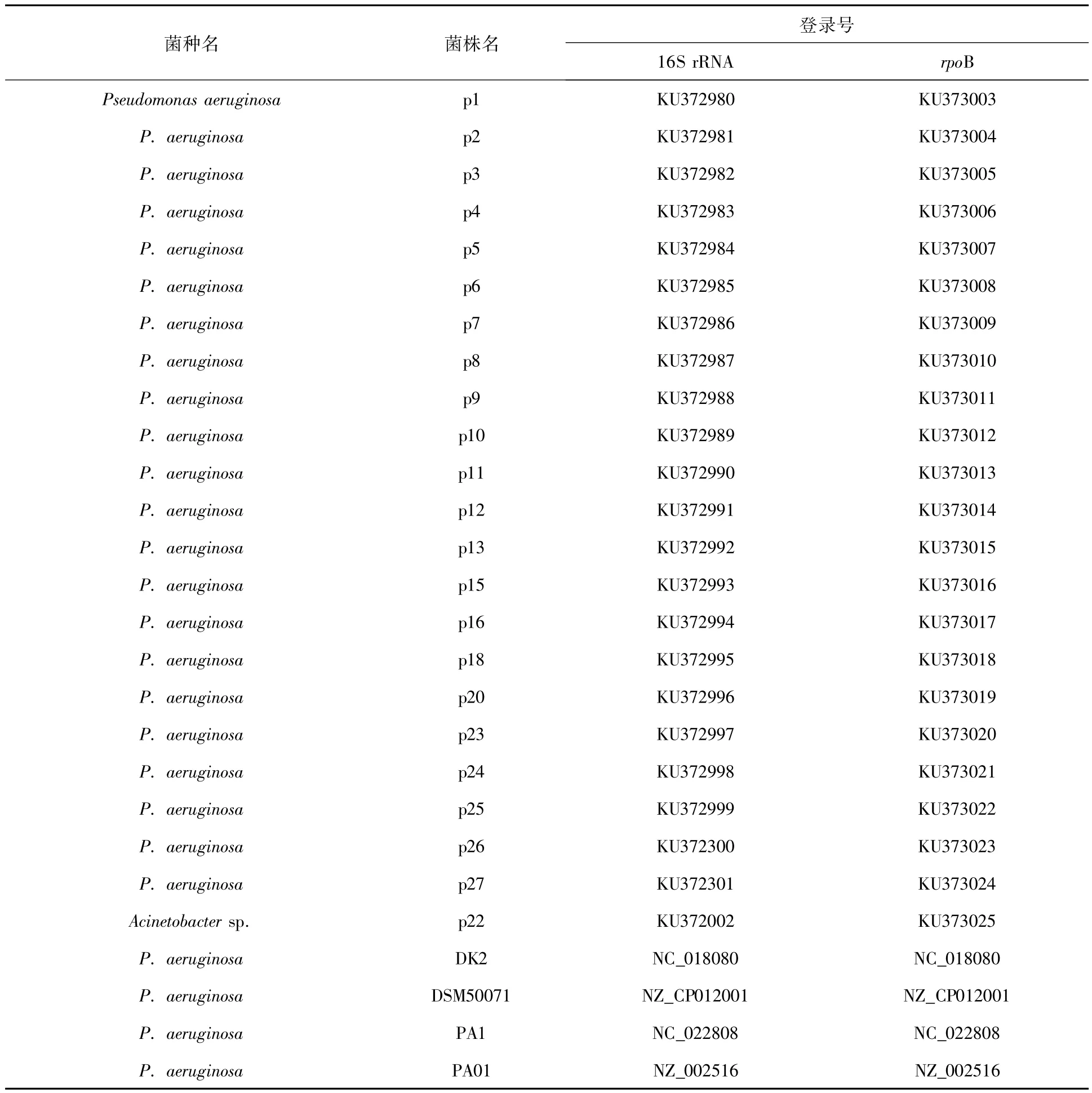
表3 用于16S rRNA和rpoB分析的種、株及GenBank登錄號Table 3List of analyzed species,strains and the GenBank accession number for 16S rRNA and rpoB

續表3
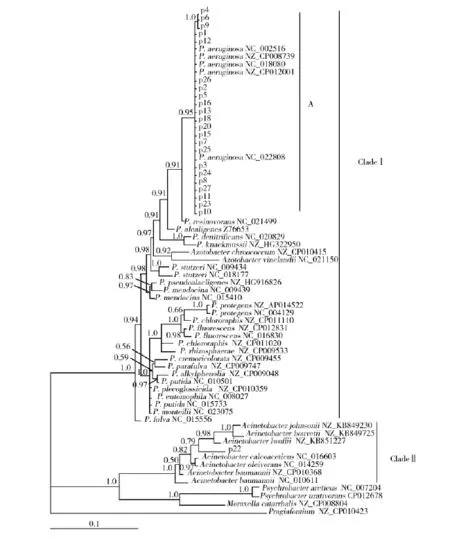
圖1 分離菌株與GenBank中相關菌株的16S rRNA系統發育樹Fig.116S rRNA phylogenetic tree of Pseudomonas strains from this study and GenBank進化樹采用MrBayes v3.2軟件進行貝葉斯分析獲得,cut-off值為50%,節點處為貝葉斯后驗概率,下圖同The 50%majority-rule consensus tree inferred from Baysain inference using MrBayes v3.2.Bayesian posterior probabilities were show at nodes,same the follow
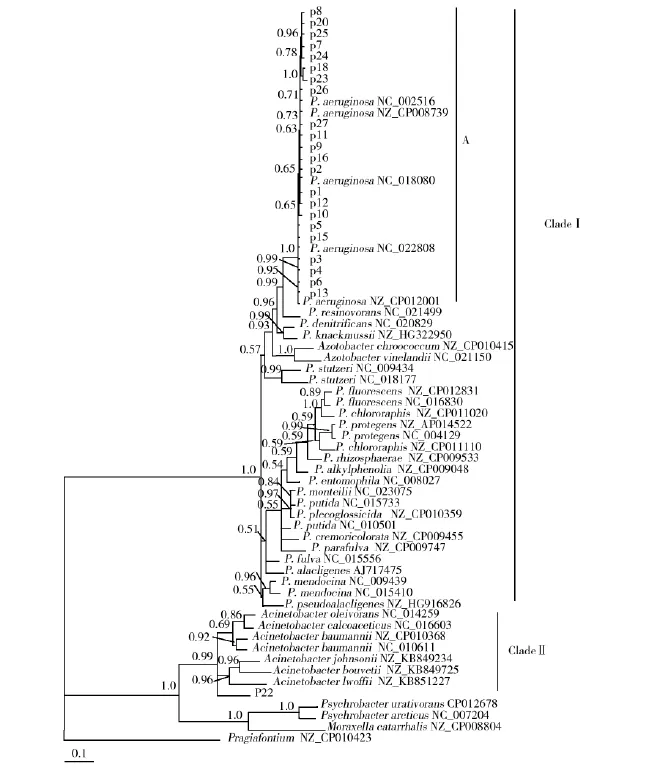
圖2 分離菌株與GenBank中相關菌株的rpoB系統發育樹Fig.2rpoB phylogenetic tree of Pseudomonas strains from this study and GenBank
2.2.2 基于rpoB基因的分子系統發育分析23株分離株均擴增出約980 bp大小的rpoB基因片段,測序獲得的序列存儲于GenBank,其編號為KU373003~KU373025(見表3)。p22的rpoB序列比對與不動桿菌未定種(Acinetobacter sp.) ADP1基因組序列同源性最高(88%),其次是鮑曼不動桿菌(Acinetobacter baumannii)6411(同源性84%),其余22株序列與銅綠假單胞菌(Pseudomonas aeruginosa)213BR基因組序列同源性最高(99%或100%)。Modeltest獲得的最優數據模型為TIM+I+G。在rpoB數據矩陣構建的貝葉斯樹中(圖2),所有假單胞菌屬序列以及2個固氮菌屬序列聚成一枝Clade I,其后驗概率為1.0。22株分離株與銅綠假單胞菌聚成Clade I中的一個亞枝A,后驗概率(posterior probability)為1.0。另一個分離株p22則與不動桿菌屬序列聚成一枝Clade II,后驗概率為0.99。
3 討論
傳統的細菌鑒定主要依靠對細菌表型特征的觀察,如菌落形態、染色后鏡下形態、生化特征等。自動微生物鑒定系統是將傳統的生化鑒定試劑微型化,制成干燥的微型鑒定試劑板條,采用數理統計原理的編碼鑒定方法,結合現代電子技術而形成的一種鑒定方法[11]。其應用簡便、快速,但鑒定系統的準確性受到系統數據庫覆蓋范圍的影響,由于微生物分類在不斷變化,使得一些鑒定系統在使用一段時間后與新的分類系統逐漸發生偏差,準確率下降。相近屬的細菌其生化特征亦較相似,這同樣影響鑒定的準確率。來自接受抗生素治療病人的分離株,其生化特性可能有所改變,鑒定結果也可能被影響[11]。
16S rRNA和rpoB基因已被廣泛應用于細菌的分子分類及鑒定研究。基于16S rRNA的分析被普遍認為能鑒定細菌至屬水平,而不能解決近緣種之間的分類問題[12-13]。rpoB基因表現出比16S rRNA更大的優越性。Marianelli等[14]通過對布魯氏菌rpoB基因的研究,認為rpoB基因能用于鑒定所有的布魯氏菌種及大部分的生物變種。Mollet等[15]通過對腸桿菌科14個種的細菌的16S rRNA和rpoB基因的研究,認為rpoB基因部分序列能準確鑒定腸道細菌。Adékambi等[16]認為,rpoB基因測序能準確反映細菌的G+C%含量,DNA-DNA雜交水平及平均核苷酸同源性(2個菌株共享的全基因組序列的比例),能在種和亞種水平解決細菌系統發育關系,可作為一個強有力的臨床微生物鑒定工具。16S rRNA和rpoB基因分析的這種差異可能是因為16S rRNA基因分子結構高度保守且在許多細菌基因組中有多個拷貝[17],因而確定其序列的準確性降低,而管家基因rpoB則只具有單一拷貝[18],且不同菌株rpoB基因變異程度明顯高于16S rRNA[15]。
本研究中,23株臨床分離株被自動微生物鑒定系統鑒定為銅綠假單胞菌,在本實驗室進行再鑒定時,該23株菌在培養特性、形態特征、生化特征等方面均符合銅綠假單胞菌的特點,與臨床鑒定結果相一致。但經進一步的基因分析,分離株p22分析結果明顯不同于臨床鑒定結果,其與不動桿菌屬更為接近。
p22的16S rRNA和rpoB基因序列在Gen-Bank中進行Blast比對時,其同源性最高的序列為不動桿菌,且在16S rRNA和rpoB數據矩陣構建的貝葉斯樹中,p22均與不動桿菌屬序列聚成一枝,而其余22株分離株則均與銅綠假單胞菌序列聚成一枝。在2棵貝葉斯樹中,p22均未有高的后驗概率支持與某種不動桿菌聚為一枝,且Clade II枝中各單倍型相距較遠(分析結果未在資料中顯示),無法構建有意義的單倍型網絡圖以確認p22與該枝中各種的親緣遠近關系。由此,p22鑒定為不動桿菌(種未定)更為準確。
在本研究中,基于16S rRNA和rpoB的貝葉斯樹拓撲結構高度相似,22株分離株序列及下載的5個銅綠假單胞菌序列被高的后驗概率支持聚為一枝,且假單胞菌屬內種間關系清晰,表明16S rRNA和rpoB基因在假單胞菌屬系統發育研究中均能較準確反映種間關系。在此,rpoB基因并未表現出對16S rRNA的明顯優勢,這可能是假單胞菌屬內最近為種間關系,而rpoB基因在親緣關系更近的菌株鑒定中優勢更為突出有關[16]。
zen等[19]曾用基于基因組序列的3種方法研究棕色固氮菌(Azotobacter vinelandii)與假單胞菌屬的關系,結果均顯示棕色固氮菌與假單胞菌屬高度相似,表明固氮菌實際應屬于假單胞菌。本研究基于16S rRNA和rpoB的貝葉斯樹中,2個固氮菌序列均與假單胞菌屬序列聚為一枝,再一次肯定了zen等的研究。
本研究對臨床分離的23個菌株用傳統表型分類方法和DNA測序分析進行了再鑒定,傳統表型分類方法鑒定23株均為銅綠假單胞菌,16S rRNA和rpoB基因序列分析鑒定22株分離株為銅綠假單胞菌,分離株p22應為不動桿菌。16S rRNA和rpoB基因能準確鑒定銅綠假單胞菌臨床分離株,并能較準確建立假單胞菌屬種間關系。
[1]金美娟,李秀紅,邢旺興.236株銅綠假單胞菌感染的臨床分析[J].中華醫院感染學雜志,2005,15(3):348-350.
[2]Farmer JJ,Davis BR,Hickman-Brenner FW,et al.Biochemical identification of new species and biogroups of Enterobacteriaceae isolated from clinical specimens[J].J ClinMicrobiol,1985,21(1):46-76.
[3]Capurro A,Artursson K,Waller KP,et al.Comparison of a commercialized phenotyping system,antimicrobial susceptibility testing,and tuf gene sequence-based genotyping for specieslevel identification of coagulase-negative staphylococci isolated from cases of bovine mastitis[J].Vet Microbiol,2009,134 (3-4):327-333.
[4]BaghalAsghari F,Nikaeen M,Mirhendi H.Rapid monitoring of Pseudomonas aeruginosa in hospital water systems:a key priority in prevention of nosocomial infection[J].FEMS Microbiol-Lett,2013,343(1):77-81.
[5]Harris KA,Hartley JC.Development of broad-range 16S rDNA PCR for use in the routine diagnostic clinical microbiology service[J].J Med Microbiol,2003,52(Pt 8):685-691.
[6]Ait Tayeb L,Ageron E,Grimont F,et al.Molecular phylogeny of the genus Pseudomonas based on rpoB sequences and application for the identification of isolates[J].Res Microbiol,2005,156(5-6):763-773.
[7]Mollet C,Drancourt M,Raoult D.rpoB sequence analysis as a novel basis for bacterial identification[J].MolMicrobiol,1997,26(5):1005-1011.
[8]Seok Y,Shin H,Lee Y,et al.First report of bloodstream infection caused by Pseudomonas fulva[J].J ClinMicrobiol,2010,48(7):2656-2657.
[9]Lane DJ.16S and 23S rRNA sequencing.In:Stackebrandt E,Goodfellow M(eds)Nucleic acids techniques in bacterial systematics[M].Chichester:John Wiley&Sons,1991:115-175.
[10]Diancourt L,Passet V,Verhoef J,et al.Multilocus sequence typing of Klebsiellapneumoniae nosocomial isolates[J].J Clin-Microbiol,2005,43(8):4178-4182.
[11]吳會桃,蔡芷荷,吳清平,等.細菌鑒定系統的應用研究進展[J].中國衛生檢驗雜志,2010,20(9):2381-2384.
[12]Harris KA,Hartley JC.Development of broad-range 16S rDNA PCR for use in the routine diagnostic clinical microbiology service[J].J Med Microbiol,2003,52(Pt 8):685-691.
[13]Stackebrandt E,Goebel BM.Taxonomic note:a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J].Int J SystBacteriol,1994,44(4):846-849.
[14]Marianelli C,Ciuchini F,Tarantino M,et al.Molecular characterization of the rpoB gene in Brucella species:new potential molecular markers for genotyping[J].Microbes Infect,2006,8(3):860-865.
[15]Mollet C,Drancourt M,Raoult D.rpoB sequence analysis as a novel basis for bacterial identification[J].MolMicrobiol,1997,26(5):1005-1011.
[16]Adékambi T,Drancourt M,Raoult D.The rpoB gene as a tool for clinical microbiologists[J].Trends Microbiol,2009,17 (1):37-45.
[17]Pei AY,Oberdorf WE,Nossa CW,et al.Diversity of 16S rRNA genes within individual prokaryotic genomes[J].Appl Environ Microbiol,2010,76(12):3886-3897.
[18]Kim BJ,Kim CJ,Chun J,et al.Phylogenetic analysis of the genera Streptomyces and Kitasatospora based on partial RNA polymerase beta-subunit gene(rpoB)sequences[J].Int J SystEvolMicrobiol,2004,54(Pt 2):593-598.
[19]zen AI,Ussery DW.Defining the Pseudomonas genus:where do we draw the line with Azotobacter[J].MicrobEcol,2012,63(2):239-248.
Molecular Phylogeny Analysis Based on 16S rRNA and rpoB Sequences for Identification of Clinical Isolates of Pseudomonas aeruginosa
HE Yan-xia1,2,XIANG Shi-fei3,LI Jiao1,HE Jin-lei1,ZHANG Jun-rong1,YUAN Dong-mei1,QIN Han-xiao1,CHEN Da-li1,CHEN Jian-ping1
(1.Teach.&Res.Div..of Parasitol.,W.China Schl.of Basic&Forensic Med.,Sichuan Uni.,Chengdu 610041;2.Teach.&Res.Div.of Parasitol.,Coll.of Med.,Hubei coll.for Minority Nationalities,3.Affil.Minda Hosp.,Hubei Uni.for National's,Enshi 445000)
16S rRNA and rpoB gene sequence analysis to identify of Pseudomonas aeruginosa was compared with conventional phenotypic methods to evaluate the application of 16S rRNA and rpoB sequence analysis to identify P.aeruginosa;23 isolated strains that were re-characterized with phenotypic classification method that was identified as P.aeruginosa by clinical automatic microbial identification system.16S rRNA and rpoB gene fragments were amplified with PCR and sequenced for molecular phylogenic analysis.The results showed that the re-characterized results were consistent with those characterized with automatic microbial identification system.Using Bayesian phylogenetic analyses based on 16S rRNA and rpoB genes,22 isolates are characterized as P.aeruginosa,whereas one isolate,p22,was classified as Acinetobacter sp.p22 should be identified as Acinetobacter sp.16S rRNA and rpoB genes can accurate identify P.aeruginosa and could fairly establish the interspecific relation within the genus of Pseudomonas.
Pseudomonas aeruginosa;16S rRNA;rpoB
Q939.11+2
A
1005-7021(2016)04-0027-09
10.3969/j.issn.1005-7021.2016.04.005
國家自然科學基金項目(31572240,81171607,J1103604);四川大學與瀘州市合作項目(2013CDLZ-S16)
何艷霞女,講師,博士研究生。研究方向為分子病原生物學。E-mail:49300492@qq.com
*通訊作者。陳達麗女,副教授,博士。研究方向為病原鑒定及分子系統發育。E-mail:cdl1978119@sina.com陳建平男,教授,博士,博士生導師。研究方向為分子病原生物學。E-mail:jpchen007@163.com
2016-01-07;
2016-06-19

