應(yīng)用DNA條形碼技術(shù)對市場苦木藥材的檢測研究*
陳 影,張景景,湯 歡,王 俊,趙李龍,孫 偉**
( 1.中國中醫(yī)科學(xué)院 中藥研究所 中藥鑒定與安全性檢測評估北京市重點實驗室 北京 100700;2.南陽理工學(xué)院張仲景國醫(yī)國藥學(xué)院 南陽 473000;3.四川農(nóng)業(yè)大學(xué)林學(xué)院 成都 611130;4.武漢理工大學(xué)化學(xué)化工與生命科學(xué)學(xué)院 武漢 430070)
應(yīng)用DNA條形碼技術(shù)對市場苦木藥材的檢測研究*
陳 影1,張景景2,湯 歡3,王 俊4,趙李龍2,孫 偉1**
( 1.中國中醫(yī)科學(xué)院 中藥研究所 中藥鑒定與安全性檢測評估北京市重點實驗室 北京 100700;2.南陽理工學(xué)院張仲景國醫(yī)國藥學(xué)院 南陽 473000;3.四川農(nóng)業(yè)大學(xué)林學(xué)院 成都 611130;4.武漢理工大學(xué)化學(xué)化工與生命科學(xué)學(xué)院 武漢 430070)
目的:利用 DNA條形碼技術(shù)對市場上中藥材苦木進行檢測,為其市場監(jiān)管及用藥安全提供保障。方法:對15份苦木藥材(每份取樣2個)共30個樣品進行DNA提取,PCR擴增其ITS2序列并雙向測序,應(yīng)用CodonCode Aligner version 6.0.2軟件對測序峰圖進行校對拼接,將得到的所有序列與GenBank以及中藥材DNA條形碼鑒定系統(tǒng)分別進行比對,選擇相似度最相近的物種在中國植物志網(wǎng)站上查詢。在此基礎(chǔ)上擴增其psbA-trnH序列以驗證結(jié)果的可靠性。結(jié)果:市場購買的苦木樣品中正品苦木Picrasma quassioides占70%、臭椿Ailanthus altissima占6.67%、板藍Baphicacanthus cusia占13.33%、楝葉吳茱萸Tetradium glabrifolium占3.33%、柘Maclura tricuspidata占6.67%。結(jié)論:市場上苦木藥材存在混偽品,DNA條形碼技術(shù)能有效鑒別藥材真?zhèn)巍?/p>
苦木 DNA條形碼 ITS2序列 psbA-trnH序列
中藥材傳統(tǒng)的鑒定方法主要是性狀鑒別、顯微鑒別和化學(xué)特征鑒別[1]。然而,性狀鑒別受藥材原植物的生長環(huán)境以及藥材是否炮制加工的影響較大,由于組織特征的相似性且組織結(jié)構(gòu)易受地理環(huán)境、生長期、儲存條件諸多因素的影響[2]。化學(xué)特征受生長環(huán)境影響較大,加上中藥大多數(shù)有效成分復(fù)雜且不明確,變異幅度大,同時其有效成分的含量受采收時間等諸多因素影響,因此難以規(guī)定一個合理的數(shù)值標準,從而影響到化學(xué)鑒定的準確性[2]。
近年來,由于限制性內(nèi)切酶的發(fā)現(xiàn)、DNA重組技術(shù)的建立、DNA序列快速測定方法的發(fā)明,使得分子生物學(xué)及其技術(shù)以迅猛的速度發(fā)展,帶動了生物學(xué)乃至整個科學(xué)的發(fā)展,鑒定中藥材的真?zhèn)我搽S之有所改善[3,4]。DNA條形碼分子鑒定技術(shù)首先于2003年由Hebert等[5]提出,即通過一段DNA序列,從基因水平對物種進行快速準確有效的鑒定。陳士林等[6]提出將ITS2序列作為通用序列來鑒定藥用植物,并輔以psbA-trnH的補充序列,推動了中藥DNA條形碼鑒定技術(shù)。目前,DNA條形碼技術(shù)已經(jīng)穩(wěn)定地應(yīng)用于紅景天[7]、冬蟲夏草[8]、羌活[9]和靈芝[10]等中藥材鑒定。
苦木為苦木科苦木屬植物苦木Picrasma quassioides (D.Don) Benn.的干燥枝和葉。夏、秋兩季采收,干燥[11]。苦木性寒,味苦,有清熱解毒、祛濕的功效,用于風熱感冒、咽喉腫痛、濕熱瀉痢、濕疹、瘡癤、蛇蟲咬傷等癥。谷華[12]、趙文娜[13]等指出苦木具有多種藥理作用以及具有很好的臨床應(yīng)用價值。胡小平[14]也指出穴位注射苦木注射液治療小兒腹瀉臨床療效較好,建議臨床推廣。因此,進行苦木的質(zhì)量控制以及混偽品的鑒別尤為重要。然而目前關(guān)于苦木摻偽的現(xiàn)象報道較少,在市場上是否有偽品存在還有待鑒定。本研究利用DNA條形碼技術(shù)對市場上的苦木進行系統(tǒng)鑒定,以保障中藥材市場的藥品安全性,維護消費者的合法權(quán)益[15,16]。
本研究運用基于ITS2和psbA-trnH序列的DNA條形碼技術(shù)嘗試對15份苦木市場樣品(每份取樣2份)進行鑒定,檢測市場上苦木藥材是否存在混偽品。
1 材料
通過藥材市場購買以及淘寶店鋪購買等方式收集了商品名為苦木的藥材共15份,產(chǎn)地分別為廣西玉林、河池、河北保定、石家莊、北京、安徽亳州、浙江溫州和上海,每份取樣2份。樣本信息及形狀見表1、圖1。

表1 樣本信息

圖1 樣品圖片
2 方法
2.1 樣品的DNA提取
樣品用浸有75%乙醇的酒精棉擦拭實驗樣品的表面以除去雜菌污染,取實驗材料30 mg,用高通量組織研磨儀,在50 Hz頻率下研磨120 s后呈粉末狀態(tài);隨后加800 μL核分離液漂洗3次;使用百泰克新型快速基因組DNA提取試劑盒(BioTeke Corporation,中國),按其說明書提取樣品總DNA,并參考“中藥材DNA條形碼分子鑒定指導(dǎo)原則”對提取步驟進行適當調(diào)整[17],如將水浴條件調(diào)整為56℃后水浴過夜以更有效地提取得到DNA。
2.2 樣品DNA的PCR擴增和測序
ITS2序列擴增[18]正向引物為ITS2F:5'-ATGC GATACTTGGTGTGAAT-3',反向引物為ITS3R:5'-GACGCTTCTCCAGACTACAAT-3';psbA-trnH序列擴增[19]正向引物為:PA:5'-GTTATGCATGAAC GTAATGCTC-3',擴增反向引物為TH:5'-CGCGC ATGGTGGATTCACAATCC-3'。
PCR反應(yīng)體系為25 μL,包括:2×Taq PCR MasterMix 12.5 μL,ITS2序列(或者psbA-trnH序列)的正向和反向引物(2.5 μmol·L-1)各1.0 μL,雙蒸餾水8.5 μL,模板DNA 2.0 μL。
PCR擴增條件:ITS2序列擴增程序[18]為:94 ℃變性5 min;94 ℃變性1 min,55℃退火1 min,72℃延伸30 s(進行40個循環(huán));72℃延伸10 min。psbA-trnH序列擴增程序[19]為:94℃變性5 min;94℃變性1 min,55℃退火1 min,72℃延伸1.5 min(進行30個循環(huán));72℃延伸7 min。
電泳檢查PCR擴增情況,經(jīng)PCR擴增成功后的產(chǎn)物經(jīng)純化后進行雙向測序。
2.3 數(shù)據(jù)處理
使用CodonCode Aligner version 6.0.2(Codon Co.,USA)對所獲得的測序序列進行質(zhì)量分析和剪切校對拼接,剪去低質(zhì)量區(qū),采用基于隱馬爾科夫模型的HMMer[20]注釋方法去除兩端5.8S 和28S區(qū)段。將最終得到的所有序列與GenBank(http:// blast.ncbi.nlm.nih.gov/Blast.cgi)和中藥材DNA條形碼鑒定系統(tǒng)(http://tcmbarcode.cn)分別進行比對,然后選擇比對的相似度最相近的物種在中國植物志網(wǎng)站(http://frps.eflora.cn/)上進行查詢,得到相應(yīng)的物種信息,與所做的樣品進行比對。
3 結(jié)果與分析
3.1 DNA提取及PCR擴增結(jié)果分析
中藥材DNA提取是開展中藥材DNA條形碼研究的首要環(huán)節(jié)[21]。在大部分中藥材都含有大量的次生代謝產(chǎn)物,在本實驗中采用核分離液洗滌3次,盡可能除去存在于細胞核中的多糖、多酚等物質(zhì),從而能有效地提高所提取的DNA的純度;由于根莖類藥材質(zhì)地較為堅硬,不易提取,為了更有效地提取得到DNA,選擇56℃條件下水浴過夜。15份樣品,每份取樣2個,提取得到的DNA進行PCR擴增,其PCR產(chǎn)物經(jīng)電泳后均能顯示單一目的條帶(獲得30條ITS2序列,28條psbA-trnH序列)。
3.2 序列比對分析
將最終得到的所有序列與GenBank(http:// blast.ncbi.nlm.nih.gov/Blast.cgi)以及中藥材DNA條形碼鑒定系統(tǒng)(http://tcmbarcode.cn)分別進行比對,然后選擇比對的相似度最相近的物種在中國植物志(http://frps.eflora.cn/)網(wǎng)站上進行查詢。
ITS2序列鑒定的結(jié)果為苦木P. quassioides (KM1-1、KM1-2、KM2-1、KM2-2、KM3-1、KM3-2、KM4-1、KM4-2、KM5-1、KM5-2、KM6-1、KM6-2、KM9-1、KM9-2、KM10-1、KM10-2、KM12-1、KM12-2、KM13-1、KM13-2、KM14-1)的占總?cè)恿康?0%,與兩個庫的相似度分別在99%和99.6%以上;臭椿A. altissima(KM7-1、KM7-2)占6.67%,與兩個庫的相似度分別在99% 和 99.6%以 上;板 藍B. canthuscusia(KM8-1、KM8-2、KM11-1、KM11-2)占13.33%,與兩個庫的相似度分別在92%和91.8%以上;楝葉吳茱萸T. glabrifolium(KM14-2)占3.33%,與兩個庫的相似度分別在99%和100%;柘M. tricuspidata(KM15-1、KM15-2)占6.67%,與兩個庫的相似度分別是98%和98.3%。
psbA-trnH序列鑒定的結(jié)果為苦木P. quassioides(KM1-1、KM1-2、KM2-1、KM2-2、KM3-1、KM3-2、KM4-1、KM4-2、KM5-1、KM5-2、KM6-1、KM6-2、KM9-1、KM9-2、KM10-1、KM10-2、KM12-1、KM12-2、KM13-1、KM13-2、KM14-1)的占總?cè)恿康?0%,與兩個庫的相似度分別在98%和99.4%以上;臭椿A. altissima(KM7-1、KM7-2)占6.67%,與GenBank的相似度分別是100%,在DNA條形碼鑒定系統(tǒng)暫未收錄;板藍B. canthuscusia(KM8-1、KM8-2、KM11-1、KM11-2)占13.33%,與兩個的相度分別在99%和99.5%;楝吳茱萸T. glabrifolium(KM14-2)占3.33%,與GenBank的相似度分別是93%,在DNA條形碼鑒定系統(tǒng)暫未收錄;此實驗中psbA-trnH序列并未做出KM15-1和KM15-2為何物。
ITS2序列和psbA-trnH序列在2個庫中分別比對的相似度大部分在99%以上,由此可認為相對應(yīng)樣品鑒定結(jié)果可靠。而KM8-1、KM8-2、KM11-1和KM11-2的ITS2序列在GenBank 和DNA條形碼鑒定系統(tǒng)的比對相似度較低,而其psbA-trnH序列在GenBank和DNA條形碼鑒定系統(tǒng)的比對的相似度均在99%以上;KM14-2的ITS2序列在GenBank和DNA條形碼鑒定系統(tǒng)的比對的相似度在99%以上,而其psbA-trnH序列在與GenBank比對的相似度僅為93%。這表明在庫中相應(yīng)的序列信息可能有識別錯誤的情況。雖然如此,通過得到的ITS2序列和psbA-trnH序列與數(shù)據(jù)庫中比對得到的相似度,還是能夠為市場上中藥材苦木是否存在混偽品及混偽品類型提供一定參考。ITS2序列檢測后不同結(jié)果占總?cè)恿康陌俜直纫妶D2,ITS2序列和psbA-trnH序列比對的具體相似度見表2。
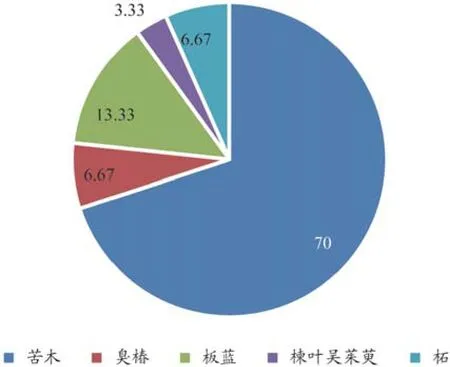
圖2 ITS2序列檢測后不同結(jié)果占總?cè)恿康陌俜直?/p>
4 討論
雖然目前暫未發(fā)現(xiàn)有關(guān)苦木混偽品的相關(guān)報道,但是通過此次研究發(fā)現(xiàn),商品藥材中存在混偽品,主要包括臭椿、板藍、楝葉吳茱萸和柘,臭椿和苦木乃同一科(苦木科)的植物,其余的樣品都不是苦木科植物。從外觀上(見圖1)觀察,臭椿和苦木性狀相似,但是臭椿的顏色較淺;板藍的性狀較苦木差距較大,而且質(zhì)地更加堅硬;但是楝葉吳茱萸和柘的飲片較苦木差別不明顯,這就對準確鑒別造成一定的影響。
陳士林團隊經(jīng)過近10年研究,對上千種中草藥及其混偽品的DNA條形碼進行研究,創(chuàng)建了“中藥材DNA條形碼生物鑒定體系”,從基因?qū)用娼鉀Q中藥材與混偽品的物種識別問題,DNA分子鑒定技術(shù)依靠這種有差異的DNA片段來做分子鑒定,不會受到藥材的外觀和是否進行加工等造成的影響,而且鑒別快速、精確,在中藥材鑒定上具有一定的準確性和優(yōu)越性,應(yīng)用前景很廣闊[22-24]。陳士林等[25]指出:DNA分子鑒定具有準確性、方法通用性、可數(shù)字化、可重復(fù)性、樣品處理簡單性和實用性等優(yōu)點,對于鑒定混偽品來說非常方便而且有效,在特定中藥鑒定中將發(fā)揮重要作用。近些年來,Wu[26]、馬孝熙[27]、凃媛[28]、樊佳佳[29]、熊超[30]、張雅琴等[31]所做的實驗研究表明應(yīng)用ITS2序列或psbA-trnH序列可以有效、準確穩(wěn)定地鑒別遠志、天仙子、竹茹、天竺黃、谷物芽類、石韋及其混偽品。本研究應(yīng)用ITS2序列和psbA-trnH序列對市場銷售的苦木商品進行檢測,結(jié)果顯示:ITS2序列和psbA-trnH序列可有效地鑒定苦木及其混偽品。在整個的實驗過程中,獲得高質(zhì)量的DNA是進行DNA條形碼研究的必要前提和關(guān)鍵環(huán)節(jié),這是因為中藥材中基本上都含有較多的次生代謝物質(zhì),往往會干擾植物DNA提取成功率,所以DNA提取方法應(yīng)當進行相應(yīng)改進[32,33]。
目前,傳統(tǒng)鑒定與分子鑒定相互獨立,如果能將二者有機整合起來,從“表型”和“遺傳型”2個層次共同鑒定,不僅可以提高中藥鑒定的準確性,而且將有利于中藥鑒定學(xué)的發(fā)展[34]。
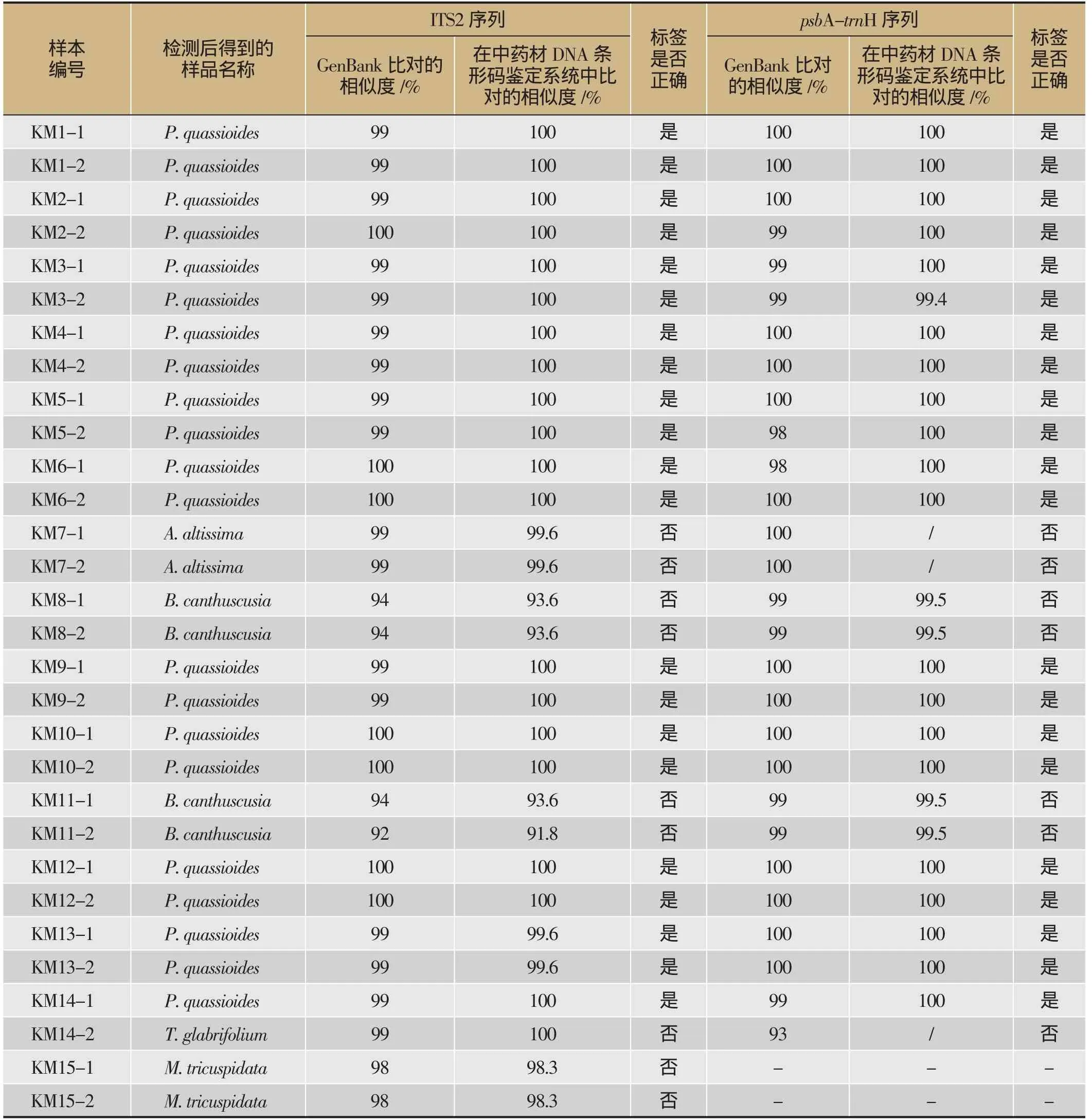
表2 基于ITS2和psbA-trnH序列的市售藥材為苦木的比對相似度
1 羅焜,馬培,姚輝,等.多基原藥材秦艽ITS2 條形碼鑒定研究.藥學(xué)學(xué)報, 2012, 47(12): 1710-1717.
2 袁慶軍,張文婧,姜丹,等.論中藥分子鑒定的方法和原則.植物分類與資源學(xué)報, 2012, 34(6): 607-613.
3 成新躍,周紅章,張廣學(xué).分子生物學(xué)技術(shù)在昆蟲系統(tǒng)學(xué)研究中的應(yīng)用.動物分類學(xué)報, 2000, 25(2): 121-133.
4 楊光明,蔡寶昌,王明艷,等.分子生物學(xué)技術(shù)在中藥鑒定中的應(yīng)用.世界科學(xué)技術(shù)-中醫(yī)藥現(xiàn)代化, 2001, 3(4): 29-35.
5 Hebert P D, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes. Proc Biol Sci, 2003, 270(1512): 313-321.
6 陳士林,龐曉慧,姚輝,等.中藥DNA條形碼鑒定體系及研究方向.世界科學(xué)技術(shù)-中醫(yī)藥現(xiàn)代化, 2011, 13(5): 747-754.
7 Xin T Y, Li X J, Yao H, et al. Survey of commercial Rhodiola products revealed species diversity and potential safety issues. Sci Rep. 2015, 5:8337.
8 Xiang L, Song J Y, Xin T Y, et al. DNA barcoding the commercial Chinese caterpillar fungus. FEMS Microbiol Lett. 2013, 347(2): 156-162.
9 辛天怡,姚輝,羅焜,等.羌活藥材ITS/ITS2條形碼鑒定及其穩(wěn)定性與準確性研究.藥學(xué)學(xué)報. 2012, 47(8): 1098-1105.
10 Liao B S, Chen X C, Han J P, et al. Identification of commercial Ganoderma (Lingzhi) species by ITS2 sequences. Chin Med. 2015, 10:22-30.
11 國家藥典委員會.中華人民共和國藥典(一部). 北京:中國醫(yī)藥科技出版社, 2015: 200.
12 谷華,王梅,王志陽.苦木的現(xiàn)代藥理與臨床應(yīng)用. 中醫(yī)研究, 2001, 14(5): 55-56.
13 趙文娜.苦木的化學(xué)成分及藥理活性研究.西安:西北大學(xué)博士學(xué)位論文, 2013: 20-23.
14 胡小平.穴位注射苦木注射液治療小兒腹瀉80 例臨床觀察.醫(yī)學(xué)信息(中旬刊), 2011, 7: 3099-3100.
15 陳士林,姚輝,宋經(jīng)元,等.基于DNA barcoding(條形碼)技術(shù)的中藥材鑒定.世界科學(xué)技術(shù)-中醫(yī)藥現(xiàn)代化, 2007, 9(3): 7-12.
16 于俊林,趙莎,任明波,等.基于ITS2條形碼鑒定柴胡與大葉柴胡.中國中藥雜志, 2014, 39(12): 2160-2163.
17 陳士林,姚輝,韓建萍,等.中藥材DNA條形碼分子鑒定指導(dǎo)原則.中國中藥雜志, 2013, 38(2): 141-148.
18 朱英杰,陳士林,姚輝,等.重樓屬藥用植物DNA 條形碼鑒定研究.藥學(xué)學(xué)報, 2010, 45(3): 376-382.
19 李西文, 凱撒·蘇來曼,等. 伊犁貝母和川貝母的trnH-psbA序列鑒定研究.全國植物化學(xué)控制與化感作用交流研討會. 2010: 102-104.
20 Keller A, Schleicher T, Schultz J, et al. 5.8S-28S rRNA interaction and HMM-based ITS2 annotation. Gene, 2009, 430(1-2): 50-57.
21 段中崗,黃瓊林,楊錦芬,等.適合中藥材DNA條形碼分析的DNA提取方法的研究.中藥新藥與臨床藥理, 2009, 20(5): 480-484.
22 丁洋. 中藥材鑒定邁入基因時代. 中國中醫(yī)藥報, 2014年12月22 日(第002版).
23 焦明超,趙大顯,歐陽珊,等. DNA 條形碼技術(shù)在生物分類學(xué)中的應(yīng)用前景.湖北農(nóng)業(yè)科學(xué), 2011, 50(5): 886-890.
24 郭艷麗,鞠愛霞,等. DNA 分子標記技術(shù)在中藥鑒定學(xué)中的應(yīng)用.黑龍江醫(yī)藥. 2012, 25(4): 545-547.
25 陳士林,郭寶林,張貴君,等.中藥鑒定學(xué)新技術(shù)新方法研究進展.中國中藥雜志, 2012, 37(8): 1043-1055.
26 Wu L, Sun W, Wang B, et al. An integrated system for identifying the hidden assassins in traditional medicines containing aristolochic acids. Sci Rep, 2015, 5: 11318-11327.
27 馬孝熙,任偉超,孫偉,等.遠志藥材及其混偽品的DNA 條形碼鑒定.世界科學(xué)技術(shù)-中醫(yī)藥現(xiàn)代化, 2014, 16(8): 1719-1723.
28 凃媛,熊超,師玉華,等.細小種子類毒性藥材天仙子的DNA 條形碼鑒定.世界科學(xué)技術(shù)-中醫(yī)藥現(xiàn)代化, 2014, 16(11): 2337-2342.
29 樊佳佳,張婉冰,向麗,等.探索psbA-trnH序列對竹茹、天竺黃及其近緣物種的鑒定.世界科學(xué)技術(shù)-中醫(yī)藥現(xiàn)代化, 2014, 16(11):2349-2354.
30 熊超,周紅,賀海波,等.基于ITS2 序列鑒定谷物芽類藥材及其混偽品.世界科學(xué)技術(shù)-中醫(yī)藥現(xiàn)代化, 2014, 16(11): 2343-2348.
31 張雅琴,石鉞,宋明,等.蕨類藥材石韋及其混偽品的psbA-trnH序列鑒定.中國中藥雜志, 2014, 39(12): 2222-2226.
32 羅焜,馬培,姚輝,等. 中藥DNA條形碼鑒定中的DNA提取方法研究.世界科學(xué)技術(shù)-中醫(yī)藥現(xiàn)代化, 2012, 14(2): 1433-1439.
33 Bi I V, Harvengt L, Chandelier A, et al. Improved RAPD amplification of recalcitrant plant DNA by the use of activated charcoal during DNA extraction. Plant Breeding, 1996, 115: 205-206.
34 崔占虎,龍平,王穎莉,等. DNA分子標記技術(shù)在中成藥鑒定中的應(yīng)用與展望.中藥材, 2015, 38(1): 188-192.
DNA Barcoding for Identification of Picrasma quassioides and Its Adulterants on the Market
Chen Ying1, Zhang Jingjing2, Tang Huan3, Wang Jun4, Zhao Lilong2, Sun Wei1
(1. Institute of Chinese Materia Medica, China Academy of Chinese Medical Sciences, Key Laboratory of Beijing for Identification and Safety Evaluation of Chinese Medicine, Beijing 100700, China; 2. Zhang Zhong-jing Traditional Chinese Medicine College, Nanyang Institute of Technology, Nanyang 473000, China; 3. College of Forestry, Sichuan Agricultural University, Chengdu 611130, China; 4. School of Chemical Engineering and Life Sciences, Wuhan University of Technology, Wuhan 430070, China)
DNA barcoding technology was employed for identifying commercial products called Picrasma quassioides on the market. This study provided a guarantee for the market supervision and drug safety.Genomic DNA (2 repeats for each sample) from 15 P. quassioides samples was extracted. The ITS2 sequences were amplified and bidirectionally sequenced. All sequences were assembled and obtained using the CodonCode Aligner version 6.0.2. All sequences were compared with sequences available in the GenBank and TCMBARCODE database. The highest similarity species were selected and searched on the website of Flora Reipublicae Popularis Sinicae. In addition, the psbA-trnH sequences were amplified to verify the reliability of the results. The results showed that P. quassioides (70%), Ailanthus altissima (6.67%), Baphicacanthus cusia (13.33%), Tetradium glabrifolium (3.33%) and Maclura tricuspidata (6.67%) were found in the commercial samples. It was concluded that there were some adulterants of P. quassioides on the market. DNA barcoding technology can effectively monitor the medicines from its adulterants.
Picrasma quassioides, DNA barcoding, ITS2 sequence, psbA-trnH sequence
10.11842/wst.2016.01.008
R931.5
A
(責任編輯:馬雅靜 張志華,責任譯審:王 晶)
2015-06-16
修回日期:2015-12-07
* 科學(xué)技術(shù)部國家重大新藥創(chuàng)制項目(2013ZX09301307):基于中醫(yī)臨床轉(zhuǎn)化的中藥創(chuàng)新品種研發(fā),負責人:陳士林。
** 通訊作者:孫偉,男,博士,助理研究員,主要研究方向:中藥資源與中藥分子鑒定。

