腸膜明串珠菌及其近緣種dnaA和rpoB基因的系統發育分析
(內蒙古農業大學乳品生物技術與工程教育部重點實驗室,呼和浩特010018)
腸膜明串珠菌及其近緣種dnaA和rpoB基因的系統發育分析
馮淑貞,徐海燕,宋宇琴,張和平,孫志宏
(內蒙古農業大學乳品生物技術與工程教育部重點實驗室,呼和浩特010018)
比較16S rRNA基因、dnaA、rpoB部分基因序列對Leuconostoc mesenteroides(Leu.mesenteroides)及其近緣種的分辨力,為腸膜明串珠菌提供快速準確的鑒定方法。以分離自自然發酵乳制品中的8株Leu.mesenteroides為研究對象,以其看家基因dnaA、rpoB部分基因序列作為分子標記,結合GeneBank數據庫中的近緣種及亞種的相應序列構建系統發育樹,并與16S rRNA基因的系統發育樹進行比較。研究發現,基于Leu.mesenteroides及其近緣種的dnaA和rpoB部分基因序列構建的系統發育樹的拓撲結構與16S rRNA基因基本一致,但分辨率高于16S rRNA基因;種間的dnaA和rpoB部分基因序列相似性分別為81.7~84.8%和85.5、87.4%,而種間16S rRNA基因相似性為98.1%~99.5%。相較于16S rRNA基因,dnaA和rpoB基因更容易鑒定腸膜明串珠菌及其近緣種,其中rpoB可明晰區分試驗菌株與腸膜明串珠菌亞種間的系統發育關系,因此rpoB基因可作為16S rRNA基因的輔助工具用于Leu.mesenteroides及其近緣種的快速鑒定。
腸膜明串珠菌;系統發育分析;16S rRNA基因;dnaA;rpoB
0 引 言
腸膜明串珠菌為革蘭氏陽性兼性厭氧且不產芽孢的球形細菌,常存在于植物體表[2],目前包括腸膜明串珠菌葡聚糖亞種、乳脂亞種、腸膜亞種和最新分離得到的Leu.mesenteroides subsp.suionicum[6-7],它們均在食品工業中扮演重要角色[3],然而傳統的生理生化反應以及16S rRNA基因序列因自身局限不足以對近緣種或亞種進行分類鑒定[2,9-10],因此,有必要為其在工業上的應用提供快速準確的分類鑒定方法,看家基因作為重要蛋白的編碼基因,進化速率快且分辨能力強,特別適用于近緣菌株的區別和鑒定[12]。
本研究利用16S rRNA基因序列結合dnaA和rpoB的部分看家基因序列分析了分離自發酵乳制品中的8株試驗菌株的系統發育關系,充實多相分類鑒定以便分類學地位準確無誤,為其在工業上的應用提供快速準確的鑒定方法。
1 實 驗
1.1 材料
(1)實驗菌株。本文所用的8株腸膜明串珠菌均由內蒙古農業大學乳品生物技術與工程教育部重點實驗室提供,試驗菌株的16S rRNA基因、dnaA和rpoB基因序列登錄號見對應系統發育樹圖括號內所示。
(2)主要試劑和儀器。Applied biosystems PCR儀,ND-1000型微量紫外分光光計,CDS8000型UPV凝膠成像分析系統,DHP-9272型電熱恒溫培養箱,HZS-H水浴震蕩器,PL303/01電子天平,Eppendorf 5810R高速冷凍離心機,ML-30L型全自動高壓蒸汽滅菌器,DYY-12電泳儀等。
(3)實驗所用引物。通過比較基因組學分析,選擇dnaA和rpoB兩個單拷貝且含有多變區的基因為目標序列,結合序列比對信息,采用Prim er 5.0設計通用引物并由上海美吉生物醫藥科技有限公司合成,引物信息如表1所示。
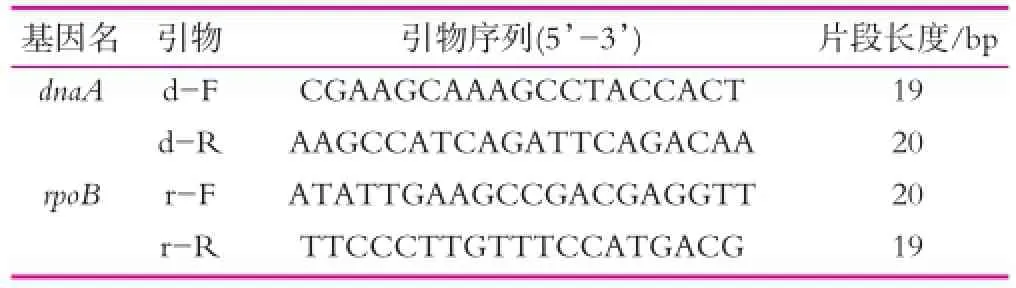
表1 PCR擴增引物
1.2 DNA提取、PCR擴增和測序
菌體DNA提取采用CTAB-液氮凍融法[13],在此基礎上針對本實驗樣品改進后具體步驟為:取適量菌體于1.5m L的EP管中,加入500μL TE緩沖液并用吸管反復吹打,使之重懸,置于液氮中反復凍融3-4次,然后加入80μL質量濃度為100 g/L的SDS和10μL質量濃度為20 g/L的蛋白酶K,混勻,于37℃搖床4 h。再加入80μL濃度為10 m o l/L的CTAB溶液及100μL濃度為5mo l/L的NaC l溶液,混勻,于65℃水浴30m in,得到粗提取液。在此粗提取液中,加入700μL酚/氯仿/異戊醇(V酚∶V氯仿∶V異戊醇=25∶24∶1),顛倒混勻,離心(12 000 g,10 m in);取上清,并加入700μL氯仿/異戊醇(V氯仿∶V異戊醇=24∶1),顛倒混勻,離心(12 000 g,10 m in),取上清,加入500μL冷的異丙醇和50μL 3 m ol/L的N aAc,輕輕混合,直到DNA沉淀下來,離心(12 000 g,10 m in)后,棄去上清,得到DNA沉淀,用500μL 70%的乙醇洗滌DNA兩次。最后用50μL TE溶解DNA,并加入適量R NaseA,4℃放置過夜后,將提取的基因組DNA稀釋到100μg/L作為PCR擴增模板,反應體系50μL。其中dnaA基因的擴增條件為:95℃預變性5 m in;95℃變性1 m in,52℃退火45 s,72℃延伸1 m in,30個循環;72℃末端延伸10 m in。rpoB基因擴增條件的除了退火溫度為56℃,其余反應條件均與dnaA一致。PCR擴增產物的質量用1.2%的瓊脂糖凝膠電泳檢測,并將擴增成功的PCR產物送上海美吉生物醫藥科技有限公司測序。
1.3 序列分析與系統發育樹的構建
8株試驗菌株的dnaA和rpoB基因擴增產物經純化、測序獲得相應的核苷酸序列。然后從GenBank數據庫中下載試驗菌株、參考菌株的16S rRNA基因序列,以及參考菌株的dnaA和rpoB基因序列(序列登錄號見對應系統發育樹圖括號內所示),采用M ega6.0軟件進行C lustalW比對,運用鄰接法(Neighour-joining,N-J)分別構建試驗菌株、參考菌株的16S rRNA基因和dnaA、rpoB部分基因序列的系統發育樹,數據自展重抽樣次數1 000次。
2 結果和分析
2.1 基于16S rRNA基因的系統發育分析
8株試驗菌株和6株參考菌株的16S rRNA基因序列下載自GenBank核酸序列數據庫。通過多重序列比對,獲得比對整齊的序列文件并構建系統發育樹。結果顯示,共劃分出A、B、C 3個較為穩定的大類群。其中,8株試驗菌株和Leu.mesenteroides subsp.mesenteroides ATCC 8293T、Leu.mesenteroides subsp.cremoris ATCC 19254T聚為A類群,它們兩兩之間的16S rRNA基因序列的平均相似性等于或大于99%;A類群與B類群Leu.pseudomesenteroides分支的平均相似性為99.5%,與C類群leu.citreum的平均相似性為98.1%,這與Saw a[14]、Elizaqunvel[15]、Laguerre[16]等人的研究結論一樣,即腸膜明串珠菌與其近緣種的16S rRNA基因序列相似性高且進化速率較低,因此當對腸膜明串珠菌亞種及其近緣種之間進行區別時,需要選用一種較16S rRNA基因分辨率高的方法及工具。

圖1 8株試驗菌株與參考菌株的16S rRNA基因系統發育樹
2.2 dnaA和rpoB部分基因序列的系統發育分析
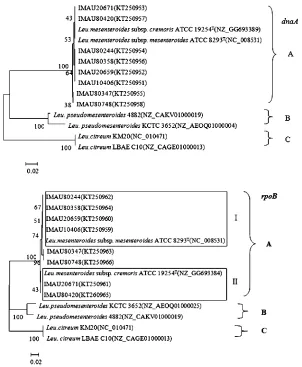
圖2 8株試驗菌株與參考菌株的dnaA、rpoB基因系統發育樹
基于dnaA部分基因序列構建的系統發育樹中(圖2),8株試驗菌株與6株參考菌株構建的系統發育樹的拓撲結構與16S rRNA基因的基本相同,仍形成A、B、C 3大類群,區別在于相似性不同,其中A類群與B類群兩兩菌株之間的基因平均相似性為84.8%,與C類群之間基因平均相似性為81.7%,這表明相較于16S rRNA基因序列,dnaA基因序列在鑒定明串珠菌屬內各近緣種的系統發育關系時分辨率更高。但遺憾的是,dnaA基因同源性不足以區分腸膜明串珠菌的亞種,模式菌株Leu.mesenteroides subsp.mesenteroides ATCC 8293T和Leu.mesenteroides subsp.cremoris ATCC 19254T的基因相似性為100%,可能的原因是由于用于本文構建系統發育樹的dnaA基因片段長度僅312 bp,未能提供足夠的序列信息造成。
基于rpoB部分基因序列構建的系統發育樹中(圖2),8株試驗菌株與6株參考菌株仍形成較為穩定的A、B、C 3大類群,其中A類群與B類群菌株兩兩之間基因的平均相似性為87.4%,與C類群之間基因平均相似性為85.5%,分辨率高于16S rRNA基因,類似地,Shevtsov等人也表明rpoB基因在分析乳桿菌屬內近緣種的親緣關系時較16S rRNA分辨率高[17]。另外在亞種水平上,試驗菌株(A類群)可明顯分為3個分支,其中IM AU 80244、IM AU 80358、IM AU 20659、IM AU 10406與模式菌株Leu.mesenteroides subsp.mesenteroides ATCC 8293T聚為Ⅰ亞群,IM AU 20671、IM AU 80420與模式菌株Leu.mesenteroides subsp.cremoris ATCC 19254T聚為II亞群。IM AU 80347與IM AU 80748未與參考菌株劃分為一類,它們與模式菌株Leu.mesenteroides subsp. mesenteroides ATCC 8293T的相似性為99.5%,與模式菌株Leu.mesenteroides subsp.cremoris ATCC 19254T的相似性為99.1%。
另對構建系統發育樹的8株試驗菌株以及參考菌株的dnaA、rpoB及16S rRNA的基因序列進行了多態性分析(表2),結果表明,2個看家基因的多態性位點比率均比16S rRNA基因高,dnaA基因的多態性位點比率最高,為27.1%,rpoB基因多態性位點比率為21.24%,遠高于16S rRNA基因的2.52%。遺傳距離可以反映同屬不同種之間基因差別的程度,結果顯示各看家基因的遺傳距離也均大于16SRNA。綜上所述看家基因相較于16S rRNA基因在區分腸膜明串珠菌近緣種及亞種時有著更高的分辨率,其中rpoB可明晰區分試驗菌株與腸膜明串珠菌亞種間的系統發育關系,因此rpoB基因可作為16S rRNA基因的輔助工具用于Leu.mesenteroides及其近緣種的快速分離鑒定。
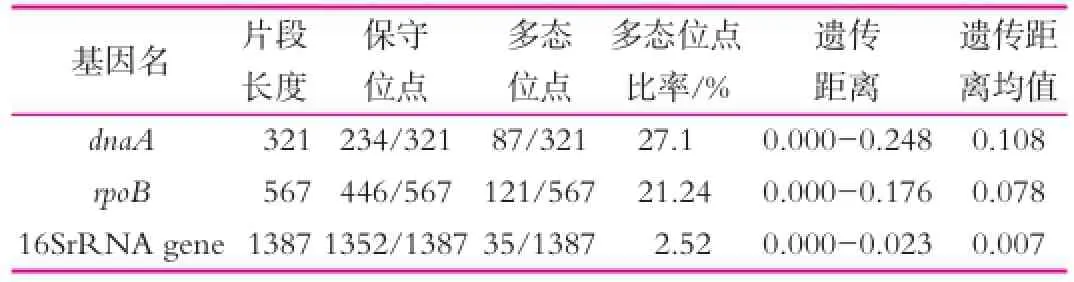
表2 腸膜明串珠菌16S rRNA基因及看家基因多態性分析
表2中,保守性位點、多態性位點數、多態位點比率為多態性位點與所有位點的比值由DNAsp5得出,各基因比對后片段長度、基因距離與基因距離平均值則由MEGA6.0計算得出。
3 討 論
細菌16S rRNA基因兼有保守區與可變區,使得它成為研究細菌分類進化的重要工具[18-19],然而隨著生物信息學的日益發展科研人員發現,16S rRNA基因在區分近緣種時往往顯示出較低的分辨率[9-10]。張文羿[9]等人利用看家基因clpx、recA將經傳統生理生化和16S rRNA基因序列鑒定的9株Enterococcus faecium和1株Enterococcus durans重新劃分為Enterococcus faecalis,成功將16S rRNA基因無法區分的菌株準確鑒定到種水平。2007年,Bruyne[20]等人對分離自埃塞俄比亞咖啡中的乳酸菌的16S rRNA基因序列進行了分析鑒定,結果表明這株菌與Leuconostoc cireum(Leu.cireum)以及Leuconostoc lactis(Leu.lactis)的16S rRNA基因序列相似性分別是99.6%和99.0%,故未能將其準確劃分到種水平,但通過聯合看家基因、DNA雜交等技術將分離得到的菌株劃分為新種Leuconostoc holzapfeliisp. nov.。類似地,呂嬙[10]等人對比研究了16S rRNA基因以及dnaA、murC和pyrG看家基因序列對Leu.cireum、Leu.lactis的區分能力,結果表明:看家基因相較于16S rRNA基因具有更高的分辨率,在研究近緣種的系統發育關系時更具有優勢。早前有研究指出腸膜明串珠菌與檸檬明串珠菌、假腸膜明串珠菌等近緣種的16S rRNA基因序列相似性大于97%[14-16],因此僅利用16S rRNA基因序列無法對其進行準確區分,本文構建的系統發育樹也證明了這一結論,因此本文研究腸膜明串珠菌及其近緣種的親緣關系選用了分辨率更高的看家基因序列作為輔助工具,這樣一來,克服了16S rRNA基因的局限性。
本研究通過比較16S rRNA基因,dnaA和rpoB基因的構建的N-J樹發現,看家基因與16S rRNA基因表現出了較高的一致性,表明本文所選的看家基因可以作為16S rRNA基因的輔助工具用于今后明串珠菌屬內近緣種的快速準確鑒定,另外利用rpoB基因構建的系統發育樹在亞種水平的區分能力高于dnaA,但遺憾的是,亞種之間的差異性小于1%,因此需要進一步聯合分辨率高的看家基因或通過傳統生理生化鑒定與分辨率高的看家基因相結合來區分腸膜明串珠菌亞種的系統進化地位。
[1]POGACIC T,CHUAT V,MADEC M N,et al.Phenotypic traits of genetically closely related Leuconostoc spp[J].International Dairy Journal,2014,39(1):96-101.
[2]HEMME D.Leuconostoc and its use in dairy technology[M].Florida:CRC Press Boca Raton,2012:73-108.
[3]李文斌,宋敏麗,高榮琨.腸膜明串珠菌的研究和應用進展[J].食品工程,2006(4):3-4.
[4]JUNG JY,LEE S H,LEEH J,et al.Effects of Leuconostoc mesenteroides starter cultures on microbial communities and metabolites during kimchi fermentation[J].International Journal of Food Microbiology,2012,153(3):378-387.
[5]KIM DC,CHAE HJ,IN M-J.Fermentation characteristics of Korean pear(Pyrus pyrifolia Nakai)puree by the Leuconostoc mesenteroides 51-3 strain isolated from Kim chi[J].African Journal of Biotechnology,2013,9(35):5735-5738.
[6]GARVIE EI.Leuconostoc mesenteroides subsp.cremoris(Knudsen and Sorensen)comb.nov.and Leuconostoc mesenteroides subsp.dextranicum(Beijerinck)comb.nov[J].International Journal of Systematic&Evolutionary Microbiology,1983,(1):118-119.
[7]GU CT,WANG F,LICY,et al.Leuconostoc mesenteroides subsp.suionicum subsp.nov[J].International Journal of Systematic and Evolutionary Microbiology,2012,62(7):1548-1551.
[8]羅靳,楊雅麟,王建華.腸膜明串珠菌右旋糖苷蔗糖酶的研究進展[J].微生物學通報,2007,34(4):787-790.
[9]張文羿,呂嬙,徐海燕,等.糞腸、屎腸球菌及相近種部分持家基因的系統發育分析[J].微生物學通報,2014,41(2):297-303.
[10]呂嬙,陳明,徐海燕,等.檸檬明串珠菌及相近種部分持家基因的系統發育分析[J].微生物學報,2013,53(7):669-676.
[11]BARRANGOU R,YOON SS,BREIDT F JR,et al.Identification and characterization of Leuconostoc fallax strains isolated from an industrial sauerkraut fermentation[J].Applied and Environmental Microbiology,2002,68(6):2877-2884.
[12]陳偉峰,洪舒音,趙秀玲.蛋白編碼基因在細菌分類學中的應用[J].生物技術通報,2012(5):42-48.
[13]烏日娜,張和平,孟和畢力格.酸馬奶中乳桿菌Lb.casei.Zhang和ZL12-1的16S rDNA基因序列及聚類分析[J].中國乳品工業,2005,33(6):4-9.
[14]SAWA N,OKAMURA K,ZENDO T,etal.Identification and characterization of novel multiple bacteriocins produced by Leuconostoc pseudomesenteroides QU 15[J].Journal of General and Applied Microbiology,2010,109(1):282-291.
[15]ELIZAQU IVEL P,CHENOLL E,AZNAR R.et al.A Taq-M an-based real-time PCR assay for the specific detection and quantification of Leuconostoc mesenteroides in meat products[J].FEMS microbiology Letters,2008,278(1):62-71.
[16]LAGUERRE S,AMARI M,VUILLEM IN M,et al.Genome sequences of three Leuconostoc citreum strains,LBAE C 10,LBAE C11,and LBAE E16,isolated from wheat sourdoughs[J].Journal of Bacteriology,2012,194(6):1610-1611.
[17]SHEVTSOV A,KUSHUGULOVA A,KOJAKHMETOV S,et al. Detection of Lactobacillus species using a gene fragment of the RNA polymerase beta subunit rpoB[J].Moscow University Biological Sciences Bulletin,2011,66(1):22-27.
[18]STACKEBRANDT E,FREDERIKSEN W,GARRITY GM,et al. Report of the ad hoc committee for the re-evaluation of the species definition in bacteriology[J].International Journal of Systematic and Evolutionary Microbiology,2002,52(3):1043-1047.
[19]楊玲玲,職曉陽,李文均.擬諾卡氏菌16S rRNA,gyrB,sod和rpoB基因的系統發育分析[J].微生物學報,2007,47(6):951-955.
[20]DE BRUYNE K,SCH ILLINGER U,CAROLINE L,et al.Leuconostoc holzapfelii sp.nov.,isolated from Ethiopian coffee fermentation and assessment of sequence analysis of housekeeping genes for delineation of Leuconostoc species[J].International Journal of Systematic and Evolutionary Microbiology,2007,57(12):2952-2959.
Phylogenetic analysis of closely related Leuconostoc mesenteroides species based on dnaA and rpoB gene homologues
FENG Shu-zhen,XU Hai-yan,SONG Yu-qing,ZHANG He-ping,SUN Zhi-hong
(Key Laboratory of Dairy Biotechnology and Engineering,Ministry of Education,Inner Mongolia Agricultural University,Hohhot 010018,China)
The study aimed to compare the efficiency of 16S rRNA,dnaA,rpoB gene on the phylogenetic relationship of Leu.mesenteroides and its related species,to provide a rapid identification method of the Leu.mesenteroides strains.Two housekeeping genes,dnaA and rpoB gene,of eight Leu.mesenteroides strains originally isolated from traditional dairy products were used as phylogenetic markers to construct the phylogenetic trees with the published corresponding gene sequences of reference strains in Genbank.The phylogenetic trees of 16S rRNA gene of tested strains and reference strains were also constructed.The topologies of the two housekeeping genes trees were consistent with that of 16S rRNA gene.How ever,homology of the close related Leu.mesenteroides species based on dnaA,rpoB and 16S rRNA gene sequences was different,ranging from 81.7%to 84.8%,85.5%to 87.4%,and 98.1%to 99.5%,respectively.Com pared with 16S rRNA gene,the dnaA and rpoB gene,were more appropriate for phylogenetic identification of Leu.mesenteroides and its related species.Meanwhile,considering the rpoB gene is competent to distinguish the test strains at the subspecies level,it can be used as an auxiliary tool for rapid classification and identification of Leu.mesenteroides and its related species.
Leu.mesenteroides;phylogenetic analysis;16SrRNA gene;dnaA;rpoB
Q93-33
:A
:1001-2230(2016)02-0008-04
2015-09-02
國家自然科學基金項目(31430066);國家高技術研究發展計劃(2011AA100901)。
馮淑貞(1990-),女,碩士研究生,研究方向為乳品生物技術。
孫志宏

