同源異型框蛋白Msx1的兩個新的磷酸化位點的鑒定
程慶靈 王敬強
(復旦大學 遺傳工程國家重點實驗室,上海 200438)
同源異型框蛋白Msx1的兩個新的磷酸化位點的鑒定
程慶靈 王敬強
(復旦大學 遺傳工程國家重點實驗室,上海 200438)
同源異型框蛋白Msx1的同系物Msx2中已鑒定出磷酸化修飾位點,并且發揮著重要的生物學功能。但是對Msx1的磷酸化修飾情況還不清楚,為了對Msx1的磷酸化修飾情況進行研究。利用生物信息學的方法對Msx1的磷酸化修飾位點進行預測,進一步利用免疫沉淀和質譜技術對其進行具體實驗驗證。C2C12細胞中的Msx1蛋白復合物經胰蛋白酶消化后,通過高效液相色譜和質譜技術進行分離和分析。結果發現兩個新的磷酸化修飾位點,分別是152位的絲氨酸和160位的絲氨酸,而且兩個磷酸化位點在人和小鼠中是高度保守的。進一步,制備針對這兩個磷酸化位點的特異性磷酸化抗體。磷酸化位點的鑒定以及特異性抗體的獲得為進一步研究Msx1的生物學功能提供了良好的條件。
同源異型框蛋白;Msx1;磷酸化修飾;質譜鑒定
蛋白質翻譯后修飾(post translation modification,PTM)是目前蛋白研究中比較受歡迎的一個重要研究方向,其中磷酸化修飾在生物體中是一種最普遍最重要的調控方式,幾乎調節著生命活動的整個過程,包括細胞的增殖、凋亡和分化、信號轉導、基因表達、細胞周期以及其他生命活動過程[1-3]。據統計,哺乳動物細胞內有1/3以上的蛋白質被磷酸化修飾[4]。真核生物中磷酸化主要發生在絲氨酸、蘇氨酸和酪氨酸等殘基,其比例大概為1 800∶200∶1[5]。大多數磷酸化的蛋白質有一個以上的磷酸化位點,并且生物體內磷酸化修飾是一個動態過程,同一種蛋白在不同的時間和空間時刻發生著磷酸化與去磷酸化的交替活動。因此,生物體內磷酸化修飾的蛋白質的鑒定和定量分析對更好的了解許多生命活動的發生發展有重要的意義[6]。
同源異型框基因廣泛存在于真核生物中,其結構中含有一個高度保守的同源異型盒,編碼一段含有60個氨基酸殘基的蛋白結構域,稱為同源異型結構域。作為一類重要的轉錄調控因子,在胚胎發育中對組織和器官的形成具有重要的調控作用。核磁共振及晶體學的研究表明,同源異型結構域可形成螺旋—轉折—螺旋結構,它可與DNA特異性結合。同源異型結構蛋白(homeodomain protein)作為轉錄因子通過與靶基因調控序列相結合來調控靶基因的轉錄。有些同源異型結構域蛋白之間具有協同作用,其不同組合方式可影響其活性[7]。
Msx1作為哺乳動物中典型的同源異型框基因在胚胎發育過程的多種組織和器官中表達,包括四肢、顱面的衍生物、神經管和乳腺等[8-11],對早期胚胎發育起到重要的調控作用。Msx1基因敲除小鼠顯示顱面發育缺陷,包括腭裂、牙齒畸形、顱骨和鼻骨發育缺陷[12],以及間腦發育缺陷[13]。Msx1基因的過量表達同樣會導致細胞分化的缺陷,Msx1轉基因小鼠出現乳腺增生[14]。Msx1基因突變則造成人類牙齒和骨骼等發育異常以及人類遺傳相關疾病(出生缺陷)如唇/腭裂、Witkop綜合癥以及缺牙癥[15]。發育過程中Msx1作為轉錄因子通過調控其靶標基因來介導上皮細胞和間質細胞之間的信號傳導,負向調控細胞分化[16-19]。Msx1只在未分化的前體細胞中表達,抑制前體細胞的終端分化[19,20]。前期研究表明,在肢體發育中Msx1的抑制性靶標基因主要分布在細胞核核周,而活化性靶標基因則均勻分布在細胞核內。在胚胎發育中Msx1和PRC2復合物(Ezh2復合物)在肢體(limb bud)和神經管(neural tube)高表達,但是Msx1只富集在肢體細胞核核周并招募PRC2復合物到細胞核核周[21],重新分布組蛋白標記物H3K27me3到細胞核核周,最終發揮轉錄抑制作用[21]。同時還發現,Msx1招募甲基化轉移酶G9a到其靶標基因并產生抑制性標記H3K9me2,抑制肌肉細胞分化[22]。經研究發現,Msx1和一系列轉錄因子如Ezh2、G9a、PIAS1等形成蛋白復合物,發揮生物學作用[19,21,22]。
Msx1的翻譯后修飾可能對Msx1在發育中的生物功能起著重要的調控作用。本研究以同源異性框蛋白Msx1的翻譯后修飾為切入點,利用生物信息學和生物質譜學對其進行分析,發現兩個新的磷酸化修飾位點,為進一步研究Msx1的生物學功能以及磷酸化修飾的動態調控在細胞分化過程中的作用奠定了良好的基礎。
1 材料與方法
1.1 材料
1.1.1 試劑 高保真聚合酶,T4連接酶,Xho I、BamH I限制性內切酶,dNTP等(TaKaRa),甲醇、無水乙醇、氯化鈉等生化試劑(國藥),10 cm培養皿、6孔細胞培養板購自上海WHB公司,DMEM培養基,胎牛血清(FBS)、胰酶/EDTA、Opti-MEM、Lipofectamine2000購自Life公司。序列級胰蛋白酶(trypsin)購于德國Boehringer Mannheim GmbH公司,乙腈為美國Fisher Scientific公司產品,三氟乙酸(TAF)為Merk公司產品。所用水為Mili-Q水。1.1.2 質粒和細胞系 pcDNA3-Flag-Msx1,pLERSIRES-GFP-Flag-Msx1[17,18],pcDNA3表達質粒用來做瞬時轉染,pLERS-IRES-GFP用來生產逆轉錄病毒。C2C12細胞和PhE細胞來源于ATCC。
1.1.3 引物 Msx1(S152A)-F:5'-TGG ATG CAG AGT CCC CGC TTC GCC CCG CCC CCA GCC AGA CGG CTG-3';Msx1(S152A)-R:5'-CAG CCG TCT GGC TGG GGG CGG GGC GAA GCG GGG ACT CTG CAT CCA-3';Msx1(S160A)-F:5'-CCG CCC CCA GCC AGA CGG CTG GCT CCC CCA GCA TGC ACC CTA CGC-3';Msx1(S160A)-R:5'-GCG TAG GGT GCA TGC TGG GGG AGC CAG CCG TCT GGC TGG GGG CGG-3'。
1.2 方法
1.2.1 突變體質粒的構建 以pcDNA3-Flag-Msx1質粒為模板,用Primer Premier 5軟件進行引物設計,PCR擴增F-Flag-Msx1(S152A)和R-Flag-Msx1(S152A) 片 段;F-Flag-Msx1(S160A) 和R-Flag-Msx1(S152A)片段。引物由蘇州金唯智公司合成,序列如上。PCR的參數設置為:98℃,預變性30 s;98℃變性10 s,58℃退火15 s,72℃延伸1 min,35個循環;72℃延伸5 min。膠回收F端PCR產物;同樣程序進行PCR,膠回收R端PCR產物;以F端和R端PCR產物為模板,SP6和T7為引物進行PCR,最后膠回收PCR產物,用BamH I和Xho I分別酶切PCR擴增產物和pcDNA3載體。回收DNA片段,并用T4連接酶16℃連接過夜。連接產物轉化大腸桿菌DH5α,在氨芐青霉素(終濃度50 μg/mL)的LB平板中培養過夜后,挑選菌落抽提質粒進行酶切和測序鑒定。
1.2.2 細胞培養 C2C12細胞在DMEM培養基(含10% FBS)中,37℃、5% CO2細胞培養箱中培養,細胞生長接近75%時,0.05%胰酶消化,吹散并加新鮮培養基傳代。
1.2.3 構建Msx1過表達的C2C12細胞 (1)按照15%-25%密度接種PhE 細胞于10 cm細胞培養板中,培養過夜。(2)將12 μg質粒DNA稀釋于0.8 mL Opti-MEM中,混勻,室溫放置5 min;另將30 μL Lipofectamine2000稀釋于0.8 mL Opti-MEM中,混勻,室溫放置5 min。(3)將Lipofectamine2000稀釋液滴入DNA 稀釋液中,混勻,室溫放置20-30 min。(4)吸去PhE 細胞的培養液,補入6.4 mL Opti-MEM。(5)將Lipofectamine2000與DNA 混合液輕柔混勻,緩慢逐滴加入PhE 細胞上,輕柔晃勻。(6)37℃,5% CO2細胞培養箱培養4-6 h,將轉染后細胞上清吸棄,1×PBS洗一遍,補入10 mL 新鮮DMEM(含10%胎牛血清)。(7)37℃,5% CO2培養24 h,檢測轉染效率。換成含有puromycin的DMEM進行篩選2 d。(8)第3天,吸去PhE 細胞的培養液,并用1×PBS洗一遍,補入10 mL 新鮮DMEM(含10%胎牛血清)。同時按照5%-10%細胞密度接種C2C12細胞于10 cm 細胞培養板中,培養過夜。(9)第4天,用熒光顯微鏡觀察細胞,熒光效率幾乎達到100%,將PhE中的病毒用0.45 μm的濾頭過濾到50 mL離心管中,加入2 mL新鮮DMEM,并加入 12 μL Polybrane(8 mg/mL),終濃度為8 μg/mL,混合均勻。吸掉C2C12細胞的培養基,將上述混合好的病毒液緩緩加入C2C12細胞中。(10)37℃,5% CO2培養24 h,觀察感染效率,并重復前一天操作過程。(11)37℃,5% CO2培養,等細胞長到90%左右收集細胞用于后期實驗。
1.2.4 免疫沉淀 用預冷的PBS洗滌細胞兩次,最后一次吸干PBS,加入適量含蛋白酶抑制劑的細胞裂解緩沖液(1×RIPA buffer,Protease inhibitor cocktail),冰上裂解30 min,細胞裂解液于4℃,最大轉速離心10 min后取上清;取少量裂解液以備Western blot分析。分別取2 mL protein A 瓊脂糖珠和2 mL Flag Bead(Sigma A-2220),用適量裂解緩沖液洗3 次,每次800×g離心3 min;將預處理過的40 μL protein A瓊脂糖珠(Sigma)加入到剩余裂解液中,4℃緩慢搖晃1 h,在4℃ 以800×g 速度離心3 min,將上清轉移到新的EP管中,加入60 μL預處理過的Flag Bead(Sigma A-2220),4℃緩慢搖晃孵育4 h。免疫沉淀反應后,在4℃ 以800×g 速度離心3 min,將瓊脂糖珠離心至管底,將上清小心吸去,瓊脂糖珠用1 mL裂解緩沖液洗3-4次;最后根據蛋白濃度加入相應體積的2×SDS 上樣緩沖液,沸水煮5 min;SDS-PAGE,Western blotting。
1.2.5 質譜 將位于33 kD處的蛋白染色條帶切下,在脫色緩沖液(25 mmol/L NH4HCO3,50%(V/V)乙腈)中脫色直至藍色消失將脫色后的凝膠條帶在2%(W/V)二硫蘇糖醇溶液中還原30 min,再加到2.5%(W/V)碘乙酰胺溶液中反應60 min,使游離的巰基全部烷基化將膠條用乙腈脫水后,加入新配制的測序級的胰蛋白酶的50 mmol/L NH4HCO3溶液,在37℃反應過夜次日加入適量10%甲酸終止反應,離心取上清液再用含有10%乙腈的25 mmol/L NH4HCO3溶液從膠條中反復抽提3次合并3次抽提液,并與初始的上清液混合,即得到蛋白的酶解多肽混合物,凍干后20℃保存。所有干燥的多肽樣品均溶解在緩沖液A(0.1%甲酸99%水+1%乙腈+0.1%甲酸)中,再經C18反相毛細管色譜柱(100 mm×0.17 mm)分離以0.1%甲酸和乙腈為流動相進行梯度洗脫,洗脫梯度為5%-40%緩沖液B(B:99%乙腈+1%水+0.1%甲酸),洗脫時間為180 min,流速為0.25 μL/min,經毛細管反相色譜洗脫出的多肽應用LTQ Orbitrap質譜分析,質核比檢測范圍為400-2 000 amu應用數據依賴方式進行二級質譜掃描,每個全掃描后進行5個二級掃描,碰撞能量為35%,動態排除時間為2 min。
1.2.6 磷酸化抗體的制備 將合成的單磷酸化多 肽1(C-QSPRFpSPPPAR-NH2)、 非 磷 酸 化多 肽1(C-QSPRFSPPPAR-NH2)、 單 磷 酸 化 多肽2(C-PARRLpSPPAC-NH2)、非磷酸化多肽2(C-PARRLSPPAC-NH2)分別注射兔子,并且分別使用單磷酸化多肽及非磷酸化多肽制備親和純化柱,對磷酸化特異性抗體進行純化,制備針對兩個位點的特異性抗體。
2 結果
2.1 Msx1磷酸化位點的生物信息學預測
鑒于同源異型框蛋白Msx1在生物體發生和發展中起著至關重要的作用,因此Msx1的翻譯后修飾尤其是磷酸化修飾必然能夠影響其功能的發揮。首先利用磷酸化位點預測軟件對Msx1的蛋白序列(圖1-A)進行預測分析,發現其有21個潛在的磷酸化位點,其中有15個絲氨酸,5個蘇氨酸和1個酪氨酸分別被磷酸化,如圖1-C所示。圖1-B 中顯示的兩個磷酸化位點處于Msx1的同源結構域中。對上述潛在的磷酸化位點進行序列分析,發現絲氨酸和蘇氨酸磷酸化位點前后6個氨基酸殘基是有區別的,但是都具有一定的保守性,該結果說明這兩種磷酸化位點的激酶識別位點是不同的。蛋白質翻譯后修飾大多數都是由酶介導催化,預測結果表明是由不同的激酶對這兩個位點磷酸化的發生進行了催化,分別是cdc2和ATM、PKA和IKK。

圖1 Msx1中磷酸化位點的預測
2.2 磷酸化位點的質譜鑒定
為了證實Msx1蛋白在小鼠C2C12細胞中的磷酸化狀態和預測結果是吻合的,首先在C2C12細胞中過表達含有Flag標簽的Msx1質粒,裂解C2C12細胞,用SDS-PAGE進行檢測,取其中一塊凝膠33 kD處IP膠塊用來做質譜鑒定,另一塊利用Western blot的方法和Flag抗體進行檢測,如圖2所示。
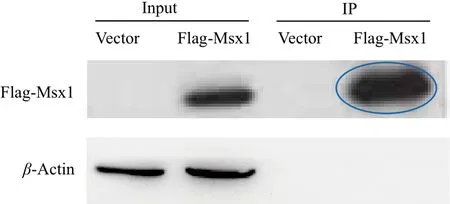
圖2 免疫共沉淀Msx1蛋白的SDS-PAGE檢測結果
質譜鑒定結果如圖3所示,圖3-A中紅色肽段為質譜鑒定出來的Msx1肽段,藍色圈里的是含有絲氨酸152位和絲氨酸160位的磷酸化肽段位置。質譜數據中有兩處荷質比變化明顯的峰,分別包含在肽段FpSPPPARR和RLpSPPACTLR中,如圖3-B和3-C的二級波譜圖所示,這兩處變化正是由于絲氨酸152和絲氨酸160位的磷酸基團引起的。
2.3 磷酸抗體的制備及其驗證
為了進一步證實這兩個磷酸化位點的存在,合成單磷酸化多肽1(C-QSPRFpSPPPAR-NH2)、非磷酸化多肽1(C-QSPRFSPPPAR-NH2)、單磷酸化多肽2(C-PARRLpSPPAC-NH2)、非磷酸化多肽2(C-PARRLSPPAC-NH2)分別注射兔子,制備針對兩個位點的特異性抗體,采用Western blot方法,利用特異性抗體檢測了第152位和第160位絲氨酸的磷酸化狀態,結果(圖4)同樣證實Msx1在這兩個位點是磷酸化的,并且這兩個位點突變成丙氨酸后則不能被磷酸化。
2.4 Msx1磷酸化位點的高度保守性
人和小鼠高覆蓋率的基因組已被測序,而且同源異型框基因還是一類高度保守的基因家族,為了證實這兩個磷酸化位點在二者之間是否有高度的保守性,利用NCBI將蛋白序列進行比對,結果(圖5)表明兩個磷酸化位點具有高度保守性。此外,利用生物信息學分析了人的蛋白序列,結果顯示這兩個位點也是磷酸化的。
3 討論
蛋白質磷酸化修飾是生物體內一類十分重要的翻譯后修飾,快速鑒定出蛋白磷酸化修飾位點成為研究磷酸化修飾的關鍵之處,本研究首先利用預測軟件對Msx1磷酸化位點進行預測,只需要直接輸入蛋白的氨基酸序列便可以進行磷酸化位點預測,方便快捷,避免繁瑣的實驗操作,為磷酸化位點的確定提供了一定的參考依據。但是預測軟件只是根據蛋白的一級結構進行預測,而生物體內蛋白質的結構復雜多樣,因此Msx1磷酸化的確定,還必須通過充分的實驗來驗證。本實驗過程中,除了Msx1,細胞裂解液中還含有其他蛋白成分,質譜分析對磷酸化肽段純度要求較高,然而免疫共沉淀能夠使蛋白富集,從而可以得到相對純化的Msx1,這有利于對Msx1的質譜鑒定。通過質譜對Msx1進行分析鑒定,發現蛋白中有潛在的磷酸化修飾肽段,蛋白質譜學的快速發展,能夠在短時間內對潛在肽段進行二級質譜分析,結果和軟件預測是吻合的。
生物體內蛋白磷酸化修飾是一個動態調控過程,不同生存環境都可能會影響蛋白的修飾狀態。磷酸化肽段非常不穩定,容易發生脫磷酸化,因此在實驗處理過程中除了加入磷酸酶抑制劑外還要注意操作環境的溫度。從免疫共沉淀技術到質譜鑒定,從專一的生物化學反應再到特異性驗證等一系列實驗,證實了Msx1的磷酸化修飾狀態,發現了絲氨酸152位和絲氨酸160位兩個新的磷酸化位點。文中的特異性抗體,利用152位和160位單磷酸化和非磷酸化的肽段注射兔子,并且分別使用單磷酸化多肽及非磷酸化多肽制備親和純化柱,對磷酸化特異性抗體進行純化,最后得到了特異性更高的抗體。特異性抗體的制備和驗證更是為后期實驗的研究提供重要依據,可以直接利用特異性抗體檢測蛋白中磷酸化位點,進一步研究生物體內磷酸化修飾的動態調控模式。Msx1磷酸化位點的實驗驗證,提供了現代生物學研究中又一個富有啟示性的案例。
本研究以同源異型框蛋白Msx1為代表研究蛋白磷酸化修飾,優勢在于Msx1基因片段較小,操作方便。運用生物信息學與質譜技術聯合的方法對蛋白翻譯后修飾進行預測和鑒定分析,方便快捷,可以通過預測軟件對其他同源異型框蛋白進行預測,進一步利用特異性抗體加以驗證,為下一步研究Msx1磷酸化修飾對其生物學功能的影響以及其他同源框蛋白的生物功能奠定了扎實的生物基礎。
本研究對人和小鼠的Msx1蛋白進行比對,二者的蛋白序列具有高度保守性,特異性抗體所用的肽段在人和小鼠中是相同的,因此可以直接用來檢測人的MSX1磷酸化狀態。但是不同物種中Msx1的磷酸化修飾是否完全相同,需要進一步驗證。蛋白的翻譯后修飾大部分是由激酶介導,不同的酶可催化同一個底物,同一個酶也可催化不同的底物。預測結果同時提供了不同的潛在磷酸激酶,后期實驗可以通過體外磷酸激酶分析驗證,通過對細胞內的激酶敲除,利用特異性的磷酸化抗體進行檢測。酶對底物的催化反應可以是間接也可以是直接結合進行催化,Msx1是否直接被酶催化,需要對Msx1的空間結構做進一步分析。
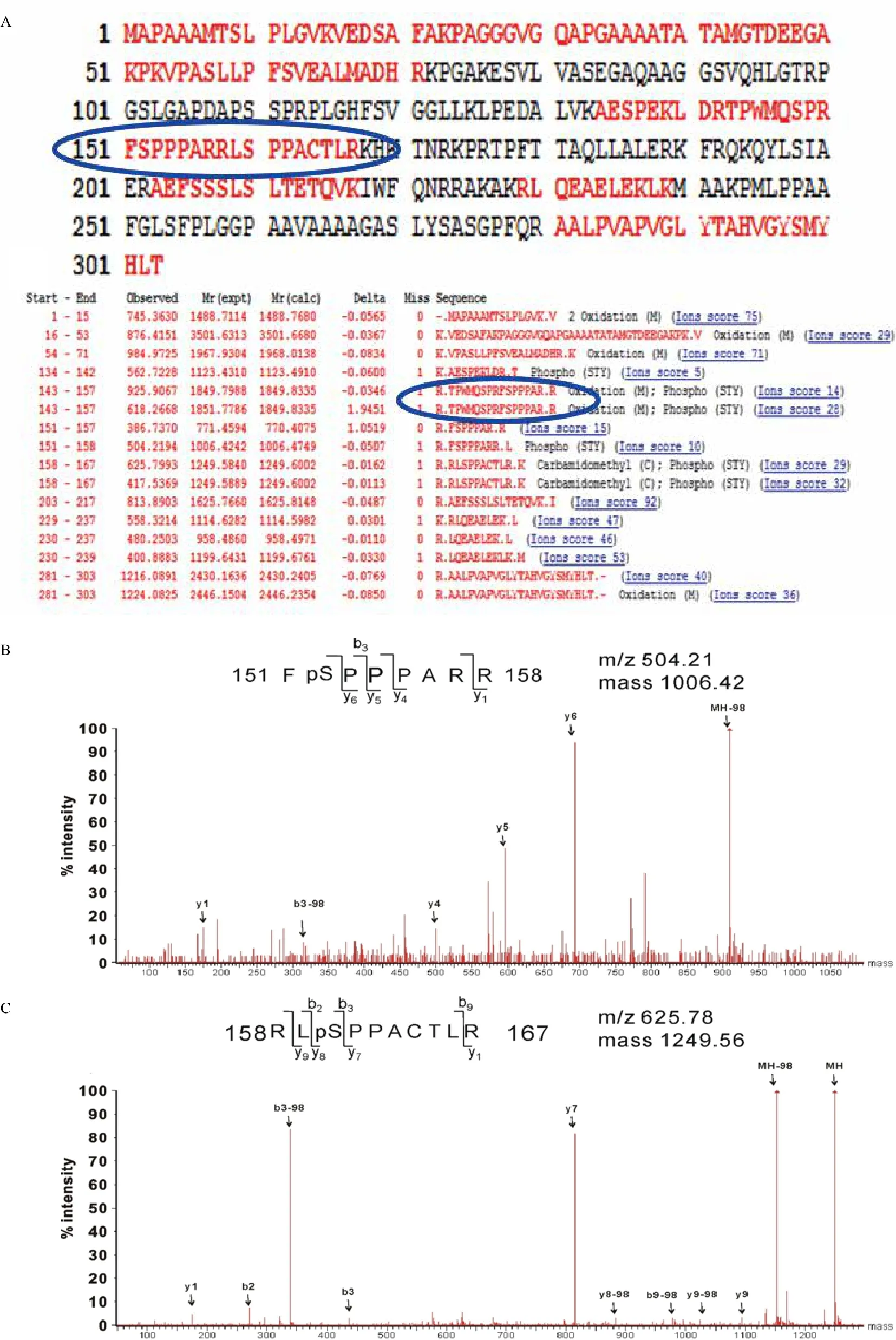
圖3 Msx1中磷酸化位點的質譜鑒定
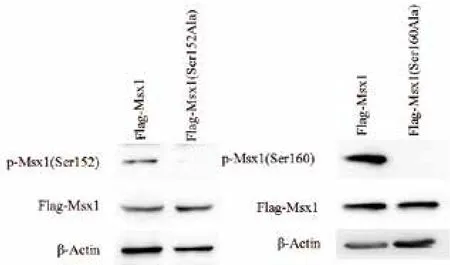
圖4 利用 Western blot 驗證特異性抗體的活性
4 結論
本研究首先運用生物信息學對同源異型框蛋白Msx1進行了預測,發現了21個潛在的磷酸化位點,進而在C2C12細胞中過量表達Msx1蛋白,并利用免疫共沉淀技術以及質譜分析技術對磷酸化位點進行鑒定,發現了兩個新的磷酸化位點,分別為絲氨酸152位和絲氨酸160位,預測結果和實驗結果具有一致性。這兩個位點的發現為研究Msx1磷酸化修飾以及其他翻譯后修飾提供了重要的參考。同源異型框基因又是一類高度保守的基因,實驗結果同時驗證了這兩個磷酸化位點在人和小鼠中都是高度保守的,從而利用生物信息學軟件對人的MSX1的磷酸化修飾位點進行預測,可以為研究MSX1在調控人類健康以及人類重大疾病相關的復雜生命過程中精確的分子作用提供參考依據。

圖5 人和小鼠的Msx1蛋白序列比對結果圖
[1]Thingholm TE, Jensen ON, Larsen MR. Analytical strategies for Phosphoproteomics[J]. Proteomics, 2009, 9(6):1451-1468.
[2]Duan JJ, Lozada AF, Gou CY, et al. Nicotine recruits glutamate receptors to postsynaptic sites[J]. Mol Cell Neurosci, 2015, 68:340-349.
[3]Linke D, Koudelka T, Becker A, Tholey A. Identification and relative quantification of phosphopeptides by a combination of multiprotease digestion and isobaric labeling[J]. Rapid Commun Mass Spectrom, 2015, 29(10):919-926.
[4]Blazek M, Santisteban TS, Zengerle R, et al. Analysis of fast protein phosphorylation kinetics in single cells on a microfluidic chip[J]. Lab Chip, 2014, 15(3):726-734.
[5]Luo R, Zhou C, Lin J, Yang D, et al. Identification of in vivo protein phosphorylation sites in human pathogen Schistosoma japonicum by a phosphoproteomic approach[J]. Proteomics, 2012, 75:868-877.
[6]Zhang B, Liu JY. Mass spectrometric identification of in vivo phosphorylation sites of differentially expressed proteins in elongating cotton fiber cells[J]. PLoS One, 2013, 8:e58758.
[7]Lelli KM, Noro B, Mann RS. Variable motif utilization in homeotic selector(Hox)-cofactor complex formation controls specificity[J]. Proc Natl Acad Sci USA, 2011, 108(52):21122-21127.
[8]Dai J, Mou Z, Shen S, et al. Bioinformatic analysis of Msx1 and Msx2 involved in craniofacial development[J]. J Craniofac Surg, 2014, 25(1):129-134.
[9]Zhang W, Qu HC, Zhang Y. Association of MSX1 and TGF-β1 genetic polymorphisms with hypodontia:meta-analysis[J]. Genet Mol Res, 2014, 13(4):10007-10016.
[10]Nassif A, Senussi I, Meary F, et al. Msx1 role in craniofacial bone morphogenesis[J]. Bone, 2014, 66:96-104.
[11]Reddy NA, Adusumilli G, Devanna R, et al. MSX1 gene variant -its presence in tooth absence - a case control genetic study[J]. J Int Oral Health, 2013, 5(5):20-26.
[12]Mundstock CA, Bortolini MC, Salzano FM, et al. MSX1 and PAX9 investigation in monozygotic twins with variable expression of tooth agenesis[J]. Twin Res Hum Genet, 2013, 16(6):1112-1116.
[13]Souza LT1, Kowalski TW, Collares MV, et al. MSX1 gene and nonsyndromic oral clefts in a Southern Brazilian population[J]. Braz J Med Biol Res, 2013, 46(7):555-558.
[14]Yilmaz A, Engeler R, Constantinescu S, et al. Ectopic expression of Msx2 in mammalian myotubes recapitulates aspects of amphibian muscle dedifferentiation[J]. Stem Cell Res, 2015, 15(3):542-553.
[15]Seo YJ, Park JW, Kim YH, et al. Associations between the risk of tooth agenesis and single-nucleotide polymorphisms of MSX1 and PAX9 genes in nonsyndromic cleft patients[J]. Angle Orthod, 2013, 83(6):1036-1042.
[16]Kim NY, Kim YH, Park JW, et al. Association between MSX1 SNPs and nonsyndromic cleft lip with or without cleft palate in the Korean population[J]. J Korean Med Sci, 2013, 28(4):522-526.
[17]Xie H, Cherrington BD, Meadows JD, et al. Msx1 homeodomain protein represses the αGSU and GnRH receptor genes during gonadotrope development[J]. Mol Endocrinol, 2013, 27(3):422-436.
[18]Rafighdoost H, Hashemi M, Narouei A, et al. Association between CDH1 and MSX1 gene polymorphisms and the risk of nonsyndromic cleft lip and/or cleft palate in a southeast Iranian population[J]. Cleft Palate Craniofac J, 2013, 50(5):e98-e104.
[19]Wang J, Abate-Shen C. Transcriptional repression by the Msx1 homeoprotein is associated with global redistribution of the H3K27me3 repressive mark to the nuclear periphery[J]. Nucleus, 2012, 3(2):155-161.
[20]Song YJ, Lee H. PIAS1 negatively regulates ubiquitination of Msx1 homeoprotein independent of its SUMO ligase activity[J]. Mol Cells, 2011, 32(3):221-226.
[21]Wang J, Kumar RM, Biggs VJ, et al. The Msx1 homeoprotein recruits Polycomb to the nuclear periphery during development[J]. Dev Cell, 2011, 21(3):575-588.
[22]Wang JQ, Abate-Shen C. The msx1 homeoprotein recruits G9a methyltransferase to repressed target genes in myoblast cells[J]. PLoS One, 2012, 7(5):e37647.
(責任編輯 李楠)
Identification of Two Novel Phosphorylated Sites of Homeoprotein Msx1
CHENG Qing-ling WANG Jing-qiang
(State Key Laboratory of Genetic Engineering,Fudan University,Shanghai 200438)
Some phosphorylated sites in the homolog Msx2 of the homeoprotein Msx1 has been identified,which plays a crucial biological role. However,the phosphorylated sites in Msx1 are not known yet,thus in order to examine the Msx1 phosphorylated status,we applied bioinformatics analysis to predict phosphorylation sites in Msx1,further used immunoprecipitation and mass spectrometry to experimentally verify the phosphorylation sites in Msx1. Tryptic digests of Msx1 protein complexes purified from C2C12 cells were separated and analyzed by nLC-MS-MS. Two novel phosphorylation sites(Ser152 and Ser160)were identified,moreover,these two sites were highly conserved in mouse and human. Furthermore,the specifically-phosphorylated antibody was prepared targeting these two phosphorylation sites.
homeoprotein;Msx1;phosphorylation;mass spectrometric identification
10.13560/j.cnki.biotech.bull.1985.2016.06.031
2015-10-27
國家自然科學基金資助項目(31471230),上海市浦江人才計劃資助(14PJ1401300)
程慶靈,女,碩士,研究方向:Msx1翻譯后修飾在發育中的分子作用;E-mail:13210700132@fudan.edu.cn
王敬強,男,博士,研究方向:Msx1在發育,出生缺陷,組織再生和癌癥中的分子作用;E-mail:Jingqiangwang@fudan.edu.cn

