骨髓間充質干細胞分化過程中表觀遺傳調控機制的研究進展
王連慶 翟俏麗 趙培慶 李濤
(淄博市中心醫院轉化醫學中心,淄博 255036)
骨髓間充質干細胞分化過程中表觀遺傳調控機制的研究進展
王連慶 翟俏麗 趙培慶 李濤
(淄博市中心醫院轉化醫學中心,淄博 255036)
骨髓間充質干細胞(bone marrow mesenchymal stem cells,BMMSCs)具有自我更新的能力和多向分化潛能,在體外可被誘導分化成多種細胞類型,在骨及軟骨組織修復中具有重要的臨床應用價值。為研發有效促進BMMSCs定向分化的藥物,將BMMSCs更合理安全地應用于臨床,有必要闡明表觀遺傳在BMMSCs分化過程中的調控機制。由于BMMSCs的異常分化可導致疾病的發生,其分化調控機制一直是研究的熱點。表觀遺傳調控,如DNA甲基化、組蛋白乙酰化、組蛋白甲基化及非編碼RNA等,對決定BMMSCs分化方向至關重要。闡明表觀遺傳在BMMSCs分化過程中的調控機制,將有助于研發有效促進BMMSCs定向分化的藥物,更合理安全地將BMMSCs應用于臨床。就BMMSCs分化中的主要表觀遺傳調控機制的最新研究進展做一綜述,并進行了總結。
骨髓間充質干細胞;細胞分化;表觀遺傳調控
骨髓間充質干細胞(bone marrow mesenchymal stem cells,BMMSCs)是骨髓造血微環境的重要組成部分,除具有支持與調控造血功能外,還具有成骨、成脂和軟骨形成等多向分化能力,是目前骨組織工程廣泛使用的主要細胞來源,可用于多種骨骼疾病的臨床治療,在其他組織器官損傷修復過程中也具有廣闊的應用前景[1,2]。BMMSCs的分化受多種因素調控,表觀遺傳作為BMMSCs分化的主要調控機制之一,在決定其分化方向上起到重要作用。
表觀遺傳是能夠調控基因表達但不改變相關基因序列的可遺傳的修飾。表觀遺傳調控BMMSCs分化相關基因的時空表達,在一定程度上代表BMMSCs的多能性。同時,表觀遺傳狀態的改變也能 影響BMMSCs的分化方向及能力[3]。常見的表觀調控主要包括DNA甲基化、組蛋白乙酰化、組蛋白甲基化和非編碼RNA調控等,本文擬從以上幾方面介紹近年來BMMSCs分化過程中表觀遺傳調控的相關研究進展。
1 組蛋白修飾
1.1 組蛋白乙酰化修飾
組蛋白乙酰化修飾是在組蛋白N端賴氨酸殘基上添加乙酰基團,是最常見的表觀遺傳修飾之一。組蛋白乙酰化能夠促進染色質結構開放從而使基因轉錄活化,組蛋白去乙酰化則與染色質轉錄抑制相關。組蛋白乙酰化水平主要受組蛋白乙酰基轉移酶(histone acetylase,HAT)和組蛋白去乙酰化酶(histone deacetylase,HDAC)的調控[4]。
相關調控基因的組蛋白乙酰化程度可反映BMMSCs的干性維持及分化狀態,H3K9和H3K14的乙酰化(H3K9ac,H3K14ac)是基因活化的標志。在BMMSCs成骨分化過程 中,成骨相關基因RUNX2和ALP的表達逐步上調,而干細胞自我更新相關的干性因子Oct4和Sox2表達顯著下降,它們的表達變化與H3K9ac和H3K14ac密切相關[5]。
根據HDAC結構域的同源性可將其分為4類。在現有研究中,第I類的HDAC1、HDAC8及第III類的SIRT1在BMMSCs的分化選擇中發揮了重要的作用。在心肌微環境下,BMMSCs可以分化形成心肌細胞,在該過程中,HDAC1的表達顯著下降,同時,敲低HDAC1能夠促進BMMSCs直接分化為心肌細胞[6]。HDAC8通過抑制H3K9的乙酰化及RUNX2的活性,使大鼠BMMSCs向成骨分化的程度降低[7]。SIRT1則可直接調控干性因子Sox2來維持BMMSCs的自我更新和多能性,其活性的降低使Sox2表達下調,從而導致BMMSCs自我更新能力和分化能力的退化,活化的SIRT1則以劑量依賴的方式促進BMMSCs克隆形成能力和成骨成脂分化能力[8]。同樣,SIRT1可通過去乙酰化β-catenin使其在核內累積進而調控BMMSCs分化相關基因的轉錄[9]。此外,SIRT1可通過活化Sox9和NF-κB的去乙酰化,促進BMMSCs的軟骨分化過程[10](圖1 右)。
組蛋白去乙酰化酶抑制劑對BMMSCs的分化有很強的影響。用組蛋白去乙酰化酶抑制劑丙戊酸(VPA)和丁酸鈉(NaBu)處理BMMSCs可增加組蛋白H3和H4乙酰化水平,顯著促進肝臟特異基因的表達,這提示去乙酰化酶抑制劑促進BMMSCs向肝臟方向的分化[11]。同時,NaBu可抑制大鼠BMMSCs中HDAC2的表達及其在平滑肌特異基因上的募集,進一步誘導高水平的H3K9ac和H4ac,促進平滑肌特異基因的表達,誘導BMMSCs向平滑肌方向分化[12]。另外一種組蛋白去乙酰化酶抑制劑TSA,可顯著抑制干性因子Oct4、Sox2及Nanog的下調,以穩定BMMSCs中多能性基因的表達[13]。其他研究發現,TSA處理增加了組蛋白H3的乙酰化水平并抑制了BMMSCs成脂分化[14](圖1左)。
1.2 組蛋白甲基化修飾
組蛋白甲基化是另一種常見的組蛋白翻譯后修飾。組蛋白可以發生單甲基化、雙甲基化或三甲基化。甲基化的增加通常能夠促進組蛋白與DNA的親和力,增加轉錄抑制的程度,例如H3K9me和H3K27me。然而也有一些重要的例外,如H3K4的甲基化常與活化的染色質相關,而H3K9三甲基化依賴于不同的基因對染色質狀態有不同的調控作用。組蛋白甲基化受組蛋白甲基轉移酶(HMT)和組蛋白去甲基化酶(HDM)共同調控[4]。
研究發現,組蛋白甲基化修飾酶能夠調控BMMSCs的分化能力。甲基轉移酶EZH2,可以三甲基化染色質H3K27,而這種抑制性的表觀遺傳標記能夠被去甲基化酶KDM6A去掉。Hemming小組[15]發現,EZH2的高表達能夠促進BMMSCs的成脂分化,抑制成骨分化,KDM6A則有相反的作用,兩者通過影響關鍵調控基因啟動子區H3K27me3的水平來調控BMMSCs分化的特異性。精氨酸甲基轉移酶CARM1,可通過組蛋白甲基化介導的染色質重塑調控基因表達,該因子進入細胞核后,可對H3R17進行甲基化,提高BMMSCs的體外成脂分化、成骨分化及成肌分化的能力[16]。
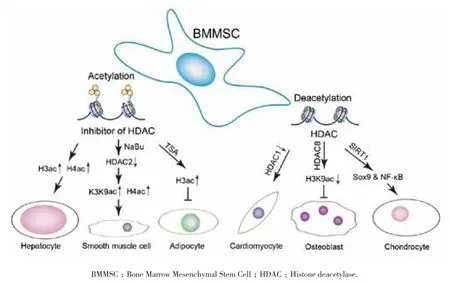
圖1 乙酰化修飾對BMMSC分化的影響
胚胎干細胞中的關鍵基因位點同時包含活化的組蛋白修飾標記(如H3K4me3)和抑制性的組蛋白修飾標記(如H3K27me3),這兩種修飾使基因處于一種抑制性但可以被迅速活化的狀態[3]。這些位點上表觀遺傳修飾的狀態可決定相關基因的最終命運。在未分化的BMMSCs中,c-Myc和cyclin D1(Wnt/βcatenin信號通路的靶點)基因上與β-catenin相結合的啟動子區同時具有抑制狀態及活化狀態的染色質標記,在BMMSCs向成骨分化過程中,啟動子區含有活化的染色質標記H3K4me,而抑制性標記H3K27me3缺失,與之相反,在BMMSCs成脂分化過程中,啟動子區則僅有抑制性的H3K27me標記[17]。
2 DNA甲基化
DNA甲基化是DNA化學修飾的一,在不改變DNA序列的前提下,改變遺傳表現。哺乳動物的DNA甲基化主要是在C-G二核苷酸(CpG)上共價添加甲基基團,具有靶向特異性,是最重要的沉默性表觀遺傳修飾之一。目前已發現的所有CpG島的DNA甲基化幾乎都沉默了相關基因,DNA甲基化抑制劑5-Aza可使基因表達很大程度上得以恢復。DNA甲基轉移酶蛋白家族(DNMTs)與可能的DNA去甲基化酶協同調控DNA甲基化水平及相關基因的表達[4]。
DNA甲基化狀態的改變關系到細胞分化命運的轉換。5-Aza導致的整體甲基化水平的改變抑制了BMMSCs的增殖和成脂分化[14],促進其成骨分化、向神經元樣細胞、心肌細胞或骨骼肌細胞的分化[18]。應用靶向DNA甲基化技術將BMMSCs中Trip10甲基化,其分化潛能受到限制,促進了BMMSCs向神經及成骨方向分化,阻斷了BMMSCs的成脂分化,說明Trip10的甲基化對BMMSCs分化方向的誘導具有特異性[19]。腫瘤抑制基因HIC1 和RassF1A的靶向甲基化促使BMMSCs轉化成腫瘤干細胞樣細胞,轉化的BMMSCs仍然能分化成不同的細胞類型,包括成骨細胞、神經細胞和脂肪細胞,同時,HIC1 和RassF1A定向甲基化后,少量轉化的BMMSCs使免疫缺陷的小鼠快速產生腫瘤,這也提示DNA的異常甲基化可導致腫瘤發生[20]。
3 非編碼RNA
3.1 長鏈非編碼RNA
長 鏈 非 編 碼 RNA(long noncoding RNAs,lncRNAs)是一類轉錄長度大于200 bp的非編碼RNA,能夠在轉錄及轉錄后水平調控基因表達,是表觀遺傳調控研究的另一個熱點。人類的lncRNA有大約30 000條不同的轉錄本,是非編碼轉錄組的主要組成成分,絕大部分lncRNA的功能尚不清楚。近年來,一些研究證實lncRNA在BMMSCs分化調控中也發揮了重要作用。Wang等[21]報道,在BMMSCs向軟骨方向分化過程中,lncRNA ZBED3-AS1 和 CTA-941F9.9表達顯著上調,并在分化28 d后仍呈高水平表達,提示它們可能參與了BMMSCs的軟骨分化過程。在BMMSCs成骨分化過程中,lncRNA-ANCR(anti-differentiation ncRNA)表達顯著降低,lncRNA-ANCR通過與EZH2結合抑制Runx2的表達,進而抑制BMMSCs的成骨分化,抑制內源lncRNA-ANCR的表達則促進了成骨分化[22]。此外,有研究報道lncRNA H19和uc022axw.1的上調表達可能也參與了BMMSCs的成骨分化[23]。這些研究結果表明lncRNA也是BMMSCs分化的一種重要調控因子,為骨相關疾病的治療提供了新的靶點。
3.2 微小RNA
微小RNA(microRNA)是一類長度為21-25 nt的內源性非編碼單鏈RNA,可通過堿基互補配對的方式與靶mRNA特異結合,剪切靶基因的轉錄產物或抑制其翻譯。miRNA也參與了基因的表觀遺傳調控,在BMMSCs的干性維持和多分化方向的過程中發揮了重要作用。miRNA-133a能夠促進BMMSCs向心肌細胞的分化[24]。而miRNA-124通過與STAT3的3'-UTR的靶向結合,抑制STAT3的翻譯,使BMMSCs向心肌細胞分化的能力減弱[25]。此外,miRNA-9與β-巰基乙醇協同誘導BMMSCs向神經方向分化[26]。miRN A-29a/b及miRNA-449a可抑制BMMSCs向軟骨方向的分化[27]。miRNA-155,miRNA-221/222則在BMMSCs的成脂分化過程中發揮了重要的負調控作用[28]。

圖2 PPARγ在BMMSC成骨分化末期調控C/EBPα表達的模式圖
4 表觀遺傳修飾的網絡調節
在生物體復雜精細的內環境下,表觀遺傳調控往往也不是以單一的方式發揮作用的,不同的組蛋白修飾之間可以相互影響,協同發揮作用,組蛋白修飾也可以與DNA甲基化相互偶聯,從而產生復雜的表觀遺傳效應。表觀遺傳修飾的網絡調節模式也參與了BMMSCs分化的精細調控。
BMMSCs的成骨分化和成脂分化之間的平衡受C/EBPα啟動子區的DNA甲基化和組蛋白乙酰化的協同調控,在成骨分化的末期,C/EBPα啟動子區域的高甲基化阻止了PPARγ的結合,HDAC1進一步結合到該區域,降低組蛋白乙酰化水平,PPARγ在C/EBPα的啟動子區建立了DNA甲基化和組蛋白乙酰化的橋梁[29]。組蛋白修飾因子YY1與轉錄共激活因子p300可通過對組蛋白乙酰化水平的調控改變BMMSCs中軟骨特異基因ChM-I的表達,在啟動子區低甲基化的細胞中抑制YY1并增加p300和基本轉錄因子Specificity protein3(Sp3)的表達能夠維持ChM-I的表達,但在高甲基化的細胞中并沒有這種作用,說明在BMMSCs軟骨分化過程中,ChM-I受組蛋白去乙酰化和組蛋白甲基化的協同負調控[30]。BMMSCs在成骨分化過程中RUNX2表達上調,同時轉錄活化相關的H3K9ac和H3K4me3修飾水平及在RUNX2啟動子區的募集均有升高,而與轉錄抑制相關的H3K9me3修飾水平及在RUNX2啟動子區的募集降低,RUNX2啟動子區DNA甲基化修飾程度降低[31]。這些研究結果說明,不同的表觀遺傳修飾之間可以協同作用調控BMMSCs的分化過程。
5 骨髓間充質干細胞分化過程中的表觀遺傳組學研究
近年來,系統性高通量的研究方法比如染色質免疫共沉淀結合高通量測序ChIP-seq(chromatin immunoprecipitation with deep sequencing) 和 芯 片ChIP-on-chip(chromatin immunoprecipitation assay)技術的廣泛應用有助于從基因組整體水平研究表觀遺傳修飾的狀況,建立BMMSCs分化過程的表觀遺傳譜。
Herlofsen 等[32]應用ChIP-Seq研究了來源于4個捐贈者的hBMMSCs體外軟骨分化前后7 d的染色質表觀遺傳譜,分析了6種組蛋白修飾(H3K4me3,H3K9ac,H327me3,H3K36me3,H3K4me1,H3K27ac)在整體基因組上的變化情況,并用亞硫酸氫鹽修飾后測序法檢測DNA甲基化和mRNA芯片檢測基因表達情況。結果表明,分化過程中上調的基因與軟骨形成密切相關。在其基因體(gene bodies)區發生了與轉錄延伸相關的H3K36me3修飾,在基因的啟動子區和5'末端區域活化染色質標記H3K4me3和H3K9ac水平大幅增加,每個上調表達的基因在至少一個增強子區域的H3K27ac和H3K4me1修飾增加,這兩種修飾也是活化染色質的標記。在7 d的時間范圍內啟動子區的DNA甲基化改變與基因表達的變化并不顯著相關。
此外,Tan 等[33]應用ChIP-on-chip技術在全基因組水平研究hBMMSCs的基因啟動子區H3K9ac和H3K9me2修飾狀況,結果表明在hBMMSCs中基因啟動子區H3K9的修飾與mRNA的表達相關性很好,功能分析顯示在hBMMSCs自我更新中多種關鍵的胞內信號轉導途徑能夠被H3K9的修飾調控。而在hBMMSCs成骨分化過程中,H3K9ac在基因啟動子區的整體富集逐漸降低,H3K9me2的整體富集升高[34]。這些結果表明H3K9ac和H3K9me2影響的基因活化和沉默可能對hBMMSCs的自我更新、多能性維持及成骨分化至關重要。
6 展望
表觀遺傳調控參與了多種細胞發育和分化過程,也是BMMSCs分化的主要調控機制。近年來已發現多種參與BMMSCs分化的表觀遺傳修飾,在此基礎上,研發有效調控這些修飾的藥物,為BMMSCs提供精確的分化條件,使BMMSCs能夠向可控可預測的方向分化。除了文中介紹的組蛋白修飾類型外,組蛋白磷酸化、泛素化等修飾在BMMSCs分化中的作用也有待深入研究。此外,隨著BMMSCs體外培養代數的增加,DNA復制、細胞周期和成脂分化相關基因甲基化水平升高[35]。這些因素對BMMSCs的衰老有重要影響,因此對DNA甲基化的合理調控是抑制BMMSCs衰老,使之更有效的應用于臨床治療的一種方法。這些研究成果對其他組織來源的間充質干細胞,如脂肪和臍帶血來源的間充質干細胞的分化機制研究及臨床應用也有重要的參考價值。BMMSCs的異常分化可導致腫瘤及其他一些疾病的發生,表觀遺傳調控的異常是其主要原因之一[3],表觀遺傳修飾試劑作為分化誘導劑的應用對選擇性癌癥治療是很重要的,它侵入性更低,并對癌細胞有更高的選擇性。表觀遺傳治療的缺點是缺乏特異性,目前能夠調控干細胞分化和增殖的小分子藥物已在檢測和開發階段,可調控觀編程和發育信號途徑的各個方面[36]。
綜上所述,表觀遺傳在BMMSCs的多能性維持和分化過程中發揮了重要的調控作用,但具體機制尚 不完全明了,新的調控修飾有待發現。這個領域的深入研究必將為提高BMMSCs的定向分化效率提供線索,在臨床組織工程和細胞移植領域有廣闊的應用前景。
[1]Pittenger MF, Mackay AM, Beck SC, et al. Multilineage p otential of adult human mesenchymal stem cells[J]. Science, 1999, 284(5411):143-147.
[2]Jin HJ, Bae YK, Kim M, et al. Comparative analysis of human mesenchymal stem cells from bone marrow, adipose tissue, and umbili cal cord blood as sources of cell therapy[J]. Int J Mol Sci, 2013, 14(9):17986-8001.
[3]Leu YW, Huang TH, Hsiao SH. Epigene tic reprogramming of mesenchymal stem cells[J]. Adv Exp Med Biol, 2013, 754:195-211.
[4]Jenuwe in T, Allis CD. Translating the histone code[J]. Science, 2001, 293(5532):1074-1080.
[5]Li Z, Liu C, Xie Z, et al. Epigenetic dysregulation in mesenchy mal stem cell aging and spontaneous differentiation[J]. PLoS One, 2011, 6(6):e20526.
[6]Lu DF, Yao Y, Su ZZ, et al. Downregulation of HDAC1 is involved in the cardiomyocyte differentiation from mese nchymal stem cells in a myocardial microenvironment[J]. PLoS One, 2014, 9(4):e93222.
[7]Fu Y, Zhang P, Ge J, et al. Histone deacetylase 8 suppresses osteogenic differentiation of bone marrow stromal cells by inhibiting hist one H3K9 acetylation and RUNX2 activity[J]. Int J Biochem Cell Biol, 2014, 54:68-77.
[8]Yoon DS, Choi Y, Jang Y, et al. SIRT1 directly regulates SOX2 to maintain self-renewal and multipotency i n bone marrow-derived mesenchymal stem cells[J]. Stem Cells, 2014, 32(12):3219-3231.
[9]Simic P, Zainabadi K, Bell E, et al. SIRT1 regulates differentiation of mesenc hymal stem cells by deacetylating beta-catenin[J]. Embo Mol Med, 2013, 5(3):430-440.
[10]Buhrmann C, Busch F, Shayan P, et al. Sirtuin-1(SIRT1)is required for promoting chondrogenic d ifferentiation of mesenchymal stem cells[J]. J Biol Chem, 2014, 289(32):22048-22062.
[11]Dong X, Pan R, Zhang H, et al. Modification of histone acetylation facilitates hepatic differen tiation of human bone marrow mesenchymal stem cells[J]. PLoS One, 2013, 8(5):e63405.
[12]Liu J, Wang Y, Wu Y, et al. Sodium butyrate promotes the differentiation of rat bone marrow mesenchymal stem cells t o smooth muscle cells through histone acetylation[J]. PLoS One, 2014, 9(12):e116183.
[13]Han B, Li J, Li Z, et al. Trichostatin A stabilizes the expression of pluripotent genes in hum an mesenchymal stem cells during ex vivo expansion[J]. PLoS One, 2013, 8(11):e81781.
[14]Zych J, Stimamiglio MA, Senegaglia AC, et al. The epigenetic modifiers 5-aza-2'-deoxycytidine and trichostatin A influence adipocyte different iation in human mesenchymal stem cells[J]. Braz J Med Biol Res, 2013, 46(5):405-416.
[15]Hemming S, Cakouros D, Isenmann S, et al. EZH2 and KDM6A act as an epigenetic switch to regul ate mesenchymal stem cell lineage specification[J]. Stem Cells, 2014, 32(3):802-815.
[16]Jo J, Song H, Park SG, et al. Regulation of differentiation potential of human mesenchymal stem cells by intracytoplasmic delivery of coactivator-associated arginine methyltransfe rase 1 protein using cell-penetrating peptide[J]. Stem Cells, 2012, 30(8):1703-1713.
[17]Krishnan V, Bryant HU, Macdougald OA. Regulation of bone mass by Wnt signaling[J]. J Clin Invest, 2006, 116(5):1202-1209.
[18]Zhou GS, Zhang XL, Wu JP, et al. 5-Azacytidine facilitates osteogenic gene expression and differentiation of mesenchymal stem cells by alteration in DNA methylation[J]. Cytotechnology, 2009, 60(1-3):11.
[19]Hsiao SH, Lee KD, Hsu CC, et al. DNA methylation of the Trip10 promoter accelerates mesenchymal stem cell lineage determination[J]. Biochem Biophys Res Commun, 2010, 400(3):305, 312.
[20]Teng IW, Hou PC, Lee KD, et al. Targeted methylation of two tumor suppressor genes is sufficient to transform mesenchymal stem cells into cancer stem/initiating cells[J]. Cancer Res, 2011, 71(13):4653-4663.
[21]Wang L, Li Z, Yu B, et al. Long noncoding RNAs expression signatures in chondrogenic differentiation of human bone marrow mesenchymal stem cells[J]. Biochem Biophys Res Commun, 2015, 456(1):459-464.
[22]Zhu L, Xu PC. Downregulated LncRNA-ANCR promotes osteoblast differentiation by targeting EZH2 and regulating Runx2 expression[J]. Biochem Biophys Res Commun, 2013, 432(4):612, 617.
[23]Wang L, Wang Y, Li Z, et al. Differential expression of long noncoding ribonucleic acids during osteogenic differentiation of human bone marrow mesenchymal stem cells[J]. International Orthopaedics, 2015, 39(5):1013-1019.
[24]Lee SY, Ham O, Cha MJ, et al. The promotion of cardiogenic differentiation of hMSCs by targeting epidermal growth factor receptor using microRNA-133a[J]. Biomaterials, 2013, 34(1):92, 99.
[25]Cai B, Li J, Wang J, et al. microRNA-124 regulates cardiomyocyte differentiation of bone marrow-derived mesenchymal stem cells via targeting STAT3 signaling[J]. Stem Cells, 2012, 30(8):1746-1755.
[26]Han R, Kan Q, Sun Y, et al. MiR-9 promotes the neural differentiation of mouse bone marrow mesenchymal stem cells via targeting zinc finger protein 521[J]. Neurosci Lett, 2012, 515(2):147-152.
[27]Paik S, Jung HS, Lee S, et al. miR-449a regulates the chondrogenesis of human mesenchymal stem cells through direct targeting of lymphoid enhancer-binding factor-1[J]. Stem Cells Dev, 2012, 21(18):3298-3308.
[28]Skarn M, Namlos HM, Noordhuis P, et al. Adipocyte differentiation of human bone marrow-derived stromal cells is modulated by microRNA-155, microRNA-221, and microRNA-222[J]. Stem Cells Dev, 2012, 21(6):873-883.
[29]Zhao QH, Wang SG, Liu SX, et al. PPARgamma forms a bridge between DNA methylation and histone acetylation at the C/EBPalpha gene promoter to regulate the balance between osteogenesis and adipogenesis of bone marrow stromal cells[J]. The FEBS journal, 2013, 280(22):5801-5814.
[30]Aoyama T, Okamoto T, Fukiage K, et al. Histone modifiers, YY1 and p300, regulate the expression of cartilage-specific gene, chondromodulin-I, in mesenchymal stem cells[J]. J Biol Chem, 2010, 285(39):29842-29850.
[31]胡曉青, 張辛, 代嶺輝, 等. 骨髓間充質干細胞成骨分化過程中Runx2的表觀遺傳學修飾[J]. 中國生物化學與分子生物學報, 2014(2):150-155.
[32]Herlofsen SR, Bryne JC, Hoiby T, et al. Genome-wide map of quantified epigenetic changes during in vitro chondrogenic differentiation of primary human mesenchymal stem cells[J]. BMC Genomics, 2013, 14:105.
[33]Tan J, Huang H, Huang W, et al. The genomic landscapes of histone H3-Lys9 modifications of gene promoter regions and expression profiles in human bone marrow mesenchymal stem cells[J]. J Genet Genomics, 2008, 35(10):585-593.
[34]Tan J, Lu J, Huang W, et al. Genome-wide analysis of histone H3 lysine9 modifications in human mesenchymal stem cell osteogenic differentiation[J]. PLoS One, 2009, 4(8):e6792.
[35]Choi MR, In YH, Park J, et al. Genome-scale DNA methylation pattern profiling of human bone marrow mesenchymal stem cells in long-term culture[J]. Exp Mol Med, 2012, 44(8):503-512.
[36]Eid JE, Garcia CB. Reprogramming of mesenchymal stem cells by oncogenes[J]. Semin Cancer Biol, 2015, 32:18-31.
(責任編輯 狄艷紅)
Research Progress on Epigenetic Regulation Mechanism in the Differentiation of Bone Marrow Mesenchymal Stem Cell
WANG Lian-qing ZHAI Qiao-li ZHAO Pei-qing LI Tao
(Center of Translational Medicine,Central Hospital of Zibo,Zibo 255036)
Bone Marrow Mesenchymal Stem Cells(BMMSCs),with the potential of self-renewal and multipotent differentiation,may differentiate to be several types of cells by induction,thus hold critical clinic values for the restoration of destroyed bones and cartilages. In order to investigate a medicine that may efficiently promote the directional differentiation of BMMSCs and thus using BMMSCs in clinic more safely and reasonably,it is necessary to illustrate the epigenetic regulation mechanism in the differentiation process of BMMSCs. Abnormal regulation of BMMSCs differentiation may lead to development of several diseases,thus appropriate control of BMMSCs differentiation mechanism has been the hotspot topic of research. Epigenetic regulation,such as DNA methylation,histone acetylation,histone methylation and noncoding RNA,play critical roles in the regulation of BMMSCs differentiation. This review summarizes the progress on major epigenetic regulation mechanisms involved in BMMSCs differentiation in recent years.
bone marrow mesenchymal stem cells(BMMSCs);cell differentiation;epigenetic regulation
10.13560/j.cnki.biotech.bull.1985.2016.07.003
2015-08-25
山東省自然科學基金項目(ZR2014HM042,ZR2015PH031,ZR2015HM031)
王連慶,男,博士,研究方向:遺傳學;E-mail:lianqing.wang@hotmail.com
李濤,男,博士,研究方向:骨科;E-mail:litaozhongguo@vip.163.com

