上調基因-11(URG11)生物信息學與功能分析
姚楊 蘇杰 劉凱歌 徐銳
(西安醫學院第一附屬醫院,西安 710077)
上調基因-11(URG11)生物信息學與功能分析
姚楊 蘇杰 劉凱歌 徐銳
(西安醫學院第一附屬醫院,西安 710077)
應用基因芯片技術獲取以穩定轉染HBx基因的肝癌細胞HepG2(HepG2-X)Y,以及非轉染的肝癌細胞HepG2的差異表達基因,利用生物信息學方法對其進行初步分析表明,該蛋白基因編碼673個氨基酸,預測分子量為17.06 kD,理論等電點為4.83,定位于細胞核,具有轉錄調控、生長因子、信號傳導的功能,同源性分析結果表明,其堿基序列與已經報道的其他12個物種的相似率為76%-97%,且符合種屬之間的進化關系。
乙肝相關性肝癌;生物信息學分析;URG11;HBx
目前,我國每年約有11-13萬人死于原發性肝癌,占全球肝癌死亡數的半數之多。在誘發肝細胞癌(hepatocellular carcinoma,HCC)的眾多因素中,乙型肝炎病毒(hepatitis B virus,HBV)感染占到50%以上[1-3]。流行病學和分子生物學研究認為HBV是HBV相關性HCC發生的高危因素。HBx(Hepatitis B virus X protein)是HBV四個開放讀碼框架中X編碼的一種病毒多功能蛋白,研究發現X基因表達產物(HBx)在肝癌形成過程中參與肝癌血管生成,但作用機理并不明確[4,5]。URG11(upregulated gene11)是一個可被HBx蛋白上調的基因,能促進肝癌細胞G1期向S期進展,與腫瘤的發生、發展和轉移密切相關的基因[6,7]。
URG11與腫瘤關系的相關的報道已經很多,但并未對其進行詳細的生物信息學分析。本實驗通過HBx基因穩定轉染的HepG2細胞為實驗組,非轉染的肝癌細胞HepG2為對照組,通過Agilent人全基因芯片篩選差異表達基因,通過生物信息學方法對其進行分析,旨在為HBV相關肝癌的發生、轉移的診斷、靶基因治療和預后評估提供參考。
1 材料與方法
1.1 材料
人肝癌細胞系HepG2及HepG2-X均為本實驗室保存。10%小牛血清(四季青生物工程工司),RPMI-1640培養基(Gibco公司);消化液(0.02%EDTA 0.25%胰蛋白酶);DEPC水;Agilent人類全基因4×44K芯片;Agilent Microarray Scanner和Agilent feature extraction數據分析軟件。
1.2 方法
1.2.1 基因芯片 用Trizol[8]法分別抽取實驗組及對照組細胞總RNA,紫外分析及電泳檢測,合成有熒光標記的cDNA探針并純化,將基因芯片和雜交探針分別在95℃水溶液中變性5 min,將探針加在芯片上,蓋玻片封片,65℃,10 r/min,滾動雜交17 h,實驗組和對照組分別雜交上樣量825 ng,60℃下用SCC和0.2%SDS分別洗2 min,室溫晾干。片結果采用Agilent Microarray Scanner進行掃描,用Feature Extraction software 10.7讀取數據,最后采用Gene Spring Software 11.0進行歸一化處理,所用的算法為Lowess。
1.2.2 序列的生物信息學分析 利用BLAST在線分析工具分別對GenBank 的非冗余核算數據庫和非冗余蛋白質數據庫序列進行比對分析;采用NCBI的ORF finder[9]對URG11開放閱讀框進行預測;利用ExPASy 的ProtParam[10]程序統計基因中各種氨基酸含量,并預測理論分子量和等電點;應用ProtScale[11]程序進行蛋白質的疏水性分析;跨膜結構域分析采用TMHMM軟件預測[12]。信號肽采用SignalP3.0server工具分析[13]。磷酸化位點分析采用蛋白質亞細胞定位NetPhos2.0 Server在線分析工具[11]。采用在線工具(http://psort nibb.ac.jp/form. html)預測,通過NCBI數據庫GenBank 分析蛋白質保守結構域;采用Protfun在線工具預測URG11的功能分類[14];采用同源模型服務軟件SWISSMDEL[15,16]預測URG11的三維結構;采用MEGA5.0軟件進行系統進化樹的構建。
2 結果
2.1 開放閱讀框分析
本研究利用NCBI ORF Finder對URG11在線分析表明其在+1閱讀框有最長的開放性讀碼框,長度為2 020 bp編碼673個氨基酸殘基(圖1)。
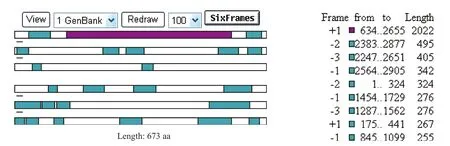
圖1 人URG11基因序列的ORF分析
2.2 保守結構域預測
利用NCBIBLAST 工具進行保守區分析,發現URG11氨基酸序列中含有4個保守的作用位點,如圖2所示。
2.3 理化性質分析
利用ExPASy 在線工具ProtParam對URG11基因編碼產物的理化性質預測結果表明,其中含量最多的兩種氨基酸是Cys(C)和Gly(G),所占比例分別為34.4%和27.8%理論分子量為17.06 kD,理論等電點為4.83。
根據蛋白質親水性/疏水性分析原理,利用ExPASy ProtScal程序,對人URG11基因編碼產物的疏水性進行分析,結果(圖3)顯示,整條多肽鏈表現為親水性,由此可知人URG11是一種可溶性蛋白。
2.4 跨膜結構域分析
TMHMM綜合了跨膜區疏水性、螺旋長度和膜蛋白拓撲學限制等性質,用馬氏模型對跨膜區及膜內外區進行整體預測。本實驗利用TMHMM對URG11進行跨膜結構的分析,結果表明此蛋白不含跨膜位點。
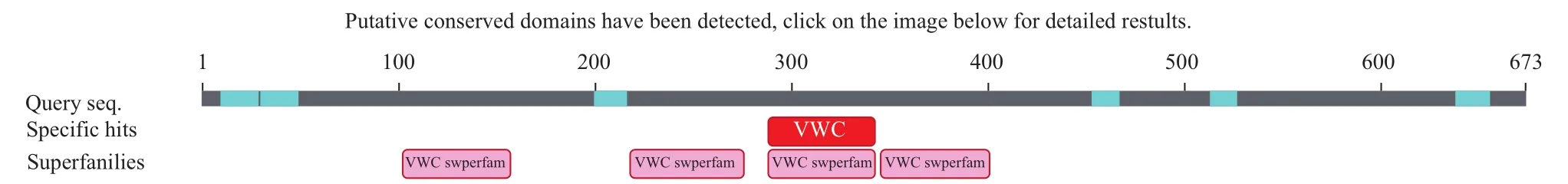
圖2 保守域預測結果
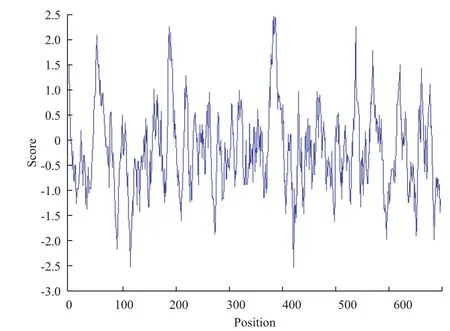
圖3 ProtScal程序分析URG11編碼產物的疏水性結果
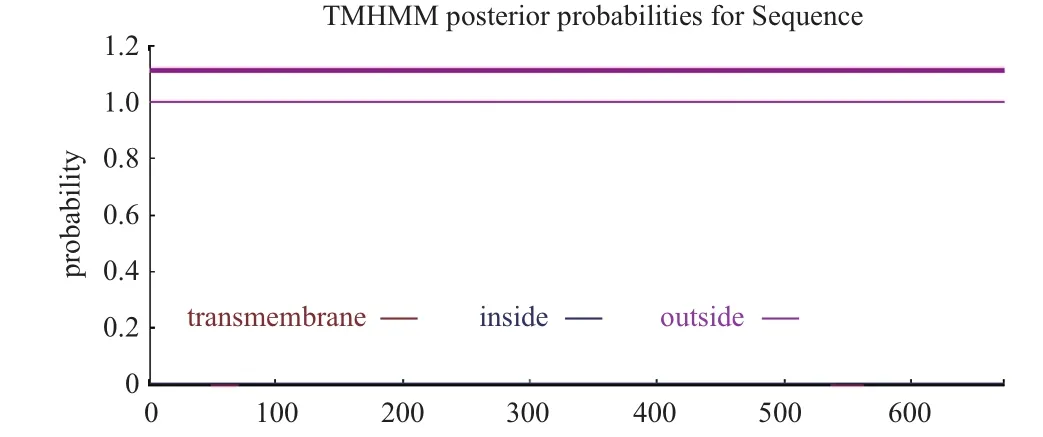
圖4 URG11跨膜結構預測產物信號肽預測
信號肽(signal peptide)是一段由3-60個氨基酸組成的短肽序列,它是引導新合成蛋白質被輸送至目的地點的識別信號,也被稱為靶標信號。利用SignalP 3.0 Server工具對人URG11信號肽進行預測,結果(圖5)表明,該蛋白不含信號肽。

圖5 URG11編碼產物氨基酸序列信號肽預測
2.5 磷酸化位點分析
磷酸化和去磷酸化在多種真核細胞中具有重要的作用,如新陳代謝、細胞分裂及信號轉導等,利用NetPhos2.0 Server在線分析工具對URG11磷酸化位點分析,結果(圖6)發現有29個絲氨酸和12個蘇氨酸可能被磷酸化。
2.6 編碼產物功能預測及分析
通過Protfun預測URG11編碼產物的功能,從分析結果(表1)可知,URG11具有轉錄調控、生長因子、信號傳導的功能,可能性分別為0.854、0.476、0.365。
2.7 URG11保守域預測與高級結構分析
通過Swiss-Modeling軟件對URG11的氨基酸序列進行蛋白質三維結構的同源建模,獲得其氨基酸序列的預測三級結構(圖7)。
2.8 同源比對和系統進化樹分析
將URG11氨基酸序列與NCBI數據庫在核苷酸和蛋白質一級結構水平上進行同源性比較,結果(表2)表明其在進化中十分保守,與其他12種物種URG11氨基酸水平的相似性為76%-97%。同時也反映了URG11編碼產物在不同物種結構上的穩定性對生物體功能的重要性。根據URG11基因特征序列和保守序列在GenBank數據庫進行檢索,物種間系統進化樹(圖8)分析表明,白頰長臂猿、蘇門答臘猩猩、獼猴的親緣關系較近。

圖6 NetPhos2.0 Server預測磷酸化位點

表1 URG11編碼產物功能分析結果
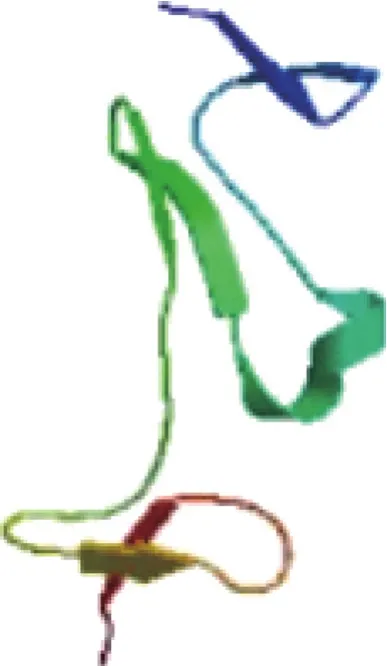
圖7 URG11氨基酸序列高級結構分析結果
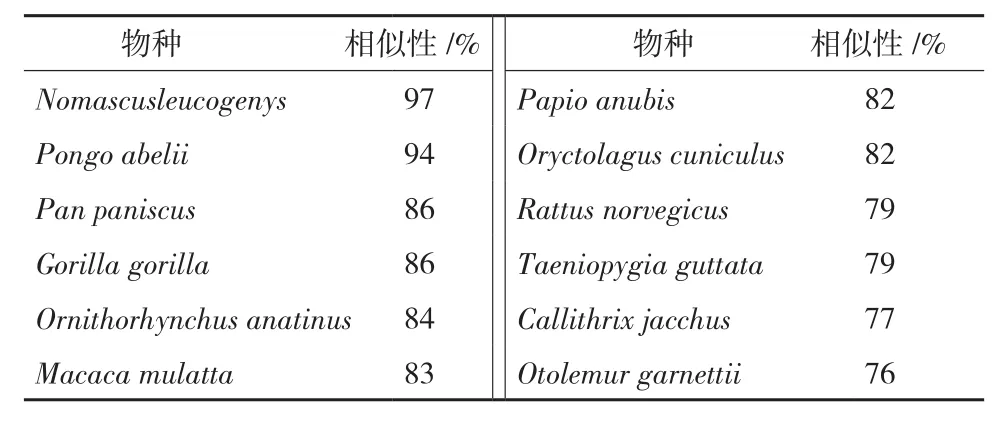
表2 URG11與其他近緣物種該蛋白基因氨基酸序列同源性
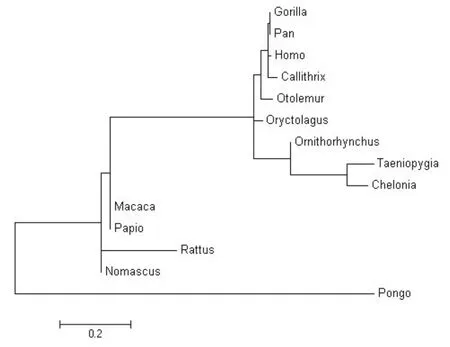
圖8 不同物種URG11編碼產物系統發育樹
3 討論
URG11最早是利用抑制消減雜交及全長PCR擴增技術克隆的一個基因,該基因定位于人11號染色體長臂,能促進肝癌細胞系的增殖、分化并促進肝癌細胞裸鼠體內的成瘤能力,更重要的是將此基因轉染入腫瘤細胞,可促進B-連接蛋白(B-catenin)的表達。B-catenin不僅在維持細胞正常形態及介導細胞運動中發揮重要作用,還是與腫瘤發生及發展密切相關的Wnt信號轉導通路中的關鍵分子[17,18]。Peng 等[19]發現URG11通過上皮間質轉化促進胰腺癌的侵襲,從而導致預后效果不佳。Fan等[20]發現抑制URG11,HCC細胞可通過下調G1-S期相關蛋白抑制細胞增殖,通過下調BCL-2誘導細胞凋亡。
雖然關于URG11基因的報道已經很多,但并未對其進行詳細的生物信息學分析。本研究通過基因芯片技術篩選出HBx參與的乙肝相關性肝細胞性癌差異表達基因URG11,對其同源性比較結果表明,其堿基序列與已經報道的其他12個物種的相似率為76%-97%,與其他物種比較,URG11基因氨基酸序列表現出高度保守性,以保證其執行重要的生物學功能。利用MeGa5.0軟件基因NJ法構建系統進化樹,人URG11與大猩猩、黑猩猩、普通狨該基因的分子進化距離最近,親緣關系最密切,這與動物學分類結果一致,這種同源性在一定程度上代表著物種親緣關系的遠近,同時也反映了URG11編碼產物在不同物種結構上的穩定性對生物體功能的重要性。隨著對URG11結構和功能的深入研究,針對URG11的干擾和抑制可能成為一種新型的臨床分子靶點抗癌藥物。
4 結論
本研究結合分子生物學技術和生物信息學分析,通過基因芯片技術篩選出HBx參與的乙肝相關性肝細胞性癌差異表達基因URG11,該基因編碼673個氨基酸,預測分子量為17.06 kD,理論等電點為4.83,同源性分析結果表明,其堿基序列與已經報道的其他12個物種的相似率為76%-97%,且符合種屬之間的進化關系。亞細胞定位于細胞核的可能性大,具有轉錄調控、生長因子、信號傳導的功能。
[1] Wang C, Yang W, Yan HX, et al. Hepatitis B virus X(HBx)induces tumorigenicity of hepatic progenitor cells in 3, 5-diethoxycarbonyl-1, 4-dihydrocollidine-treated HBx transgenic mice[J]Hepatology, 2012, 55(1):108-120.
[2] Yuan W, Huang T, Yu J, et al. Comparative analysis of viral protein interaction networks in Hepatitis B Virus and Hepatitis C Virus infected HCC[J]. Biochim Biophys Acta, 2013, 6(2):512-516.
[3] Gong P, Zhang X, Zhang J, et al. Comparative analysis of viral protein interaction networks in Hepatitis B Virus and Hepatitis C Virus infected HCC[J]. Front Biosci, 2013, 18:1256-65.
[4] Tang RX, Kong FY, Fan BF, et al. HBx activates FasL and mediates HepG2 cell apoptosis through MLK3-MKK7-JNKs signal module[J]. World Journal of Gastroenterology, 2012, 18(13):1485-1495.
[5] Holotnakova T,Tylkova L, Takacova M. Role of the HBx oncoprotein in carbonic anhydrase 9 induction[J]. J Med Virology, 2010, 82(1):126-129.
[6] Tong GD, Zhang X, Zhou DQ, et al. Efficacy of early treatment on 52 patients with preneoplastic hepatitis B virus-associated hepatocellular carcinoma by compound Phyllanthus urinaria L. [J]. Chin J Integr Med, 2014, 20(4):263-271.
[7] Yuan K, Lian Z, Sun B, et al. Role of miR-148a in hepatitis B associated hepatocellular carcinoma[J]. PLoS One, 2012, 7(4):1-12.
[8] Reno C, Marchuk L, Sciore P, et al. Rapid isolation of total RNA from small samples of hvpocellular dense connective tissues[J]. Bio Techniques, 1997, 22:1082-1086
[9] Altschul SF, Madden TL, Sch?ffer AA, et al. Gapped BLAST and PSI-BLAST:A new generation of protein database search programs[ J ]. Nucleic Acids Res, 1997, 25(17):3389-3402.
[10] Wilkins MR, Gasteiger E, Bairoch A, et al. Protein identification and analysis tools on the ExPASy server[J] . Methods Mol Biol,1999, 112:531-52.
[11] 王慶浩, 陳愛華, 張伯禮.丹參:一種中藥研究的模式生物[J].中醫藥學報, 2009, 37(4):1-3.
[12]Chen Y, Yu P, Luo J, Jiang Y. Secreted protein prediction system combining CJ-SPHMM, TMHMM, and PSORT[J]. Mammalian Genome, 2003, 14(12):859-865
[13]Mukhopadhyay D, Riezman H. Proteasome-independent functions of ubiquitin in endocytosis and signaling[J]. Science, 2007,315:201-205.
[14] Nielsen H, Engelbrecht J, Brunak S, von Heijne G. A neural network method for identification of prokaryotic and eukaryoticsignal peptides and prediction of their cleavage sites[ J ]. International Journal of Neural Systems, 1997, 8(5):581-599.
[15] Arnold K, Bordoli L, Kopp J, Schwede T. The SWISS-MODEL workspace:a web-based environment for protein structure homology modeling[J]. Bioinformatics, 2006, 22(2):195-201.
[16] Kiefer F, Arnold K, Kunzli M, et al. The SWISS-MODEL repository and associated resources[J]. Nucleic Acids Research, 2009,37:D387-D392.
[17] Lian Z, Liu J, Li L, et al. Enhanced cell survival of Hep3B Cell by the hepatitis B x antigen effector, URG11, is associated with upregulation of beta-catenin [J]. Hepatology, 2006, 43(3):415-424.
[18] Du R, Zhao L, Xia L. Association of URG11 and twist with clinical pathological characteristics and prognosis in patients with IgA nephropathy [J]. Nephrol Dial Transplant, 2013, 28(9):2268-2276.
[19] Peng W, Zhang J, Liu J. URG11 predicts poor prognosis of pancreatic cancer by enhancing epithelial-mesenchymal transitiondriven invasion [J]. Med Oncol, 2014, 31(7):64.
[20] Fan R Li X, Du W, et al. Adenoviral-mediated RNA interference targeting URG11 inhibits growth of human hepatocellular carcinoma[J]. Int J Cancer, 2011, 128(12):2980-2993.
.
(責任編輯 李楠)
Function Analysis of Up-regulated Gene(URG11)Based on Its Bioinformatics
YAO Yang SU Jie LIU Kai-ge Xu Rui
(The First Affiliated Hospital of Xi'an Medical University,Xi'an 710077)
This work was to obtain differentially-expressed genes with hepatoma cell HepG(HepG2-X)Y of stable transfected HBx and non-transfected hepatoma cell HepG2using gene chip technology. The preliminary bioinformatic analysis demonstrated that the gene of the protein encoded 673 amino acids with a predicted molecular weight of 17.06 kD and pI 4.83. URG11 were mainly localized in the nuclear and played the role in transcription regulation,growth factor and signal transducer,and it showed 76%-97% identity with the others reported 12 species,as well as was consistent with the evolutionary relationship between species.
HBV-related hepatocellular carcinoma;bioinformatics;URG11;HBx
10.13560/j.cnki.biotech.bull.1985.2016.03.032
2015-06-04
西安市科技計劃項目(SF1418(6)),西安醫學院科研項目(12FZ15),西安醫學院附屬醫院科研項目(XYFY11-14)
姚楊,碩士研究生,研究方向:生化與分子;E-mail:yaoyang1220@sina.com

