3種不同來源側耳品種親緣關系差異化分析
趙同心, 趙洪新, 王升厚
(1.沈陽師范大學 化學與生命科學學院,遼寧 沈陽 110034;2.浙江理工大學 生命科學學院,浙江 杭州 310018;3.沈陽師范大學 實驗教學中心,遼寧 沈陽 110034)
?
3種不同來源側耳品種親緣關系差異化分析
趙同心1, 趙洪新2, 王升厚3*
(1.沈陽師范大學 化學與生命科學學院,遼寧 沈陽 110034;2.浙江理工大學 生命科學學院,浙江 杭州 310018;3.沈陽師范大學 實驗教學中心,遼寧 沈陽 110034)
采取拮抗實驗、酯酶同工酶、基因組進行多態性分析(RAPD)、內部轉錄間隔區序列(ITS)對來源不同的特大鳳尾菇(Pleurotussajor-caju)、夏秀990(Pleurotusgeesteranus)和黑平A3三個側耳品種的親緣關系進行了對比分析。實驗結果表明:供試的3個側耳品種從形態學水平、蛋白質水平和分子水平上看,其品種間親緣關系界定具有一致性。表現為特大鳳尾菇與夏秀990的親緣關系較近,與黑平A3的親緣關系較遠,夏秀990與黑平A3親緣關系較遠。本研究從不同的水平,為食用菌菌種屬間親緣關系的鑒定提供了綜合分析方法,對區分同種異名、雜交親本選擇以及判定雜交結果具有指導意義。
側耳;親緣關系;酯酶同工酶;隨機擴增多態性DNA(RAPD);轉錄間隔區序列(ITS)
側耳屬真菌常見的品種有糙皮側耳(Pleurotusostreatus,俗稱平菇),金頂側耳(PleurotuscitrinopileatusSing,俗稱榆黃蘑),漏斗狀側耳(Pleurotussajor-caju,俗稱鳳尾菇),刺芹側耳(Pleurotuseryngi,俗稱杏鮑菇),阿魏側耳(PleurotusferulaeLanzi,俗稱白靈菇)等,全世界約有50多種,我國現有36種[1],是目前人工栽培數量較大、范圍較廣的食用大型真菌。隨著人工栽培規模的不斷擴大,特別是選育新品種的種植與推廣,新的商品名稱或代號的菌種不斷出現,出現了來源各異、同種異名或異種同名的混亂現象。由于不同種源對環境適應能力的差異,給引種菇農造成損失現象時有發生。為了對菌種做到科學管理、保護品種產權、杜絕生產領域的菌種亂引亂種現象,許多研究人員采取不同方法對側耳屬進行鑒別。黃奕存[2]提出了不同個體間的拮抗反應,從菌絲的形態學方面進行不同鑒定。李榮春等[3]采用酯酶同工酶方法對不同菌株間的相似程度進行鑒定分析。鄭素月等[4]、張慶華等[5]、湯衛真等[6]分別分析了菌株之間酯酶同工酶的差異。隨著分子生物學的發展,RAPD技術和ITS技術用于平菇分類。詹才新等[7]、陳明杰等[8]驗證了RAPD可用于金針菇和香菇親緣關系的鑒定,隨后譚琦等[9]用RAPD技術分析并建立了遺傳相關聚類圖,分析出香菇雜交非對稱雜交后代相似關系。White等[10]為真菌rRNA基因的ITS設計3對特異引物,即ITS1、ITS4、ITS5,可用于大多數擔子菌和子囊菌。譚琦等[11]分析了中國6個白靈菇商業菌株的DNA圖譜,證明6個菌株為同一個種。為提高品種間親緣關系鑒定方法的有效性、可靠性和實用性,本研究選擇不同來源側耳屬的3個品種為供試菌株,采取拮抗鑒定、酯酶同工酶分析、RAPD多態性分析和ITS間隔區序列比對,從表型、蛋白和分子3個水平進行綜合運用,為菌種間親緣關系鑒定提供一種更為科學有效的綜合方法。
1 材料與方法
1.1 材料
1.1.1 供試菌株 夏秀990(秀珍菇,Pleurotusgeesteranus)、黑平A3(Pleurotusostreatus)、特大鳳尾菇(Pleurotussajor-caju),由沈陽師范大學特種菌業研究所保存。
1.1.2 培養基 改良PDA培養基:葡萄糖20 g,蛋白胨5 g,磷酸二氫鉀 2 g,硫酸鎂1 g,水1 000 mL。
1.2 方法
1.2.1 培養方法 ①拮抗實驗:取活化好的菌種,在改良PDA培養基上打孔,進行兩兩拮抗實驗, 25 ℃培養箱中培養1周。②菌種培養:在液體PDA培養基中,200 r/min、25 ℃培養1周,取菌絲體提取蛋白質和DNA。酯酶同工酶的方法參考文獻[12]。DNA提取用CTAB法。
1.2.2 RAPD 和ITS引物篩選與擴增 引物如表1所示。引物采取設計隨機引物的方法,從引物庫中進行篩選。引物合成和PCR產物測序,由上海生工生物公司完成。 RAPD PCR反應體系:10 ×TaqPCR Buffer 2.5 μL,隨機引物2 μL(2 μmol/L),TaqDNA聚合酶 0.5 μL,DNA 0.5 μL,dNTP 0.5 μL(10 mmol/L),去離子無菌水 14 μL。PCR反應程序: 94 ℃預變性 5 min,94 ℃變性1 min,36 ℃退火1 min,72 ℃延伸80 s,30個循環,72 ℃保持10 min,4 ℃保存。樣品取出置于冰箱-20 ℃中保藏。用0.8%瓊脂糖凝膠電泳,切膠分離PCR產物,回收,送生工生物工程(上海)股份有限公司測序。
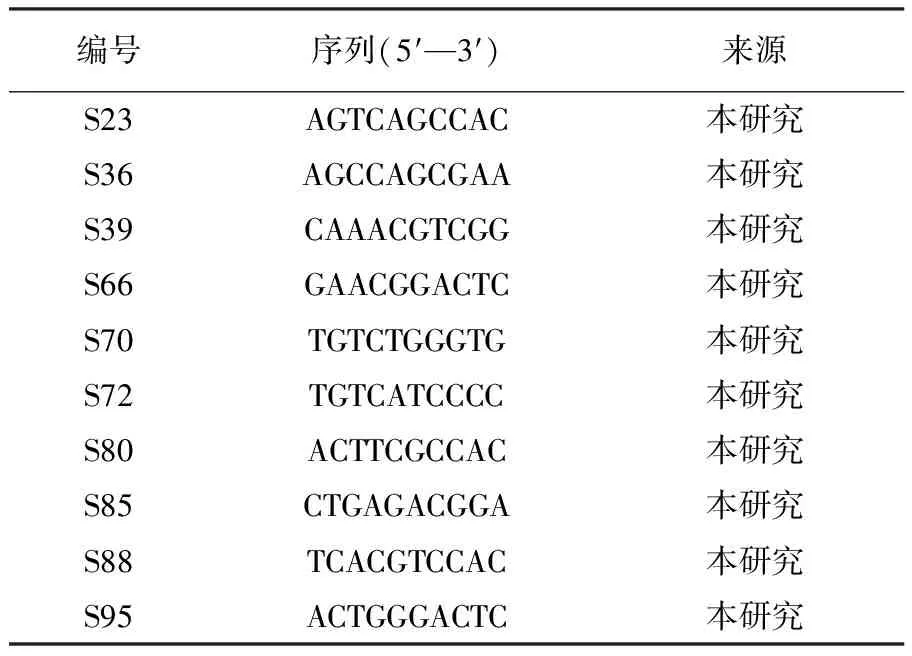
表1 RAPD 引物
ITS聚合酶鏈式反應PCR 擴增參考文獻[7],用2個真菌引物ITS4和ITS5進行擴增。PCR反應體系 10 ×TaqPCR Buffer 2 μL,引物ITS4 0.5 μL,引物ITS5 0.5 μL,TaqDNA聚合酶 0.5 μL,DNA 2 μL,dNTP 0.5 μL(10 mmol/L),去離子無菌水 14 μL。
PCR反應條件:94 ℃ 預變性 5 min,94 ℃變性1 min,55 ℃退火1 min,72 ℃延伸90 s,30個循環,72 ℃保持10 min,4 ℃保存,取出樣品冰箱-20 ℃保藏。用0.8%瓊脂糖凝膠電泳分離PCR產物。切膠回收PCR產物,將回收的產物連接到T載體上,轉化到大腸埃希菌E.coli5α的感受態細胞中,Amp100抗性篩選,用菌落PCR篩選陽性的單菌落。用鼎國質粒提取試劑盒(NEP011-1)提取純化質粒后,送生工生物工程(上海)股份有限公司進行測序。
1.2.3 數據分析 用SPASS進行數值分析,MAGE 6.0進行ITS序列分析親緣關系。
2 結果與分析
2.1 拮抗實驗
不同菌種之間均出現拮抗線(圖1),說明3種平菇屬于不同的種。
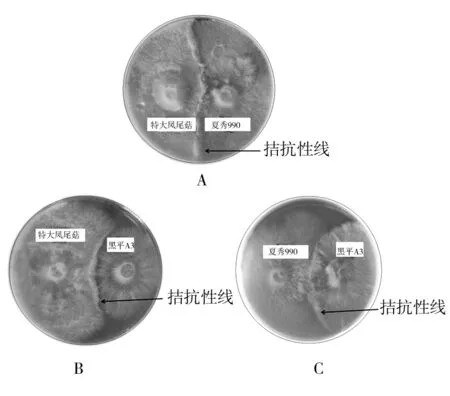
圖1 3個平菇品種的拮抗反應
2.2 酯酶同工酶譜帶分析
2.2.1 酶譜的類型 3個平菇品種共有18條酯酶同工酶條帶(圖2),分子量大約在70~170 kDa。 遷移率不同的為13條,其中黑平A3有4條,與其他兩個品種相比,沒有相同分子量的條帶,說明與其他兩個品種之間差異較大。夏秀990有5條酶帶,與特大鳳尾菇相同,說明二者親緣關系較近;另外,特大鳳尾菇有自己特異的4條帶,與夏秀990不同。可能由于特大鳳尾菇與夏秀990存在進化程度上的差異,或者特大鳳尾菇進化的快些,導致了較多的特異性條帶。
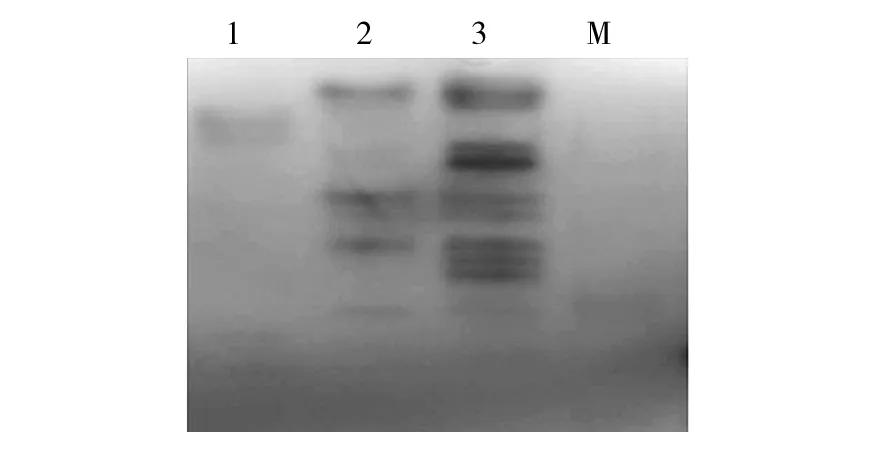
圖2 酯酶同工酶電泳圖
2.2.2 聯合系數 根據sneath和sokal(1973年)提出的聯合系數,可以清楚地反映品種間的親緣關系。聯合系數在0~1之間變化,其值越大兩品種親緣關系越近,越小親緣關系越遠。特大鳳尾與夏秀990聯合系數為0.556,說明二者親緣關系較近,黑平菇的親緣關系較其他兩個關系較遠,見表2。
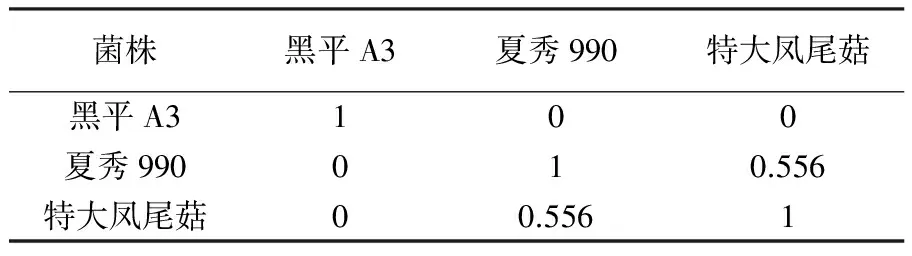
表2 3個平菇品種之間的聯合系數
2.3 RAPD 條帶分析
2.3.1 RAPD電泳條帶分析 10個RAPD 引物對3種側耳進行PCR擴增,篩選出S23、S88引物進行電泳分析。S23引物擴增結果,3個品種中有2條相同的條帶,特大鳳尾菇與夏秀990帶型一致,其中夏秀990的3條帶和特大鳳尾菇相同,特大鳳尾菇2條特異性條帶,黑平A3特異性條帶2條與另外兩種明顯不同。S88引物結果顯示,特大鳳尾菇5條帶其中2條與夏秀990相同,特異性條帶2條,夏秀990特異性條帶1條。黑平A3有1條與夏秀990相同,2條特異性條帶。結果見圖3。總之,根據RAPD結果分析可知夏秀990和特大鳳尾菇的條帶較為一致,親緣關系較近,在分子水平上可以看出它們有共同的特征條帶,不同品種間存在著差異。
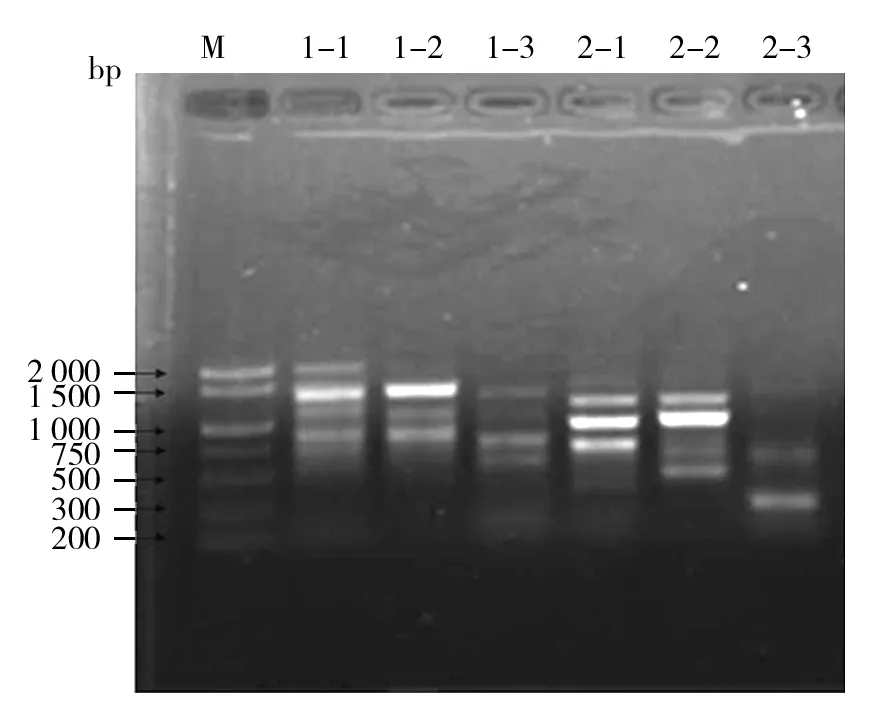
圖3 3個平菇品種之間的RAPD條帶
2.3.2 聚類分析 用SPASS進行數值分析,有條帶的為1,無條帶的為0,得到的聚類分析圖見圖4。由圖4可以看出,特大鳳尾菇和夏秀990親緣關系較近,與黑平A3進化距離遠。
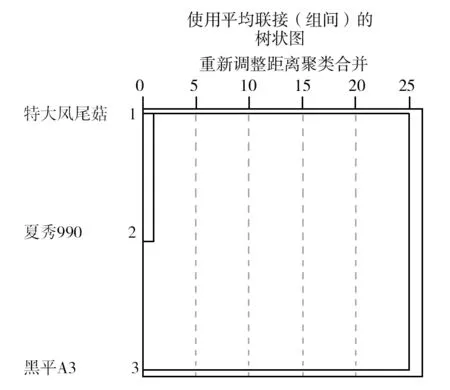
圖4 3個平菇品種之間的聚類分析Fig.4 Cluster analysis among three kinds Pleurotus spp.
2.4 ITS序列分析
2.4.1 目的片段的擴增 用引物ITS4和ITS5擴增3個品種的ITS區,電泳圖譜如圖5所示,可以看出,PCR條帶基本一致,分子量約為680 bp。
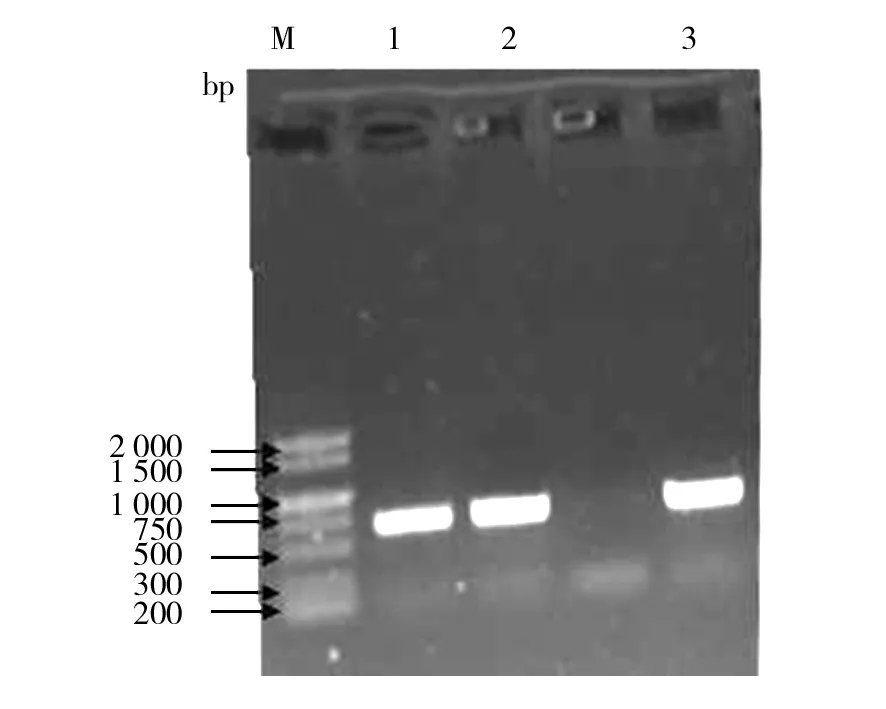
圖5 3個平菇品種ITS PCR產物Fig.5 ITS PCR product among three kinds Pleurotus spp.1:特大鳳尾菇;2:夏秀990;3:黑平A31:Phoenix mushroom;2:Xiaxiu 990;3:Black oyster mushroom A3
2.4.2 ITS分析 對3種平菇的ITS序列分析比對發現,夏秀990和特大鳳尾菇的序列完全相同,無堿基差別,而黑平A3與夏秀990、特大鳳尾菇堿基序列差別較大。1~54 bp為18S RNA,55~286 bp為轉錄間隔區1即ITS1,287~444 bp為5.8S RNA,445~642 bp為轉錄間隔區2即ITS2,643~703 bp為28S RNA。
由序列比對可以看出,在ITS1發生了堿基替換,第161 bp的C→T,第71 bp的T→C。在ITS2區序列出現堿基的替換和插入,在第466 bp的A→T,第 469 bp和第496 bp的T→C,第481 bp的T→A,第514 ~515 bp的TG→CT, 第517 bp和第521 bp A→G,在469 bp和470 bp之間插入了序列GGTTTTTTTCC。
2.4.3 平菇的進化樹 用MAGE 6.0進行親緣關系遠近分析,結果顯示:1號的特大鳳尾菇和2號的夏秀990親緣關系較近,3號黑平A3親緣關系較遠。本研究通過對ITS區域的擴增測序, 分析平菇不同品種間的親緣關系并將平菇ITS區域序列登錄到核苷酸序列數據庫中,登錄號分別為KP877605、KP877606、KP877607,見圖6。
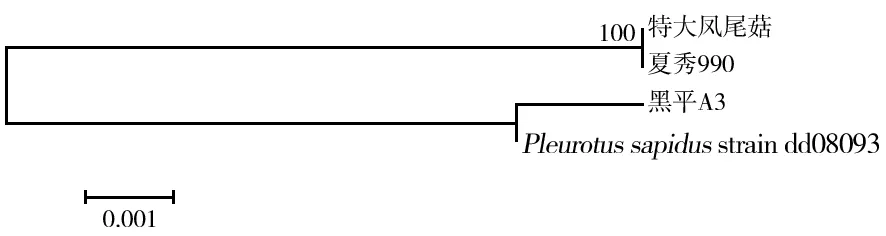
圖6 3個平菇品種ITS聚類分析Fig.6 ITS Cluster Analysis among three kinds Pleurotus spp.
3 討 論
本研究結合形態學的拮抗反應,蛋白質水平酯酶同工酶,分子水平的RAPD和ITS區序列分析得出了供試3種側耳屬品種間的親緣關系。拮抗反應是真菌鑒定的常用手段,但往往不能有效區分出兩個在遺傳上有相似背景或親緣關系很近的菌株,可作為初級分類的方法;同工酶是由不同等位基因或不同基因位點編碼的[13],特別是在屬內種間及品種之間的分類鑒定是非常有效的。同工酶技術也具有易受培養時間、培養基配方及培養溫度的影響而表現出不一致性的缺點,并且不能直接反映 DNA 水平的情況[14]。因此做好分子水平的鑒定就成為完整實驗方法的重要組成。本研究從ITS序列比對結果看,特大鳳尾菇和夏秀990序列完全相同,說明它們的進化程度一致,同時也說明了ITS區序列長度有限,相對全基因組而言,有限的序列信息要想真正反映物種的進化規律,還需要其他的形態學等方面的數據。從序列比對可以看出的ITS區域的差異主要表現在ITS2區域,與司源等[15]的ITS1相對變化、ITS2相對保守的觀點不同,進化的快慢可能是由于不同的生物ITS區,但是ITS2相對變化這一觀點與Yao等[16]將ITS2區域作為動植物的DNA標記觀點一致。
本研究從宏觀水平、蛋白質水平和DNA水平等多個層面說明了平菇之間的進化程度與親緣關系,同時也證明進行分類時應結合多種手段來說明它們之間的親緣關系。
[1] 圖力古爾,李玉.我國側耳屬真菌的種類資源及其生態地理分布[J].中國食用菌,2001,20(5):8-9.
[2] 黃亦存.高等擔子菌的性非親和性系統和體細胞非親和性系統在遺傳多樣性保存中的作用[J].生物多樣性,1996,4(1):41-44.
[3] 李榮春,陳嚴平.五種牛肝菌酯酶和過氧化物酶的同工酶研究[J].中國食用菌,1999,18(3)1:5-19.
[4] 鄭素月,張金霞,黃晨陽,等.中國栽培平菇的酯酶同工酶分析[J].食用菌學報,2003,10(4):1-6.
[5] 張慶華,趙新海,鐘麗娟,等.酯酶同工酶分析鑒別生產用平菇菌種真實性研究[J].山東農業科學,2009,26(6):10-12.
[6] 湯衛真,鄭國揚,畢志樹.利用同工酶酶譜法鑒別側耳屬的種和菌株[J].廣西植物,1987,7(2):143-145.
[7] 詹才新,朱蘭寶,楊新美.RAPD技術在金針菇雜交育種中的應用[J].食用菌學報,1995,2(1):1-7.
[8] 陳明杰,譚琪,汪昭月,等.應用RAPD技術對香菇融合菌株進行遺傳鑒定[J].食用菌學報,1997,4(4):17-20.
[9] 譚琦,潘迎捷,陳明杰,等.香菇遺傳研究和菌種選育的現狀及發展[J].食用菌學報,1998,5(3):6-11.
[10]White T J,Bruns T D, Lee.S. ,et al.Analysis of phytogenetic relationships by amplification and direct sequencing of ribosomal RNA genes[C].INNISMA.PCR Protocols:A Guide to Methods and Applications.New York Academic,1990:315-322.
[11]譚琦,楊建明,陳明杰,等.用RAPD技術對香菇非對稱雜交后代遺傳性狀的分析[J].食用菌學報,2001,8(2):10-14.
[12]胡能書,萬國賢.同工酶技術及應用[M].長沙:湖南科學技術出版社,1985:49-181.
[13]熊全沫.同工酶電泳數據的分析及其種群遺傳上的應用[J].遺傳,1980,8(1):1-5.
[14]Gottlieb L D. Conservation and duplication of isozymes in plants[J].Science,1982,216(23):373-380.
[15]司源,郭亦琦,孔航輝.基于ORF Finder方法的植物ITS片段結構特點分析[J].華北農學報,2005, 20(5):54.
[16]Yao H,Song J,Liu C,et al.Use of ITS2 region as the universal DNA barcode for plants and animal[J].PLoS ONE,2010,5(10):13102.
Found projecd: National Natural Science Foundation of China (31160371, 30860165); National Key Technology Research and
Development Program of China (2012BAD19B02); National High Technology Research and Development Program of China (863
Program) ( 2011AA10A206)
Brief introduction to the writer: Cheng liang male, Ph D student. Research field: natural product.
Tel:0971-5313283, E-mail:liangcheng1979@163.com
Draft accepted date: 2015-04-03; Revision returned date: 2015-08-31
Alliance Variance Analysis of ThreePleurotusSpeciesfrom Different Sources
ZHAO Tong-xin1, ZHAO Hong-xin2, WANG Sheng-hou3
(1.Coll.ofChem. &LifeSci.ShenyangNormalUni.,Shenyang110034; 2.Coll.ofLifeSci.ZhejiangSci-Tech.Uni.,Hangzhou310018; 3.Exper’lTeach.Ctr.,ShenyangNormalUni.,Shenyang110034)
In this study the alliance among threePleurotusofphoenixtail mushroom,xiaxiu990 andblackoyster mushroom A3 from different sources with different morphology were carried out contrast analysis. The results showed that the tested threePleurotusspecies possessed the congruity among species alliance definition in morphology level, protein level and molecular level. The alliance betweenphoenixtail mushroom andxiaxiu990 displayed closer, but further fromblackoyster mushroom A3, and the alliance betweenxiaxiu990 andblackoyster mushroom A3 was further. This study provided comprehensive methods to analyze the characterization of the alliance of edible fungi of inter-genus in different levels, and had directive significance to separate different name of the same species, to select hybrid parents, and to judge the results of hybridization.
Pleurotusostreatus; alliance; esterase isozyme; random amplified polymorphic DNA (RAPD); transcribed spacer sequence (ITS)
遼寧省科學事業公益項目(2014003020)
趙同心 女,碩士研究生。研究方向為微生物制藥。E-mail:txzhao@sina.com
* 通訊作者。男,教授,碩士生導師。主要從事藥用真菌資源的開發與利用。E-mail:wshhwq2009@163.comn
2015-06-01;
2015-08-03
Q93-331
A
1005-7021(2016)02-0062-05
10.3969/j.issn.1005-7021.2016.02.011

