基于鰓的microRNA轉錄組研究中國蛤蜊對重金屬鎘的響應
張晶晶 ,李宏俊 ,秦艷杰,劉敏 ,葉晟
(1. 國家海洋環境監測中心 海洋生態室,遼寧 大連 116023;2. 大連海洋大學 水產與生命學院,遼寧 大連 116023)
?
基于鰓的microRNA轉錄組研究中國蛤蜊對重金屬鎘的響應
張晶晶1,2,李宏俊1*,秦艷杰2,劉敏1,2,葉晟1,2
(1. 國家海洋環境監測中心 海洋生態室,遼寧 大連 116023;2. 大連海洋大學 水產與生命學院,遼寧 大連 116023)
中國蛤蜊(Mactrachinensis)是一種重要的經濟貝類,分布于我國的遼寧和山東。近年來,生境惡化和過度捕撈導致我國蛤蜊資源量減少。我們利用Illumina Hiseq-2500 平臺對中國蛤蜊鰓的microRNA轉錄組進行測序,運用差異表達尋找對鎘刺激有響應的功能microRNA。結果顯示對照組獲得了14 415 256條clean reads,實驗組獲得了15 570 111條clean reads。在這些reads中一共存在14 584 077小RNA,包括1 898 035條unique小RNA,其中對照組和實驗組共有187 859條unique小RNA。兩個組的小RNA文庫的片段長度分布是相似的,大部分小RNA長度分布在26~27 nt。在對照組文庫中最豐富的片段長度是27 nt,其次是28 nt、26 nt和23 nt。在實驗組文庫中,數量最豐富的片段長度是26 nt,其次是27 nt、28 nt和23 nt。經過對序列前體以及結構特征的分析,發現這兩個組中一共有50個microRNA,其中已知的是38個,新發現的是12個。對照組和實驗組microRNA含量最豐富的片段長度是23 nt。通過差異表達分析,5個microRNA基因具有顯著性差異且從對照組到實驗組的表達量是上調的,其余45個沒有顯著性的差異。把這50個序列與中國蛤蜊轉錄組進行比對,一共找到了542個靶基因。5個具有表達差異的基因一共比對到11個靶基因。把這11個靶基因與NCBI數據庫比對,4個靶基因在COG中得到注釋,1個靶基因在GO中得到注釋,6個靶基因在KEGG中得到注釋,11個靶基因在nr數據庫中得到注釋,注釋到的基因有泛素蛋白連接酶E3,Wnt信號通路,G蛋白信號調控等。
中國蛤蜊;鎘;microRNA;差異表達;功能基因
1 引言
中國蛤蜊(Mactrachinensis),又名飛蛤,屬軟體動物門,瓣鰓綱,簾蛤目,蛤蜊科,蛤蜊屬,生活在潮間帶中沙區的細砂灘,至水深60 m的淺海區,在我國分布于遼寧、山東,在日本、朝鮮也有分布[1]。中國蛤蜊肉味鮮美,出肉率高,是中國重要的經濟貝類。由于缺乏苗種,中國蛤蜊的灘涂養殖尚處于自然繁衍、保護資源的狀態[2]。近幾年來,海水的污染以及過度捕撈使中國蛤蜊野生資源缺乏,種質資源退化。
鎘(Cadmium)是一種具有金屬光澤的非典型過渡型重金屬,是一種非必須元素。它是一種生物蓄積性強、具有“三致”作用、毒性持久的劇毒元素,攝入過量的鎘對生物體的危害極其嚴重,會導致腎臟、肺部、肝臟、骨骼、生殖器官的損傷,對免疫系統、心血管系統等具有毒性效應,進而引發多種疾病。已有研究證明鎘對野生生物來說是一個高毒性的物質,并且能使人致畸致癌[3]。工業發展帶來了經濟的騰飛,但同時工業發展的廢棄物,如未經處理的或者處理不達標的包含有重金屬,有機化合物等的廢水等毀壞了中國東部海域的水質[4-5]。中國生態標準(2001—2005)記載黃、渤海鎘污染嚴重,2013年海洋環境質量公報報道遼東灣主要超標要素之一是鎘。雙殼貝類習固著型的濾食生活且自身的用于代謝的混合氧化系統存在缺陷,導致體內很容易富集環境中存在的重金屬離子[6-9]。貝類體內富集的鎘含量與環境中鎘離子的濃度呈正相關[10-11,13]。張傳永和孫振興[14]等通過鎘對中國蛤蜊的急性試驗研究表明鎘對中國蛤蜊的24 h、48 h、72 h、96 h的半致死濃度(LC50)分別為12.48 mg/L、7.06 mg/L、5.52 mg/L、4.22 mg/L;Cd2+對中國蛤蜊的安全質量濃度為0.04 mg/L。Cd2+對中國蛤蜊的急性毒性作用較強;在Cd2+的慢性毒性脅迫下,中國蛤蜊的鰓和消化盲囊的SOD活性受到了抑制。
MicroRNA又稱miRNA,是一類長度約為19~25 nt的內源性非編碼單鏈小分子RNA,屬于小RNA的一種,廣泛存在與真核生物中,主要以單拷貝、多拷貝或基因簇的形式存在,具有高度保守性、時序性和組織特異性[15-16]。MiRNA的生成始于核內,通過Micro-processor復合體(Drosha和DGCR8)剪切后生成單鏈RNA前體。然后在Exportin5-Ran-GTP蛋白出核轉運體的輔助下轉至胞質。在胞質中由Dicer酶剪切加工形成長度在19~25 nt的成熟的miRNA。起初,成熟的miRNA與其互補的序列結合成雙螺旋結構,隨后雙螺旋結構解旋,其中一條結合到RNA誘導的基因沉默復合物(RNA-induces Silencing Complex,RISC)上,該復合物以不完全配對的方式結合到靶mRNA的3`非編碼區(UTR)上,從而導致該基因被誘導或者抑制,甚至沉默。近年來也有研究發現,miRNA還可以與mRNA的5’UTR和氨基酸編碼區(Coding Sequences,CDS)結合[17]。1993年,Lee等[18]在對秀麗新小桿線蟲(Caenorhabditiselegan)進行突變體的遺傳分析時,發現了一種可時序調控胚胎后期發育且長度約為22 nt的小分子非編碼RNA——lin-4,但是這個發現在當時并沒有引起人們的關注;直至2000年,Reinhart[19]等又在線蟲(C.Elegan)中發現了let-7,miRNA才被科學界廣泛關注,let-7成為第二個被發現的miRNA基因。在過去的幾十年間,從不同的物種中鑒定出了大量的miRNA。據尹福強[20]介紹,從2002至2012年,miRNA的數量由215個增至25 141個,僅2011—2012年,測出miRNA的物種新增加了40個,說明miRNA已經受到人們的關注,成為熱門研究領域。
miRNA通常在轉錄后水平負調控其靶基因的表達,在機體生命活動的各個過程,比如細胞增殖和分化,凋亡疾病的發生發展等中,發揮著重要的調節作用[15]。盡管軟體動物作為海鮮在農業中有著它們重要的價值,但對它們的miRNA的研究并沒有如植物、昆蟲和重要動物寄生蟲的間接宿主研究的多[21]。在軟體動物中,長牡蠣(Crassostreagigas)[22]、泥蚶(Tegillarcagranosa)[23]和馬氏珠母貝(Pinctadamartensii)[24]等已經得到了miRNA的轉錄組并且得出一些與免疫反應或者抗性有關的miRNAs,以及得出miRNA可能啟動與之相關的反應,比如吞噬、凋亡、氧化還原反應,讓生物更好的適應環境和生存的結論。本文通過鎘離子刺激中國蛤蜊的急性毒性試驗以及運用Illumina Hiseq2500 SE50測中國蛤蜊鰓的miRNA轉錄組,期望通過對照組和實驗組的差異性表達分析,找到響應鎘離子刺激的miRNA,并且希望為尋找中國蛤蜊響應鎘離子刺激的抗性機制提供數據。
2 材料與方法
2.1 材料
2014年6月,于大連長興市場購買成熟的大小一致的中國蛤蜊,放置到4×100 L的水箱中暫養。7 d之后,對中國蛤蜊進行Cd2+急性毒性試驗,投毒試劑是CdCl2。100只中國蛤蜊平均分到2個水箱中,每個水箱代表一個處理,分別是空白對照組和實驗組,其濃度分別是0和2.76(1/2 LC50)mg/L。在養殖期間,每天投喂螺旋藻一次,換水兩次,溫度和鹽度分別控制在(20±1)℃和30。經過48 h處理,分別解剖對照組和實驗組的3只中國蛤蜊,獲得鰓并組內混合,分別命名為S01和S02,浸泡在RNA-EZ Reagents D RNA-Be-Locker A(購于上海生工),4℃過夜后,放于-80℃儲存,然后送北京百邁克測miRNA轉錄組。
2.2 小RNA文庫的構建,庫檢和深度測序
用Trizol試劑提取總的RNA,隨后用瓊脂糖電泳和NanoDrop測試檢測RNA的純度,Qubit2.0檢測RNA的濃度,Agilent 2100 Bioanalyzer 檢測RNA的數量。運用NEB Multiple Small RNA Library Pre Set Kit for Illumina 試劑盒構建小RNA文庫,質量比較高的總RNA被用作構建小RNA的原始材料。根據小RNA的序列末端的特征,其3’端被連接上與之配對的適用于Illumina測序的接頭,然后反轉錄得到第一條鏈;之后在序列的5’端加上另一個合適的接頭,以第一條鏈為模板反轉錄,完成了cDNA的合成。接下來用和接頭互補的引物,進行PCR擴增,獲得PCR產物;把產物通過6%的聚丙烯酰胺凝膠,分離目標基因,切膠,然后用Plastic回收試劑盒純化回收得到小RNA文庫。得到小RNA文庫之后,首先運用Qubit 2.0簡單的計算小RNA文庫的濃度,當濃度達到2 ng/μL,說明用Agilent 2100檢測得到的文庫中的插入序列的大小的結果是正確的。這時,為了確保文庫的質量,運用ECO實時熒光定量PCR精確的計算文庫中基因的數量(有效的文庫濃度大于2 nmol/L)。如果質量達標,就運用Illumina Hiseq2500 SE50對小RNA文庫進行測序。
2.3 測序數據的生物信息學分析
運用Illumina Hiseq2500 SE50測序之后需要對得到的小片段進行加工。首先是過濾掉低質量的片段、接頭序列、片段長度大于30 bp和小于18 bp的序列,以及序列片段中含有N的序列。這些clean片段與Rfam和GenBank數據庫比對,得到了ncRNA、rRNA、tRNA、snRNA、scRNA和snoRNA的注釋信息;與現有的中國蛤蜊基因組文庫進行比對獲得與miRNA有聯系的基因組序列。運用miRDeep 2,依據miRNA的生物學的特性并且結合其環狀結構的特征及預測,在獲得的基因組序列中鑒定總miRNA及其保守和新的miRNA。運用RNAhybrid[25]和miRanda[26]預測miRNA的靶基因。
2.4 MiRNA的差異表達分析和預測的靶基因的注釋
為了得出S01和S02的差異表達數據,DESeq[27]被用于篩選具有差異表達量的miRNA序列,篩選的條件是同時滿足假陽性率(FDR)小于0.01和fold change≥2,并且用Benjamini Hochber矯正過的P值校正FDR。Hierarchical cluster analysis被用于估算協同表達的趨勢和樣品之間miRNA的不同表達量。被預測到目的基因與NCBI常用數據庫比對得到注釋信息。
3 結果
3.1 小RNA的測序
運用Illumina Hiseq-2500 SE50平臺,一共得到34 590 000條原始reads,登錄號SRP075745,經過過濾得到29 990 000條clean reads,其中S01和S02分別得到14 415 256和15 570 111條clean reads(表1)。把中國蛤蜊的鰓的S01和S02的clean reads與中國蛤蜊mRNA轉錄組,Rfam和GenBank數據庫比對之后,分別發現了1 124 747和623 018條genome,594 070和377 193條rRNA,322和266條snRNA,27和58條snoRNA,5 682和6 408條tRNA,3 457和2 520條重復序列,其他的分別是S01和S02的獲得的小RNA的88.01%和93.52%(表2)。rRNA在S01和S02文庫中clean reads的百分比分別是4.12%和2.42%,說明這兩個文庫的質量是可靠的。經過拼接和比對,這兩個文庫一共得到14 584 077條小RNA,S01和S02共同擁有12 584 077條,S01特有1 306 643條,S02擁有772 379條(圖1)。聚類小RNA(Uniq小RNA)一共1 898 035條,S01和S02共同擁有187 859條,S01特有1 039 738條,S02特有670 438條(圖2)。這兩個文庫的小RNA的片段長度大部分分布在26~27 nt(表3),說明這兩個文庫的片段的長度分布是相似的。在S01文庫中最豐富的片段大小是在27 nt(173 373 條),其次是28 nt(137 879條),26 nt(135 169條)和23 nt(109 334條)。在S02文庫中,數量最豐富的片段長度是26 nt(93 571條),其次是27 nt(135 169條),28 nt(73 418條)和23 nt(73 099條)。經過與中國蛤蜊轉錄組文庫比對,S01文庫中產生1 124 747 clean 片段,S02文庫中產生623 018 clean片段。這兩個文庫的疑似miRNA片段長度分布在18~25 nt之間,S01和S02含量最豐富的片段是23 nt,分別是96 043條和64 491條,其次是22 nt(1 771和1 773條)和21 nt(1 133條和1 279條)。

表1 中國蛤蜊小RNA數據統計
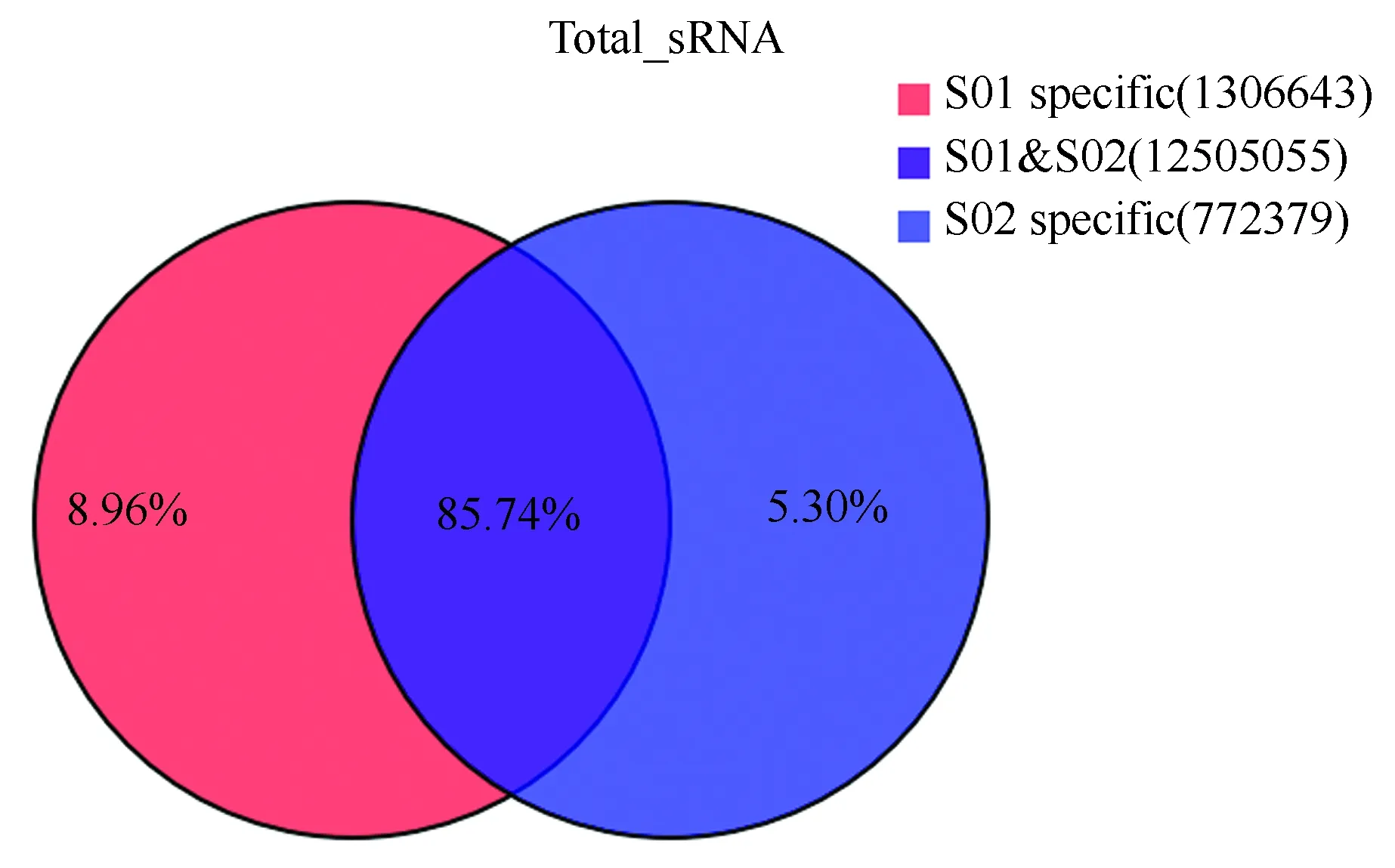
圖1 中國蛤蜊總的小RNA的韋恩分布圖Fig.1 Wayne figure of total small RNA distribution of the Chinese surf clam
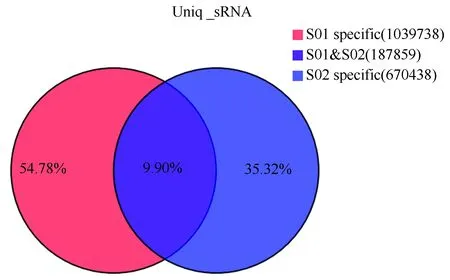
圖2 中國蛤蜊獨特的小RNA的維恩分布圖Fig.2 Wayne figure of unique small RNA distribution of the Chinese surf clam
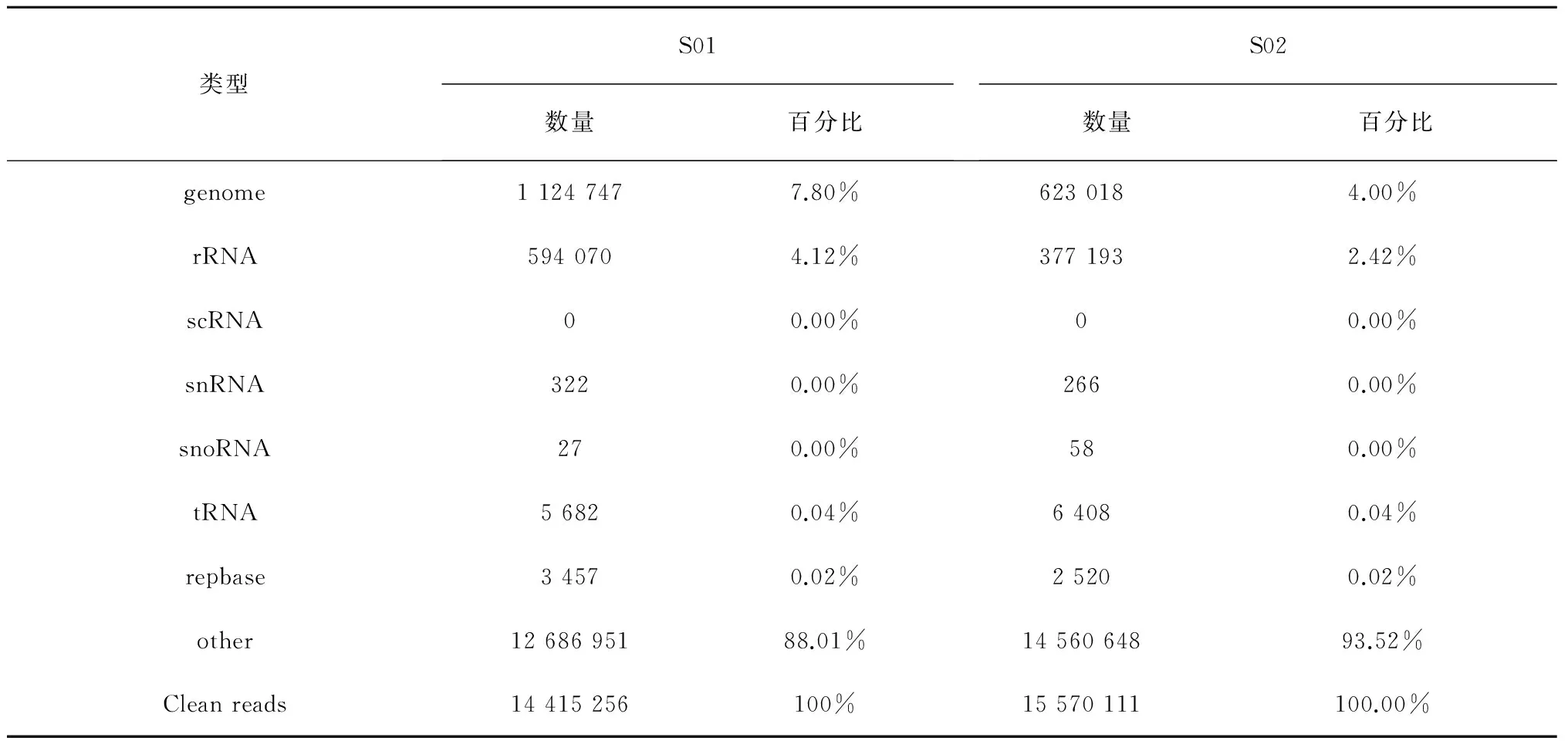
類型S01數量百分比S02數量百分比genome11247477.80%6230184.00%rRNA5940704.12%3771932.42%scRNA00.00%00.00%snRNA3220.00%2660.00%snoRNA270.00%580.00%tRNA56820.04%64080.04%repbase34570.02%25200.02%other1268695188.01%1456064893.52%Cleanreads14415256100%15570111100.00%
2.2 miRNA及其靶基因的確認
一共預測出了50個miRNA,包括38條保守的和12條新的miRNA。其中25條miRNA的長度是22 nt,14條miRNA的長度是21 nt,8條miRNA的長度是23 nt。這些新的miRNA被預測了前體結構,并且用miRDeep軟件進行估計前體的分值,圖3是一個新發現的miRNA預測的前體結構。在38條保守的序列中,其中的37條比對到了478條靶基因,12條新預測到的miRNA序列比對到了129條靶基因(表4)。
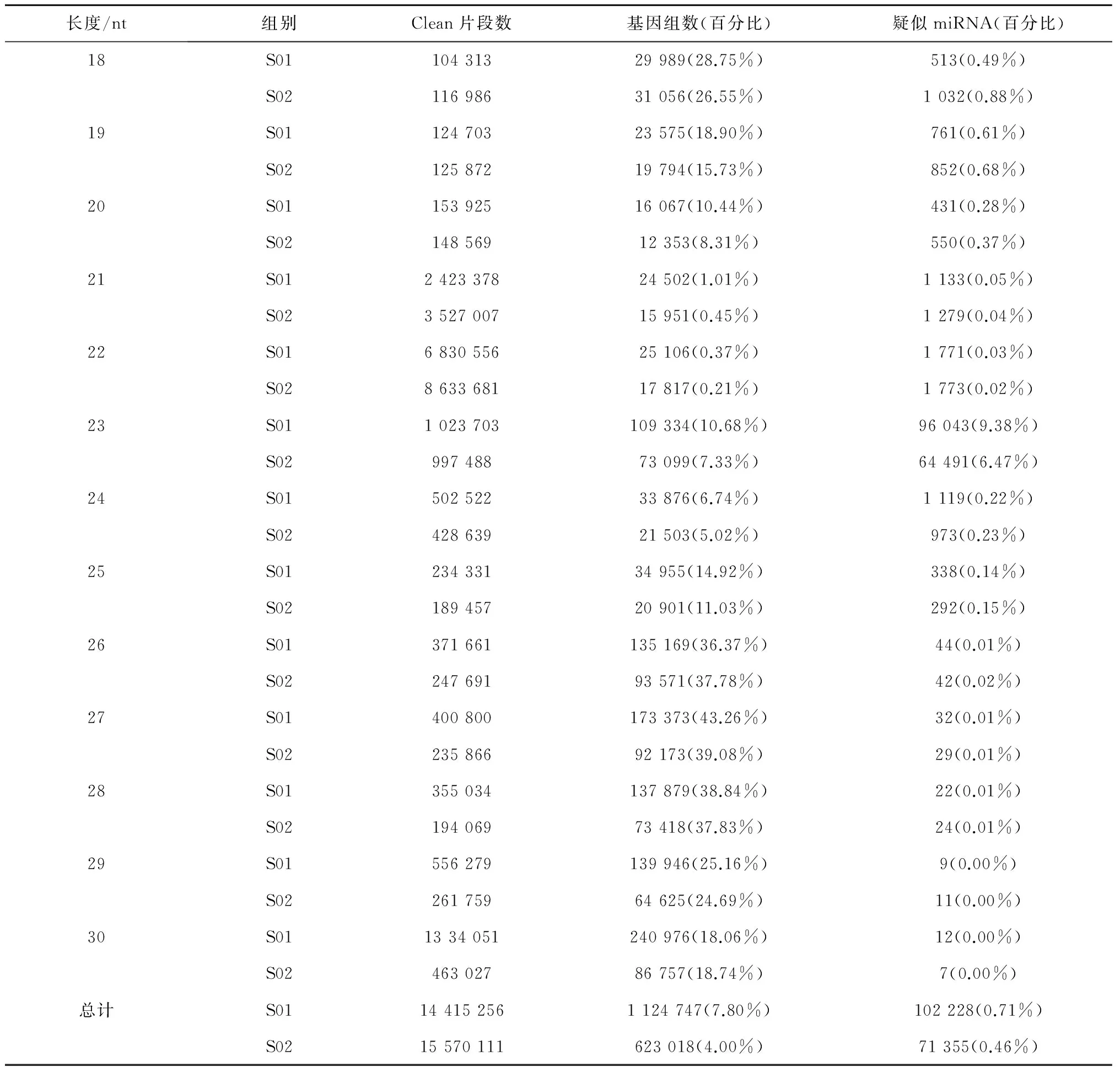
表3 疑似miRNA 長度統計表

圖3 新發現的miRNA預測的前體結構Fig.3 The predicted precursor structure of one novel miRNA
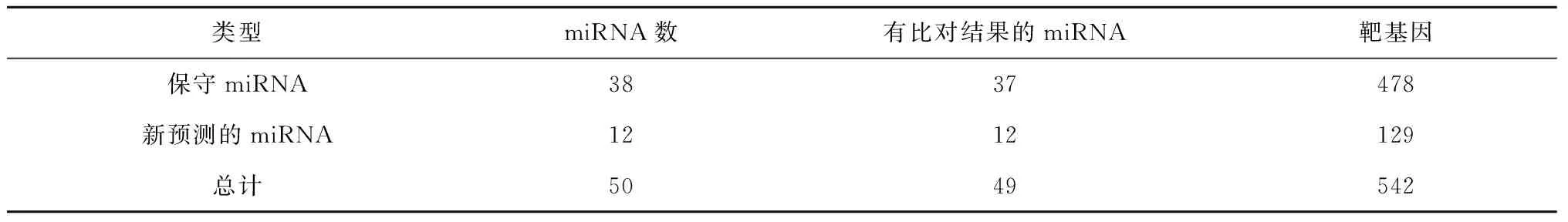
類型miRNA數有比對結果的miRNA靶基因保守miRNA3837478新預測的miRNA1212129總計5049542
2.3 miRNA的差異表達
運用EcoTM實時熒光定量PCR估計miRNA的表達量,在這50個miRNA中有5個差異表達基因,包括3個保守的miRNA基因(sme-miR-2202-5p,dme-miR-963-3p和 mmu-miR-216c-3p)和2個新的基因,并且這些基因的表達量是升高的(表5)。其余的45個基因中,17個表達趨勢是降低的,28個表達趨勢是升高的,但不符合FDR<0.01且fold change≥2(P<0)條件,即差異顯著性不明顯。圖4是篩選出的5個差異表達miRNA表達量的柱狀圖,從圖中可以看出S01的表達量高于S02的表達量。圖5是這兩個文庫表達水平的分析和展示,S02的log10rpkm的值高于S01,這也說明這5個基因在S02的表達量要高于S01的表達量。

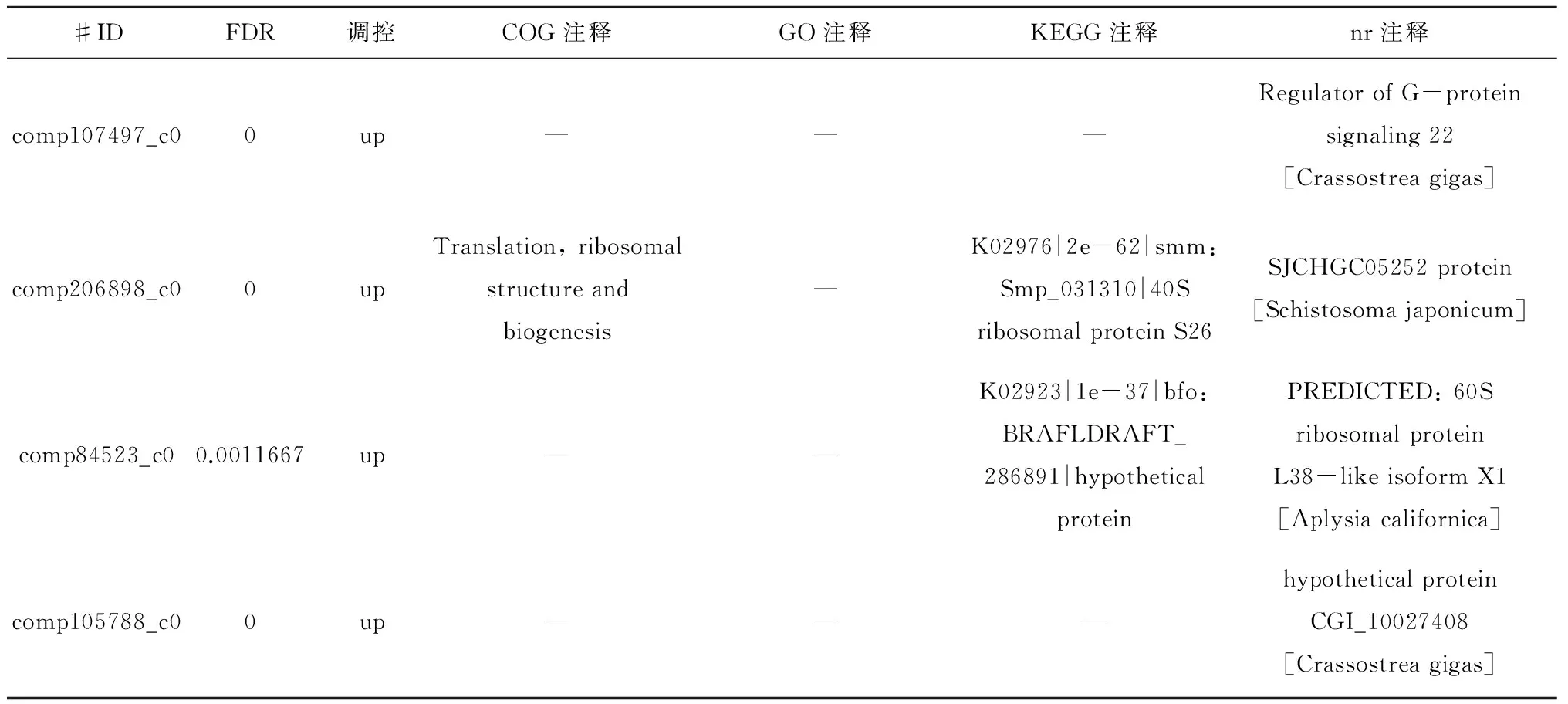
表5 5個顯著性差異的miRNA的靶基因的注釋
續表5

圖4 5個差異表達miRNA的柱狀圖Fig. 4 Expression profiles of the 5 differential expression miRNA
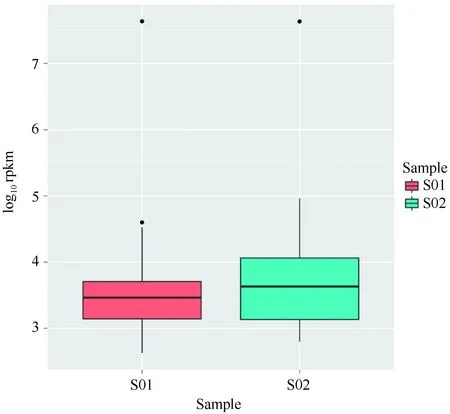
圖5 5個差異表達的基因在兩個文庫miRNA表達水平的分析與展示Fig.5 The analysis and show of the expression level of the 5 differential expression in two miRNA library
2.4 靶基因的注釋
使用BLAST軟件將預測得到的542個靶基因序列與NR、GO、COG、KEGG數據庫比對,獲得靶基因序列的注釋信息。最終獲得注釋信息的靶基因有91個。MiRNA靶基因注釋的統計結果見表6,在COG中注釋24條,GO中注釋23條,KEGG注釋34條,得到注釋的基因的長度在1 000 bp之上的有61條,這個數值是片段300~1 000 bp被注釋數量的2倍。其中具有顯著表達差異的基因的注釋統計結果如表6,在COG數據庫中比對到的靶基因數是4條,GO中1條,KEGG 5條,在nr數據庫比對到的一共是11條。

表6 所有miRNA靶基因注釋統計表

表7 注釋的差異miRNA靶基因數量統計
2.4.1 差異表達miRNA靶基因的GO分類
GO數據庫適用于各個物種,能對基因、蛋白質進行限定和描述。GO分析按照細胞組分、分子功能、生理過程對基因進行分類,從而展示出具有差異表達的與生物學功能顯著相關的miRNA靶基因。樣品S01與S02間差異表達miRNA靶基因GO分類統計結果見圖6。兩種不同的顏色代表所有靶基因以及差異表達靶基因的GO分類統計。差異表達miRNA分子功能中的catalytic activity、binding和生理過程的代謝過程,細胞過程,刺激響應以及信號的靶基因的富集趨勢比所有靶基因的高,尤其是刺激響應和信號轉導增高的趨勢更顯著,說明這些靶基因參與中國蛤蜊對鎘離子的刺激響應的過程。
2.4.2 差異表達miRNA靶基因的COG注釋
COG數據庫是基于細菌、藻類、真核生物的系統進化關系構建得到的,利用COG數據庫可以對基因產物進行直系同源分類。樣品S01與S02間差異表達miRNA靶基因COG分類統計結果見圖7。其中E、J、K、L、O、R、T對應的功能基因表現出差異表達,分別代表的是氨基酸的轉運和代謝,翻譯、核糖體結構和生物轉化,轉錄本,復制、重組和修復,后轉錄修復,蛋白質折疊和分子伴侶,通用功能預測和信號轉導代謝。
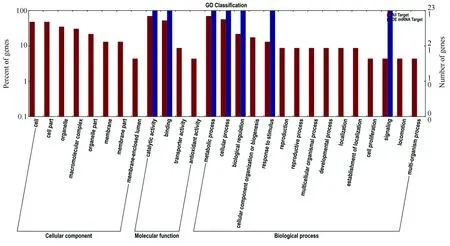
圖6 5個差異miRNA靶基因GO注釋聚類圖Fig.6 The GO annotation cluster of 5 differential expression
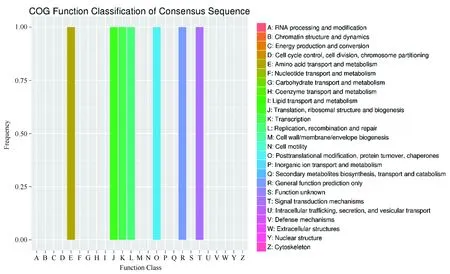
圖7 5個差異miRNA靶基因COG注釋分類圖Fig.7 COG annotation of the target genes of 5 differential expression
2.4.3 差異miRNA靶基因的KEGG注釋
在生物體內,通過不同基因相互協調來行使生物學功能。基于Pathway分析有助于進一步解讀基因的功能。KEGG是有關Pathway的主要公共數據庫。把5個具有顯著差異的miRNA的靶基因與KEGG數據庫進行比對,最后的數據總結如表8。在KEGG通路中一共比對到5條通路,分別是Lysosome,Wnt signaling pathway,Ribosome biogenesis in eukaryotes,Ribosome,Ubiquitin mediated proteolysis。
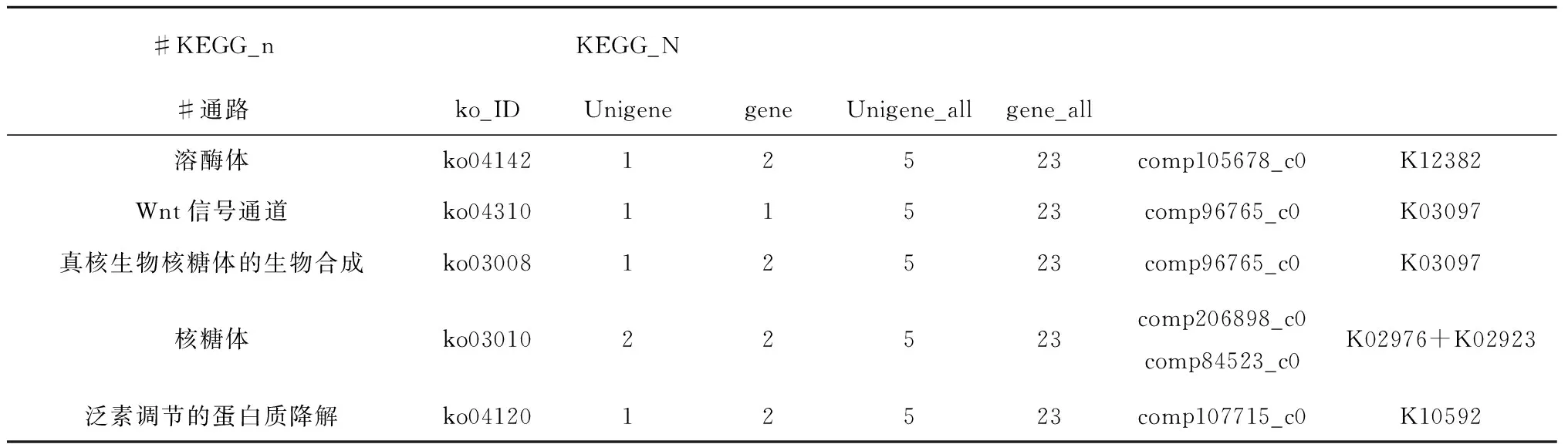
表8 5個差異性表達miRNA的靶基因的KEGG注釋
3 討論
MiRNA是一大類小的非編碼的RNA,它們通過使目標基因信使RNA的降解,抑制翻譯的方式阻止基因的表達。眾所周知,miRNA可以調節很多生理和病理的過程,比如宿主免疫反應和應激反應[28-29]。在本研究中,中國蛤蜊兩個處理組,經Illumina Hiseq2500平臺測序,一共得到了34.59 M原始reads。經過去掉低質量的序列,接頭和長度小于18 nt和大于30 nt的序列,獲得了29.99 M高質量的序列。去掉rRNA、scRNA、snRNA、snoRNA、tRNA等,得到疑似是miRNA的序列173 583條,其中S01有102 228,S02有71 355條,最后一共預測得到了50個miRNA。最豐富的miRNA序列的長度是22 nt(25條),其次是21 nt(14條),23 nt(8條),24 nt(2條)和20 nt(1條)。這些結果和海參(Apostichopusjaponicus)[30]、 長牡蠣(C.gigas)[22]、泥蚶(T.granosa)[23]和馬氏珠母貝(P.martensii)[24]基本一致。其他的研究認為同一大類物種中的最主要的miRNA的長度是一樣的,比如昆蟲Blattellagermanica[31]、Locustamigratoria[32]和Aedesalbopictus[33]的最優勢片段的大小是22 nt,植物Citrustrifoliate[34]、Cucumissativus[35]和Vitisvinifera[36]的最優勢的片段是24 nt。但是在貝類中,馬氏珠母貝(P.martensii)[24]的最優勢的片段長度是22 nt,泥蚶(T.granosa)[23]的是21nt,長牡蠣(C.gigas)[22]的是22 nt。
50個miRNA中38條可以注釋的序列稱為已知的miRNA,12條不能得到注釋稱為新的miRNA。中國蛤蜊鰓中的miRNA的數量與目前已得到miRNA的貝類相比較是最少的,長牡蠣(C.gigas)[22]血細胞中含有199條miRNA,包括71條已知的和128條新的;馬氏珠母貝(P.martensii)[24]中含有258條miRNA,包括205條已知的和53條新的;泥蚶(T.granosa)[23]血細胞中含有254條miRNA,包括215條已知的和39條新的,這些差異除了與物種本身所含有的miRNA有關之外,可能也與小RNA不同的測序深度有關。這12條新的基因可能是中國蛤蜊獨有的,因為在其他報道中并沒有與這些基因相同的序列。在所有的miRNA中,cel-miR-34-5p,sme-miR-2202-5p,sma-miR-8478-5p,mmu-miR-216c-3p是對照組和實驗組中排名前5的最豐富的基因,它們的高的表達量暗示他們在維持鰓的生理功能方面起到重要的作用[37]。但是,由NCBI小RNA數據庫得出miR-2202-5p僅存在于渦蟲中,cel-miR-34-5p存在于13種物種中,miR-8478-5p僅存在于曼氏裂體吸蟲中,miR-216c-3p僅存在于小鼠中。有趣的是,中國蛤蜊的miRNA基因有6個miRNA至少有一個前體,暗示它們可以從不同的位點進行轉化并且有很多的啟動子,這就賦予他們在多樣條件下的轉錄水平中有更多的調控機制,并且有更多的機會參與生理學功能的調節[38]。
MiRNAs調控已經被認為在脊椎動物的免疫和適應階段是一個重要的調控原則[39]。為了檢測miRNA在中國蛤蜊免疫反應中的潛在的作用,經過鎘離子刺激之后,估算了鰓中經過鎘離子極性毒理刺激之后的miRNA的表達水平。在50個miRNA中有18個表達水平是降低的,32個是升高的。其中有5個miRNA具有顯著差異,并且都是上調的,說明這5個miRNA在中國蛤蜊抵御鎘離子反應的過程中起到作用,猜測他們可能在免疫反應中起到重要的調控的作用[40- 41]。
為了描述miRNA和靶基因在鎘離子刺激下的作用網絡圖以及獲得差異表達miRNA的更深刻的生物學意義,我們把與其對應的靶基因與GO、KEGG、COG、nr數據庫進行比對,分析它們潛在的功能。50個miRNA在COG數據庫中比對到了7條具有顯著性差異的基因功能束。說明在鎘離子刺激下,中國蛤蜊的這些結構和功能基因發生響應。這7條基因功能束分別代表氨基酸的轉運和代謝,翻譯、核糖體結構和生物轉化,轉錄本,復制、重組和修復,后轉錄修飾,蛋白質折疊和分子伴侶,通用功能預測和信號轉導代謝。1個miRNA對應的基因在GO數據庫中得到了注釋,注釋為分子功能:protein serine/threonine kinase activity(GO:0004674)和ATP binding(GO:0005524)以及生理過程:protein phosphorylation(GO:0006468)和Wnt signaling pathway(GO:0016055),這個基因在nr數據庫中對應的是hypothetical protein CAPTEDRAFT_162678基因,可以猜測這個基因在GO注釋的這些功能中都起到了作用。Wnt信號通路是參與胚胎及器官發育的主要4大類信號傳導途徑之一,對胚胎及器官發育起著不可替代的作用,此外還參與一些疾病,比如腫瘤,心血管疾病和肝纖維化的調控[42]。絲氨酸/蘇氨酸蛋白磷酸酶,是由催化亞基C、結構亞基A和多種調控B亞基組成的復合物。蛋白磷酸酶2A(protein phosphatase 2A, PP2A)是真核生物中最主要的絲氨酸/蘇氨酸蛋白磷酸酶。PP2A在細胞信號轉導通路包括DNA損傷修復、細胞周期進程、轉錄激活、細胞轉化、細胞凋亡等過程中起著很重要的作用。研究發現PP2A活性下降導致細胞對重金屬的毒性敏感性增強,并證實PP2A通過介導AMP-激活蛋白激酶α(AMPKα)的去磷酸化來調控熱休克蛋白(heat shock proteins,HSP)中HSP70和HSP27的表達并參與細胞應激反應。通過熒光定量發現,hypothetical protein CAPTEDRAFT_162678表達量是增加的,說明這個基因在抵御鎘離子的刺激中起到重要的作用。這個基因所在的這些通路的相互作用形成中國蛤蜊抵御鎘離子刺激的調控網絡圖。
5個基因注釋到KEGG,包括泛素和溶酶體等。泛素-蛋白酶體途徑(Ubiquitin—proteasome pathway)和溶酶體途徑是真核細胞內的蛋白質主要的蛋白酶解降解系統。通常情況下,細胞內膜相關蛋白和某些在應激狀態下產生的蛋白質以及通過內吞過程從胞外攝取的蛋白質等主要經溶酶體降解;而泛素—蛋白酶體途徑則高選擇性地降解細胞在應激和非應激條件下產生的異常蛋白質,對維持細胞正常生理功能具有十分重要的意義。
泛素是一個由76個氨基酸殘基組成的高度保守的多肽,因其廣泛分布于各類細胞中而得名,被泛素標記的蛋白質將被特異性地識別并被迅速降解.細胞中蛋白質的降解是一個復雜的、被嚴密調控的過程,此過程在涉及細胞基本進程(如DNA修復,細胞周期調控,細胞凋亡等),抗原呈遞,炎癥反應等一系列通路中扮演重要角色[43].泛素連接酶E3是泛素化過程中的關鍵酶之一,介導活化的泛素從結合酶E2轉移到底物,不同的泛素連接酶作用于不同的底物蛋白,決定了泛素化修飾的特異性。
在nr數據庫中比對到了G蛋白信號調控蛋白(RGS),RGS負責蛋白質的快速避開G protein-coupled受體信號通路的主要機制。RGS蛋白通過GTPase蛋白激活RGS區域對G蛋白進行負調控。分子伴侶是在生物大分子的折疊、組裝、轉運及降解等過程中起協助作用,參與協助抗原的呈遞和遺傳物質的復制、轉錄及構象的確立,但自身并不發生任何變化的一大類廣泛存在于生物體內的蛋白質分子。總之,這5個具有顯著差異的基因在中國蛤蜊抵御鎘離子刺激的作用下都能起到重要的作用。
[1] 齊鐘彥. 中國經濟軟體動物[M]. 北京:中國農業出版社, 1998.
Qi Zhongyan. Economic Mollusca of China[M]. Beijing:China Agriculture Press, 1998.
[2] 王海濤, 王浩, 蘇海巖, 等. 中國蛤蜊生態養殖技術[J]. 科學養魚, 2013(1):45-46.
Wang Haitao, Wang Hao, Su Haiyan, et al. Ecological breeding technology ofMactrachinensis[J]. Scientific Fish Farming, 2013(1):45-46.
[3] Ranald P, Black B. Privatising water in the driest state:The impacts on employees and industrial relations of the corporatisation and outsourcing of metropolitan water and sewerage services in south Australia[J]. Labour & Industry:a Journal of the Social and Economic Relations of Work, 2000, 11(2):17-38.
[4] Wang Shuailong, Xu Xiangrong, Sun Yuxin, et al. Heavy metal pollution in coastal areas of South China:a review[J]. Marine Pollution Bulletin, 2013, 76(1/2):7-15.
[5] Wu Zhihao, He Mengchang, Lin Chunye. Environmental impacts of heavy metals (Co, Cu, Pb, Zn) in surcial sediments of estuary in Daliao River and YingKou Bay (Northeast China):concentration level and chemical fraction[J]. Environmental Earth Sciences, 2012, 66(8):2417-2430.
[6] Roesijadi G. Metallothioneins in metal regulation and toxicity in aquatic animals[J]. Aquatic Toxicology, 1992, 22(2):81-113.
[7] Engel D W, Brouwer M. Trace metal-binding proteins in marine molluscs and crustaceans[J]. Marine Environmental Research, 1984, 13(3):177-194.
[8] 劉明星, 李國基, 顧宏堪. 渤海魚類、甲殼動物、軟體動物的痕量金屬含量[J]. 環境科學學報, 1983, 3(2):149-155.
Liu Mingxing, Li Guoji, Gu Hongkan. Trace metal concentration in fish, crestacea and mollusea of the Bohai Bay[J]. Acta Scientiae Circumstantiae, 1983, 3(2):149-155.
[9] 蔡立哲, 劉瓊玉, 洪華生. 菲律賓蛤仔在高濃度鋅鉛水體中的金屬積累[J]. 臺灣海峽, 1998, 17(4):456-461.
Cai Lizhe, Liu Qiongyu, Hong Huasheng. Metal-accumulation ofRuditapesphilippinarumin high zinc and lead experimental waters[J]. Journal of Oceanography in Taiwan Strait, 1998, 17(4):456-461.
[10] Amiard-Triquet C, Berthet B, Metayer C, et al. Contribution to the ecotoxicological study of cadmium, copper and zinc in the musselMytilusedulis[J]. Marine Biology, 1986, 92(1):7-13.
[11] 王曉麗, 孫耀, 張少娜, 等. 牡蠣對重金屬生物富集動力學特性研究[J]. 生態學報, 2004, 24(5):1086-1090.
Wang Xiaoli, Sun Yao, Zhang Shaona, et al. Experiment researches on the kinetic characteristics of bioconcentration of heavy metals inO.gigasThunberg[J]. Acta Ecologica Sinica, 2004, 24(5):1086-1090.
[12] 陸超華, 謝文造, 周國君. 近江牡蠣作為海洋重金屬鎘污染指示生物的研究[J]. 中國水產科學, 1998, 5(2):79-83.
Lu Chaohua, Xie Wenzao, Zhou Guojun. Studies onCrassostrearivularisas a biological indicator of cadmium pollution[J]. Journal of Fishery Sciences of China, 1998, 5(2):79-83.
[13] 陸慧賢, 徐永健. 縊蟶(Sinonovaculaconstricta)對海水中鎘富集規律的研究[J]. 生態科學, 2013, 32(4):434-438.
Lu Huixian, Xu Yongjian. Study on cadmium bioaccumulation inSinonovaculaconstricta[J]. Ecological Science, 2013, 32(4):434-438.
[14] 張傳永, 孫振興. Cd2+對中國蛤蜊的急性毒性及SOD活性的影響[J]. 安徽農業科學, 2010, 38(1):193-195.
Zhang Chuanyong, Sun Zhenxing. Effect of Cd2+on SOD activity and the acute toxicity of clamMactrachinensis[J]. Journal of Anhui Agricul Agricultural Science, 2010, 38(1):193-195.
[15] 李潔, 秦性良, 邵寧生. MicroRNA及其靶基因的時空特異性與動態變化[J]. 生物化學與生物物理進展, 2013, 40(7):617-626.
Li Jie, Qin Xingliang, Shao Ningsheng. Time and space specificity and dynamic change of microRNA and its targets[J]. Progress in Biochemistry and Biophysics, 2013, 40(7):617-626.
[16] 金吉春. MicroRNA的概述及其研究[J]. 醫學研究生學報, 2013, 26(10):1109-1112.
Jin Jichun. Overview and research of microRNA[J]. Journal of Medical Postgraduates, 2013, 26(10):1109-1112.
[17] Tay Y, Zhang J Q, Thomson A M, et al. MicroRNAs to Nanog, Oct4 and Sox2 coding regions modulate embryonic stem cell differentiation[J]. Nature, 2008, 455(7216):1124-1128.
[18] Lee R C, Feinbaum R L, Ambros V. TheC.elegansheterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5):843-854.
[19] Reinhart B J, Slack F J, Basson M, et al. The 21-nucleotide let-7 RNA regulates developmental timing inCaenorhabditiselegans[J]. Nature, 2000, 403(6772):901-906.
[20] 尹福強. 煙草苗期干旱脅迫誘導根系mRNA和miRNA快速響應機理研究[D]. 雅安:四川農業大學, 2013.
Yin Fuqiang. Characterize of roles of mRNAs and miRNAs in responding to drought stress in tobacco seedling roots[D]. Ya’an:Sichuan Agriculture University, 2013.
[21] Owens L, Malham S. Review of the RNA interference pathway in molluscs including some possibilities for use in bivalves in aquaculture[J]. Journal of Marine Science and Engineering, 2015, 3(1):87-99.
[22] Zhou Zhi, Wang Lingling, Song Linsheng, et al. The identification and characteristics of immune-related microRNAs in haemocytes of oysterCrassostreagigas[J]. PLoS One, 2014, 9(2):e88397.
[23] Bao Yongbo, Zhang Lili, Dong Yinghui, et al. Identification and comparative analysis of theTegillarcagranosahaemocytes microRNA transcriptome in response to Cd using a deep sequencing approach[J]. PLoS One, 2014, 9(4):e93619.
[24] Jiao Yu, Zheng Zhe, Du Xiaodong, et al. Identification and characterization of microRNAs in pearl oysterPinctadamartensiiby solexa deep sequencing[J]. Marine Biotechnology, 2014, 16(1):54-62.
[25] Rehmsmeier M, Steffen P, H?chsmann M, et al. Fast and effective prediction of microRNA/target duplexes[J]. RNA, 2004, 10(10):1507-1517.
[26] Enright A J, John B, Gaul U, et al. MicroRNA targets inDrosophila[J]. Genome Biology, 2004, 5(1):R1.
[27] Anders S, Huber W. Differential expression analysis for sequence count data[J]. Genome Biology, 2010, 11(10):R106.
[28] Izar B, Mannala G K, Mraheil M A, et al. MicroRNA response to listeria monocytogenes infection in epithelial cells[J]. International Journal of Molecular Sciences, 2012, 13(1):1173-1185.
[29] O’Connell R M, Rao D S, Baltimore D. MicroRNA regulation of inflammatory responses[J]. Annual Review Immunology, 2012, 30:295-312.
[30] Li Chenghua, Feng Weida, Qiu Lihua, et al. Characterization of skin ulceration syndrome associated microRNAs in sea cucumberApostichopusjaponicusby deep sequencing[J]. Fish & Shellfish Immunology, 2012, 33(2):436-441.
[31] Cristino A S, Tanaka E D, Rubio M, et al. Deep sequencing of organ-and stage-specific microRNAs in the evolutionarily basal insectBlattellagermanica(L.) (Dictyoptera,Blattellidae)[J]. PLoS One, 2011, 6(4):e19350.
[32] Wei Yuanyuan, Chen Shuang, Yang Pengcheng, et al. Characterization and comparative profiling of the small RNA transcriptomes in two phases of locust[J]. Genome Biology, 2009, 10(1):R6.
[33] Skalsky R L, Vanlandingham D L, Scholle F, et al. Identification of microRNAs expressed in two mosquito vectors,AedesalbopictusandCulexquinquefasciatus[J]. BMC Genomics, 2010, 11:119.
[34] Song Changnian, Wang Chen, Zhang Changqing, et al. Deep sequencing discovery of novel and conserved microRNAs in trifoliate orange (Citrustrifoliata)[J]. BMC Genomics, 2010, 11:431.
[35] Martínez G, Forment J, Llave C, et al. High-throughput sequencing, characterization and detection of new and conserved cucumber miRNAs[J]. PLoS One, 2011, 6(5):e19523.
[36] Wang Chen, Wang Xicheng, Kibet N K, et al. Deep sequencing of grapevine flower and berry short RNA library for discovery of novel microRNAs and validation of precise sequences of grapevine microRNAs deposited in MiRBase[J]. Physiologia Plantarum, 2011, 143(1):64-81.
[37] Christodoulou F, Raible F, Tomer R, et al. Ancient animal microRNAs and the evolution of tissue identity[J]. Nature, 2010, 463(7284):1084-1088.
[38] Starega-Roslan J, Koscianska E, Kozlowski P, et al. The role of the precursor structure in the biogenesis of microRNA[J]. Cellular and Molecular Life Sciences, 2011, 68(17):2859-2871.
[39] Leung A K L, Sharp P A. MicroRNA functions in stress responses[J]. Molecular Cell, 2010, 40(2):205-215.
[40] Schmidt W M, Spiel A O, Jilma B, et al.Invivoprofile of the human leukocyte microRNA response to endotoxemia[J]. Biochemical and Biophysical Research Communications, 2009, 380(3):437-441.
[41] Chen Youhai. MicroRNA immunobiology:When microRNA chemists meet immunologists[J]. Cell & Molecular Immunology, 2011, 8(5):369-370.
[42] 韓萍萍, 鄭若男. Wnt信號通路及其與疾病的關系[J]. 生物技術通報, 2009(11):13-15.
Han Pingping, Zheng Ruonan. Wnt signal pathway and its role in disease[J]. Biotechnology Bulletin, 2009(11):13-15.
[43] Pickart C M. Back to the future with ubiquitin[J]. Cell, 2004, 116(2):181-190.
Identification and differential expression of gill microRNA in the Chinese surf clam (Mactra chinensis) with Cd2+exposure
Zhang Jingjing1,2, Li Hongjun1, Qin Yanjie2, Liu Min1,2, Ye Sheng1,2
(1.MarineEcologyDepartment,NationalMarineEnvironmentalMonitoringCenter,Dalian116023,China; 2.CollegeofFisheriesandLifeScience,DalianOceanUniversity,Dalian116023,China)
The Chinese surf clam (Mactrachinensis) is an economically important clam, distributed in Liaoning Province and Shandong Province. In recently years, because of coastal environmental deterioration and overfishing, the natural population ofM.chinensishave considerably declined. In this paper, we studied the microRNA transcriptome of gills, and control and experimental group were sequenced through Illumina Hiseq 2500 CE. The differential expression andlysis was used to find the functional microRNA response to the Cd2+exposure. Through Illumina Hi-seq 2500 CE, a total of 14 415 256 clean reads and 15 570 111 clean reads were yielded in the gill of control and experimental group respectively. A total of 14 584 077 small RNA, including 187 859 unique small RNA were yielded. The distribution of the small RNA length in the two library was similar, most of them were 26-27 nt. 27 nt was the most abundant length in control group, followded by 28 nt, 26 nt, and 23 nt; 26 nt was the most abundant length in experimental group, and followed by 27 nt, 28 nt and 23 nt. 50 microRNA was found in unique small RNA, including 38 conserved and 12 novel genes. The most abundant length of microRNA in the two library was the same, 23 nt. Through the analyze of differential expression analysis, the expression of 5 microRNA was induced with significantly difference, other 45 microRNA was regulated up or down without significantly difference. 542 target genes were yielded when the 50 microRNA were hit to mRNA genome. And the target genes of differential expression microRNA were annotated by hitting to the NCBI database, and 4 genes hit to the COG, 1 genes hit to the GO, 5 genes hit to the KEGG and 11 genes hit to the nr database. The genes hit to the NCBI database included E3 ubiquitin-protein ligase, Wnt signaling pathway and Regulator of G-protein signaling 22.
Mactrachinensis; cadmium ion; microRNA; differential expression; functional genes
2016-06-30;
2016-09-15。
國家自然科學基金(31572595);國家海洋局近岸海域生態環境重點實驗室開放基金(201511)。
張晶晶(1989— ),女,河南省周口市人,研究方向為分子生態學。E-mail:liutian.3090@163.com
*通信作者:李宏俊,博士,副研究員,研究方向為分子生態學。E-mail:hjli@nmemc.org.cn
10.3969/j.issn.0253-4193.2016.12.012
S917.4
A
0253-4193(2016)12-0118-14
張晶晶,李宏俊,秦艷杰,等. 基于鰓的microRNA轉錄組研究中國蛤蜊對重金屬鎘的響應[J].海洋學報,2016,38(12):118—131,
Zhang Jingjing, Li Hongjun, Qin Yanjie, et al. Identification and differential expression of gill microRNA in the Chinese surf clam (Mactrachinensis) with Cd2+exposure[J]. Haiyang Xuebao,2016,38(12):118—131, doi:10.3969/j.issn.0253-4193.2016.12.012

