人源肺細胞cDNA文庫構建及與流感病毒NP 互作宿主蛋白的篩選
羅維玉,朱鵬陽,張 杰,胡永浩,孔暉暉,梁立濱,周 圓,李呈軍,姜 麗,陳化蘭
?
人源肺細胞cDNA文庫構建及與流感病毒NP 互作宿主蛋白的篩選
羅維玉1,2,朱鵬陽2,張 杰2,胡永浩1,孔暉暉2,梁立濱2,周 圓2,李呈軍2,姜 麗2,陳化蘭1,2
(1甘肅農業大學動物醫學院,蘭州730070;2中國農業科學院哈爾濱獸醫研究所獸醫生物技術國家重點實驗室/農業部動物流感重點開放實驗室,哈爾濱150001)
【目的】構建肺組織細胞Calu-3及A549的高質量酵母cDNA文庫并篩選與流感病毒NP蛋白相互作用的宿主因子,為深入研究流感病毒NP蛋白功能、病毒復制及致病機制奠定基礎。【方法】提取等量Calu-3及A549細胞的總RNA,混合后反轉錄生成cDNA,利用長距離PCR(LD-PCR)擴增合成dscDNA,用CHROMA SPINTM+TE-400 純化柱純化dsDNA,按照Clontech公司的Make Your Own“Mate &Plate”Library System操作程序,將帶有同源臂的dscDNA與線性化pGADT7-Rec共同轉化Y187酵母感受態細胞,涂布SD/-Leu平板后于30℃培養4d左右,收集所有菌落,混勻分裝即為Calu-3和A549 細胞cDNA的酵母文庫,并對文庫庫容、滴度及多樣性進行分析。利用RⅠ和HⅠ雙酶切將A/Auhui/2/2005(H5N1)NP定向插入pGBKT7載體,構建高致病性流感病毒NP的誘餌質粒pGBKT7-NP,經驗證該質粒無自激活活性,并進一步采用構建完成的Calu-3/A549 細胞酵母文庫進行雜交篩選,篩選得到的正確閱讀的獵物質粒與誘餌質粒共轉Y2H Gold酵母菌,分別以BD-P53/AD-T7作為陽性對照和BD-Lam/AD-T7作為陰性對照,挑取最終在SD/-Trp/-Leu/-Ade/-His/X-α -gal/AroA(SD/-4/X/A) 固體培養板上生長良好且變藍的菌落,即為候選與目標蛋白互作陽性的蛋白,提取酵母質粒,進行測序分析、序列比對和Gene Ontology分析。【結果】提取兩種細胞RNA 28S與18S條帶清晰,5S條帶暗淡,表明所提RNA質量較高,基本無降解;對提取的RNA反轉錄純化獲得dscDNA,dscDNA條帶呈彌散狀,片段大小分布于500—2 000bp之間,說明不同豐度及大小的RNA均成功反轉錄;構建的dscDNA文庫庫容為1.5×107,滴度為2.2×108cfu/mL,重組率為88%,PCR鑒定文庫插入片段,條帶大小不一、多樣性好;利用誘餌質粒與文庫進行雙雜交篩選,回交驗證后得到11個與NP蛋白互作的宿主因子。經Gene Ontology分析顯示,11個宿主因子參與的生物過程包括:細胞凋亡、胚胎發育、可變剪接、轉錄調節及細胞增殖等;涉及的分子功能包括:GTP結合活性、金屬離子結合活性、DNA結合活性及轉錄因子活性。【結論】成功構建同時含有Calu-3和A549兩種人源肺細胞cDNA的酵母文庫,文庫覆蓋cDNA更全面,為后期篩選與流感病毒其它蛋白互作的宿主蛋白奠定基礎,篩選得到與NP蛋白存在相互作用的宿主因子為進一步深入研究NP功能提供了可靠的前期數據。
流感病毒;NP蛋白;cDNA文庫構建;酵母雙雜交系統;宿主因子
0 引言
【研究意義】流感病毒是重要的人獸共患傳染病病原,可以在不同宿主間循環存在,不斷發生突變及重組,嚴重威脅人類的生命和健康,并對經濟造成沉重負擔[1]。酵母雙雜交技術在篩選與研究與已知蛋白相互作用的未知蛋白方面得到了廣泛的應用,利用該技術構建覆蓋面廣的酵母雙雜交cDNA文庫,并鑒定與流感病毒蛋白發生直接相互作用的宿主蛋白,對深入理解流感病毒的復制和致病機制,創新抗流感病毒藥物和療法具有重要意義。【前人研究進展】A型流感病毒的基因組由8條單股負鏈RNA組成,已經報道可編碼多達18種蛋白[2],根據表面蛋白血凝素(HA)和神經氨酸酶(NA)抗原性的不同分為18個HA亞型和11個NA亞型[3]。流感病毒的基因組極易發生變異,一方面,病毒基因組的分節段特性使得不同病毒在感染同一細胞時會發生病毒間基因片段的交換而產生新的基因重組病毒[4],另一方面,由于病毒的RNA聚合酶缺乏校正功能導致病毒在復制的過程中很容易發生基因突變[5]。流感病毒的高突變率和易于發生基因重組的特性導致流感病毒的抗原變異頻繁,為以疫苗免疫為主的防控措施帶來嚴重挑戰。藥物治療是除了疫苗以外的另一種抗流感病毒策略。目前,針對流感病毒NA蛋白設計的藥物奧塞米韋和扎那米韋在臨床治療方面發揮了顯著的作用[6-7]。但在用藥過程中,病毒的神經氨酸酶基因可能會發生突變而產生耐藥性毒株,導致藥物失去治療效果[8-9]。為此,需要加強流感病毒的復制周期調控機制研究,揭示流感病毒生命周期中所依賴的宿主蛋白,為抗流感藥物的研發提供新思路和潛在的靶標。截至目前,利用不同方法,已經發現了多種與流感病毒蛋白互作而影響病毒復制的宿主蛋白[10-11]。對于NP蛋白而言,它在病毒RNA合成、復制復合體運輸和子代病毒包裝等過程中發揮關鍵作用。NP蛋白作為流感病毒主要的結構蛋白,是組成病毒核糖核蛋白復合體vRNP的成分[12],在整個流感病毒生命周期發揮重要的作用,參與vRNP的入核[13]、病毒基因組的轉錄與復制、與M1蛋白和NEP蛋白一起介導vRNP出核[13-15]。而且,NP蛋白在不同亞型流感病毒中非常保守[16],可以作為抗流感藥物的良好靶標。因此,與流感病毒NP蛋白互作宿主因子的研究也就自然成為一個研究熱點。目前,利用不同方法發現的與NP蛋白互作的宿主蛋白包括有:DDX3[17]、MOV10[18]、importin α[19]、CRM1[20]、UAP56[21]和Tat-SF1[22]等。【本研究切入點】鑒于NP蛋白是流感病毒粒子中除M1蛋白以外含量最高的病毒蛋白且在病毒復制周期的多個環節發揮重要作用,挖掘與流感病毒NP蛋白互作的宿主蛋白具有重要意義。因此,本研究以流感病毒的NP蛋白為研究對象,力圖發掘病毒復制周期中與NP蛋白互作的新的宿主因子。【擬解決的關鍵問題】系統深入的了解與NP作用的宿主蛋白,需要構建覆蓋基因更全面的文庫。為此,本研究成功構建了同時含有肺泡上皮細胞A549和肺腺癌細胞Calu-3兩種肺臟細胞類型的酵母雙雜交cDNA文庫,并利用酵母雙雜交系統篩選與流感病毒NP蛋白互作的宿主因子,為研究流感病毒復制和致病機制及設計針對NP和宿主因子的抗流感藥物奠定基礎。
1 材料與方法
試驗于2015年在中國農業科學院哈爾濱獸醫研究所完成。
1.1 主要試劑
Rneasy Plus Mini kit RNA提取試劑盒購自QIAGEN公司;酵母質粒小提取試劑盒購自碧云天生物公司;Ex Taq DNA聚合酶購自TaKaRa公司;膠回收試劑盒購自AXYGEN 公司;酵母文庫構建試劑盒、各種酵母培養基及Aureobasidin A和X-α-gal均購自Clontech公司。
1.2 RNA提取及第一鏈cDNA合成
采用RNeasy Plus Mini kit試劑盒,按說明書步驟提取等量A549細胞和Calu-3細胞總RNA。在無RNA酶的1.5 mL離心管中分別加入2 μg新提取的細胞總RNA和1 μmol·L-1primer,用RNase-free H2O補至總體積4 μL;72℃水浴熱激2 min,冰上冷卻2 min,13 000×g瞬時離心10 s;加入2 μL 5×First-Strand Buffer、1 μL 100 mmol·L-1DTT、1 μL 10 mmol·L-1dNTP、1 μL SMART MMLV,混勻,42℃水浴10 min;加入1 μmol·L-1SMART III-modified oligo,混勻后42℃繼續孵育1 h,然后75℃ 10 min 終止反應;室溫冷卻后,加入1 μL RNase H(2 個活性單位),37℃孵育20 min。
1.3 dscDNA 的長距離PCR(LD-PCR)擴增合成及其純化
按照表1加入cDNA模板和各種反應試劑。按照以下擴增程序:95℃預變性30s;95℃變性10 s,68℃ 6 mina,26個循環(a表示每增加一個循環延伸時間都要增加5 s);68℃延伸5min,反應結束后4℃保存。最后,用CHROMA SPINTM+TE-400 純化柱純化dsDNA。

表1 LD-PCR擴增體系
1.4 Y187酵母感受態制備
Y187 酵母菌株劃線于YPDA固體培養板上,30℃培養;挑取單菌落于3 mLYPDA液體培養基中,30℃ 250 r/min震蕩培養,待OD600≈0.3時,700×g室溫離心5 min,棄去上清;以100 mL新鮮YPDA液體培養基重懸菌體,繼續培養至OD600≈0.5,700×g離心5 min后以30 mL滅菌水重懸菌體,700×g離心5min收集菌體;1.5 mL 1.1×TE/ LiAc溶液重懸菌體,13 000×g高速離心30s,用600 μL 1.1×TE/ LiAc的溶液重懸即為酵母感受態。
1.5 dscDNA與pGADT7-Rec轉化及同源重組
Carrier DNA 95℃ 5 min后置于冰上10 min,如此反復2次備用;將pGADT7-Rec、純化的dscDNA、預變性的Carrier DNA及制備好的Y187感受態于無菌15 mL離心管中混勻;加入2.5 mLPEG/LiAc混勻后于30℃孵育45 min(每15 min搖勻一次);加入160 μL DMSO混勻,42℃水浴20 min(每10 min搖勻一次);室溫700×g離心5 min后用3 mL YPD Plus重懸沉淀;30℃震蕩培養90 min后700×g離心收集菌體,再以15 mL 0.9% NaCl溶液重新懸浮。取100 μL進行10倍倍比稀釋,分別取100 μL稀釋10倍及100倍的菌液涂于SD/-Leu 100 mm固體培養板,計算文庫容量,剩余樣品涂布于70 個150 mm的SD/-Leu 培養板上。30℃倒置培養3d左右直至菌落出現。
1.6 文庫收集及滴度測定
將1.5中已經長出菌落的平板置于4℃過夜冷卻;每個平板加入15個玻璃珠及5 mL的凍存培養液,輕輕搖晃收集所有菌體,用細胞計數板計算酵母細胞個數,使細胞密度達到2×107/mL,1 mL分裝凍存。取100 μL文庫稀釋10-2、10-4、10-5、10-64個稀釋度,各取100 μL涂于100 mm SD/-Leu平板上,30℃培養3 d,菌落計數,計算文庫滴度:克隆數目/稀釋混合物涂板體積×稀釋倍數=cfu/mL。隨機挑取24個菌落以ExTaq進行PCR鑒定,判定文庫多樣性及計算重組率。
1.7 構建酵母雙雜交誘餌質粒pGBKT7-NP及誘餌菌株
以A/Anhui/2/2005(H5N1)病毒的NP表達質粒pBD-NP為模板,用NP基因正向引物BD-NP-F(gcatatGAATTCATGGCGTCTCAAGGCAC)及反向引物BD-NP-R(gcaaatGGATCCTTAATTGTCATACT CCTCTGC),用prime STAR高保真DNA聚合酶擴增NP全長;對NP擴增片段及pGBKT7載體進行RI和HI雙酶切,連接轉化后涂于卡那抗性(50 μg·mL-1)的LB固體培養板上,挑取單菌落進行菌液PCR及質粒測序鑒定。測序正確的誘餌質粒轉化酵母菌株Y2HGold,涂布SD/-Trp固體培養板,單菌落加入500 μL含有25%甘油的YPDA培養液中凍存備用。共轉化pGBKT7-53和pGADT7-T作為陽性對照,共轉化pGBKT7-Lam和pGADT7-T作為陰性對照,共轉化pGADT7和pGBKT7-NP后分別涂布到SD/-Trp/-Leu和SD/-Trp/-Leu/X-α-gal/AbA檢測誘餌質粒自激活。
1.8 誘餌菌株與文庫雜交
凍存的誘餌菌株于SD/-Trp固體培養板上劃線培養2—3 d;挑取單菌落于50 mL SD/-Trp培養液中至OD600≈0.8,離心收集誘餌菌株后用5—6 mLSD/ -Trp培養液重懸,于2 L錐形瓶中同時加入一支1 mL的文庫,再加入45 mL 2×YPDA,混勻;避光,30℃、40 r/min搖晃培養進行雜交,24 h后離心收集菌體;10 mL 0.5×YPDA重懸菌體,均勻涂布40個150 mm的SD/-Trp/-Leu/-Ade/-His(SD/-4)固體培養板上;挑取單個菌落重新劃線到SD/-Trp/ -Leu/-Ade/-His/X-α-gal/AroA(SD/-4/X/A)固體培養板上,挑取變藍且長粗的菌落到4—5 mL SD/ -Trp/-Leu(SD/-2)液體培養基中,30℃、250 r/min培養,準備提取質粒。
1.9 酵母質粒的提取及回交驗證
按照碧云天酵母質粒小提試劑盒說明書提取酵母質粒,進一步轉化至DH5α,利用Axygen 質粒小量DNA提取試劑盒提取質粒,進行測序,Blast序列比對。篩選正確閱讀的獵物質粒,將獵物質粒與誘餌質粒各500ng共同轉化Y2HGold酵母感受態菌株中,涂布SD/-2、SD/-4和SD/-4/X/A固體培養板,30℃培養箱中靜置培養3—7d,觀察生長情況。
2 結果
2.1 Calu-3細胞及A549細胞總RNA電泳
提取calu-3及A549細胞RNA濃度分別為1 023和967 ng?μL-1,OD260/OD280≈1.8,OD260/OD230≈2.0說明提取RNA純度較高,蛋白以及其他離子污染較少;28S與18S條帶清晰,5S條帶暗淡,說明RNA基本沒有降解,適合文庫構建的后續試驗(圖1)。

M:DL2000 Marker;1:Calu-3細胞總RNA;2:A549細胞總RNA
2.2 dscDNA的鑒定
提取RNA反轉錄獲得cDNA,以LD-PCR擴增合成dscDNA,利用CHROMA SPIN+TE-400對其進行純化。1%核酸電泳顯示dscDNA條帶于500—2 000 bp之間呈彌散狀(圖2),說明不同豐度和不同大小的RNA均被反轉錄,可以用于文庫構建。
2.3 庫容及滴度測定
將收集的文庫以10倍倍比稀釋后,取10-2、10-4、10-5、10-6四個稀釋度各100 μL涂于100 mm SD /-Leu平板上。菌落計數106稀釋度有22個菌落,計算所建文庫滴度為2.2×108cfu/mL(圖3)。轉化pGADT7-Rec和dscDNA的Y187菌液做1﹕10及1﹕100稀釋,涂于SD/-Leu平板上,計算所建文庫的庫容為1.5×107,滿足酵母雙雜交試驗的建庫要求。

M:DL2000 Marker;1:純化的dscDNA

酵母菌液分別稀釋至(A)10-2、(B)10-4、(C)10-5和(D)10-6在SD/-Leu平板上的生長情況,用于計算文庫滴度
2.4 文庫PCR鑒定
一方面,利用多種形式宣傳造勢企業管理制度。移動網絡的普遍,給中小企業領導者開辟了新的管理方式。可以利用移動網絡形式開展管理制度學習的比賽,或收集對企業管理制度的意見和對企業組織發展的想法建議等,以求企業員工理解管理制度,明確制度的意義,認可制度,使得企業管理工作正常進行,管理制度能夠得以貫徹落實。
隨機挑取24個單菌落進行PCR鑒定,結果顯示擴增片段大小分布在250—2 000 bp之間,且片段長度并不均一,24個克隆僅有3個沒有檢測到插入片段,重組率達到88%(圖4),上述結果表明構建的文庫質量較高,可用于后續的酵母雙雜交試驗。
2.5 誘餌質粒pGBKT7-NP的PCR鑒定和序列分析
挑取單菌落進行擴大培養,而后提取質粒,以NP特異性引物進行PCR擴增,獲得與NP基因全長(1 565 bp)相近的DNA片段,對擴增大小正確的質粒進一步測序分析,結果表明誘餌質粒構建成功。
2.6 回交驗證
篩選得到的正確閱讀的獵物質粒與誘餌質粒共轉Y2H Gold酵母菌,分別以BD-P53/AD-T7作為陽性對照和BD-Lam/AD-T7作為陰性對照,經過篩選鑒定共發現11個獵物質粒和誘餌質粒,共轉后在SD/-Trp/- Leu/-Ade/-His/X-α-gal/AroA(SD/-4/X/A)培養板上呈現陽性結果(圖6),即蛋白間存在相互作用。所篩選得到的與流感病毒NP蛋白互作的宿主蛋白的編碼基因信息見表2。
2.7 Gene Ontology分析
利用在線網站https://david.ncifcrf.gov/對篩選得到的宿主因子進行Gene Ontology(GO)分析,結果顯示11個宿主因子共參與7個生物過程,分別是:細胞凋亡調節、胚胎發育、可變剪接、多態性、金屬結合、轉錄調節以及細胞增殖調節;分子功能有4種,包括GTP結合活性、金屬離子結合活性、DNA結合活性及轉錄因子活性。具體信息見表3。

M:DL2000 Marker;1-24:樣品編號 M:DL2000 Marker;1-24:Sample number

表2 篩選得到與流感病毒NP互作蛋白的編碼基因

表3 與NP蛋白相互作用蛋白GO分析
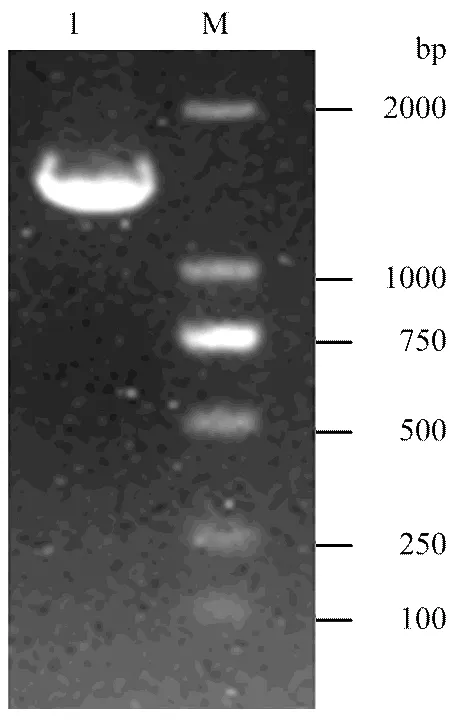
M:DL2000 Marker;1:誘餌質粒PCR產物

圖6 獵物與誘餌在Y2H Gold中的相互作用
3 討論
酵母雙雜交技術是一種被廣泛用于研究蛋白質相互作用的成熟方法,該方法不需純化蛋白,避免蛋白變性;接近于體內環境,能夠比較真實反應機體內蛋白相互作用的情況;靈敏度非常高,對于蛋白之間非常微弱、瞬間發生的互作也能夠篩選到;具有篩選量大、效率高及應用廣泛等優點[23]。
流感病毒是有囊膜的單股負鏈RNA病毒,其病毒基因組分為8個節段,可編碼多達18種蛋白。核蛋白NP由vRNA片段5編碼,全長498個氨基酸,在中性pH條件下帶正電荷,對RNA具有很強的結合力[24]。NP蛋白在結構上分為頭部結構域、主體結構域和尾部結構域[25]。NP蛋白作為流感病毒核糖核蛋白復合體vRNP的重要組分,在不同亞型的流感病毒中相當保守,在病毒復制周期的多個環節發揮重要作用[26]。因此,通過挖掘與流感病毒NP蛋白互作的宿主蛋白,不僅可以深入了解宿主蛋白參與流感病毒復制周期調控的機制,而且有可能發現潛在的藥物靶標,為研制新型抗流感病毒藥物以及干預治療提供理論依據。本研究構建誘餌質粒pGBKT7-NP進行酵母雙雜交篩選,回交驗證得到11個與其互作的宿主因子。這些蛋白具有多種分子功能,參與多個生物過程,但與病毒相關的研究仍相對較少。對這些NP互作蛋白的深入研究,將深化人們對NP蛋白功能、流感病毒感染和致病機制等的認知,并促進防控策略和手段的創新。
4 結論
本研究成功構建了高質量A549/Calu-3細胞cDNA的酵母雙雜交文庫,并以誘餌質粒pGBKT7-NP篩選到11個與流感病毒NP蛋白存在相互作用的宿主蛋白,為進一步研究NP蛋白功能及流感病毒致病機制奠定了基礎。
References
[1] MOLINARI N A, ORTEGA-SANCHEZ I R, MESSONNIER M L, THOMPSON W W, WORTLEY P M, WEINTRAUB E, BRIDGES C B. The annual impact of seasonal influenza in the US: measuring disease burden and costs.2007, 25(27): 5086-5096.
[2] YAMAYOSHI S, WATANABE M, GOTO H, KAWAOKA Y. Identification of a novel viral protein expressed from the PB2 segment of influenza a virus.2016, 90(1): 444-456.
[3] WU Y, TEFSEN B, SHI Y, GAO G F. Bat-derived influenza-like viruses H17N10 and H18N11.2014, 22(4): 183-191.
[4] JAGGER B W, WISE H M, KASH J C, WALTERS K A, WILLS N M, XIAO Y L, DUNFEE R L, SCHWARTZMAN L M, OZINSKY A, BELL G L, DALTON R M, LO A, EFSTATHIOU S, ATKINS J F, FIRTH A E, TAUBENBERGER J K, DIGARD P. An overlapping protein-coding region in influenza A virus segment 3 modulates the host response., 2012, 337(6091): 199-204.
[5] INCE W L, GUEYE-MBAYE A, BENNINK J R, YEWDELL J W. Reassortment complements spontaneous mutation in influenza A virus NP and M1 genes to accelerate adaptation to a new host.2013, 87(8): 4330-4338.
[6] BAIGENT S J, BETHELL R C, MCCAULEY J W. Genetic analysis reveals that both haemagglutinin and neuraminidase determine the sensitivity of naturally occurring avian influenza viruses to zanamivir1999, 263(2): 323-338.
[7] BARNETT J M, CADMAN A, BURRELL F M, MADAR S H, LEWIS A P, TISDALE M, BETHELL R.selection and characterisation of influenza B/Beijing/1/87 isolates with altered susceptibility to zanamivir., 1999, 265(2): 286-295.
[8] KROL E, RYCHLOWSKA M, SZEWCZYK B. Antivirals--current trends in fighting influenza., 2014, 61(3): 495-504.
[9] SAMSON M, PIZZORNO A, ABED Y, BOIVIN G. Influenza virus resistance to neuraminidase inhibitors., 2013, 98(2): 174-185.
[10] MATSUOKA Y, MATSUMAE H, KATOH M, EISFELD A J, NEUMANN G, HASE T, GHOSH S, SHOEMAKER J E, LOPES T J, WATANABE T, WATANABE S, FUKUYAMA S, KITANO H, KAWAOKA Y. A comprehensive map of the influenza A virus replication cycle.2013 (7): 97.
[11] WATANABE T, KAWAKAMI E, SHOEMAKER J E, LOPES T J, MATSUOKA Y, TOMITA Y, KOZUKA-HATA H, GORAI T, KUWAHARA T, TAKEDA E, NAGATA A, TAKANO R, KISO M, YAMASHITA M, SAKAI-TAGAWA Y, KATSURA H, NONAKA N, FUJII H, FUJII K, SUGITA Y, NODA T, GOTO H, FUKUYAMA S, WATANABE S, NEUMANN G, OYAMA M, KITANO H, KAWAOKA Y. Influenza virus-host interactome screen as a platform for antiviral drug development.2014, 16(6): 795-805.
[12] NAYAK D P, BALOGUN R A, YAMADA H, ZHOU Z H, BARMAN S. Influenza virus morphogenesis and budding.2009, 143(2): 147-161.
[13] BOULO S, AKARSU H, RUIGROK R W, BAUDIN F. Nuclear traffic of influenza virus proteins and ribonucleoprotein complexes., 2007, 124(1-2): 12-21.
[14] AKARSU H, BURMEISTER W P, PETOSA C, PETIT I, MULLER C W, RUIGROK R W, BAUDIN F. Crystal structure of the M1 protein- binding domain of the influenza A virus nuclear export protein (NEP/NS2).2003, 22(18): 4646-4655.
[15] BAUDIN F, PETIT I, WEISSENHORN W, RUIGROK R W.dissection of the membrane and RNP binding activities of influenza virus M1 protein.2001, 281(1): 102-108.
[16] KUKOL A, HUGHES D J. Large-scale analysis of influenza A virus nucleoprotein sequence conservation reveals potential drug-target sites.2014, (454-455):40-47.
[17] RAMAN S N T, LIU G, PYO H M, CUI Y C, XU F, AYALEW L E, TIKOO S K, ZHOU Y. DDX3 interacts with influenza A NS1 and NP proteins and exerts antiviral function through regulation of stress granule formation.2016, 90(7):3661-3675.
[18] ZHANG J, HUANG F, TAN L, BAI C, CHEN B, LIU J, LIANG J, LIU C, ZHANG S, LU G, CHEN Y, ZHANG H. MOV10 is a restriction factor of influenza a virus by inhibiting the nuclear import of the viral Nnucleoprotein.2016, 90(8): 3966-3980.
[19] CROS J F, GARCIA-SASTRE A, PALESE P. An unconventional NLS is critical for the nuclear import of the influenza A virus nucleoprotein and ribonucleoprotein.2005, 6(3): 205-213.
[20] ELTON D, SIMPSON-HOLLEY M, ARCHER K, MEDCALF L, HALLAM R, MCCAULEY J, DIGARD P. Interaction of the influenza virus nucleoprotein with the cellular CRM1-mediated nuclear export pathway.2001, 75(1): 408-419.
[21] MOMOSE F, BASLER C F, O'NEILL R E, IWAMATSU A, PALESE P, NAGATA K. Cellular splicing factor RAF-2p48/NPI-5/BAT1/ UAP56 interacts with the influenza virus nucleoprotein and enhances viral RNA synthesis.2001, 75(4): 1899-1908.
[22] NAITO T, KIYASU Y, SUGIYAMA K, KIMURA A, NAKANO R, MATSUKAGE A, NAGATA K. An influenza virus replicon system in yeast identified Tat-SF1 as a stimulatory host factor for viral RNA synthesis.2007, 104(46): 18235-18240.
[23] ZHOU M, LI Q, WANG R. Current experimental methods for characterizing protein-protein interactions., 2016, 11(8):738-756.
[24] YAMANAKA K, ISHIHAMA A, NAGATA K. Reconstitution of influenza virus RNA-nucleoprotein complexes structurally resembling native viral ribonucleoprotein cores., 1990, 265(19): 11151-11155.
[25] YE Q, KRUG R M, TAO Y J. The mechanism by which influenza A virus nucleoprotein forms oligomers and binds RNA.2006, 444(7122): 1078-1082.
[26] PORTELA A, DIGARD P. The influenza virus nucleoprotein: a multifunctional RNA-binding protein pivotal to virus replication.2002, 83(Pt 4): 723-734.
(責任編輯 林鑒非)
Construction of cDNA Library Derived from Human Lung Epithelial Cell Lines and Screening for Host Cellular Proteins Interacting with Influenza Virus Nucleoprotein
LUO Wei-yu1,2, ZHU Peng-yang2, ZHANG Jie2, HU Yong-hao1, KONG Hui-hui2, LIANG Li-bin2, ZHOU Yuan2, LI Cheng-jun2, JIANG Li2,CHEN Hua-lan1,2
(1College of Veterinary Medicine, Gansu Agricultural University, Lanzhou 730070;2State Key Laboratory of Veterinary Biotechnology, Harbin Veterinary Research Institute/Animal Influenza Laboratory of the Ministry of Agriculture, Chinese Academy of Agricultural Sciences, Harbin 150001)
【Objective】In order to study the biological function of influenza virus nucleoprotein and provide a basis for the understanding of mechanism of influenza virus replication and pathogenesis, a yeast two-hybrid library derived from human lung epithelial cell lines Calu-3and A549 was constructed, and the host factors that interact with nucleoprotein (NP) of influenza virus were screened. 【Method】Total RNA was extracted from equal numbers of Calu-3 and A549 cells and their cDNA was synthesized by reverse transcription. Double strand cDNA (dscDNA) was amplified by long-distance PCR (LD-PCR) and purified through CHROMA SPINTM+TE-400 column according to the user manual of Make Your Own “Mate & Plate” Library System (Clontech). The purified dsDNA containing homologous arms was transformed into component Y187 yeast cells together with linearized pGADT7-Rec plasmid, and then the samples were incubated on SD/-Leu plates at 30℃ for about 4 days. All colonies were harvested and aliquoted, resulting in the construction of yeast two-hybrid library of Calu-3 and A549 cells. The library quality was evaluated by capacity, titer, recombination efficiency and diversity. Meanwhile, theRI andHI digested NP gene from influenza virus A/Anhui/2/2005 (H5N1) was inserted into pGBKT7 vector to generate the bait plasmid, which was further demonstrated without self-activating activity. The bait plasmid pGBKT7-NP was transformed into Y2H-Gold yeast strain, and then used to screen for interacting proteins from the yeast two-hybrid library. The selected pray plasmids with correct encoding insert and bait plasmids were co-transformed into the yeast cells, with BD-P53/AD-T7 and BD-Lam/AD-T7 plasmids as positive and negative controls, respectively. The blue colonies that grew well in medium containing SD/-Trp/-Leu/-Ade/-His/X-α-gal/AroA(SD/-4/X/A) were selected as candidate containing potential interacting partners of NP. After plasmid extraction and sequencing analysis, these candidates were annotated and classified by Blast and Gene Ontology (GO) analyses. 【Result】Analysis of RNA extracted from the two types of cells showed that both 28S and 18S RNA bands were clear whereas 5S band was faint, indicating that high quality RNA was obtained with almost no degradation. The dscDNA reverse transcribed from RNA was evenly dispersed on the gel with sizes ranged mostly between 500 and 2000 bp, demonstrating that RNA with different abundance and sizes were successfully reverse transcribed. The capacity and titer of the established yeast two-hybrid library were 1.5×107and 2.2×108cfu/mL, respectively, with 88% recombination efficiency and sufficient diversity. The screening of the library with the NP bait revealed 11 candidate interacting proteins. Through gene ontology analysis, it was found that these proteins are involved in several biological processes including regulation of cell apoptosis, embryonic development, alternative splicing, transcription regulation translation, metabolic process, regulation of cell proliferation. In addition, the molecular functions of these proteins included GTP binding, metal ion binding, DNA binding and transcription factor activity.【Conclusion】In conclusion, the yeast two-hybrid library containing Calu-3 and A549 cells was successfully constructed, which laid a foundation for the screening of host factors interacting with other influenza virus proteins, and the identification of 11 potential interacting host factors provided preliminary data for further study of the biological function of influenza virus NP protein.
influenza virus; NP protein; cDNA library construction; yeast two-hybrid (Y2H) system; host factor
2016-04-22;接受日期:2016-09-18
國家自然科學基金項目(31521005)
羅維玉,E-mail:lwy365946758@163.com。通信作者陳化蘭,E-mail:chenhualan@caas.cn;通信作者姜麗,E-mail:jiangli@caas.cn

