粘質沙雷氏菌幾丁質酶基因ChiA克隆及生物信息學分析
孟利強, 沙長青, 張先成, 張淑梅, 趙曉宇, 曹 旭, 李 晶*
(1.黑龍江省科學院 微生物研究所 生物工程重點實驗室,黑龍江 哈爾濱 150010;2.黑龍江省科學院高技術研究院,黑龍江 哈爾濱 150020;3.黑龍江省科學院 條件財務處,黑龍江 哈爾濱 150001)
?
粘質沙雷氏菌幾丁質酶基因ChiA克隆及生物信息學分析
孟利強1,2, 沙長青1,3, 張先成1, 張淑梅1,2, 趙曉宇1,2, 曹 旭1,2, 李 晶1,2*
(1.黑龍江省科學院 微生物研究所 生物工程重點實驗室,黑龍江 哈爾濱 150010;2.黑龍江省科學院高技術研究院,黑龍江 哈爾濱 150020;3.黑龍江省科學院 條件財務處,黑龍江 哈爾濱 150001)
通過生物信息學技術對ChiA基因序列進行分析預測,了解ChiA的基因結構及蛋白質性質。從自有菌株(粘質沙雷氏菌SerratiamareescensS68)中克隆到幾丁質酶基因ChiA,利用相關軟件對ChiA基因序列進行分析預測。ChiA基因全長1 714 bp,開放閱讀框編碼563個氨基酸,推測其編碼的蛋白質分子量為60 983.8 Da,等電點為6.35,是一種穩定的親水性蛋白質。預測ChiA可能存在信號肽,切割位點在第23~24位氨基酸之間,1~23位氨基酸為其跨膜結構,其余肽鏈位于細胞外。ChiA主要存在3種二級結構元件,在二級、三級結構中都有體現。該ChiA是一種水溶性蛋白質,結構穩定且可以分泌到胞外。
粘質沙雷氏菌;幾丁質酶;基因克隆;生物信息學
幾丁質(chitin)又稱甲殼素,是由N-乙酰-2-脫氧-D-葡萄糖聚合而成的大分子多糖,廣泛存在于自然界藻類、細菌、放線菌等體內的多聚物,是僅次于纖維素居第二位的可再生資源[1]。幾丁質酶(chitinase)可以催化水解幾丁質的β-1,4糖苷鍵生成幾丁寡糖和N-乙酰-氨基葡萄糖,是非常重要的保健品和醫療原材料[2]。粘質沙雷氏菌(Serratiamarcescens)是一種可以產生幾丁質酶的菌株,早在1986年,Jone等[3]就克隆到了粘質沙雷氏菌的ChiA和ChiB基因,并測定了全序列。1999年,Tanaka等[4]從濕熱古細菌ThermococcuskodakaraensisKOD1中克隆到了幾丁質酶基因ChiA。國內對微生物幾丁質酶學的研究比較晚,2003年,張表等[5]構建了含有ChiA基因的克隆載體,并完成測序和序列分析。本研究所用菌株粘質沙雷氏菌S68為本研究室自有菌株,具有較強的幾丁質酶活性。通過實驗設計引物,克隆到目的基因ChiA,并對其幾丁質酶蛋白質結構進行生物信息學分析,為了解微生物幾丁質酶系及構建高產幾丁質酶工程菌提供參考。
1 材料與方法
1.1 材料
粘質沙雷氏菌(Serratiamarcescens)由本研究室保藏,菌株編號S68;克隆載體pZeroback/ blunt vector及感受態細胞DH5α(140326)購于天根生化科技有限公司。
1.2 方法
1.2.1 引物設計及PCR擴增 分析比對GenBank中10個Serratiamarcescens的ChiA 基因序列,在其共同保守區設計特異性引物:上游引物:5′-GGAATTCATGCGCAAATTTAATAAACCG-3′;下游引物:5′-CCCAAGCTTAACCGATTATTGAACGCCG-3′。PCR反應體系(25μL):模板DNA 1 μL,上下游引物各 1 μL,2×PCR Master Mix (TIANGEN) 12.5 μL,ddH2O 9.5 μL。PCR擴增條件:95 ℃預變性5 min;95 ℃變性45 s,66.2 ℃復性30 s,72 ℃延伸45 s,30個循環;最后72 ℃延伸10 min。
1.2.2ChiA 基因克隆與序列測定分析 PCR產物的瓊脂糖電泳、純化、連接、轉化及陽性克隆子的篩選均參照文獻[6]。陽性克隆子由北京三博遠志生物技術有限公司完成測序,將測得基因序列提交GenBank進行BLAST比對分析。
1.2.3ChiA 基因生物信息學分析 選用ProtParam軟件分析ChiA 的氨基酸組成、序列、分子量及等電點,用ProtScale軟件分析其疏水性,ChiA 信號肽的查找和細胞定位分別由SignalP 4.1和TMHMM v. 2.0完成,ChiA 的二級結構和三級結構分別由Psipred v3.3和Phyre2完成,ChiA 的進化分類由Prosite完成,本研究涉及軟件的生物信息學網站見表1[7]。

表1 生物信息學軟件網站
2 結果與分析
2.1ChiA基因克隆和序列分析
ChiA 基因PCR產物經瓊脂糖電泳檢測為單一條帶,分子量在1 700 bp左右,符合目的片段的大小(圖1)。將單一條帶回收、連接、轉化得到陽性克隆子,并委托三博遠志測序,得到ChiA 基因核苷酸序列(GenBank accession number KP065496)如圖2。序列全長1 714 bp,用ORF Finder 軟件分析,得到1條1 692 bp的開放閱讀框(圖3),起始密碼子ATG位于第8位置,終止密碼子TAA位于1699位置,共編碼563個氨基酸。
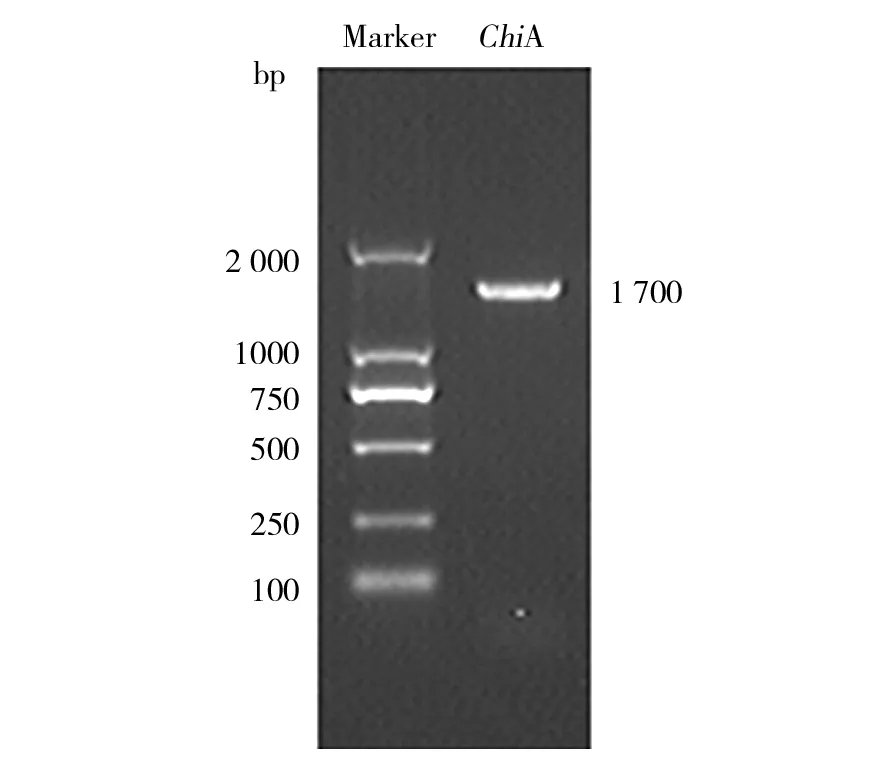
圖1 ChiA基因PCR產物電泳

圖2 ChiA基因核苷酸序列Fig.2 Nucleotide sequence of ChiA gene
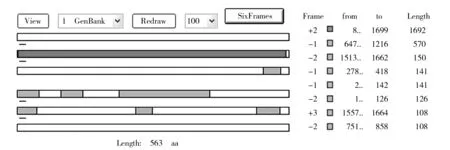
圖3 ChiA基因核苷酸開放閱讀框分析Fig.3 AnaJysis of open reading frame of ChiA
將ChiA基因序列提交NCBI網站進行BLAST比對,發現該基因序列與GenBank中登錄號為DQ990373.1的S.marcescensChiA 基因高度相似,同源性達99%以上,兩者僅在196、649、1037和1486位置的4個核苷酸堿基不同(加粗標記),由于氨基酸密碼子的兼并性,二者對應的氨基酸序列僅344位置不同,由GenBank中的Thr(T)變成了Ala(A)。2個氨基酸都屬于帶有極性的中性氨基酸,不會造成ChiA 的蛋白質結構的改變[8]。
2.2ChiA理化性質分析
ChiA基因包含一個全長1 692 bp的完整閱讀框,編碼563個氨基酸(圖4),用ProtParam軟件對其進行分析,分子量為60 983.8 Da,等電點為6.35,分子式為C2739H4205N727O822S16。如圖5所示,ChiA中含量比較多的氨基酸有甘氨酸Gly(61,10.8%)、丙氨酸Ala(55,9.8%)、亮氨酸Leu(45,8%)及賴氨酸Lys(42,7.5%),含量最少的為半胱氨酸Cys(5,0.9%)。總的帶負電荷的氨基酸(Asp+Glu)為56,總的帶正電荷的氨基酸(Arg+Lys)為54。在體外環境下ChiA的半衰期為30 h,不穩定指數為15.33(小于40),屬于穩定蛋白質。疏水性分析由ProtScale軟件完成,如圖6所示,ChiA疏水性分析第12和13分值最高為2.178,疏水性最強,第151位的脯氨酸Pro分值最低為-3.178,親水性最強。計算ChiA的平均疏水性指數為-0.282,此外由疏水性分析曲線也可以看出,基準線(0)的下半部分曲線部分明顯多于上半部分,所以預測ChiA為水溶性蛋白質[9]。

圖4 ChiA氨基酸序列Fig.4 Amino acid sequence of ChiA gene
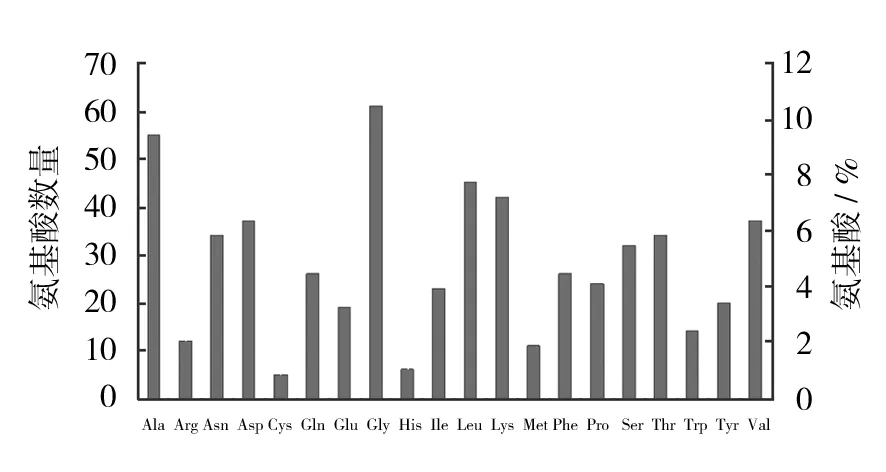
圖5 ChiA氨基酸組成及比例Fig.5 Composition and proportion of amino acid of ChiA
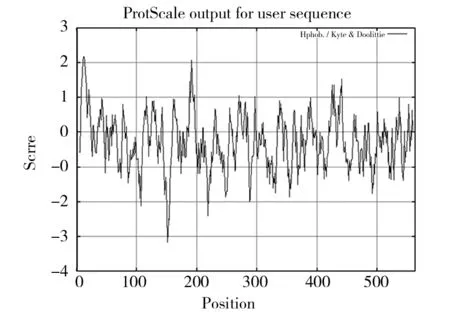
圖6 ChiA疏水性分析Fig.6 AnaJysis of hydmphobicity for ChiA
2.3ChiA信號肽分析及細胞定位
信號肽是位于分泌蛋白N端的一段氨基酸序列,將蛋白質導向細胞的正確位置,之后在信號肽酶的作用下被切除[10]。用SignalP 4.1對ChiA信號肽進行分析,結果如圖7,ChiA中可能存在信號肽,并且信號肽切割位點在23~24之間(AQA~AA),信號肽存在的可能性為0.846,推測ChiA為一種分泌蛋白。
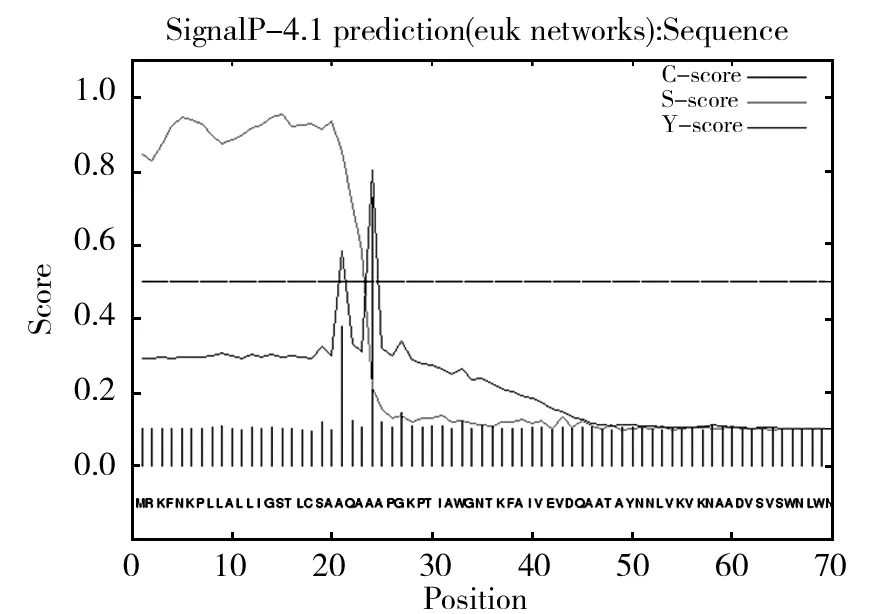
圖7 ChiA信號肽分析Fig.7 Analysis of signal peptide for ChiA
分泌蛋白的跨膜結構對其信號轉導、細胞識別及最終的細胞定位均具有重要的作用[11],通過TMHMM v. 2.0對ChiA分析發現在1~23之間存在一個跨膜結構(圖8),其余整條肽鏈都位于胞外,這與信號肽預測結果相吻合,推測ChiA的信號肽是具有疏水性的跨膜區,最終將ChiA分泌到胞外。
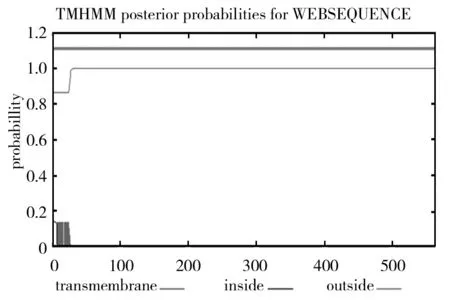
圖8 ChiA跨膜結構域分析Fig.8 Analysis of trans-membrane segments for ChiA
2.4ChiA二級結構預測
蛋白質分子的多肽鏈(一級結構)通常會以多種二級結構折疊、盤曲和延伸成特有的空間構象,并具有自身的理化性質和生物學活性,因此蛋白質二級結構的分析和預測對其空間構象的了解具有重要意義[12-13]。應用SOPMA軟件對ChiA的二級結構進行預測,結果如圖9,ChiA二級結構中共包含4種結構元件:α螺旋、延伸連、β轉角、無規則卷曲,其中無規則卷曲、α螺旋及延伸連是ChiA的主要結構元件,分別占36.94%、27.89%和24.33%,而β轉角所占比例最少僅為10.83%。
2.5ChiA三級結構及保守結構域分析
應用Phyre2軟件對ChiA的三級結構進行預測,如圖10所示,ChiA主要由無規則卷曲、α螺旋及延伸連組成,這和二級結構預測結果相符。通過搜索Prosite家族數據庫,ChiA含有1條Chitinase 18保守結構域(307~315),屬于幾丁質酶18基因家族。
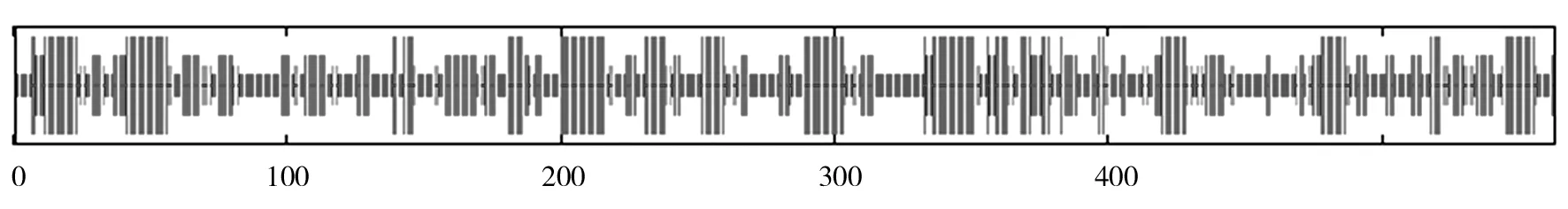
圖9 ChiA二級結構預測線性圖Fig.9 Linear map of secondary structure prediction for ChiA豎線長度(由長到短)分別表示:α螺旋、延伸連、β轉角、無規則卷曲Length of vertical (from long to short) respectively: helix, strand, angle, coil
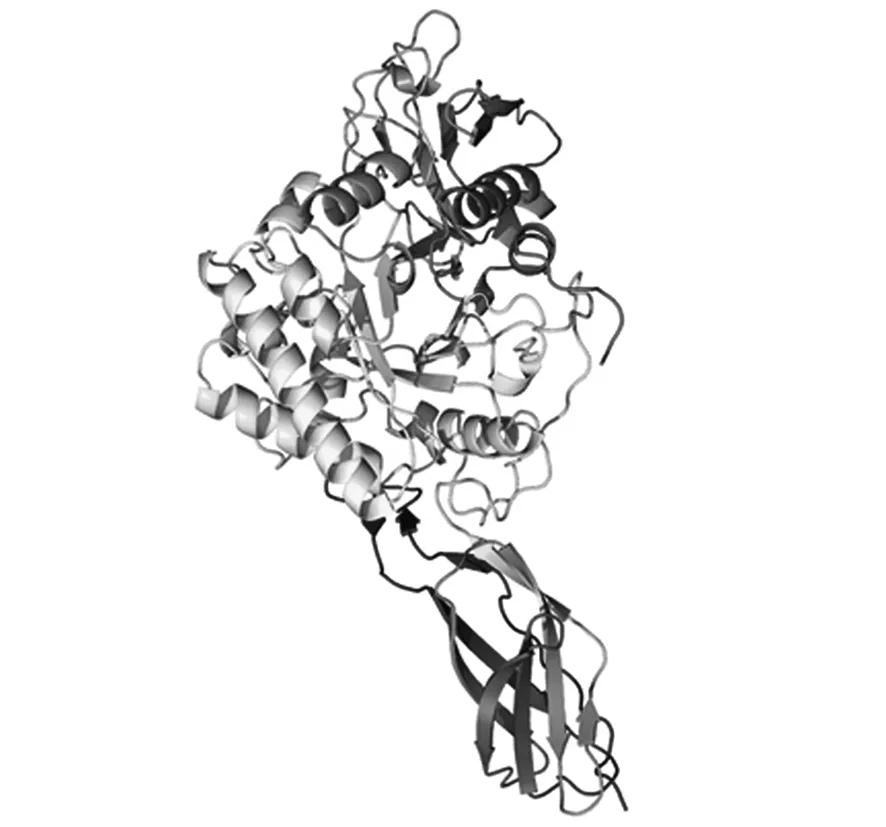
圖10 ChiA三級結構Fig.10 Tertiary structure of ChiA
3 討 論
本研究從菌株粘質沙雷氏菌(Serratiamarcescens) S68克隆到幾丁質酶基因ChiA,該基因全長1 714 bp,有1個1 692 bp的開放閱讀框,編碼563個氨基酸,這與張表等[5]報道的ChiA大小一致。該基因序列與GenBank中登錄號為DQ990373.1的S.marcescensChiA基因高度相似(99%),僅有4個堿基發生突變,最終導致了1個氨基酸的突變(Thr-Ala)。但由于這2個氨基酸都屬于帶有極性的中性氨基酸,不會造成ChiA的蛋白質結構的改變。通過一級結構分析發現ChiA中含量較高的氨基酸有甘氨酸、丙氨酸、亮氨酸和賴氨酸,半胱氨酸含量最少,分子式為C2739H4205N727O822S16,分子量為60 983.8 Da,等電點為6.35。此外,ChiA的不穩定指數和平均疏水性指數分別為15.33和-0.282,說明它屬于水溶性的穩定蛋白質。通過蛋白質預測軟件分析發現,ChiA可能是一種分泌蛋白,并且存在一個跨膜結構。ChiA的二級結構與三級結構預測結果相符[14-15]。本研究首次對自有菌株粘質沙雷氏菌S68的幾丁質酶基因ChiA進行了系統的生物信息學預測,對幾丁質酶ChiA的各級結構有了進一步認識,這將為該菌及幾丁質酶的研究和應用,如幾丁質酶的克隆表達體系的建立及定向進化的研究等方面提供參考。
[1] Dutta J,Tripathi S,Dutta P K,et al.Progress in antimicrobial activities of chitin, chitosan and its oligosaccharides: a systematic study needs for food applications[J].Food Science and Technology International,2012,18(1):3-34.
[2] 李春霞,佟永薇,侯世潔,等.微生物幾丁質酶的研究進展[J].食品研究與開發,2008,29(8):155-157.
[3] Jones J D G, Grady K L, Suslow T V, et al. Isolation and characterization of genes encoding two chitinage enzymes fromSerratiamarcescens[J].EMBOJ,1986,5(3):467-473.
[4] Tanaka T,Fukui T,Imanaka T,et al.Different cleavage specificities of the dual catalytic domains in chitinase from the hyperthermophilic archaeonThermococcuskodakaraensisKOD1[J].The Journal of Biological Chemistry,2001,276(38):35629-35635.
[5] 張表,趙曉瑜,喬環宇,等.粘質沙雷氏菌(Serratiamarcescens)幾丁質酶基因克隆的篩選及序列分析[J].河北大學學報(自然科學版),2003,23(2):184-187.
[6] Sambrook J, Fritsh E F, Maniat is T. Molecular cloning: a laboratory manual[M].New York, Cold Spring Harbor Laboratory,1989.
[7] 徐建華,朱家勇.生物信息學在蛋白質結構與功能預測中的應用[J].醫學分子生物學雜志,2005,2(3):227-232.
[8] 張久敏.例析基因突變與性狀的關系[J].試題與研究:新課程論壇, 2012,(13):69-69.
[9] 魏巍,賀淹才,方柏山,等.粘質沙雷氏菌幾丁質酶(ChiC)基因克隆及其生物信息學分析[J].江西農業大學學報,2006,28(3):444-448.
[10]Chen W.B,Nie Y,Xu Y,et al.Signal peptide-independent secretory expression and characterization of pullulanase from a newly isolatedKlebsiellavariicolashn-1 inEscherichiacoli[J].Applied Biochemistry and Biotechnology. Part A, enzyme engineering and biotechnology,2013,169(1):41-54.
[11]邱建丁,梁汝萍,譚學才,等.膜蛋白跨膜區段的預測分析[J].高等學校化學學報,2004,25(5):831-836.
[12]Osadchy M,Kolodny R.Maps of protein structure space reveal a fundamental relationship between protein structure and function[J].Proceedings of the National Academy of Sciences of the United States of America,2011,108(30):12301-2306.
[13]葉輝,程備久,朱蘇文,等.粘質沙雷氏菌幾丁質酶chiB基因的克隆與序列分析[J].激光生物學報,2007,16(3):299-303.
[14]Watanabe T, Kimura K, Sumiya T, et al. Genetic analysis of the chitinase system ofSerratiamarcescens2170[J]. J. Bacteriol.,1997,(179):7111-7117.
[15]Francesco M,Sushmita M.Natural computing methods in bioinformatics: A survey[J].Information Fusion,2009,10(3):211-216.
Cloning & Bioinformatics Analysis of Chitinase Gene ChiA from Serratia marcescens
MENG Li-qiang1, 2, SHA Chang-qing1, 3, ZHANG Xian-cheng1, ZHANG Shu-mei1, 2,ZHAO Xiao-yu1, 2, CAO Xu1, 2, LI Jing1,2
(1.KeyLab.ofBiotech.,Inst.ofMicrobiol.ofHeilongjiangAcad.ofSci.,Harbin150010;2.Inst.ofAdvancedTechnol.,HeilongjiangAcad.ofSci.,Harbin150020;3.ConditionandFinancialDiv.,HeilongjiangAcad.ofSci.,Harbin150001)
In order to learn the structure of chitinase gene (ChiA) and the protein properties ofChiA, the sequence ofChiA was analyzed and predicted by bioinformatics techniques.ChiA was cloned fromSerratiamarcescensstrain 41003, its sequence was analyzed and predicted by bioinformatics software. The results showed that the length ofChiA was 1 714 bp having an open reading frame encoding 563 amino acids inferring their encoded protein molecular weight at 60 983.8 Da with isoelectric point at 6.35, it was a stable hydrophilic protein. It was predicted that it might exist a signal peptide inChiA with its cutting site between 23rd to 24th amino acid. 1st to 23rd amino acid might be the transmembrane construction and the rest peptide chains located outside the cell.ChiA mainly existed three kinds of secondary structure elements, which embodied in both secondary structure and tertiary structure. Therefore,ChiA was a hydrosoluble protein with stable structure and could be secreted outside the cell.
Serratiamarcescens; chitinase gene; gene cloning; bioinformatics
黑龍江省科學院學科團隊創新能力提升專項(2014ws09)
孟利強 男,副研究員,碩士。研究方向為微生物資源及基因工程。E-mail:mengliqiang83420@163.com
* 通訊作者。女,研究員,博士。主要研究方向為農業微生物及分子生物學。E-mail:lj0706@sohu.com
2014-11-06;
2015-01-02
Q781
A
1005-7021(2016)01-0062-07
10.3969/j.issn.1005-7021.2016.01.011

