洋蔥S-腺苷甲硫氨酸合成酶基因的克隆及分析
王 晶,劉恩科,王永勤
(1.呂梁學院生命科學系,山西呂梁033001;2.山西省農業科學院旱地農業研究中心,山西太原030031;3.北京市農林科學院蔬菜研究中心,農業部華北地區園藝作物生物學與種質創制重點實驗室,北京100097)
洋蔥S-腺苷甲硫氨酸合成酶基因的克隆及分析
王 晶1,劉恩科2,王永勤3
(1.呂梁學院生命科學系,山西呂梁033001;2.山西省農業科學院旱地農業研究中心,山西太原030031;3.北京市農林科學院蔬菜研究中心,農業部華北地區園藝作物生物學與種質創制重點實驗室,北京100097)
根據洋蔥轉錄組測序結果設計了S-腺苷甲硫氨酸合成酶基因(AcSAMS)引物,利用RT-PCR技術和RACE技術克隆了洋蔥S-腺苷甲硫氨酸合成酶基因的cDNA全長,命名為AcSAMS。該cDNA全長1 475 bp,ORF為1 191 bp,編碼396個氨基酸的多肽。生物信息學分析表明,AcSAMS氨基酸序列與紫萼同源性為95%,與短花藥野生稻為94%,與桔梗和粳稻為93%。系統發育樹結果顯示,AcSAMS與唐菖莆SAMS的親緣關系最近。該基因的克隆可為進一步研究AcSAMS在抗旱、抗凍、耐鹽等抗逆過程中的作用機理提供理論依據,為開展抗逆基因工程奠定基礎。
洋蔥;S-腺苷甲硫氨酸合成酶基因;克隆;分析
S-腺苷甲硫氨酸合成酶(S-adenosylmethionine synthetase,SAMS,EC:2.5.1.6)是生物體內S-腺苷甲硫氨酸代謝途徑中的關鍵酶,它催化三磷酸腺苷(ATP)和L-甲硫氨酸(Met)反應合成S-腺苷甲硫氨酸(S-adenosylmethionine,SAM)[1],也是目前已知的生物體內合成SAM的唯一途徑。SAM是廣泛存在于生物中的一種生理活性分子,也是S-腺苷甲硫氨酸代謝途徑關鍵的中間產物,參與轉甲基、轉硫、轉氨丙基等反應[2-4]。SAM作為主要的甲基供體,在大多數甲基化反應中起著核心作用,如核酸、蛋白質、脂類和多糖的甲基化[5]。它參與多種不同的代謝過程,其中主要有合成多胺(精胺、亞精胺和腐胺等)、乙烯及谷胱甘肽[6]。此外,SAM還可與RNA結合參與體內基因表達的調控[6-7]。因此,研究SAMS基因功能對植物具有重要的意義。
目前,多種植物中SAMS的cDNA已被克隆出來,如擬南芥[7-8]、小麥[9]、楊樹[10]、石蒜[11]、蕪菁[12]、松樹[13]、大白菜[14]、芥菜[15-16]和杜梨[17],但是未見洋蔥SAMS基因的報道。由于SAMS的重要生物學功能,存在多種類型的SAMS基因,它由一個多基因家族所編碼,共同調節著SAM的合成速率。因此,克隆不同物種的SAMS基因具有重要的意義。
洋蔥(Allium cepa L.)為蔥科(Liliaceae)蔥屬(Allium)2年生草本蔬菜作物,是重要的世界性蔬菜,也是我國主要栽培和出口蔬菜品種,具有較強的抗凍、抗旱能力。本研究擬以洋蔥為材料,克隆SAMS基因,并對其序列特征進行分析,旨在為研究AcSAMS在洋蔥抗旱和耐凍過程中的作用機理提供理論依據,為開展洋蔥抗性分子育種奠定基礎。
1 材料和方法
1.1 材料
以紅皮洋蔥葉片為材料提取RNA。
根據洋蔥轉錄組測序獲得的SAMS基因的中間片段序列,設計了RACE引物。本研究所需引物如表1所示。

表1 引物及序列
1.2 總RNA提取及第1鏈cDNA合成
用多糖多酚植物RNA提取試劑盒(北京華越洋生物科技有限公司)提取洋蔥葉片總RNA。提取總RNA的操作步驟按照試劑盒的說明書進行。
利用B26作為引物,以3 μg總RNA作為模板,合成cDNA第1鏈。第1鏈cDNA合成的反應體系及具體操作步驟參照PrimeScript II 1stStrand cDNA Synthesis Kit(TaKaRa)說明書進行。
1.3 RACE
1.3.1 利用3′-RACE技術擴增AcSAMS基因3′端序列 以合成的第1鏈cDNA為模板,利用C1與B26引物進行PCR擴增3′末端序列。PCR反應程序為:95℃預變性2 min;94℃40 s,55℃30 s,72℃2 min,共35個循環;72℃延伸7 min;4℃保存備用。
1.3.2 利用5′-RACE技術擴增AcSAMS基因5′端序列 其參照李洪有等[18]的方法,略有改進。
TdT加Poly(C)尾反應:在加入加尾反應試劑前,將純化好的20 μLcDNA在94℃下變性6 min,之后再在冰上冷卻1 min,按照如下體系加入反應試劑:5×TdT Buffer(10 μL),0.1%BSA(5 μL),100 mmol/L dCTP(0.8 μL),TdT(15 U)和ddH2O(補至總體積50 μL),置于PCR儀中37℃1 h,65℃10 min進行加尾反應。
PCR擴增:采用巢式PCR策略,進行2輪PCR擴增。第1輪擴增以加尾的cDNA為模板,以引物AP和基因特異引物C2為上下游引物。PCR反應程序:95℃預變性2 min;94℃30 s,62~54℃30 s(62~54℃每次降2℃,其中,54℃前每個溫度2個循環,最后54℃27個循環),72℃1 min,共35個循環;72℃10 min;4℃保存備用。第2輪PCR反應以第1輪PCR擴增產物為模板,以巢式引物NP和基因特異引物C2為上下游引物。PCR擴增程序為:95℃預變性1 min;94℃30 s,60℃30 s,72℃1 min,35個循環;72℃延伸10 min;4℃保存備用。
PCR產物經瓊脂糖凝膠電泳,用瓊脂糖凝膠回收試劑盒(天根生化科技(北京)有限公司)回收目的條帶。回收的產物連接到 pEASY-T1載體(TransGen Biotech公司)上,轉化大腸桿菌DH5α,藍白斑篩選,挑白斑搖菌過夜培養。經檢測為陽性菌株送北京三博生物技術公司進行測序。
1.4 序列分析及其系統進化樹的構建
將一步RT-PCR及RACE獲得的cDNA序列利用DNAMAN軟件進行拼接,得到目的基因的全長cDNA序列。將該基因推測的氨基酸序列和來自NCBI數據庫中其他物種的同源序列進行多序列比對和系統發育分析。蛋白序列比對用Clustalx2.0軟件進行。系統發育樹用MEGA5.0軟件中的鄰近相連法進行構建,并進行Bootstrap檢測。
2 結果與分析
2.1 基因克隆及序列分析
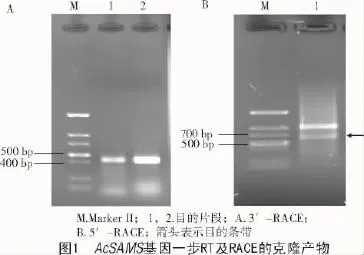
采用一步RT-PCR擴增獲得一條386 bp的3′端目的片段(圖1-A),測序結果表明,該片段長度約為400 bp。利用TdT加尾5′-RACE方法,PCR擴增后得到一個約為540 bp的5′端目的片段(圖1-B)。利用軟件DNAMAN將3條序列進行比對去除重疊部分并進行拼接,得到長度為1 475 bp的基因全長cDNA序列,命名為AcSAMS。
對AcSAMS全長序列進行分析,結果顯示,該基因全長cDNA序列包含1個80 bp的5′非編碼區;1個1 191 bp完整的開放閱讀框(ORF),編碼396個氨基酸,推測蛋白分子量為43.13 kD,等電點為5.568;1個204 bp的3′非編碼區,該非編碼區含有植物mRNA典型的Poly(A)尾序列(圖2)。

2.2 氨基酸序列比對及其系統發育分析
AcSAMS基因編碼396個氨基酸殘基,其中,堿性(+)氨基酸(K,R)42個、酸性(-)氨基酸(D,E)54個、疏水氨基酸(A,I,L,F,W,V)133個、極性氨基酸89個。對AcSAMS基因編碼蛋白序列進行二級結構預測,其中,26.52%的氨基酸殘基構成α-螺旋(alpha helix),14.39%的氨基酸殘基構成β-折疊(beta sheet),59.09%的氨基酸殘基構成隨機卷曲(randomcoil)。
將AcSAMS基因推測的氨基酸序列在NCBI數據庫中執行BLASTx比對檢索,結果發現,該氨基酸序列與紫萼同源性為95%;與短花藥野生稻為94%;與桔梗和粳稻為93%。將洋蔥AcSAMS的氨基酸序列與其他來源于不同物種的SAMS氨基酸序列進行多序列比對發現,AcSAMS包含SAMS的特征序列,分別為GHPDK(第17~21位氨基酸殘基)、GAGDQGhmfGY(第122~132位氨基酸殘基)和GGGAFSgKD(第269~277位氨基酸殘基)。
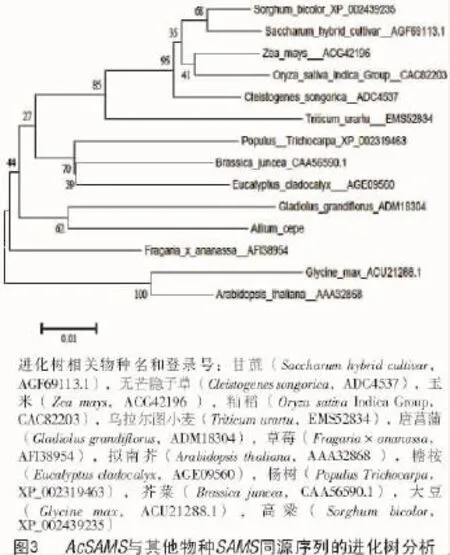
為了進一步研究洋蔥種子AcSAMS基因與其他物種SAMS基因之間的關系,用MEGA 5.0軟件采用鄰近相連法構建了AcSAMS的系統進化樹(圖3)。系統進化樹分析結果顯示,洋蔥AcSAMS基因與唐菖莆的SAMS聚在一起,表明它們的親緣關系最近。
3 討論
腺苷甲硫氨酸,在動植物體內廣泛存在,具有重要的生理生化作用。它參與滲透調節物質多胺的形成,對植物抗逆性具有重要作用。大量數據表明,SAMS基因受干旱和鹽脅迫的誘導表達,例如糜子[19]的SAMS基因受干旱脅迫的誘導表達,鹽地堿蓬[20]、大洋洲濱藜[21]、煙草[22]、陸地棉[23]等的SAMS基因受鹽脅迫的誘導表達。蝴蝶蘭SAMS基因參與了蝴蝶蘭低溫脅迫的分子調控[24];SAMS基因在甘蔗抗寒、抗旱、抗鹽和抗氧化等脅迫過程中也起某種作用[25]。將鹽地堿蓬的SsAMS2轉化煙草,轉基因植株中多聚胺含量顯著高于未轉基因煙草,且其耐鹽性也比未轉基因煙草提高[26-27]。在洋蔥的抗旱和耐鹽過程中,AcSAMS基因的組織表達模式和作用方式都有待進一步研究。
[1]張建國,李新華,袁中一.腺苷甲硫氨酸合成酶的基因及結構研究進展[J].工業微生物,2005,35(3):39-44.
[2]MatoJ,AlvarezL,Ortiz Pajares MA.S-adenosylmethionine synthesis:molecular mechanisms and clinical implications[J].Pharmacol Therapeutics,1997,73:265-280.
[3]Fontecave M,Atta M,Mulliez E.S-adenosylmethionine:nothing goes to waste[J].Trends BiochemSci,2004,29:243-249.
[4]Belbahri L,Chevalier L,Bensaddek L,et al.Different expression of an S-adenosylmethionine synthetase gene in transgenic tobacco callus modifies alkaloid biosynthesis[J].Biotechnology Bioengineering,2000,69:11-20.
[5]易樂飛,王萍,周向紅,等.條斑紫菜SAMS基因克隆與生物信息學分析[J].中國生物工程雜志,2009,29(7):43-49.
[6]柴興蘋,張玉秀,譚金娟,等.Zn脅迫下小麥腺苷甲硫氨酸代謝途徑關鍵基因表達模式分析 [J].植物生理學報,2013,49:375-384.
[7]Peleman J,Saito K,Cottyn B,et al.Structure and expression analyses of the S-adenosylmethionine synthetase gene family in Arabidopsis thaliana[J].Gene,1989,84:359-369.
[8]Sedira M,Butler E,Gallagher T,et al.Verification of auxin-induced gene expression during adventitious rooting in rol B-transformed and untransformed apple Jork[J].Plant Sci,2005,168:1193-1198.
[9] Sodabeh J G,Zahra S,Elnaz R N.Analysis of bioinformatical S-adenosylmethionine synthetase and cold-regulated(COR)proteins in wheat under cold stress[J].J Inform Technol,2013(1):12-19.
[10]Van Doorsselaere J,Gielen J,Van Montagu M,et al.A cDNA encoding S-adenosyl-L-methionine synthetase from poplar[J].Plant Physiol,1993,102:1365.
[11]Li X D,Xia B,Wang R,et al.Molecular cloning and characterization of S-adenosylmethionine synthetase gene from Lycoris radiata [J].Mol Biol Rep,2013,40:1255-1263.
[12]Kim S B,Yu J G,Lee G H,et al.Characterization of Brassica rapa S-adenosyl-L-methionine synthetase gene including its roles in biosynthesis pathway[J].Hortic Environ Biote,2012,53:57-65.
[13]Lindroth AM,Saarikoski P,Flygh G,et al.Two S-adenosylmethionine synthetase-encodinggenes differentially expressed during adventitious root development in Pinus contorta[J].Plant Mol Biol,2001,46:335-346.
[14]Yu J G,Lee G H,Park Y D.Physiological role of endogenous S-adenosyl-L-methionine synthetase in Chinese cabbage[J].Hortic Environ Biote,2012,53:247-255.
[15]Lim C C,Liu J Z,Pua E C.Characterization of S-adenosylmethionine synthetase genes and its expression is associated with ethylene synthesis in mustard(Brassica juncea)[J].Physiol Plantarum,2002,116:522-530.
[16]Mo H,Pua E C.Up-regulation of arginine decarboxylase gene expression and accumulation of polyamines in mustard(Brassica juncea)in response to stress[J].Physiol Plantarum,2002,114:439-449.
[17]張梅,王然,馬春暉,等.杜梨S-腺苷甲硫氨酸脫羧酶基因的克隆與生物信息學分析[J].華北農學報,2013,28(1):82-87.
[18]李洪有,王嬋,李麗林,等.洋蔥花器官B類MADS-box基因AcPI的克隆及表達分析 [J].中國農業科學,2012,45(23):4759-4769.
[19]林凡云,王士強,胡銀崗,等.糜子SAMS基因的克隆及其在干旱復水中的表達模式分析 [J].作物學報,2008,34(5):777-782.
[20]Ma X,Wang Z,Qi Y,et al.Isolation of S-adenosylmethionine synthetase gene from Suaeda salsa and its diferential expression under NaCl stress[J].Acta Botanica Sinica,2003,45(11):1359-1365.
[21]Schrfder G,Eichel J,Breinig S,et al.Three differentially expressed S-adenosylmethionine synthetases from Catharanthus roseus:molecular and functional characterization[J].Plant Molecular Biology,1997,33(2):211-222.
[22]Tabuchi T,Kawaguehi Y,Azuma T,et al.Similar regulation patterns of choline monooxygenase,phosphoethanolamine N-methyltransferase and S-adenosyl-l-methionine synthetase in leaves of the halophyte Atriplex nummularia L[J].Plant and Cell Physiology,2005,46(3):505-513.
[23]周凱,宋麗艷,葉武威,等.陸地棉耐鹽相關基因GhSAMS的克隆及表達[J].作物學報,2011,37(6):1012-1019.
[24]袁秀云,雷志華,梁芳,等.蝴蝶蘭S-腺苷甲硫氨酸合成酶基因的克隆及低溫下的表達分析 [J].植物生理學報,2015(5):754-762.
[25]宋修鵬,張保青,黃杏,等.甘蔗S-腺苷甲硫氨酸合成酶基因(ScSAM)的克隆及表達[J].作物學報,2014,40(6):1002-1010.
[26]戚元成,馬雷,王菲菲,等.過量表達腺苷甲硫氨酸合成酶基因能提高轉基因煙草中多聚胺的生物合成[J].植物生理學通訊,2009,45(8):791-793.
[27]戚元成,王菲菲.鹽脅迫下過量表達腺苷甲硫氨酸合成酶基因的轉基因煙草的生長 [J].植物生理學通訊,2010,46(2):150-152.
Molecular Cloning and Bioinformatic Analysis of S-adenosylmethionine Synthetase Gene from Allium cepa
WANG Jing1,LIU En-ke2,WANG Yong-qin3
(1.Department of Life Sciences,Lyuliang University,Lyuliang033001,China;2.Research Center of Arid Farming,Shanxi Academy of Agricultural Sciences,Taiyuan 030031,China;3.Key Laboratory of Biology and Genetic Improvement of Horticultural Crops(North China),Ministry of Agriculture,Beijing Vegetable Research Center,Beijing Academy of Agriculture and Forestry Sciences,Beijing100097,China)
Primers of S-adenosylmethionine synthetase gene were designed based on the result of onion transcriptome sequencing, and full-length cDNA of S-adenosylmethionine synthetase gene in onion was cloned using RT-PCR combined with RACE technology, named AcSAMS.The total length ofcDNA was 1 475 bp and its ORF encoding a polypeptide of396 amino-acid residues was 1 191 bp. Bioinformatics analysis showed that the homology between the amino acid sequence of AcSAMS and grimmia was 95%,and the short anthers of wild rice was 94%,and the japonica and platycodon grandiflorum were 93%,respectively.The dendrogram result indicated that the closest genetic relationship existed in AcSAMS and SAMS Gladiolus grandiflorus.The cloning of the gene lays a foundation for further research on the functional mechanism of AcSAMS in the process of drought resistance and salt tolerance and the development of genetic improvement.
onion;AcSAMS;cloning;analysis
S633.2
A
1002-2481(2016)05-0575-04
10.3969/j.issn.1002-2481.2016.05.02
2016-03-17
國家自然科學基金項目(31372066);北京市農林科學院科技創新能力建設專項(KJCX201101010)
王 晶(1993-),女,山西興縣人,在校學生,研究方向:分子生物學。劉恩科、王永勤為通信作者。

