基于解卷積的網絡優化算法研究及應用*
哈爾濱醫科大學衛生統計學教研室(150081) 王文杰 謝宏宇 侯 艷 李 康
基于解卷積的網絡優化算法研究及應用*
哈爾濱醫科大學衛生統計學教研室(150081) 王文杰 謝宏宇 侯 艷 李 康△
目的探討網絡解卷積算法對網絡結構的優化效果。方法模擬研究采用四種網絡算法對具有金標準的DREAM 5平臺數據進行網絡構建,并評價解卷積優化前后的網絡準確性。實例研究使用RF回歸對卵巢癌晚期化療敏感性患者的基因表達數據構建網絡,再通過網絡解卷積算法優化。結果模擬研究結果表明,四種網絡構建方法推斷出來的網絡結構在解卷積算法優化后,其準確性均有不同程度的提高,其中基于線性相關的網絡構建方法提高幅度明顯大于CLR和RF算法;實例分析結果表明,采用RF-ND方法構建的網絡移除了部分間接邊,其優化后能得到與現有數據庫較為一致的網絡結構。結論應用解卷積算法能夠優化不同網絡構建方法得到的網絡,實際中能得到準確度較高的網絡結構。
調控網絡 解卷積 網絡優化
網絡研究能夠直觀地反映變量間的作用關系,有助于特征標志物的篩選,并能從分子水平闡述復雜的生物過程,因此成為了近年的研究熱點之一。目前有很多基因調控網絡的構建方法,如基于變量間相關或偏相關系數的方法,計算變量間信息熵的互信息法,基于圖形及信息傳遞的高斯圖論模型,以及基于因果概率的貝葉斯網絡模型等[1-4]。由于高維組學數據變量間具有復雜的關聯傳遞效應,一般的網絡構建方法很難識別出變量間真正的直接調控關系,如由于A→B→C→D強相關,使得A→C、A→D這種本身并無真正調控關系的間接邊也表現出強相關(圖1左);或者由于A→B→D和A→C→D的兩個傳遞效應,使得A→D的關系增強(圖1右)。隨著網絡中變量個數的增多和級聯關系的增加,這種由傳遞效應產生的間接邊會隨之增加,容易出現假陽性結果,使得網絡推斷的準確性大幅降低。本文引入一種基于解卷積的網絡優化算法,可以進一步優化推斷出來的網絡結構,移除間接邊,解決上述問題。

圖1 四個節點在兩種傳遞效應機制下的網絡構建可能出現假陽性結果
原理與方法
網絡解卷積算法是一種網絡優化方法,其基本思想是:根據不同網絡推斷方法(如Pearson相關系數)得到網絡結構數據,建立一個網絡拓撲圖的鄰接矩陣Gobs,并將其分解為具有直接關聯和各種長度的間接關聯矩陣之和,即

而Gindir根據關系矩陣的傳遞閉包運算有
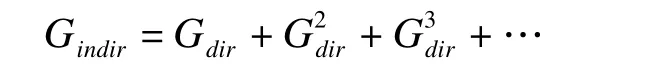

其中U和U-1分別為直接關聯矩陣Gdir的特征向量矩陣及其逆陣,Λdir為相應特征值的對角矩陣。通過對關聯矩陣進行線性尺度變換,可以將所有特征值的取值范圍限定在1至-1,可以根據無窮泰勒級數,導出近似公式:
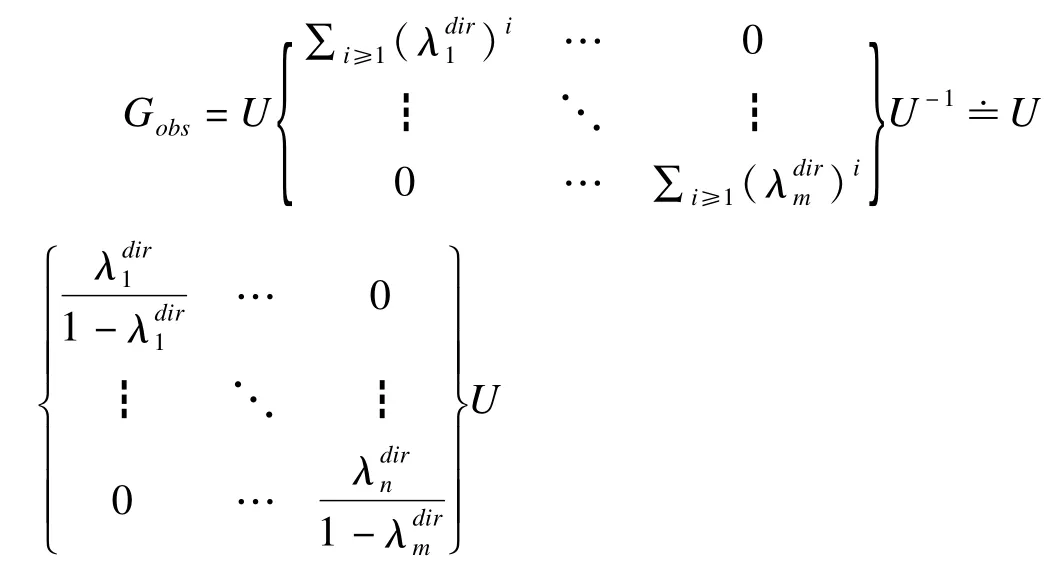
實際數據獲得的關聯矩陣同樣可以分解為

上述基于特征分解的網絡解卷積運算需滿足兩個假設:間接邊權重等于直接邊權重的乘積;觀察到的邊權重等于直接邊和間接邊之和。Feizi研究證明,理想情況下,當網絡結構的所有邊完全滿足這兩個假設時,基于特征分解的解卷積運算能移除所有間接邊;而當網絡結構不全滿足假設時,該算法也能準確推斷出87%的直接邊[5]。
網絡解卷積算法先通過對網絡結構圖的鄰接矩陣的特征分解,利用無窮泰勒系數和,最終完成對網絡各邊權重的重新賦值。通過限定一個閾值,可以把權重較高,置信度較強的邊篩選出來。從而可以移除間接邊,準確推斷出直接網絡結構。
模擬研究
1.研究目的
使用解卷積算法對四種不同的網絡構建方法得到的網絡進行優化,并評價其效果。四種方法分別采用基于Pearson相關系數、Spearson相關系數、互信息的環境相關似然度算法(context likelihood of relatedness,CLR)及隨機森林回歸算法。對三組數據進行網絡構建,然后應用解卷積算法優化,得到優化后的網絡結構,最后評價解卷積算法優化前后的效果。
2.數據來源
使用基因逆向工程評估與方法對話平臺(dialogue on reverse engineering assessment and methods project,DREAM)的數據進行評價。該平臺用于評價網絡推斷性能的數據主要包括In silico、E.coli和S.cerevisiae等,其中In silico是通過Genenetweaver軟件模擬出來的具有金標準的調控網絡數據,其余兩個是用生物學實驗測序得到的調控網絡數據[6]。

表1 DREAM 5網絡數據驗證平臺
3.評價指標
(1)ROC曲線下面積AUC,即綜合評價真陽性率和假陽性率的指標。其中真陽性率也稱回召率,即TPR(k)=TP(k)/P,TP(k)為網絡算法得出的k條邊在與金標準比較后正確邊的數量,k值可根據網絡構建時選擇不同的閾值而改變,P為金標準中陽性邊的數量。假陽性率為FPR(k)=FP(k)/N,FP(k)為網絡算法得出的k條邊在與金標準比較后錯誤邊的數量,N為金標準中陰性邊的數量。FPR(k),c為根據構建網絡的閾值得到的網絡邊數。
(2)準確度-回召率曲線下面積(area under the precision-recall curve,AUPR),即綜合評價準確度和回召率的指標。準確度為PRE=TP(k)/k,即網絡算法得出的k條邊中正確邊的比例。
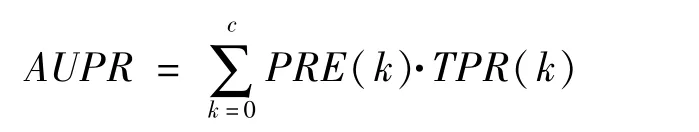
4.網絡優化的評價結果
四種網絡構建方法、三組網絡數據的評價結果如表2。結果顯示:對于三組不同的網絡數據,其還原程度不盡相同,In silico的AUROC與AUPR均最高,表明構造出來的網絡結構相比另外兩個更接近于金標準結構;對于四種不同的網絡構建方法,基于隨機森林回歸算法的AUROC,AUPR均比其他構建方法要高,尤其是在In silico數據集上表現遠高于其他算法,ROC值達到0.815,顯示了該方法良好的網絡重構性能;對于不同的網絡構建方法,解卷積優化前后其AUROC,AUPR在三個數據集上均有不同程度的提高。總體而言,經由解卷積算法優化后,基于相關的網絡構建方法,其AUROC與AUPR的提高幅度明顯大于CLR和RF算法。
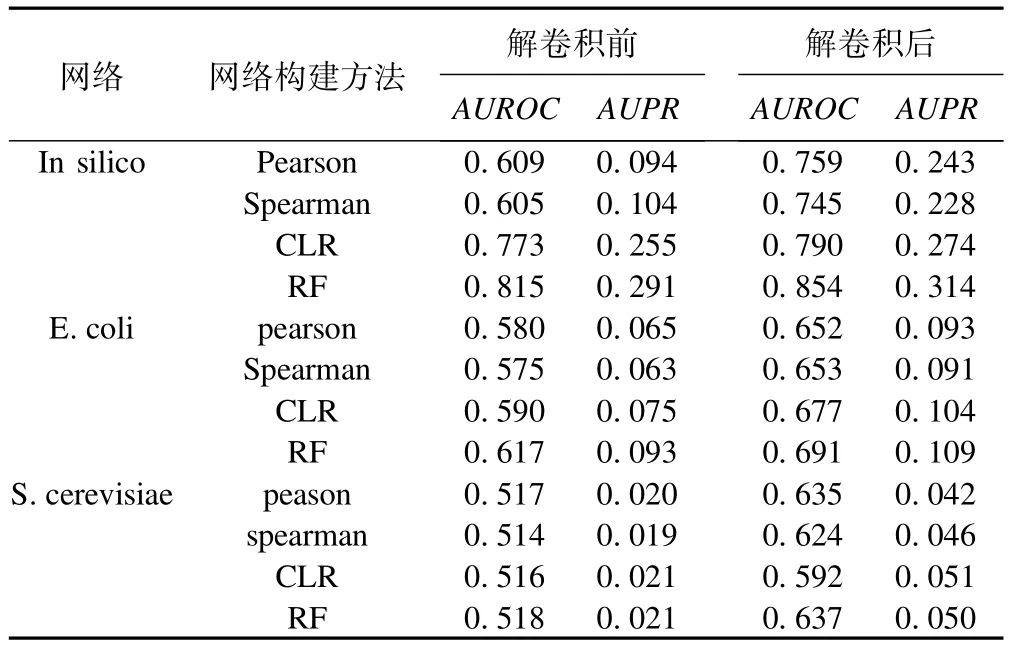
表2 不同網絡構建方法及不同數據,解卷積優化前后性能比較
實例分析
本研究通過對卵巢癌晚期化療患者的基因表達數據進行分析,使用RF回歸方法構建網絡,再通過網絡解卷積算法優化,得出基因間的調控關系網絡。最后,結合生物學知識、通路數據庫及文獻查詢,對網絡進行生物學解釋,從基因組學的角度,為卵巢癌化療敏感性的生物學機制研究提供依據。
本研究從TCGA數據庫下載348例基于卡鉑-紫杉醇化療方案的晚期卵巢癌患者的基因表達譜數據,根據化療藥物反應的敏感程度分為化療藥物敏感組310例和化療藥物不敏感組38例。全基因組表達譜數據一共測得12042個基因的表達值,使用基于W ilcoxon秩和檢驗的置換檢驗,進行1000次置換,篩選出P<0.05(校正后)的基因431個,并將這部分數據進行KEGG通路富集分析,結果有10個基因顯著富集在Wnt信號通路以及溶酶體通路。對所富集的10個基因的表達數據,采用RF回歸方法構建網絡,通過置換檢驗方法確定網絡閾值,經100次隨機置換后的VIM值的99%分位數為0.1148,從而確定邊數目的閾值為c(VIM)=0.1148,獲得20條可能具有調控關系的邊,網絡結構如圖2左所示。再通過網絡解卷積算法對這20條邊所構成的網絡進行優化,重新對各邊權重賦值,經由置換檢驗得到的新閾值為0.2016,獲得16條關系邊,移除了4條邊,解卷積優化后的網絡結構如圖2右所示。
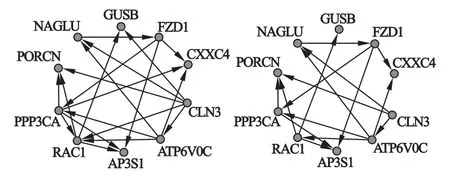
圖2 卵巢癌患者化療敏感性相關的基因調控網絡解卷積前后結構(左為ND前,右為ND后)
通過查詢GeneMANIA及KEGG基因數據庫,發現這10個基因中,有8條邊出現在數據庫中。例如ATP6V0C調控NAGLU、CLN3、RAC1和PPP3CA四個基因。NAGLU和CLN3兩個基因與ATP6V0C同屬于溶酶體通路,RAC1、PPP3CA基因與ATP6V0C同屬Wnt信號轉導通路。并且已有文獻報道ATP6V0C、NAGLU和CLN3基因在溶酶體的內吞以及物質轉運過程中發揮重要作用[7];ATP6V0C調控的RAC1和PPP3CA基因編碼合成Wnt通路中重要的反應酶,參與調節多個細胞活動,如控制細胞生長、細胞骨架重組,以及激活蛋白激酶等[8-9]。該結果與隨機森林回歸構建的網絡結構一致,對于這些重要的調控關系,基于隨機森林回歸的網絡構建算法均能很好地還原出來,并且在經解卷積算法優化后,這些調控關系都得以保留。經由解卷積算法移除的4條邊,即PPP3CA、RAC1、CLN3、GUSB基因間的間接調控關系,在數據庫中均未查詢到,并且暫時也無相應文獻報道,這就體現了解卷積算法在移除間接調控關系的優化作用。而在剩余的邊中,與ATP6V0C有關的基因還有CXXC4,此調控關系在geneMANIA并沒有找到相應的調控關系,提示這一調控關系需要進一步研究。
討 論
本文在簡要介紹解卷積算法原理的基礎上,通過DREAM 5數據驗證平臺的模擬數據,研究其對不同網絡構建方法,不同網絡數據集的優化性能。研究使用目前常見的四種基因調控網絡構建方法:基于兩種相關系數的網絡分析方法,基于互信息的構建方法,基于隨機森林(RF)回歸的方法等。其中,基于線性相關的調控網絡模型計算任意兩基因表達水平的相關系數(Pearson或Spearman相關系數),對相關系數進行排序,進而構建出網絡;基于互信息(M I)的方法主要通過計算變量間的邊際概率和聯合概率,從而得出變量間的互信息值,并根據其構建網絡;基于隨機森林回歸的算法,通過回歸樹對每個目標基因都擬合了回歸模型,計算出變量重要性評分(VIM),可得到兩基因間調控關系的大小,并根據VIM值排序從而重建整個網絡。最后將基于RF-ND的方法應用到實際卵巢癌化療敏感性的基因表達數據,并作出生物學解釋。關于網絡解卷積優化方法的實際應用,需要注意以下幾個問題:
1.本研究使用的網絡解卷積算法是基于網絡鄰接矩陣的特征分解及無窮泰勒級數和得到,如果所優化的網絡鄰接矩陣不可進行特征分解,則無法繼續使用本方法優化。這種情況下,可以使用基于迭代共軛梯度遞減的算法來進行網絡解卷積的優化運算[5,10-11],其結合了共軛性和最速下降兩種方法,不僅解決了一般網絡結構優化問題,還可處理高維情況下的大型計算問題。
2.解卷積算法屬于網絡優化方法,最終得到的網絡結構的準確性也受所選擇網絡構建方法的影響。在模擬研究中,四種網絡構建方法所重構的網絡結構,在解卷積優化后均有不同程度的提高,其中基于線性相關的方法優化幅度最大,其主要原因是線性相關類方法在衡量任意兩基因間的相關性時,并未考慮其他中介基因存在時造成的間接傳遞效應,因此其構建出來的網絡可能包含較多的假陽性間接邊,而解卷積算法正好解決該問題,因此獲得較佳的優化效果。而RF回歸算法,在計算兩變量間的VIM值時,考慮了其他變量的相互影響,構造出來的網絡結構較為準確,因此其優化前得分本身較高,優化后提升幅度并不大,但總的準確度仍然最高。說明使用隨機森林(RF)回歸構建網絡,再使用網絡解卷積進行優化,即RF-ND方法,是一種值得推薦的網絡構建方法。
3.使用RF-ND方法的最大特點是對變量的數目沒有限制,可以在高維數據上構建網絡,而且可以給出基因之間的調控方向。
4.由于實際基因組數據往往缺乏完整準確的調控關系(即金標準),因此僅基于目前的數據庫和文獻查詢只能驗證部分調控關系,用于評價一個構建方法的優劣本身并不夠嚴謹全面,對此尚需進一步研究。另外,如何構造既包含差異基因也包含非差異基因的全局調控網絡,還原生物體內完整的基因調控過程也極具挑戰性。
[1]Tibshirani R.Regression shrinkage and selection via the lasso.J R Stat Soc Series B Stat Methodol,1996,58:267-288.
[2]Pesch R,Lysenko A,Hindle M,et al.Graph-based sequence annotation using a data integration approach.Journal of integrative bioinformatics,2008,5(2),doi:10.2390/biecoll-jib-2008-94.
[3]Butte AJ,Kohane IS.Mutual information relevance networks:functional genom ic clustering using pairw ise entropy measuerments,2000,5:418-429.
[4]Mani S,Cooper GF.A Bayesian local causal discovery algorithm,2004:731-735.
[5]Feizi S,Marbach D,Medard M,et al.Network deconvolution as a generalmethod to distinguish direct dependencies in networks.Nat Biotechnol,2013,31(8):726-733.
[6]Stolovitzky G,Monroe D,Califano A.Dialogue on reverse-engineering assessment and methods:the DREAM of high-throughput pathway inference.Ann.NY Acad.Sci,2007,1115:1-22.
[7]You H,Jin J,Shu H,et al.Small interfering RNA targeting the subunit ATP6L of proton pump V-ATPase overcomes chemoresistance of breast cancer cells.Cancer Lett,2009,280(1):110-119.
[8]Ji J,Feng X,ShiM,et al.Rac1 is correlated with aggressiveness and a potential therapeutic target for gastric cancer.Int JOncol,2015,46(3):343-353.
[9]Dokmanovic M,Hirsch DS,Shen Y,et al.Rac1 as a potential therapeutic target for the treatment of target for thetreatment of trastuzumab-resistant breast cancer.Mol Cancer Ther,2009,8(6):1557-1569.
[10]Horn R,Johnson C.Matrix analysis.Cambridge Univ Pr,1990.
[11]Faith JJ,Hayete B,Thaden JT,etal.Large-scalemapping and validation of Escherichia coli transcriptional regulation from a compendium of expression profiles.PLoSBiol,2007,5(1):e8.
(責任編輯:郭海強)
Network Optim ization Algorithm Based on Network Deconvolution and its Application
Wang Wenjie,Xie Hongyu,Hou Yan,et al
(Department of Health Statistics,School of Public Health,Harbin Medical University(150081),Harbin)
ObjectiveTo investigate the performance of the network optim ization based on network deconvolution.MethodsIn simulation studies,we performed four network reconstructionmethods to construct the gene regulatory network on the data from DREAM 5 platform which have contained the gold standard.Then we compared the accuracy of before and after optim ization based on network deconvolution algorithm.In pritical studies,we applied random forest regression to construct an original network on gene expression data which comes from the advanced ovarian cancer patients thatwas susceptible to chem ical therapy.Finally,we performed the network deconvolution method to optim ize the structure of it.ResultsSimulation studies demonstrated that the accuracy of networks that reconstructed by fourmethods was increased to some degree.For the range of improvement,method that based on linear correlation was greater than CLR and RF.In practice,themethod based on RF-ND removes some indirected edges and achieves satisfactory network structure that consistent to the existing database.ConclusionThe algorithm of network deconvolution could optimize the structure of network constructed by the differentmethods and obtain the network w ith higher accuracy.
Regulatory network;Network deconvolution;Network optimization
國家自然科學基金資助(81473072,81573256);中國博士后基金資助(2015M571445)
△通信作者:李康,E-mail:likang@ems.hrbmu.edu.cn

