α-芋螺毒素GIC與Ac-AChBP共結晶條件的篩選與優化
林波 劉坤 朱明月 李偉 董栩 李孟森
(海南醫學院海南省腫瘤發生與干預重點實驗室,海口 571199)
α-芋螺毒素GIC與Ac-AChBP共結晶條件的篩選與優化
林波 劉坤 朱明月 李偉 董栩 李孟森
(海南醫學院海南省腫瘤發生與干預重點實驗室,海口 571199)
旨在篩選及優化α-芋螺毒素與乙酰膽堿結合蛋白(AChBPs)共結晶的條件,得到高分辨率的共結晶晶體。在昆蟲表達系統中分泌表達海兔乙酰膽堿結合蛋白(Ac-AChBP),并用凝膠色譜進行純化,把純化的乙酰膽堿結合蛋白與α-芋螺毒素GIC共結晶,利用結晶機器人及HAMPTON RESEARCH 結晶試劑盒進行結晶及結晶條件的篩選與優化。結果顯示,在0.6 mol/ L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.8的條件下生長的晶體較好,其分辨率可以達到2.1 ?。篩選與優化結晶條件(Ammonium citrate dibasic的摩爾濃度及0.1 mol/L Sodium acetate trihydrate的pH值)可以得到高分辨率的α-芋螺毒素GIC與Ac-AChBP共結晶晶體。
α-芋螺毒素GIC;乙酰膽堿結合蛋白;共結晶;結晶條件優化
α-芋螺毒素是煙堿型乙酰膽堿受體(nAChRs)的拮抗劑,而乙酰膽堿結合蛋白(AChBPs)與nAChRs 的胞外配體結合結構域同源性較高,AChBPs與nAChRs的藥理特性和離子通道激活機制相似,因此AChBPs 通常作為研究nAChRs的結構模板[1,2]。多個AChBPs的x-ray結構已被解析,通過其晶體結構,我們更清楚了解nAChRs的結構。AChBPs有3種,分別是Lymnaea stagnalis、Aplysiacalifornica、Bulinus truncatus, 簡 稱 為Ls-AChBP、Ac-AChBP、Bt-AChBP,它們結構相類似[3,4],α-芋螺毒素主要與Ac-AChBP 結合。α-芋螺毒素與Ac-AChBP共結晶結構不僅幫助我們剖析 nAChRs的生理和病理功能,而且有助于發現藥效更好的α-芋螺毒素[4,5]。
目前已有5個α-芋螺毒素與AChBPs共結晶結構被解析,它們分別是PnIA(A10L D14K)(PDB:2BR8)[5]、ImI(PDB:2C9T,2BYP)[6,7]、TxIA(A10L)(PDB:2UZ6)[8]、BuIA(PDB:4EZ1)和GIC(PDB:5CO5)[9]與Ac-AChBP共結晶的結構。其中GIC與Ac-AChBP的復合物結構是我們報道的分辨率較高的共結晶結構。α-芋螺毒素GIC是從殺手芋螺(Conus geographus)中發現的作用于α3β2 nAChRs的毒素。α3β2 nAChRs是位于腦部的神經受體,與記憶、認識相關。α-芋螺毒素GIC作用于α3β2 nAChRs的IC50 最低濃度為1.1 nmol/L,是目前發現最強作用于α3β2 nAChRs的毒素,這一點使它的結構與功能倍受關注,另外,它與Ac-AChBP結合的IC50值也較低(29 nmol/L),它與Ac-AChBP的共結晶的方法可為其他新型α-芋螺毒素與Ac-AChBP或nAChRs共結晶提供借鑒,為開發治療疼痛、成癮、帕金森癥、阿爾茨海默氏癥和癲癇等與nAChRs相關疾病的藥物提供理論依據[9]。
蛋白結構的解析關鍵是要獲得高分辨率的蛋白晶體。要獲得高質量的蛋白晶體,必須對蛋白結晶條件進行篩選與優化[10-12]。蛋白結晶過程通常是把沉淀劑(池液)加到蛋白質溶液表面,沉淀劑從蛋白質溶液中吸走水分而引起蛋白結晶。結晶條件是指不同的沉淀劑及其摩爾濃度、pH值等因素。這些條件(因素)有上萬種,因此需要利用機器人進行對結晶條件的大量篩選和優化,才能得到分辨率高的晶體。本研究采用正交實驗,并利用機器人及HAMPTON RESEARCH試劑盒對α-芋螺毒素GIC與Ac-AChBP共結晶條件進行篩選與優化,以期獲得高分辨率的晶體。
1 材料與方法
1.1 材料
Ac-AChBP(AChBP from Aplysia californica) 基因[13]根據NCBI 提供的序列(NM_001204599)設計并由上海生工公司合成,在兩端分別引入BamH I、Xho I酶切位點。大腸桿菌宿主菌DH10Bac,昆蟲表達載體 pFastBac1,昆蟲細胞Sf9(Spodoptera frugiperda),HAMPTON RESEARCH結晶試劑 盒PEG/Ion1,PEG/Ion2,Index,Crystal Screen,SaltRX等由本實驗室保存。
1.2 方法
1.2.1 蛋白表達與純化 在昆蟲細胞中分泌表達Ac-AChBP,分泌表達的培養基上清液經鎳柱純化后,用凝膠色譜進一步純化。本實驗所用凝膠色譜柱是 superdex200(GE Healthcare),利用 AKTA(GE Healthcare)系統分離純化,流動相HBS溶液(10 mmol/L Hepes,150 mmol/L NaCl,pH7.2)流速為1 mL/min,將蛋白樣品濃縮到1 mL后上樣,利用AKTA程序過柱,自動收集樣品。將收集樣品利用SDS-PAGE檢測,并用液氮速凍后置于 -80℃保存[13-16]。
1.2.2 α-芋螺毒素GIC的合成與純化 α-芋螺毒素GIC由上海吉爾生化公司合成,并用C18柱(Vydac,Hesperia,CA,USA)經高效液相色譜純化,收集純化的樣品(與標準品比較達到99.8%)冷凍干燥備用[9]。
1.2.3 Ac-AChBP蛋白結晶 將純化的Ac-AChBP濃縮至約20 mg/mL。使用液-液擴散法在291 k長晶體。具體如下:在96孔板中,用HAMPTON RESEARCH試劑PEG/Ion1,PEG/Ion2,Index,Cystal Screen,SaltRX當池液,用結晶機器人CRYSTAL GRYPHON(Art robbins instrusmets)點樣,將濃縮蛋白液與池液按1∶1點坐滴,點樣量為0.15 μL,在顯微鏡下觀察晶體生長[9]。
1.2.4 α-芋螺毒素與Ac-AChBP共結晶 α-芋螺毒素與 Ac-AChBP 共結晶前,先把它們混合,然后把混合好的溶液去點晶體。已知1 mol Ac-AChBP亞基與1 mol α-芋螺毒素結合(雖然理論上它們按摩爾比1∶1結合,但為了使α-芋螺毒素能充分結合到Ac-AchBP中,把α-半螺毒素的量增加了10倍)。利用ExPASy Protparam tool分析Ac-AChBP吸光指數為1.5,其分子量為27 kD,取出Ac-AChBP蛋白儲備液(濃度為1.0 mg/mL)1 mL。按以下公式計算Ac-AChBP蛋白亞基儲備液的mol數為:
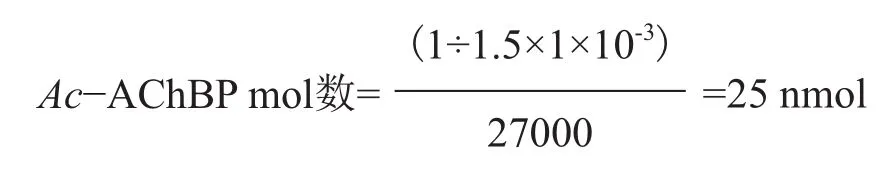
α-芋螺毒素GIC的C端已酰胺化,其分子量為1 610.4 kD,用純化的α-芋螺毒素GIC重量除以其分子量可得到GIC的摩爾數。 把α-芋螺毒素GIC按摩爾比為10∶1與Ac-AChBP亞基共混。把混和液在室溫放置2 h使其結合,然后13 000 r/min離心15 min,再用凝膠色譜純化。收集Ac-AChBP與α-芋螺毒素結合的凝膠純化色譜峰片段,將其濃縮至約20 mg/mL。使用坐滴蒸氣擴散法在291 k長晶體,點完晶體后,在顯微鏡下觀察到晶體生長[13-16]。
2 結果
2.1 Ac-AChBP的制備
在昆蟲細胞中分泌表達Ac-AChBP,并用凝膠色譜純化。Ac-AChBP的凝膠純化色譜(圖1-A)顯示,其出峰位置在12.5 mL,根據Superdex200柱的說明,峰位置為12.5 mL表示約為135 kD的相對分子量。從不同的峰位置a-d收集組分作進一步的SDSPAGE膠分析(圖1-B)。圖1-B中a-d是Ac-AChBP蛋白非變性的SDS-PAGE膠圖,即上樣時,沒有加還原劑和煮沸樣品。Ra-Rd所示是變性的SDSPAGE膠圖,即上樣時,加還原劑DDT和煮沸樣品5 min。a-d條帶在SDS-PAGE膠頂部(圖1-B),顯示它們非變性狀態是五聚體,因為其是球狀蛋白,電泳圖只顯示分子量為116 kD,比實際線型分子量(135 kD)要小。R a-d所示是變性的Ac-AChBP,其由五聚體變為單體,分子量約為27 kD;正好是表達出來的重組蛋白五聚體分子量的1/5,這一點也說明變性的Ac-AChBP狀態為單體。活性狀態為五聚體。
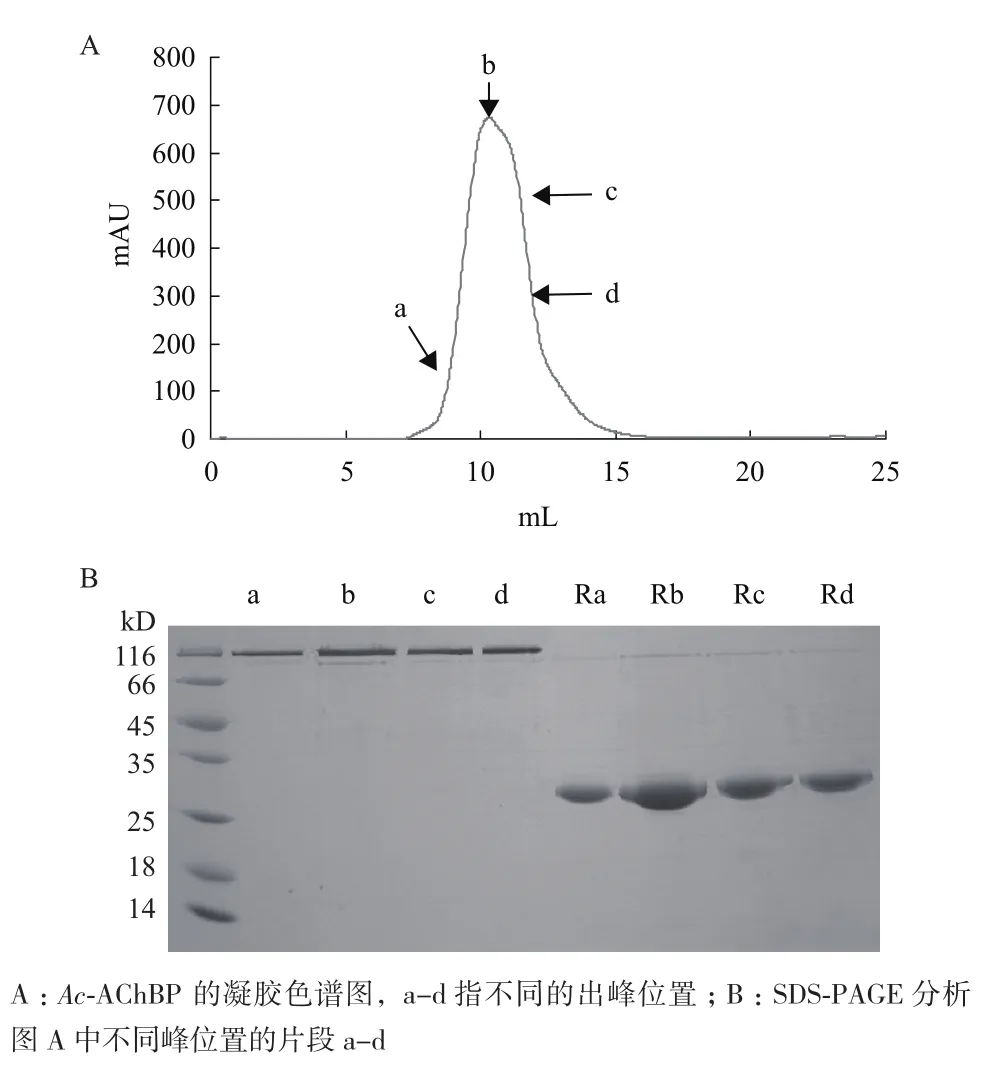
圖1 凝膠色譜(A)和SDS-PAGE(B)分析Ac-AChBP蛋白
2.2 Ac-AChBP結晶及優化
在研究α-芋螺毒素GIC與Ac-AChBP共結晶條件之前,先研究Ac-AChBP的結晶條件。原因是共結晶中Ac-AChBP是個五聚體的大蛋白,而α-芋螺毒素GIC只結合在Ac-AChBP上的小多肽。因此Ac-AChBP結晶的條件是研究共結晶的前提。Ac-AChBP結晶條件可以借鑒在共結晶中。
利用機器人初步篩選Ac-AChBP的結晶,選用結晶試劑盒PEG/Ion1,PEG/Ion2,Index,Crystal Screen,Wizard 1,SaltRX均能得到晶體,把得到的晶體送往上海同步輻射光源收集數據,SaltRX的4個條件(表1和圖2)得到的晶體分辨率較好。在這4個條件中,15號(圖2-A)晶體條件得到的晶體數據最好,分辨率為3 ?。

表1 SaltRX試劑盒篩選出來長晶體的條件
接著對15號晶體條件進行優化,優化實驗設計如表2所示,對36個結晶條件進行實驗(兩因素,六水平),并把得到的晶體送去收集數據,其中最好的晶體數據分辨率為2.4 ?,其生長條件為:0.8 mol/L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.6。
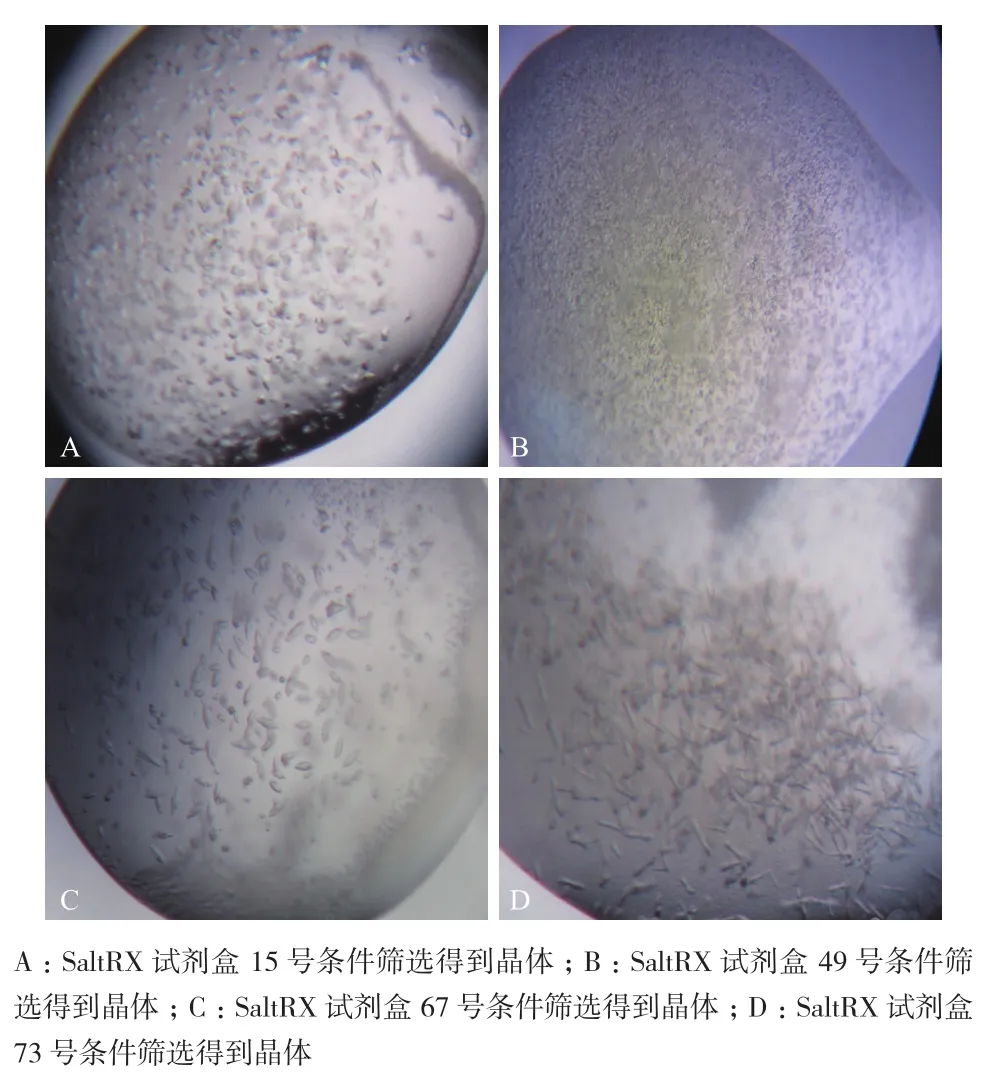
圖2 SaltRX試劑盒初步篩選得到晶體
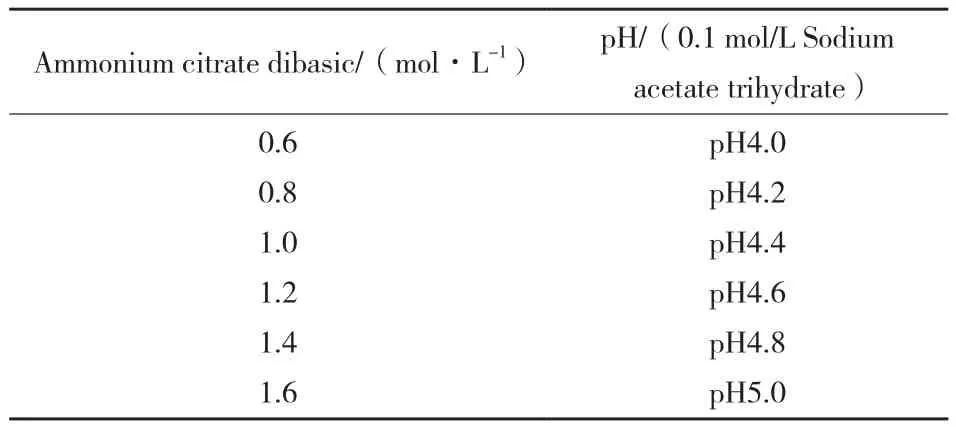
表2 SaltRX的15號結晶條件優化因素及水平
2.3 α-芋螺毒素與Ac-AChBP共結晶
2.3.1 α-芋螺毒素與Ac-AChBP結合 得到2.4 ?的Ac-AChBP晶體后,進一步研究其與α-芋螺毒素共結晶,共結晶前,先把它們共結合,結合的方法按1.2.4所示,α-芋螺毒素與Ac-AChBP結合后,用凝膠色譜純化。圖3-A藍線顯示Ac-AChBP與α-芋螺毒素GIC結合的凝膠純化色譜,紅線是結合前的Ac-AChBP。圖中藍線比紅線分子量大,說明Ac-AChBP已與α-芋螺毒素GIC結合。圖3-B是收集圖3-A藍線峰組分進行SDS-PAGE 膠分析,條帶1是活性五聚體Ac-AChBP結合α-芋螺毒素GIC,其分子量顯示為100-130 kD之間。而條帶2是變性的藍線組分,變性后,五聚體Ac-AChBP變為單體,分子量為27 kD。變性后,α-芋螺毒素GIC也解離出來,顯示在SDS-PAGE膠底部。
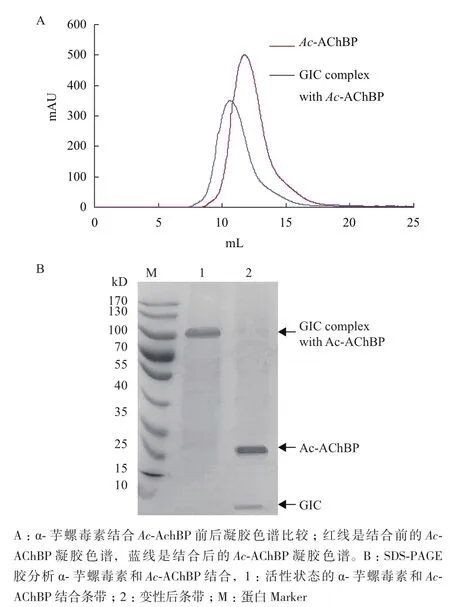
圖3 凝膠色譜(A)、SDS-PAGE膠(B)分析α-芋螺毒素和Ac-AChBP結合
2.3.2 α-芋螺毒素GIC與Ac-AChBP共結晶 收集以上共結合的凝膠色譜片段(藍線峰部分),用機器人去點樣長晶體,結晶池液采用優化Ac-AChBP晶體所得的的條件:0.8 mol/L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.6,也得到α-芋螺毒素GIC與Ac-AChBP共結晶的晶體,但由于受α-芋螺毒素GIC的影響,其晶體長得不如Ac-AChBP好,還需進一步優化。
優化采用以上優化Ac-AChBP的方法(表2),即改變不同沉淀劑的濃度和pH值,以期獲得高質量晶體。表2優化條件下長出的共結晶晶體如圖4所示(遴選),把優化條件下長出來的晶體送往上海同步輻射光源收集數據,其中最好的共結晶數據分辨率為2.1 ?,晶體如圖5所示,其生長條件為0.6 mol/L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.8。收集一套完整的數據,經HKL2000 處理后數據其他指標也較好。
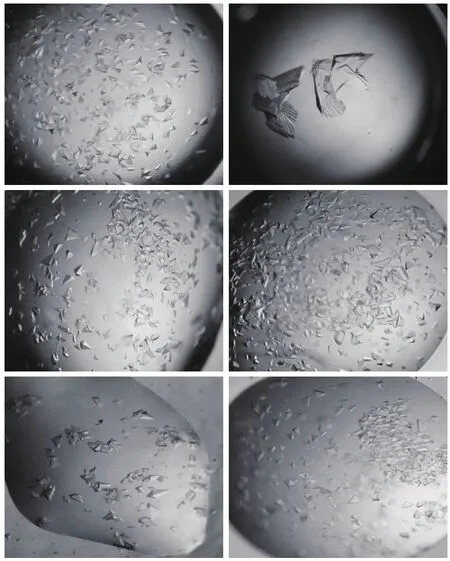
圖4 共結晶條件優化得到的晶體(遴選)

圖5 優化的α-芋螺毒素GIC與Ac-AChBP共結晶2.1 ?晶體
3 討論
目前已發表的α-芋螺毒素與AChBPs共結晶結構采用的結晶條件都有所不同,PnIA(A10L D14K)采用的結晶條件是:17%-18%(W/V)PEG 3350,170-200 mmol/L Na2SO4,100 mmol/L Bis-Tris propane,pH7.5[5];TxIA(A10L)采用的結晶條件是:20% polyethyleneglycol 3350 and bistrispropane at pH8.5[8];ImI在兩個條件都得到晶體,它們分別是:11%-14% PEG-4000,0.1 mol/L Tris,pH7.5[6]和100 mmol/L sodium acetate(pH5.5)and polyethylene glycol 5000 monomethylether[7];我們也在兩個條件下得到GIC的共結晶,分別是:1.5 mol/L lithium sulfate monohydrate,0.1 mol/L Tris,pH8.5[9]和0.6 mol/L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.8。以上看出,不同的α-芋螺毒素與AChBPs共結晶其結晶條件不同,同一α-芋螺毒素得到高分辨率的結晶條件也可以是多個。結晶條件千變萬化,這可能與結晶的方法及蛋白的性質、純度、緩沖液等因素有關。
本實驗采用的結晶方法是液-液擴散法(Liquidliquid diffusion),其原理如圖6所示,當把沉淀劑滴加到蛋白質溶液表面,它通過與蛋白質溶液接觸面擴散到蛋白質溶液中,沉淀劑從蛋白質溶液中吸走水分,從而使蛋白質溶液濃度不斷增大。因此在兩溶液接觸面形成濃度梯度,隨著沉淀劑擴散進入的量越多,蛋白質溶液濃度就越大。蛋白質由不飽和溶液變成飽和溶液,再變成過飽和溶液,從而開始結晶[10]。
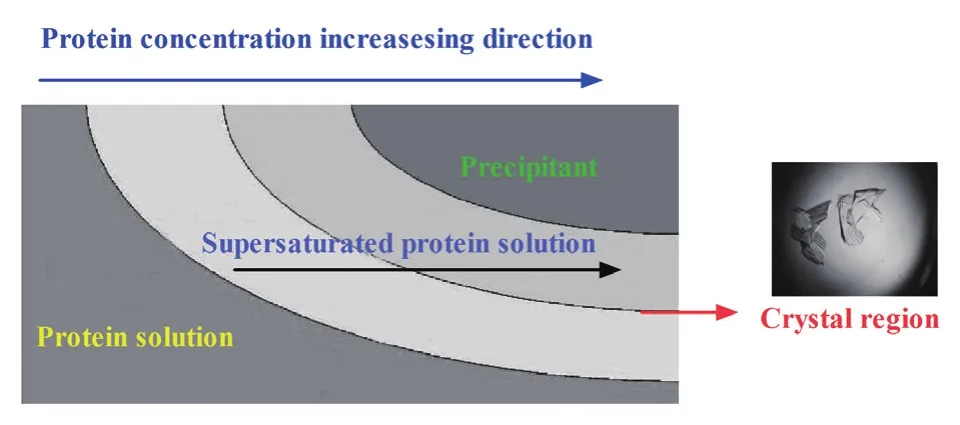
圖6 液-液擴散法結晶原理
我 們 利 用LtIA、TxIB、LvIA、TxID、RegIA、GIC 等多種α-芋螺毒素與Ac-AChBP共結晶[9,17-20],在以上條件下有LtIA、LvIA和GIC得到共結晶晶體。優化后GIC分辨率較高,LtIA和LvIA共結晶晶體的優化條件待進一步探討。不同的α-芋螺毒素對Ac-AChBP結晶影響,需要不同的結晶條件才能得到分辨率高的晶體。另外,每種α-芋螺毒素的溶解度不同,它們的溶解度對共結晶也有影響。
共結晶前采用濃度較稀的Ac-AChBP 蛋白液與濃度較稀的α-芋螺毒素溶液先共混好,這種方法可以使α-芋螺毒素更好溶解,因為它們先混合在較稀的溶液中,結合后還要經過凝膠色譜純化,所以這種方法不僅使Ac-AchBP和α-芋螺毒素充分溶解和結合,還可以除去α-芋螺毒素凍干粉中的雜質對結晶的影響。
我們試用另外一種共結晶方法,即把得到的Ac-AChBP晶體浸泡在α-芋螺毒素溶液中,浸泡時間為兩周,觀察α-芋螺毒素能否結合到晶體內部。但晶體送往上海同步光源收集數據后,分析結果顯示α-芋螺毒素沒有結合上Ac-AChBP,這說明α-芋螺毒素與Ac-AChBP共結晶的方法最好還是Ac-AChBP與α-芋螺毒素溶液先混合好再結晶,這樣它們才容易結合并共結晶。
我們先研究Ac-AChBP蛋白的結晶條件,然后在此基礎上再研究共結晶條件,可以避免浪費α-芋螺毒素,而且還可以分析每種α-芋螺毒素對共結晶條件的影響。
4 結論
本研究表達與純化海兔乙酰膽堿結合蛋白(Ac-AChBP),并把它與α-芋螺毒素GIC共結晶,采用正交實驗,并利用結晶機器人及HAMPTON RESEARCH試劑盒對其結晶條件進行篩選與優化,在0.6 mol/L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.8的條件下生長出較好質量的晶體,其分辨率達到2.1 ?。
[1] Van Dijk WJ, Klaassen RV, Schuurmans M, et al. Crystal structure of an ACh-binding protein reveals the ligand-binding domain of nicotinic receptors[J]. Nature, 2001, 411(6835):2692-2676.
[2] Smit AB, Syed NI, Schaap D, et al. A glia-derived acetylcholinebinding protein that modulates synaptic transmission[J]. Nature, 2001, 411(6835):261-268.
[3] Hansen SB, Talley TT, Radic Z, et al. Structural and ligand recognition characteristics of an acetylcholine-binding protein from Aplysia californica[J]. The Journal of Biological Chemistry, 2004, 279(23):24197-24202.
[4] Celie PH, Klaassen RV, van Rossum-Fikkert SE, et al. Crystal structure of acetylcholine-binding protein from Bulinus truncatus reveals the conserved structural scaffold and sites of variation in nicotinic acetylcholine receptors[J]. The Journal of Biological Chemistry, 2005, 280(28):26457-26466.
[5] Celie PH, Kasheverov IE, Mordvintsev DY, et al. Crystal structure of nicotinic acetylcholine receptor homolog AChBP in complex with an α-conotoxin PnIA variant[J]. Nature Structural & Molecular Biology, 2005, 12(7):582-588.
[6] Hansen SB, Sulzenbacher G, Huxford T, et al. Structures of Aplysia AChBP complexes with nicotinic agonists and antagonists reveal distinctive binding interfaces and conformations[J]. The EMBO Journal, 2005, 24(20):3635-3646.
[7] Ulens C, Hogg RC, Celie PH, et al. Structural determinants of selective alpha-conotoxin binding to a nicotinic acetylcholine receptor homolog AChBP[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(10):3615-3620.
[8] Dutertre S, Ulens C, Büttner R, et al. AChBP-targeted α-conotoxin correlates distinct binding orientations with nAChR subtype selectivity[J]. The EMBO journal, 2007, 26(16):3858-3867.
[9] Lin B, Xu M, Zhu X, et al. From crystal structure of alpha-conotoxin GIC in complex with Ac-AChBP to molecular determinants of its high selectivity for alpha3beta2 nAChR[J]. Scientific Reports, 2016, 6:22349.
[10] Gorrec F. Protein crystallization screens developed at the MRC Laboratory of Molecular Biology[J]. Drug Discovery Today, 2016, 21(5):819-825.
[11] Liu J, Yin DC, Guo YZ, et al. Selecting temperature for protein crystallization screens using the temperature dependence of the second virial coefficient[J]. PLoS One, 2011, 6(3):e17950.
[12] Juarez-Martinez G, Steinmann P, Roszak AW, et al. Highthroughput screens for postgenomics:studies of protein crystallization using microsystems technology[J]. Analytical Chemistry, 2002, 74(14):3505-3510.
[13] Lin B, Meng H, Bing H, et al. Efficient expression of Acetylcholine-binding protein from Aplysia californica in Bac-to-Bac System[J]. Biomed Res Int, 2014, 2014:691480.
[14] Wang N, Shi X, Jiang L, et al. Structure of MERS-CoV spike receptor-binding domain complexed with human receptor DPP4[J]. Cell Research, 2013, 23(8):986-993.
[15] 林波, 孟海玲, 吳勇, 等. 轉染乙酰膽堿結合蛋白基因到昆蟲細胞的研究[J]. 生命科學研究, 2014, 18(1):1-5.
[16] 林波, 孟海玲, 吳勇, 等. 靜水椎螺乙酰膽堿結合蛋白在Bacto-Bac系統中的表達、純化與結晶[J]. 生物技術通報, 2014(8):126-131.
[17] Luo S, Akondi KB, Zhangsun D, et al. Atypical alpha-conotoxin LtIA from Conus litteratus targets a novel microsite of the alpha3beta2 nicotinic receptor[J]. The Journal of Biological Chemistry, 2010, 285(16):12355-12366.
[18] Luo S, Zhangsun D, Zhu X, et al. Characterization of a novel alpha-conotoxin TxID from Conus textile that potently blocks rat alpha3beta4 nicotinic acetylcholine receptors[J]. Journal of Medicinal Chemistry, 2013, 56(23):9655-9663.
[19] Luo S, Zhangsun D, Wu Y, et al. Characterization of a novel alpha-conotoxin from conus textile that selectively targets alpha6/ alpha3beta2beta3 nicotinic acetylcholine receptors[J]. The Journal of Biological Chemistry, 2013, 288(2):894-902.
[20] Kompella SN, Cuny H, Hung A, et al. Molecular basis for differential sensitivity of alpha-Conotoxin RegIIA at rat and human neuronal nicotinic acetylcholine receptors[J]. Molecular Pharmacology, 2015, 88(6):993-1001.
(責任編輯 馬鑫)
Screening and Optimization of Co-crystallization Condition of α-Conotoxin GIC Complex with Ac-AchBP
LIN Bo LIU Kun ZHU Ming-yue LI Wei DONG Xu LI Meng-sen
(Hainan Provincial Key Laboratory of Carcinogenesis and Intervention,Hainan Medical College,Haikou 571199)
The aim is to screen and optimize the co-crystallization condition of α-conotoxin complex with acetylcholine binding protein(AChBPs),and to obtain co-crystals with high resolution. AChBP from Aplysia californica(Ac-AChBP)was expressed in insect expression system and purified by gel filtration chromatography. Then,the purified Ac-AChBP was co-crystalized with α-conotoxin GIC,further,the HAMPTON RESEARCH crystal kit and the crystal robot were applied screening and optimization of the growth condition of crystallization. Results showed that crystal grew well under the growth condition of 0.6 mol/L ammonium citrate dibasic and pH4.8 of 0.1 mol/L sodium acetate trihydrate,and the resolution of the crystal reached 2.1 ?. In conclusion,the co-crystal of α-conotoxin GIC complex with Ac-AChBP with a high resolution may be acquired via screening and optimization of crystallization condition(The molar concentration of ammonium citrate dibasic and the pH of 0.1 mol/L Sodium acetate trihydrate).
α-conotoxin GIC;acetylcholine-binding proteins;co-crystallization;optimization of crystal condition
10.13560/j.cnki.biotech.bull.1985.2017.02.028
2016-06-02
國家自然科學基金項目(31560243,81560450),海南醫學院科研培育基金項目(HY2013-05)
林波,男,助理研究員,研究方向:蛋白質結構;E-mail:linbo_752@163.com
李孟森,男,研究員,研究方向:肝癌耐藥;E-mail:mengsenli@163.com

