SCI論文的信息技術應用于小麥抗病育種領域發展態勢分析
胡春芳,李 敏,馬曉萍,閔文江,呂軍海,馮改靜,楊蘭偉
(河北省農林科學院農業信息與經濟研究所,河北 石家莊 050051)
國際標準化組織(ISO)關于信息技術的定義為:針對信息的采集、描述、處理、保護、傳輸、交流、表示、管理、組織、儲存和補救而采用的系統和工具的規范、設計及其開發[1]。隨著計算機技術和通訊技術突飛猛進的發展,信息技術對社會活動各領域影響越來越大。在20世紀70年代已經開始應用計算機技術開展農作物育種工作,如種質資源管理等,并且在育種領域應用得越來越廣泛和深入,如分子標記、基因芯片、遙感監測、紅外光譜測定、模型診斷、自動化考種機械設備普遍應用于農作物育種程序各環節,極大地推動了育種工作的進程。
在大數據時代,應用文獻計量學方法進行海量信息的提取挖掘,已經被越來越多的學者關注和采用。顏志輝等[2]對基于SCI數據庫2009~2013年全球關于作物轉基因育種領域的文獻發表數量、被引頻次、地域分布和研究機構分布進行了分析,揭示了轉基因研究的前3種作物及重點研究領域。許麗等[3]論述了2006~2015年國內關于基因編輯育種技術的論文和專利發文量變化趨勢、地域分布、研究進展及各國監管政策。王慧媛等[4]利用文獻計量學和可視化圖譜軟件,對2003~2013年國內外關于作物分子設計育種專利和論文文獻從“分子設計育種”概念提出,領域發展的知識和技術演化,以及當前的研究熱點與發展趨勢進行了全面分析。靳軍寶等[5]利用國際專利數據庫對生物育種專利技術進行了發展態勢分析,結果顯示,我國在生物育種領域專利申請量排名第2位,但專利總體質量不高,排名前10位的專利權人中沒有中國企業。但是,截至目前,通過文獻計量學對信息技術應用于作物育種發展態勢的相關研究尚未見報道。從國內外發表文獻的視角,分析了信息技術在小麥抗病育種領域的發展概況和研究進展,旨為科研人員提供參考的同時,也為科研管理工作者確定發展學科方向提供情報依據。
1 數據收集與研究方法
文獻資料來源于科學引文索引數據庫(SCI),檢索時間為2017年4月,檢索范圍為2000~2016年,檢索的主題詞為信息技術應用于小麥作物抗病育種領域主要技術詞匯。結合SCI的學科分類,通過人工清洗與判讀等,終選出文獻數量2 580篇。由于關鍵詞是反映文章主旨和涉及領域最有效的標志,運用TDA(Thomson Data Analyzer)軟件(湯森路透公司)對關鍵詞進行合并與歸類統計,探究信息技術在小麥抗病育種應用領域研究的主要內容;并從發文量、地域分布等方面對文獻進行計量分析與可視化研究。
2 結果與分析
2.1 文獻量變化趨勢分析
通過對2000~2016年SCI數據庫關于信息技術在小麥抗病育種領域應用的相關文獻進行統計發現,2016年發文量是2000年的3.6倍,平均年增長率為8.74%,總體呈平穩增長趨勢(圖1)。表明信息技術在小麥抗病育種領域應用處于穩步成長階段,有較大的發展空間。
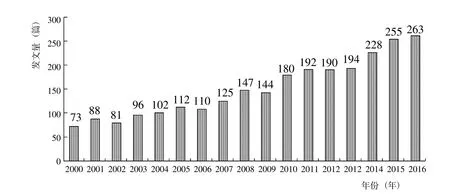
圖1 2000耀2016年SCI數據庫關于信息技術在小麥抗病育種應用的發文量Fig.1 The papers number of information technology applied to wheat disease resistance breeding research based on SCI database from 2000 to 2016
2.2 重點國家發文量
通過對2 580篇文獻進行國別統計發現,發文量>200篇的國家分別為美國(694篇)、中國(599篇)、澳大利亞(302篇)和加拿大(208篇),其余國家發文量為100~200篇。對排名前3位國家的2000~2016年發文數量變化趨勢分析發現,2000~2012年美國的發文量一直保持最高,中國次之;2013~2016年中國的發文量躍居第1位,平均年增長為17.4%,增長速率最高(圖2)。表明近幾年信息技術應用于小麥抗病育種領域仍然是研究的熱點,且中國在該領域的研究處于迅速發展時期。
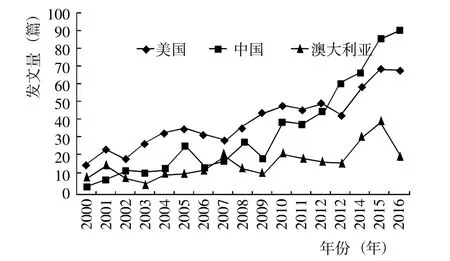
圖2 2000耀2016年SIC數據庫有關信息技術在小麥抗病育種應用領域研究排名前3位國家的發文量Fig.2 The papers number of the top three countries in information technology applied to wheat disease resistance breeding research based on SCI database from 2000 to 2016
2.3 主要研究機構分析
信息技術應用于小麥抗病育種領域發文量排名前20位的機構共發表文獻1 878篇,其中,美國農業部農業研究局、國際玉米小麥改良中心、中國農業科學院、堪薩斯州立大學、加拿大農業與農業食品部和澳大利亞聯邦科學與工業研究組織植物工業部6個機構的發文量>100篇(表1)。這20個研究機構中,美國有9個,發文量占到總數的46.2%;中國有5個,發文量占到總數的20.6%。可以看出,美國在信息技術應用于小麥抗病育種領域占有絕對優勢。

表1 2000耀2016年信息技術應用于小麥抗病育種領域發文量排名前20位的研究機構Table 1 The top 20 research institutions in the paper quantity in information technology applied to wheat disease resistance breeding research from 2000 to 2016
2.4 研究主題分析
2.4.1 基于SCI學科分類的分析 根據Web of Science類別對2 580篇關于信息技術在小麥抗病育種領域應用的文獻進行分類,結果(圖3)顯示,前5位研究領域主要分布在Plant Science(植物科學)、Agronomy(農學)、Genetics&Heredity(基因遺傳)、Horticulture(園藝)和Biochemistry&Molecular Biology(生物化學、分子生物學)領域,其中,Plant Science(植物科學)和Agronomy(農學)的發文量均>1 200篇,Genetics&Heredity(基因遺傳)、Horticulture(園藝)、Biochemistry&Molecular Biology(生物化學、分子生物學)和Food Science&Technology(食品科學與技術)的發文量在689~224篇之間;其他學科還涉及到Multidisciplinary Sciences(交叉學科)和En tomology(昆蟲學)等。
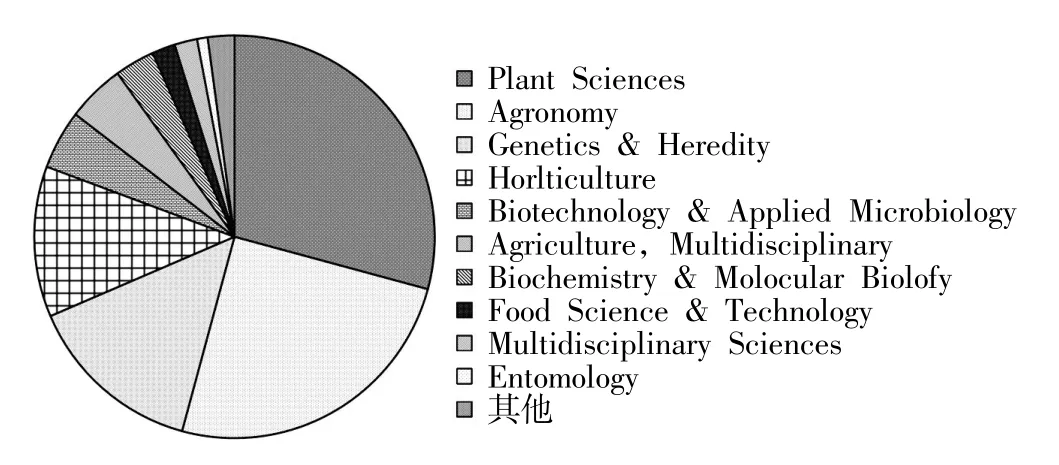
圖3 2000耀2016年信息技術在小麥抗病育種領域應用的SCI學科分類Fig.3 The subject classification of information technology applied to wheat disease resistance breeding research from 2000 to 2016
2.4.2 基于關鍵詞的研究主題分析 通過TDA軟件對2 580篇關于信息技術在小麥抗病育種領域應用文獻的關鍵詞進行分析,結果(表2)顯示,研究方向主要集中分子標記和遺傳標記,病害,病菌毒素,抗病性,以及抗冷和耐熱生理5個方面。采用的研究技術主要包括各種軟件應用、SNP芯片技術、SSR、AFLP、RAPD等分子標記技術;分析方法主要包括模型分析和Meta-analysis分析等。其中,QTL(quantitative trait loci)詞頻最高。
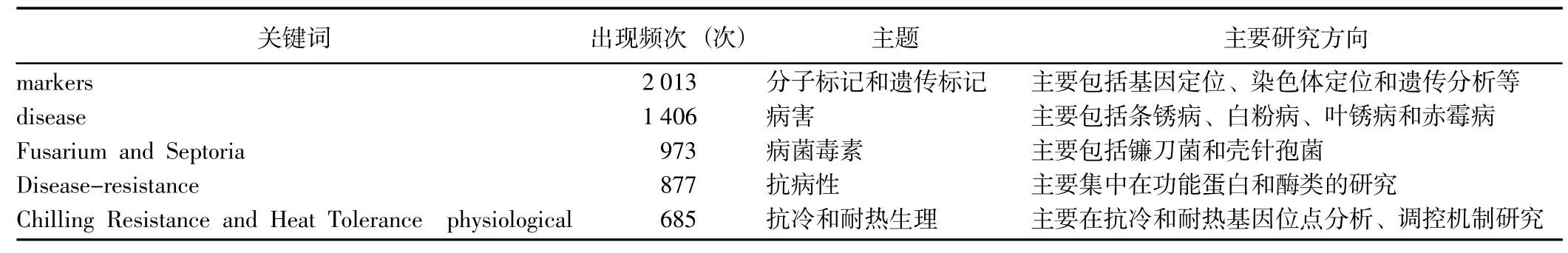
表2 基于關鍵詞的研究主題分析Table 2 The analysis on the research topics based on key words
2.5 研究性論文文獻被引頻次分析
研究性論文的被引頻次能夠反映出該研究領域的技術熱點及技術演進情況。通過對2 580篇關于信息技術在小麥抗病育種領域應用文獻的被引頻次進行統計分析,結果(表3)顯示,被引頻次排在前10位的文獻有4篇來自美國,加拿大、中國、以色列、澳大利亞、法國和墨西哥各1篇。Somers D J等[6](加拿大)2004年在《Theoretical and Applied Genetics》 上發表的論文“A high-density microsatellite consensus map for bread wheat(Triticum aestivum L.)”被引頻次最高(1 040次),作者提出了應用MapMaker V3.0和joinMapV3.0軟件對不同面包小麥雜交后代群體的特異位點GWM、GDM、CFA、CFD和BARC等開發了高密度微衛星基因定位遺傳圖譜。O'Donnell K等[7](美國) 2000年在 《Proceedings oftheNational Academy of Sciences of the United States of America》上發表的論文“Genegenealogiesrevealglobal phylogeographic struct ure and reproductive isolation among lineages of Fusarium graminearum,the fungus causing wheat scab”被引頻次居第2位,作者分析了鐮刀菌屬包含的7 120個核苷酸序列組合數據集的差異,通過基因重組揭示了自然界中7個譜系的菌種在自然界進化分支和地理變化及全球多樣性關系。Kashkush K等[8](以色列)2002年在《Genetics》上發表的論文“Gene loss,silencing and activation in a newly synthesized wheat allotetraploid”被引頻次居第3位,主要介紹了小麥異源四倍體包括抗病基因的DNA序列在遠緣雜交過程中引起基因沉默和激活的機理。Jia J(中國)等[9]2013年在《Nature》上發表的論文“Aegilops tauschii draft genome sequence reveals a gene repertoire for wheat adaptation”被引頻次居第4位,作者應用生成的全面的六倍體小麥RNA-Seq數據鑒定了43 150個蛋白質編碼基因,并對其中的30 697個基因定位到高密度基因圖譜染色體上。分析了面包小麥中抗病性基因家族、小麥品質性狀基因家族以及生物脅迫耐受性基因家族的表達關系。Cavanagh C R等[10](澳大利亞) 2013年在 《Proceedings ofthe National Academy of Sciences of the United States of America》上發表的論文“Genome-wide comparative diversity uncovers multiple targets ofselection for improvement in hexaploid wheat landraces and cultivars”被引頻次居第5位,作者介紹開發了1種高通量陣列,對全球范圍內的2 994個份小麥品種基因相關單核苷酸多態性(SNP)進行了檢測,使用基于SNP的多樣性圖譜描述了作物改良對遺傳多樣性的基因組和地理模式的影響。
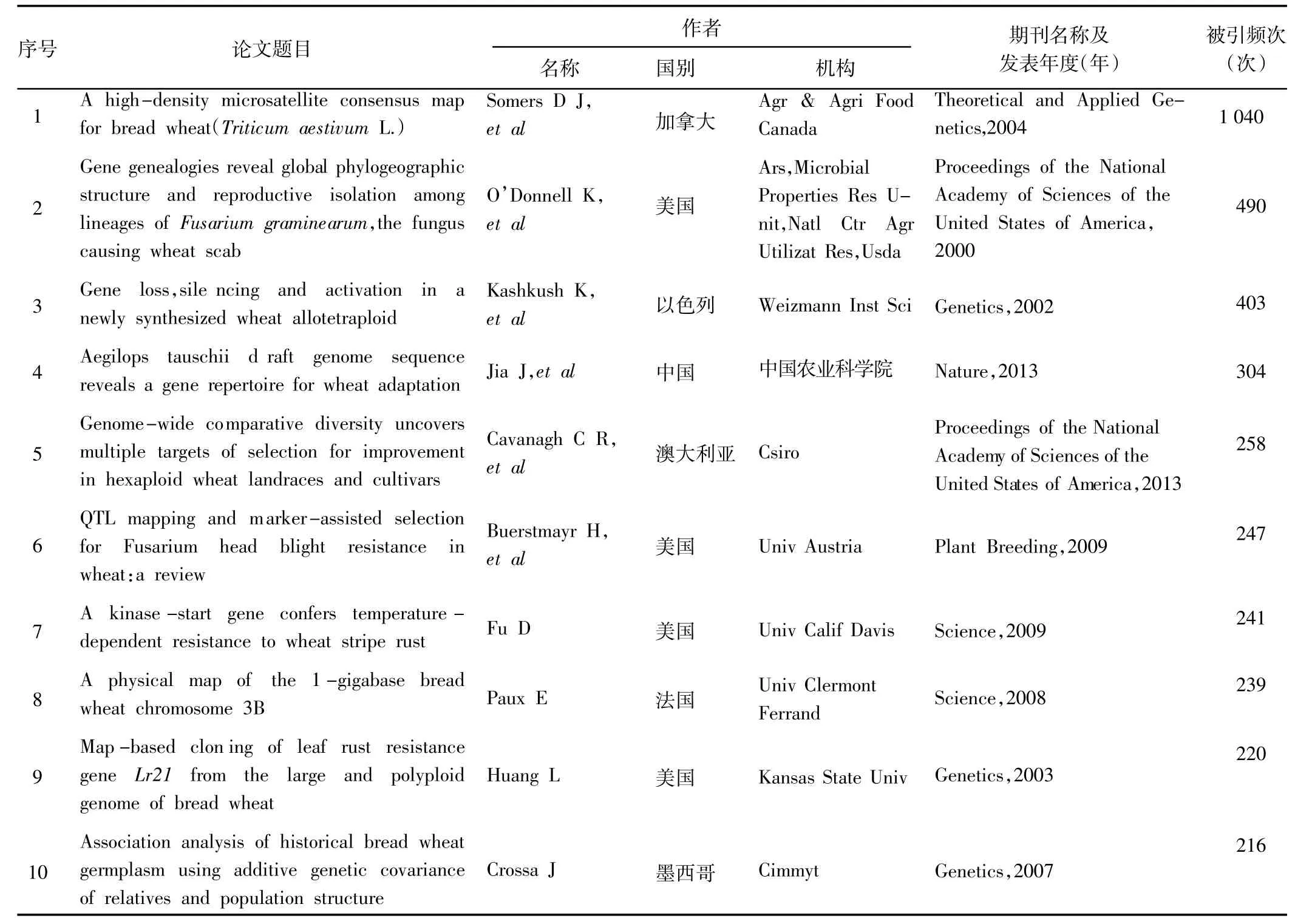
表3 小麥抗病育種單篇論文被引頻次排名前10位的論文Table 3 The top 10 cited papers of information technology applied to wheat disease resistance breeding research
3 結論與討論
通過對SCI數據庫2000~2016年關于信息技術應用于小麥抗病育種領域的發文量進行分析,得出以下結論:
(1)2000~2016年該領域論文數量平均年增長率為8.74%,其中,2016年我國總發文量居第1位,平均年增長速率達17.4%,增長速率最高。前20位研究機構中,我國有5個機構,且僅中國農業科學院排名第3位,其他國內機構排名均在11~19位。
(2)通過對主題分析和高被引文獻分析,信息技術應用于小麥抗病育種領域研究,采用的信息技術及分析方法主要為應用軟件、SNP芯片技術及SSR、AFLP、RAPD等基因分析方法,進行小麥條銹病、白粉病、葉銹病和赤霉病等病害的QTL(quantitative trait loci)分析及基因型分析,包括基因定位、基因功能分析、遺傳圖譜分析等。
綜上所述,近年來信息技術在小麥抗病育種領域的研究活躍度較高。我國各機構發文量較為分散,除中國農業科學院擁有較強優勢外,其他機構研究力量比較薄弱。應重點支持應用軟件技術、SNP芯片技術開展的小麥抗病高密度遺傳圖譜基因定位研究,以及功能基因調控機理挖掘的深度研究。
[1]張才明.信息技術的概念和分類問題研究[J].北京交通大學學報:社會科學版,2008,7(3):89-92.
[2]顏志輝,鄭懷國,趙靜娟,串麗敏,張曉靜,譚翠萍,孫素芬.基于SCI論文的作物轉基因育種領域發展態勢分析[J].中國農業科技導報,2016,18(2):208-215.
[3]許 麗,王 玥,李禎祺,蘇 燕.基因組編輯育種技術及國內外發展態勢分析[J].農業生物技術,2016,(4):46-51.
[4]王慧媛,阮梅花,王 方,劉 曉.作物分子設計育種的發展態勢分析[J].農業生物技術,2013,(6):42-50.
[5]靳軍寶,高 峰,古志文,鄭玉榮,田曉陽.基于DII的生物育種專利技術國際態勢分析[J].中國農業科技導報,2015,17(4):176-180.
[6] Somers D J,Lsaac P,Edwards K.A high-density microsatellite consensus map for bread wheat(Triticum aestivum L.)[J].Theoretical and Applied Genetics,2004, 109 (6):1105-1114.
[7] O'Donnell K,Kistler H C,Tacke B K,Casper H H.Gene genealogies reveal global phylogeographic structure and reproductive isolation among lineagesofFusarium graminearum, the fungus causing wheat scab[J].Proceedings of the National Academy of Sciences of the U-nited States of America,2000,97 (14):7905-7910.
[8] Kashkush K,Feldman M,Levy A A.Gene loss,silencing and activation in a newly synthesized wheat allotetraploid [J].Genetics,2002,160 (4):1651-1659.
[9] Jia J Z,Zhao S C,Kong X Y,Li Y R,Zhao G Y,He W M,Appels R,Pfeifer M,Tao Y,Zhang X Y.Aegilops tauschii draft genome sequence reveals a gene repertoire for wheat adaptation[J].Nature,2013,496(7443):91-95.
[10] Cavanagh C R,Chao S M,Wang S C.Genome-wide comparative diversity uncovers multiple targets of selection for improvement in hexaploid wheat landraces and cultivars[J].Proceedings of the National Academy of Sciences of the United States of America,2013,110(20):8057-8062.

