EPAS1、ANGPTL4基因多態性與慢性高原病遺傳易感性※
胥 瑾,王 展,胡 蕾,張 婉,楊應忠
(1.青海大學醫學院基礎醫學部;2.青海大學附屬醫院肝膽外科;3.四川大學2014級華西臨床醫學院護理系;4.青海大學醫學院中醫系13級中醫班;5.青海大學醫學院高原醫學研究中心)
EPAS1、ANGPTL4基因多態性與慢性高原病遺傳易感性※
胥 瑾1,王 展2,胡 蕾3,張 婉4,楊應忠△5
(1.青海大學醫學院基礎醫學部;2.青海大學附屬醫院肝膽外科;3.四川大學2014級華西臨床醫學院護理系;4.青海大學醫學院中醫系13級中醫班;5.青海大學醫學院高原醫學研究中心)
目的 探討EPAS1和ANGPTL4基因的遺傳多態性是否和慢性高原病(Chronic mountain sickness,CMS)易感性有關。方法 采用病例對照研究方法,將30例漢族CMS患者(CMS-s)、51例健康漢族者(CMS-r)和131例健康藏族者(HLT)作對照研究,所有研究對象都來自青海平均海拔3 760米的玉樹地區。采用Sequenom MassARRAY 法檢測研究對象EPAS1基因的SNP位點rs4953388和ANGPTL4基因的SNP位點rs2278236基因型分布,比較各SNP位點在三組之間的差異。結果EPAS1基因的SNP位點rs4953388基因型GG在CMS-s和 CMS-r兩組分別有28例(93.4%)、32例(62.7%),P=0.004。rs4953388的基因型AA在HLT組的頻率顯著高于CMS-s、CMS-r組,分別為57例(44.2%)、1例(3.3%),P=0.0001;57例(44.2%)、5例(9.8%),P=0.0001。ANGPTL4基因的SNP位點rs2278236的基因型CC在CMS-s和 CMS-r兩組的分布頻率有顯著性差異,分別為28例(93.4%)、39例(78.5%),P=0.027。基因型CT在HLT組的分布頻率明顯高于CMS-s組,分別為28例(21.7%)、1例(3.3%),P=0. 022。結論EPAS1基因和ANGPTL4基因的單核苷酸多態性不僅在CMS患者與同海拔健康漢族之間存在很大差異,而且在健康藏族和漢族人群中也存在巨大差異。這兩個基因的遺傳易感性既和CMS的易感性相關,又和藏、漢族對低氧環境的適應差異有關。
多態性EPAS1ANGPTL4 慢性高原病 易感性
青藏高原是世界上海拔最高的高原之一,慢性高原病(Chronic mountain sickness,CMS)發生在5%~18%的青藏高原居民中[1],其中大部分為移居高原的漢族[2]。雖然高原低氧環境被認為是高原居民發生CMS的始動因素,但CMS的具體發病機制還不十分清楚。
EPAS1、ANGPTL4(Endothelin receptors type A)基因是被XP-EHH(Cross Population Extended Haplotype Homozogysity)法和iHS(integrated haplotype score)法檢測出與藏族低氧適應相關的正選基因[3]。是否CMS患者、移居高原的漢族和世居高原的藏族健康者具有不同的EPAS1、ANGPTL4基因基因型,是否EPAS1、ANGPTL4基因的多態性和CMS的易感性相關尚不十分清楚。本研究為了明晰EPAS1、ANGPTL4基因的多態性與CMS易感性間的可能關系,檢測了青海玉樹地區(海拔3700米以上)30例CMS患者(Chronic mountain sickness-susceptibility,CMS-s)、51例漢族健康者(Chronic mountain sickness-resistance,CMS-r)和131例藏族健康者(High altitude Tibetan,HLT)的EPAS1基因的SNP位點rs4953388、ANGPTL4基因的SNP位點rs2278236。
1 對象與方法
1.1 研究對象選擇
收集青海玉樹地區57例漢族CMS者(CMS-s,男性27例,女性30例,平均年齡42.89±8.17歲)、51例漢族健康者(CMS-r,男性44例,女性7例,平均年齡40.51±10.07 歲)和129例藏族健康對照者(HLT,男性74例,女性55例,平均年齡45.14±11.78歲)資料,漢族CMS和漢族健康者都為移居玉樹地區時間≥3年以上者,藏族健康者都為世居玉樹的青藏高原康巴藏族。CMS患者是在2011年5月至2013年6月在玉樹州人民醫院被診斷為CMS者。納入標準為青海標準[2],男性血紅蛋白濃度≥21 g/dL,女性血紅蛋白濃度≥19 g/dL,并且伴有三種或更多以下癥狀:氣短、心悸、睡眠紊亂、紫紺、頭疼、耳鳴及感覺異常等。隨機選擇年齡、性別、工作條件和CMS組病例一致的在門診常規體檢的健康漢族、藏族者作為對照組。用邁德血液分析儀(BC-2300,深圳,中國)檢測靜脈血的血紅蛋白(Hb)、紅細胞壓積(HCT);脈搏氧飽和度儀(Ohmeda 3700 Pulse Oximeter,Datex-Ohmeda,Boulder,Colorado,USA)檢測血氧飽和度。所有的研究對象都沒有呼吸病史和心臟病史。該項研究獲得青海大學醫學院倫理委員會批準。
1.2 DNA提取和分型檢測
采用Gentra Puregene全血試劑盒(Qiagen, Germany),按照標準步驟從受試者的靜脈血中提取基因組DNA。用分光光度計定量,瓊脂糖凝膠電泳質檢,基因組DNA電泳條帶通常不小于20 kb。將質檢合格的DNA將濃度調整到 50 ng/μL,轉移至 384 孔板,-20 ℃儲存備用。采用Sequenom MassARRAY?SNP(博奧生物集團有限公司)分型檢測方法,經過多重PCR技術、MassARRAY iPLEX單堿基延伸技術和基質輔助激光解吸附電離飛行時間質譜分析質譜技術進行分型檢測。將包含SNP位點區域的DNA模板通過PCR技術擴增,再使用特異的延伸引物與PCR產物進行單堿基延伸反應。由于多態性位點堿基不同,延伸產物不同的末端堿基將導致延伸后的產物分子量的差異,由SNP多態性引起的堿基差異通過分子量的差異體現出來,使用基質輔助激光解吸附電離飛行時間質譜分析質譜技術檢測延伸產物分子量的大小進行SNP分型。使用Sequenom公司Genotyping Tools、MassARRAY Assay Design軟件設計待測SNP位點的PCR擴增引物及單堿基延伸引物。
1.3 統計學方法
2 結果
2.1 一般表型資料結果
CMS患者和對照組的平均年齡、性別及動脈氧飽和度(SaO2)、血紅蛋白(Hb)和血細胞比容(HCT)見表1。三組年齡做顯著性檢驗無差異,性別做顯著性檢驗發現男性CMS的發病率比女性高,符合已知的CMS在男性中高發的傾向[2]。CMS-s組和CMS-r組、HLT組比較,SaO2明顯降低,Hb和HCT明顯升高(P<0.05)。
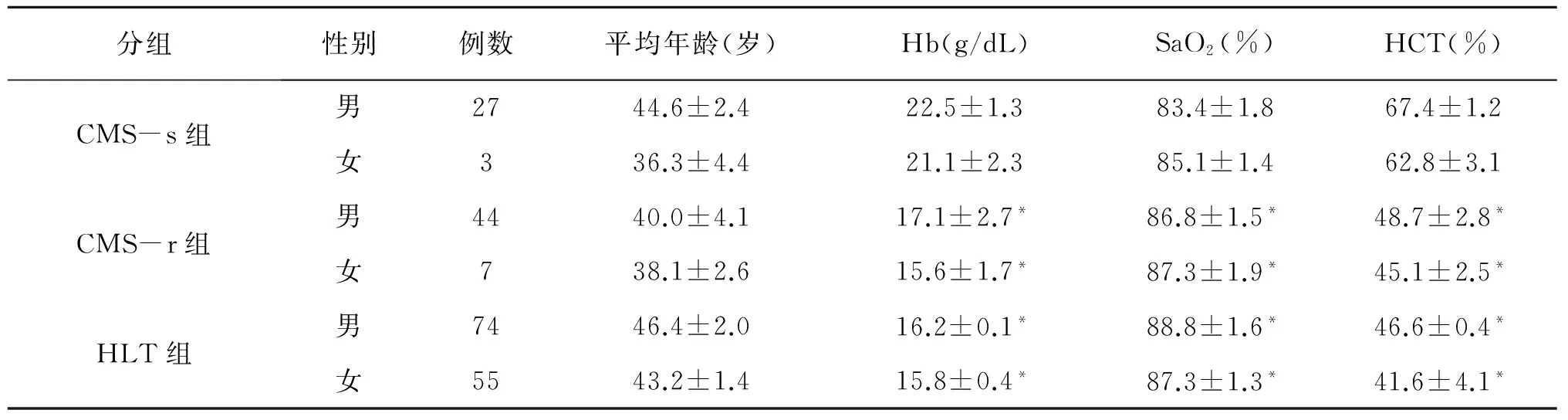
表1 CMS-s組、CMS-r組和HLT組的一般表型資料Table 1 Phenotype parameters of the CMS-s,CMS-r and HLT group
*:與CMS-s組同性別比較,P<0.05.
2.2 基因型和等位基因的分布結果
CMS-s組和對照組都符合Hardy-Weinberg平衡分布。我們比較了基因型、等位基因頻率分布及比值比(OR)、95%可信區間(95%CI)發現,EPAS1基因的SNP位點rs4953388的基因型GG在CMS-s和CMS-r兩組之間有明顯差異,見表2。該基因型人群CMS發病的風險率高[OR(95%):12.250(1.513-99.158),P=0.004]。同時,等位基因G在CMS-s組中的頻率明顯高于CMS-r組[OR(95%):5.846(1.679-20.362),P=0.002]。rs4953388的基因型AA在HLT組的頻率顯著高于CMS-s、CMS-r組,分別為[133.000(16.458-1074.797),P<0.0001]和[30.400(9.825-94.064),P<0.0001]。等位基因A在HLT組中的頻率顯著高于CMS-s、CMS-r ,分別為[39.357(11.975-129.348),P<0.0001]、[6.732(3.977-11.397),P<0.0001]。
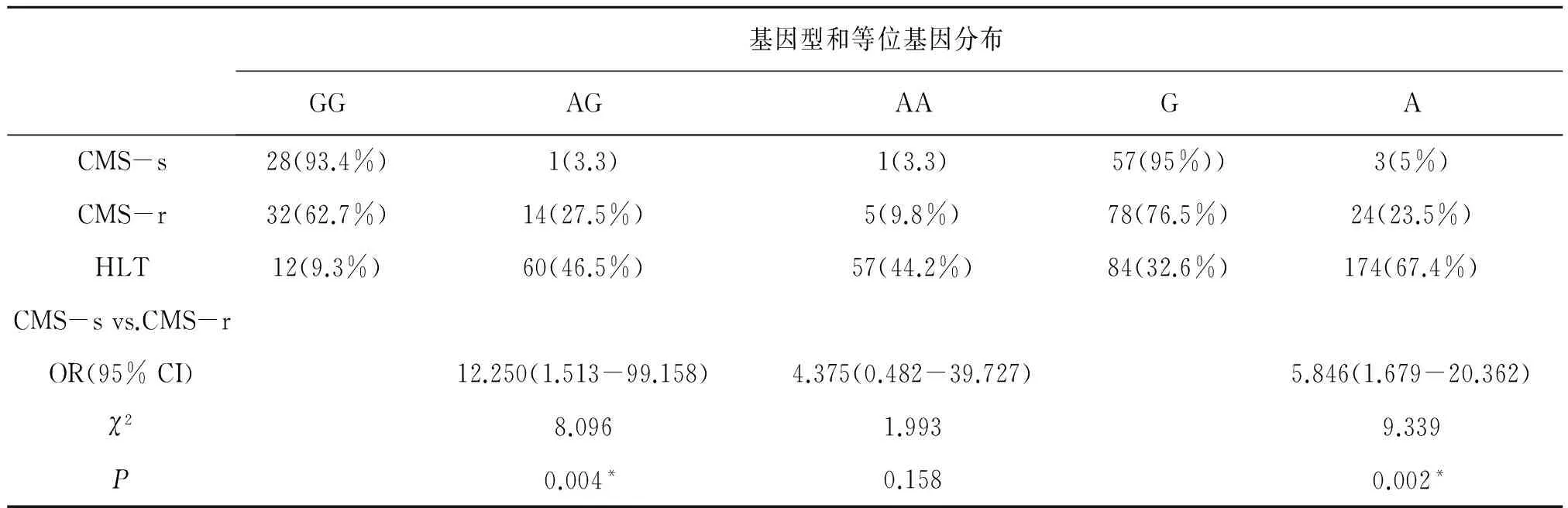
表2 rs4953388在CMS-s組、CMS-r組和HLT組基因型和等位基因的頻率分布及比較[例(%)]Table 2 Comparison of rs4953388 genotype distributions,allele frequencies in CMS-s,CMS-r and HLT groups
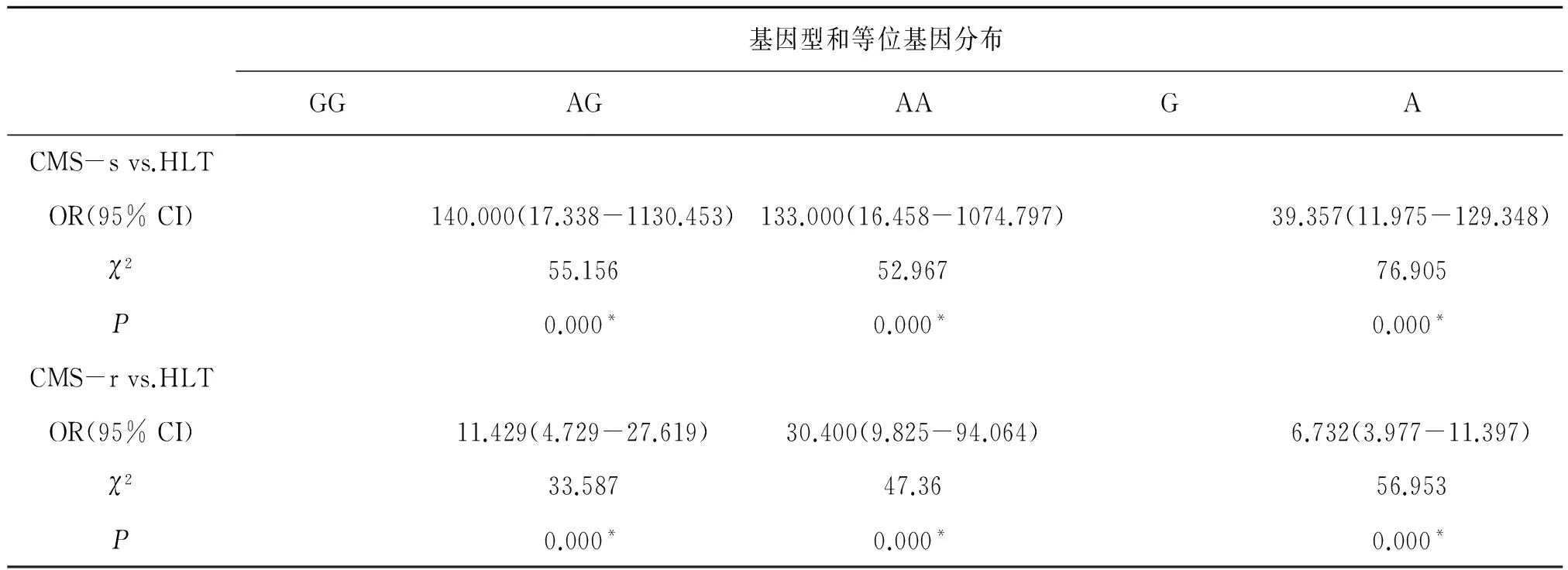
續表:
*:P<0.05.
ANGPTL4基因的SNP位點rs2278236的基因型CC在CMS-s和 CMS-r之間有顯著性差異,見表3。該基因型人群CMS發病的風險率高[OR(95%):7.897(0.963-64.744),P=0.027]。基因型CT在HLT組的頻率顯著高于CMS組,為7.762(1.011-59.585),P<0.022。
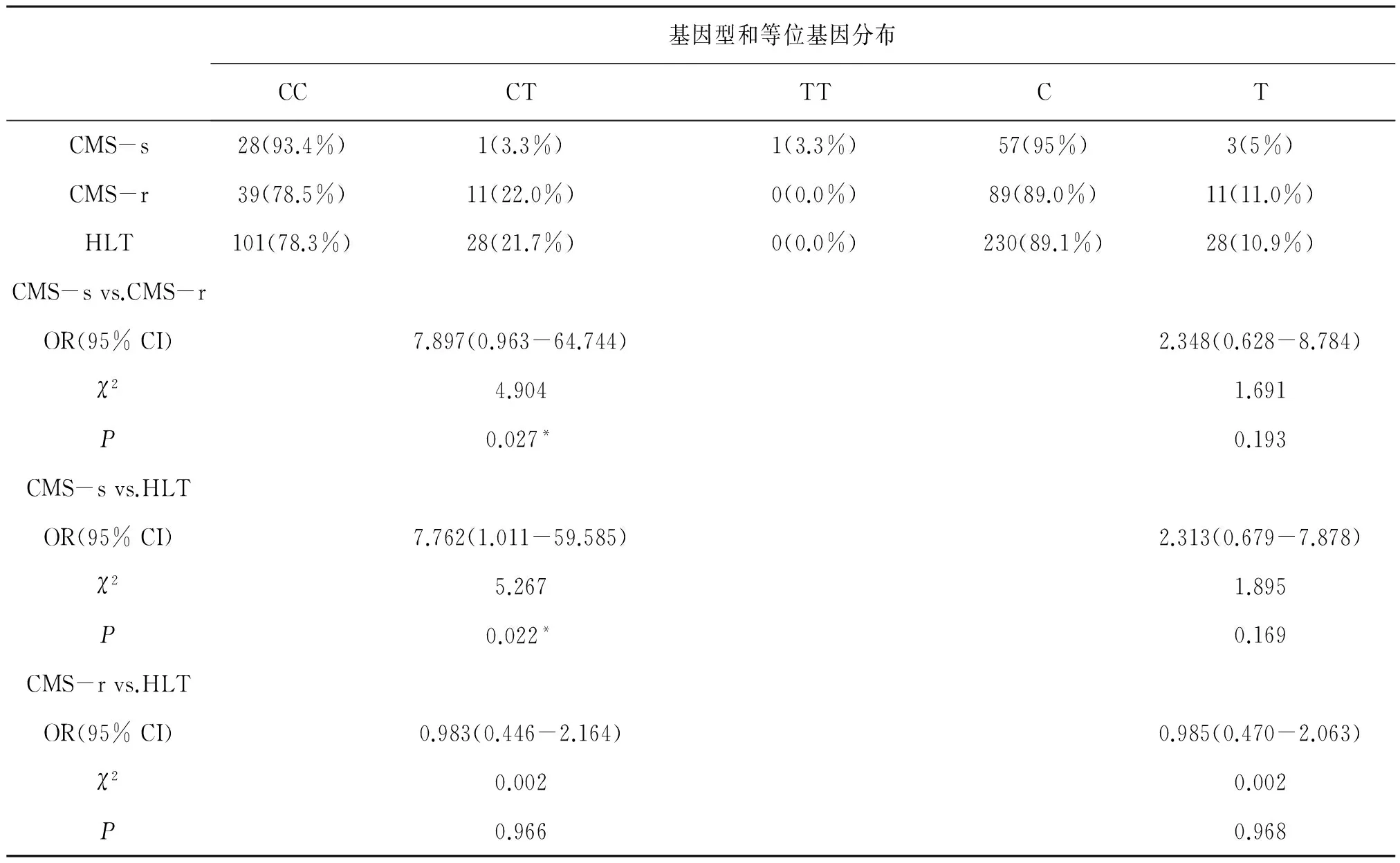
表3 rs2278236在CMS-s組、CMS-r組和HLT組基因型和等位基因的頻率分布及比較[例(%)]Table 3 Comparison of rs2278236 genotype distributions,allele frequencies in CMS-s,CMS-r and HLT groups
*:P<0.05.
3 討論
我們在青海玉樹地區采集了明確臨床診斷為CMS患者和同海拔漢族健康、藏族健康對照者的外周血白細胞DNA,檢測了三組研究對象的DNA序列中EPAS1基因的SNP位點rs4953388和ANGPTL4基因的SNP位點rs2278236的多態性,通過統計學分析(比較基因型和等位基因分布的兩組差異)發現,這兩個SNP的分布頻率在CMS患者組和對照組中存在顯著差異。國內外報道認為,無論是移居青藏高原的漢族還是移居南美洲的西班牙人,CMS在這些人群中的發病率明顯高于當地的世居者。相反,青藏高原的世居的藏族和夏爾巴人極少發生CMS,提示CMS發病具有明顯的種族差異和個體易感傾向[4]。因此,本研究比較世居藏族和移居高原漢族CMS的遺傳易感基因多態性正是從遺傳學角度解釋CMS發病率的差異。另外,誘發CMS形成的基因編碼產物在CMS患者和高原健康人群之間的表達水平差異明顯,造成這種差異的最根本原因可能在于這些基因CMS易感者體內存在特異的基因型[5]。與CMS發病相關的基因大多在呼吸和循環系統的氧感受作用中發揮重要功能,而這些基因也和藏族的高原適應性密切相關,其中就包括EPAS1基因和ANGPTL4基因。
人的EPAS1基因位于2p16-21,是重要的低氧相關基因。藏族包含EPAS1的基因組區域表現出明顯的多態性[6],多篇報道證實了其多態性和藏族體內較低的血紅蛋白濃度密切相關[7-9]。最近的研究顯示,EPAS1基因與高原世居藏族人群高原適應性密切相關,與血紅蛋白濃度高度關聯的EPAS1基因單核苷酸多態位點在藏族和漢族人群中存在巨大差異[10]。我們的研究也證實了這一點:EPAS1基因的SNP位點rs4953388的基因型AA在健康藏族(HLT)組的頻率顯著高于CMS-s、CMS-r組,分別為[140.000(17.338-1130.453),P<0.0001]、[11.429(4.729-27.619),P<0.0001]。
同時,等位基因A在HLT組中的頻率顯著高于CMS-s組和CMS-r,分別為[39.357(11.975-129.348),P<0.0001]、[6.732(3.977-11.397),P<0.0001]。我們還發現了EPAS1基因的SNP位點rs4953388的基因型GG和等位基因G在CMS-s和CMS-r兩組之間有明顯差異,分別為[OR(95%):12.250(1.513-99.158),P=0.004]、[OR(95%):5.846(1.679-20.362),P=0.002]。說明EPAS1基因的單核苷酸多態性不僅在藏族健康和漢族健康人群中存在巨大差異,同時在CMS患者和同海拔漢族健康者之間也存在很大差異,這和Hanaoka等[11]的關于EPAS1基因的多態性與高原疾病、高原適應的關聯性研究結果一致。CMS患者和健康藏族的EPAS1基因上有幾個SNP位點的等位基因分布表現出明顯的不同[12],其中rs12619696、rs13419896、rs4953354和rs1868092等SNP位點被多次報道[13,14]。EPAS1基因的啟動子功能區域核苷酸序列特異性和藏族的高原適應性顯著相關[15],說明EPAS1基因的多態性的確能夠影響機體在高原低氧下的生理反應,CMS患者群體由于在EPAS1基因的某些SNP位點上存在和高原適應民族顯著不同的基因型,使他們不能夠適應長期低壓低氧環境,從而出現血紅蛋白濃度過高的現象。
人ANGPTL4基因位于19p13.3,參與機體血管生成、脂肪代謝等過程[16]。Buroker等[17]發現ANGPTL4基因的多個SNP位點和急性高原病密切相關,我們的研究證實該基因SNP位點rs2278236和CMS的易感性也是密切相關的:ANGPTL4基因的SNP位點rs2278236的CC基因型人群的CMS發病的風險率高:[OR(95%):7.897(0.963-64.744),P=0.027]。同時,基因型CT在HLT組的頻率明顯高于CMS-s組[7.762(1.011-59.585),P=0.022]。說明ANGPTL4基因遺傳易感性既和CMS的易感性相關,又和藏、漢族對低氧環境的適應差異有關。
本研究采用的Sequenom基因分型技術是中等通量SNP分型的代表技術,其核心技術為基質輔助激光解吸電離飛行時間質譜(MALDI-TOF MS)技術。檢測原理為PCR擴增后的產物,加入SNP序列特異延伸引物,在需要檢測的SNP位點上,延伸1個堿基,再用質譜儀檢測其分子量。同一SNP位點不同等位基因的延伸產物至少具有16Da的分子量差異,因此可以檢測到該位點的不同基因型。這種分型方法分析準確:可重復性高,直接檢測待測物分子量,準確度超過99.7%。同時該法定制靈活,樣本量的大小、SNP位點的位置和數量等都可以自由選擇。
CMS是高原低氧的環境因素和許多微效致病基因共同作用的結果,其中誘導CMS形成的基因產物在CMS患者和高原習服人群之間的表達存在顯著性差異[18]。本研究證實了EPAS1基因的SNP位點rs4953388及ANGPTL4基因的SNP位點rs2278236的多態性和CMS的易感性密切相關,為CMS的進一步預防和治療提供了新的理論線索。這兩個和CMS遺傳易感性密切相關的基因,需要進一步在動物實驗、細胞學實驗等多層面深入研究,從而更深層次闡明其在CMS發生機制中發揮的作用。
[1]Windsor JS,Rodway GW.Heights and haematology:the story of haemoglobin at altitude[J].Postgrad Med J,2007,83:148-151.
[2]Murphy WG.The sex difference in haemoglobin levels in adults-mechanisms,causes,and consequences[J].Blood Rev,2014,28:41-47.
[3]Simonson TS,Yang Y,Huff CD,et al.Genetic evidence for high-altitude adaptation in Tibet[J].Science,2010,329:72-75.
[4]Bigham A,Bauchet M,Pinto D,et al.Identifying signatures of natural selection in Tibetan and Andean populations using dense genome scan data[J].PLoS Genet,2010,6(9):e1001116.
[5]Scheinfeldt LB,Soi S,Thompson S,et al.Genetic adaptation to high altitude in the Ethiopian highlands[J].Genome Biol,2012,13:R1.
[6]Huerta-Sanchez E,Jin X,Asan,et al.Altitude adaptation in Tibetans caused by introgression of Denisovan-like DNA[J].Nature,2014,512:194-197.
[7]Wang B,Zhang YB,Zhang F,et al.On the origin of Tibetans and their genetic basis in adapting high-altitude environments[J].PLoS One,2011,6:e17002.
[8]Xu S,Li S,Yang Y,et al.A genome-wide search for signals of high-altitude adaptation in Tibetans[J].Mol Biol Evol,2011,28:1003-1011.
[9]Yi X,Liang Y,Huerta-Sanchez E,et al.Sequencing of 50 human exomes reveals adaptation to high altitude[J].Science,2010,329:75-78.
[10]Peng Y,Yang Z,Zhang H,et al.Genetic variations in Tibetan populations and high-altitude adaptation at the Himalayas[J].Mol Biol Evol,2011,28:1075-1081.
[11]Hanaoka M,Droma Y,Basnyat B,et al.Genetic variants inEPAS1 contribute to adaptation to high-altitude hypoxia in Sherpas[J].PLoS One,2012,7:e50566.
[12]Buroker NE,Ning XH,Zhou ZN,et al.EPAS1 and EGLN1 associations with high altitude sickness in Han and Tibetan Chinese at the Qinghai-Tibetan Plateau[J].Blood Cells Mol Dis,2012,49:67-73.
[13]Xu J,Yang YZ,Tang F,et al.EPAS1 Gene Polymorphisms Are Associated With High Altitude Polycythemia in Tibetans at the Qinghai-Tibetan Plateau[J].Wilderness Environ Med,2015,26,288-294.
[14]Chen Y,Jiang C,Luo Y,et al.AnEPAS1 haplotype is associated with high altitude polycythemia in male Han Chinese at the Qinghai-Tibetan plateau[J].Wilderness Environ Med,2014,25:392-400.
[15]Xu XH,Huang XW,Qun L,et al.Two functional loci in the promoter ofEPAS1 gene involved in high-altitude adaptation of Tibetans[J].Sci Rep,2014,4:7465.
[16]Catoire M,Alex S,Paraskevopulos N,et al.Fatty acid-inducibleANGPTL4 governs lipid metabolic response to exercise[J].Proc Natl Acad Sci U S A,2014,111:E1043-1052.
[17]Buroker NE,Ning XH,Zhou ZN,et al.AKT3,ANGPTL4,eNOS3,and VEGFA associations with high altitude sickness in Han and Tibetan Chinese at the Qinghai-Tibetan Plateau[J].Int J Hematol,2012,96:200-213.
[18]王亞平,格日力.高原衰退癥的研究進展[J].中華醫學雜志,2012,92:2298-2300.
Association ofEPAS1 andANGPTL4 gene polymorphisms with susceptibility of CMS
XU Jin1,WANG Zhan2,HU Lei3,ZHANG wan4,YANG Ying-zhong△5
(1.Department of Basic Medical Science,Qinghai University School of Medicine; 2.Department of Hepatic-biliary-pancreatic Surgery,Qinghai university affiliated hospital; 3.Sichuan University Huaxi clinical medical college nursing department,Grade 2014; 4.Qinghai University School of Medicine,traditional Chinese medicine department,Grade 2013; 5.Research Center for High Altitude Medical Sciences,Qinghai University School of Medicine)
Objective To test whether the polymorphisms inEPAS1 gene andANGPTL4 gene are associated with susceptibility to CMS(Chronic mountain sickness,CMS).Methods We enrolled 30 Han CMS patients(CMS-s),51 and 131 healthy,age-and gender-matched control Hans(CMS-r)and Tibetans(HLT).All subjects are from the Yushu area of Qinghai where the altitude is over 3 500 meters,rs4953388 ofEPAS1 gene and rs2278236 ofANGPTL4 gene were genotyped by the Sequenom Mass ARRAY SNP assays.Results We discovered GG genotype of rs4953388 inEPAS1 gene were significantly different between CMS-s group and CMS-r group[28(93.4%)vs 32(62.7%),P=0.004].AA genotype of rs4953388 in HLT group had higher prevalence than that in CMS-s group and CMS-r group[57(44.2% vs 1(3.3%),P<0.0001;57(44.2%)vs 5(9.8%),P<0.0001 ].Additionally,CC genotype of rs2278236 inANGPTL4 gene were significantly different between CMS-s group and CMS-r group[28(93.4%)vs 39(78.5%),P=0.027].CT genotype of rs2278236 in HLT group had higher prevalence than that in CMS-s group[28(21.7%)vs 1(3.3%),P=0. 022].Conclusions Genetic variations inEPAS1、ANGPTL4 gene are not only associated with susceptibility to CMS but also nay have adaptive benefits for high altitude environment in Tibetans.
PolymorphismEPAS1ANGPTL4 CMS susceptibility
※:國家自然科學基金項目(81641078),青海省科技廳自然科學青年基金項目(2016-ZJ-924Q); 青海大學醫學院中青年基金項目(2013-KY-1).Δ:通信作者,教授,碩士生導師,E-mail:81420209@qq.com 胥瑾(1987~),女,土族,青海籍,博士
R363.1
A
10.13452/j.cnki.jqmc.2017.01.004
2015-12-13

