飛蝗內表皮蛋白基因LmAbd-5的表達與功能分析
趙小明,賈盼,2,勾昕,2,劉衛敏,馬恩波,張建珍
(1山西大學應用生物學研究所,太原 030006;2山西大學生命科學學院,太原 030006)
飛蝗內表皮蛋白基因LmAbd-5的表達與功能分析
趙小明1,賈盼1,2,勾昕1,2,劉衛敏1,馬恩波1,張建珍1
(1山西大學應用生物學研究所,太原 030006;2山西大學生命科學學院,太原 030006)
【目的】基于飛蝗(Locusta migratoria)轉錄組數據庫獲得內表皮蛋白(endocuticle structural glycoprotein)基因LmAbd-5的cDNA序列,分析該基因的序列特征和mRNA表達特性,采用RNAi方法分析其生物學功能,探討其在飛蝗表皮形成中的作用,為害蟲防治提供新的分子靶標。【方法】采用生物信息學方法搜索飛蝗轉錄組數據庫,獲得LmAbd-5 cDNA全長序列并克隆驗證;采用SignalP在線軟件分析蛋白的信號肽,利用SMART網站預測其功能域,使用MEGA 7.0軟件中neighbor-joining(NJ)方法,與其他昆蟲同源序列進行聚類分析;采用reverse-transcription quantitative PCR(RT-qPCR)方法檢測LmAbd-5在飛蝗5齡第2天若蟲不同組織部位和5齡不同發育時期體壁組織中的表達情況,揭示其組織和時期表達模式;采用RNA干擾(RNAi)技術及透射電鏡技術(TEM)觀察沉默LmAbd-5后對飛蝗生長發育和表皮結構的影響。【結果】通過搜索得到LmAbd-5 cDNA全長序列并進行了克隆和測序驗證,獲得520 bp全長cDNA序列,其中ORF為303 bp;基因結構分析顯示該基因含有3個外顯子;功能域分析發現其含有1個信號肽和1個幾丁質結合域(chitin binding domain 4,ChtBD4),與沙漠蝗、白蟻、赤擬谷盜等的Abd-5結構類似;BLAST分析結果表明Abd-5在昆蟲中高度保守,飛蝗與沙漠蝗Abd-5序列一致度高達81%;對其保守基序進行WebLogo分析發現LmAbd-5屬于表皮蛋白CPR家族中的RR-1亞類;聚類分析結果顯示,LmAbd-5與沙漠蝗和白蟻的Abd-5顯示出較近的親緣關系;RT-qPCR結果顯示LmAbd-5在前腸、后腸、氣管和體壁等由外胚層形成的組織中高表達,而在胃盲囊、中腸、馬氏管、脂肪體和翅芽中低表達或不表達;不同時期表達分析發現,LmAbd-5在4齡若蟲蛻皮后0—72 h(5齡早期)具有高表達,其中蛻皮后72 h時達到最大表達量,隨后表達量急劇降低(96—168 h),其表達時期與內表皮形成時間一致;采用RNAi技術分析該基因的生物學功能,對5齡第2天若蟲分別注射等量的dsLmAbd-5和dsGFP(對照),發現注射dsLmAbd-5的5齡若蟲和對照組相同,均可正常蛻皮,發育至成蟲第2天目的基因表達量顯著降低,但未出現肉眼可見的異常表型。分別取成蟲第2天處理組和對照組成蟲表皮進行超微結構觀察,發現與對照組相比,處理組成蟲內表皮片層結構較為疏松,導致內表皮片層變厚,最終表現為整個內表皮變厚。【結論】根據轉錄組數據庫分析獲得1個CPR家族表皮蛋白LmAbd-5,該蛋白含有1個信號肽和1個幾丁質結合域ChtBD4,屬于RR-1亞類;LmAbd-5主要在外胚層起源的組織中高表達,沉默LmAbd-5后飛蝗沒有肉眼可見的表型,但超微結構分析發現其參與飛蝗內表皮片層結構的形成。
飛蝗;表皮蛋白;LmAbd-5;RNAi;透射電鏡
0 引言
【研究意義】中國是一個農業生物災害頻發、生態環境脆弱的農業大國,毀滅性的農作物病蟲害頻繁暴發,導致農業經濟損失與環境破壞。飛蝗(Locusta migratoria)作為世界性農業害蟲,具有暴發性、群集性和遷飛性等特點,蝗災發生面積廣,致災嚴重。因此,對飛蝗的研究和蝗災的綜合治理對經濟和民生發展具有重要意義。昆蟲體壁具有重要的保護作用和生理功能,能夠保護蟲體內部結構,是抵御外界環境的第一道防線,在抵御水分蒸發和應對外界不良環境的影響等方面起著重要作用。表皮蛋白是昆蟲體壁的重要組成成分,其成分的缺失或含量變化都可引起表皮功能異常,從而影響昆蟲的生長發育。因此,從飛蝗表皮蛋白分子特性和生理功能入手,探索表皮蛋白在飛蝗表皮結構中的作用,可篩選蝗蟲防治新分子靶標,從而為蝗災有效治理提供新的思路和方法。【前人研究進展】昆蟲體壁具有保護和防御等功能,在生命過程中發揮著重要作用。昆蟲體壁由上表皮、原表皮(內表皮和外表皮)和真皮細胞構成,昆蟲蛻皮包括皮層溶離和蛻去舊表皮兩個連續的過程,昆蟲表皮在蛻皮液作用下首先發生皮層溶離,最先形成表皮質層,然后形成上表皮和外表皮,在上表皮開始沉積的同時,舊表皮逐漸被消化,在舊表皮蛻去后開始合成內表皮[1-3]。原表皮主要成分是幾丁質和蛋白質,它們隨蛻皮過程而周期性的合成和降解。幾丁質是一種多糖類物質,是由N-乙酰胺基葡糖以β-l,4糖苷鍵組成的線性生物聚合物,從真菌界到節肢動物門都大量存在[4]。在自然界中,幾丁質與結構蛋白、酶和抗菌蛋白序列中的幾丁質結合域(chitin-binding domains,CBDs)以非共價鍵形式結合形成糖蛋白,糖蛋白在外表皮中經鞣化后形成堅固穩定的結構。昆蟲表皮蛋白(insect cuticular proteins,ICP)是結構蛋白,根據幾丁質結合域的不同可分為兩類:第一類幾丁質結合域為R&R consensus(Rebers and Riddiford consensus),具有RR-1、RR-2、RR-3 等 3種形式[5-6],此類表皮蛋白富含組氨酸,缺失半胱氨酸一段68個氨基酸的序列,也稱為CPR家族。隨著果蠅(Drosophila melanogaster)、家蠶(Bombyx mori)和赤擬谷盜(Tribolium castaneum)等模式昆蟲全基因組測序相繼完成,ICP的鑒定也隨之展開。學者已在黑腹果蠅[7]、赤擬谷盜[8]、家蠶[9]和煙草天蛾(Manduca sexta)[10]等昆蟲基因組中分別鑒定獲得101、102、148和207個屬于CPR家族的表皮蛋白。目前含有R&R consensus結構域的表皮蛋白與幾丁質的結合能力已得到實驗證實[5,11]。另一類幾丁質結合域是含有6個半胱氨酸殘基,形成3個二硫鍵的保守序列,稱為the type 2 chitin-binding domain (ChtBD2 domain),此類表皮蛋白主要存在于昆蟲表皮和圍食膜中[12-13]。目前,對昆蟲CPR家族表皮蛋白的研究中,以家蠶、果蠅和赤擬谷盜等模式昆蟲的研究較為深入,如日本學者從翅原基化蛹前cDNA庫中隨機選出cDNA進行序列測定,鑒定了10種不同的表皮蛋白基因,命名為BmWCPs[14],韓國全南國立大學Yasuyuki Arakane課題組以赤擬谷盜為對象,研究鞘翅中高豐度的表皮蛋白功能[15-18],這些研究為表皮蛋白在昆蟲變態發育和表皮形成過程的作用機制研究奠定了重要基礎。然而,盡管國內外多個課題組正在開展表皮蛋白在昆蟲表皮重塑過程中的作用及其機制研究,但尚未在超微結構水平開展昆蟲內外表皮蛋白的作用機制研究工作。【本研究切入點】20世紀90年代,JESPERSEN等通過質譜分析方法從飛蝗和沙漠蝗(Schistocerca gregaria)的腹部表皮中分別鑒定出了1個內表皮結構糖蛋白(endocuticle structural glycoprotein),分別命名為LmAbd-5和SgAbd-5,并對其氨基酸序列進行了初步分析[19],然而其生物學功能尚不清楚。【擬解決的關鍵問題】了解飛蝗蛻皮過程中內外表皮的形成規律,合理設計取樣點研究LmAbd-5在不同發育時期的表達特性,明確該基因的表達與內表皮形成的相關性。進一步采用 RNAi技術和透射電鏡技術分析其對飛蝗內表皮發育的影響,為篩選新型分子靶標提供理論和實踐依據。
1 材料與方法
試驗于 2015—2016年在山西大學應用生物學研究所完成。
1.1 試驗材料
供試昆蟲:將筆者實驗室保存的飛蝗蟲卵置于人工氣候箱內孵化,待其孵化為1齡若蟲后轉移至紗籠內置于人工氣候箱中,控制人工氣候箱內環境:溫度為(30±2)℃,相對濕度為(40±10)%,光周期為14 h﹕10 h(L﹕D),以新鮮小麥苗飼喂,3齡后開始輔以麥麩飼喂。
試驗試劑:RNAisoTMPlus,cDNA反轉錄試劑購自TaKaRa公司;pMD18 Cloning Kit購自TransGen Biotech公司;SYBR?Premix EX TaqTM購自TaKaRa公司;PCR Master Mix購自天根公司;E.Z.N.A.? Gel Extraction Kit試劑盒購于Omega公司;T7 RiboMAXTMExpress RNAi System試劑盒購于Promega公司。
1.2 LmAbd-5序列搜索及cDNA全長序列獲得
在筆者實驗室構建的飛蝗轉錄組數據庫中進行搜索,通過NCBI進行BLAST比對、分析,確定LmAbd-5的cDNA部分序列。根據已報道的飛蝗翅轉錄組數據庫對LmAbd-5 cDNA序列進行比對分析[20],獲得cDNA全長序列并設計引物驗證(引物見表1,由上海生工生物公司合成),序列已上傳至 NCBI (GenBank登錄號:KX503039)。LmAbd-5的cDNA序列用ExPASy網站translate tool翻譯后(http://web. expasy.org/translate/),SignaIp4.1Server對其信號肽進行分析(http://www.cbs.dtu.dk/services/ SignalP/),利用SMART分析其結構域(http://smart. embl-heidelberg. de/),利用WebLogo進行保守基序分析(http://weblogo. berkeley.edu/logo.cgi)。
1.3 昆蟲Abd-5系統進化樹的構建
從NCBI網站下載昆蟲Abd-5氨基酸序列,使用MEGA 7.0軟件中neighbor-joining(NJ)方法,將飛蝗LmAbd-5的氨基酸序列與其他昆蟲Abd-5進行聚類分析,進行1 000次獨立分析,數值代表bootstrap估算值。用于構建系統進化樹的物種和 Abd-5的GenBank登錄號見表2。
1.4 LmAbd-5 組織部位和發育時期表達特性
1.4.1 LmAbd-5的組織特異性表達 選用發育整齊的飛蝗5齡第2天若蟲,雌、雄各半,設置3個生物學重復,每個重復4頭蟲體,解剖體壁、前腸、胃盲囊、中腸、后腸、馬氏管、氣管、脂肪體和翅芽等9個組織部位,凍存于液氮中。利用RNAisoTMPlus提取試劑(TaKaRa公司)提取上述不同組織的總RNA,1.5%的瓊脂糖凝膠電泳檢測所提取 RNA的質量,Nanodrop 2000對總RNA進行定量,然后以1 μg總RNA為模板,依據TaKaRa公司反轉錄試劑說明書合成cDNA模板。采用RT-qPCR檢測LmAbd-5在飛蝗不同組織部位的表達情況,RT-qPCR反應體系為20 μL:2×SYBR?Premix EX TaqTM10 μL,50×ROX Reference Dye 0.4 μL,cDNA模板(20×)2 μL,上下游引物2 μL(2 μmol·L-1),補dH2O 至20 μL。擴增程序為95℃ 30 s,95℃ 5 s,60℃ 31 s,40個循環,熔解曲線程序為ABI Prism 7300 SDS 1.1軟件自動添加。采用ABI Prism 7300 SDS 1.1軟件對數據進行記錄,以β-actin作為內參(KX276642),引物見表1(由上海生工生物公司合成)。
1.4.2 LmAbd-5在飛蝗5齡若蟲體壁不同發育時期的表達特性 解剖5齡剛蛻皮(0 h)、蛻皮后24、48、72、96、120、144 和168 h若蟲的第2、3腹節處體壁,并凍存于液氮中,4頭蟲體為1個生物學重復,共設3個重復。總RNA提取和cDNA模板合成、反應體系及方法同1.4.1。采用RT-qPCR檢測LmAbd-5在飛蝗5齡若蟲體壁不同發育時期的表達特性,引物見表1(由上海生工生物公司合成)。

表1 本研究中所用引物Table 1 Primer sequences used in this study
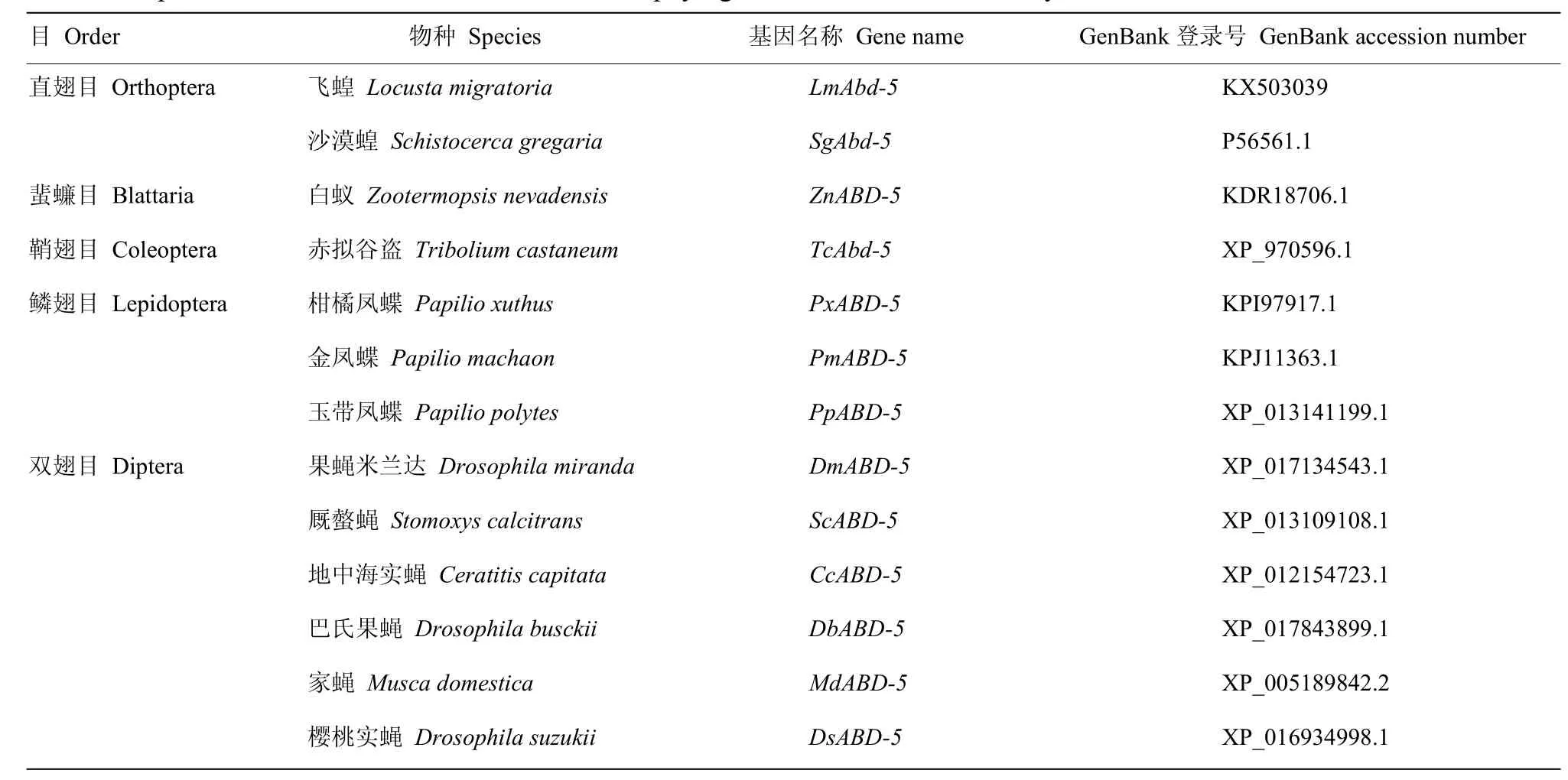
表2 本研究中用于構建系統進化樹的物種和Abd-5的GenBank登錄號Table 2 Species and GenBank accession number for phylogenetic tree used in this study
1.5 基于RNAi和透射電鏡技術的生物學功能分析
根據LmAbd-5和綠色熒光蛋白(GFP)的cDNA序列,設計合成雙鏈引物。使用PCR Master Mix(天根公司)進行PCR擴增,反應體系:12.5 μL PCR Master Mix,5 μL引物(上游/下游,2 μmol·L-1,引物見表1),2 μL表皮cDNA,加去離子水至25 μL,PCR程序:95℃預變性5 min;95℃ 15 s,60℃ 15 s,72℃ 30 s,72℃延伸10 min。產物用E.Z.N.A.?Gel Extraction Kit按照說明書進行回收,然后參照 T7 RiboMAXTMExpress RNAi System試劑盒說明書體外合成dsRNA。隨機挑選發育整齊的飛蝗5齡第2天若蟲進行 dsRNA注射,每頭蟲體注射 10 μg dsRNA,對照組注射等量dsGFP,每組注射30頭蟲體。正常飼喂至成蟲第2天,觀察表型并剪取腹部第2、3腹節表皮,同一蟲體的表皮一部分用于提取總RNA后,反轉錄為cDNA模板,RT-qPCR檢測沉默效率,另一部分(每頭蟲體均取相同部位)經3%戊二醛固定,用于透射電鏡觀察,透射電鏡樣品切片制備和染色等工作委托青島大學醫學院電鏡室完成。
1.6 數據分析
LmAbd-5的相對表達量用2-ΔΔCt方法進行分析[21],采用student t-test方法進行表達差異分析,*表示差異顯著(P<0.05),**表示差異極顯著(P<0.01)。
2 結果
2.1 LmAbd-5的cDNA序列及基因結構分析
基于飛蝗轉錄組搜索獲得 1條 LmAbd-5基因Unigene序列。根據已報道的飛蝗翅轉錄組數據庫對LmAbd-5 cDNA序列進行比對分析,獲得cDNA全長序列并進行克隆驗證。結果顯示,該基因 cDNA序列全長為520 bp,ORF長303 bp,編碼100個氨基酸,預測等電點為4.58,分子量為10.7 kD,是一種偏酸性小分子量的表皮蛋白。結合飛蝗基因組序列對其基因結構進行分析[22],結果顯示該基因含有3個外顯子,與大多數物種表皮蛋白基因結構類似[9],第1外顯子阻斷信號肽(圖1-A);將得到的LmAbd-5基因編碼的氨基酸進行功能域分析,發現其含有1個信號肽和1個幾丁質結合域ChtBD4(圖1-B)。

圖1 LmAbd-5基因結構和序列分析Fig. 1 Gene structure and sequence analysis of LmAbd-5
2.2 LmAbd-5蛋白結構域分析、序列比對及系統進化分析
LmAbd-5的全長氨基酸序列信息及功能域分析見圖 2,結果顯示飛蝗與沙漠蝗、白蟻和赤擬谷盜等Abd-5類似,不同的是沙漠蝗SgAbd-5僅為部分序列,缺少信號肽(圖2-A);通過NCBI進行BLAST分析顯示飛蝗 LmAbd-5與沙漠蝗 SgAbd-5高度同源,氨基酸序列一致度高達81%,其中幾丁質結合域序列完全一致(39—94 aa),與白蟻Abd-5一致度次之(70%),而與赤擬谷盜 Abd-5序列一致度較差(52%)(圖2-B)。將其保守基序進行WebLogo分析發現,LmAbd-5屬于表皮蛋白 CPR家族中的RR-1亞類,該基因的RR-1基序與前人在家蠶、按蚊和赤擬谷盜等昆蟲中研究的 RR基序在關鍵氨基酸的位點上是保守的,但 RR基序之外的氨基酸位點變化較大(圖2-C)。
從NCBI網站下載不同物種Abd-5氨基酸序列,物種名和登錄號如表2所示,主要包括雙翅目、鱗翅目、鞘翅目、蜚蠊目和直翅目昆蟲。將得到的飛蝗LmAbd-5全長序列與其他物種Abd-5氨基酸全長序列構建系統發育樹,聚類分析結果顯示,雙翅目和鱗翅目的Abd-5蛋白分別聚為一支,鞘翅目僅有赤擬谷盜一個Abd-5蛋白,直翅目飛蝗LmAbd-5與沙漠蝗、蜚蠊目白蟻的Abd-5聚為一支,并且與沙漠蝗Abd-5有最近的親緣關系(圖3)。
2.3 LmAbd-5的組織部位和發育時期表達特性
為了探討LmAbd-5在飛蝗不同組織部位和發育時期的表達特性,首先利用 RT-qPCR方法對LmAbd-5在飛蝗5齡第2天若蟲不同組織部位表達進行了分析(圖4)。結果顯示LmAbd-5在飛蝗前腸、后腸、氣管和體壁等由外胚層形成的組織中高表達,而在胃盲囊、中腸、馬氏管、脂肪體和翅芽中低表達或不表達(圖4-A),表明LmAbd-5可能參與表皮的形成;其次,選用5齡不同發育時期體壁組織進行表達分析,發現LmAbd-5在4齡若蟲蛻皮后0—72 h(5齡早期)具有高表達,其中蛻皮后72 h達到最大表達量,隨后表達量急劇降低(蛻皮后96—168 h)(圖4-B),LmAbd-5的表達時期與內表皮形成時間一致,推測其可能參與飛蝗內表皮結構的形成。

圖2 LmAbd-5蛋白結構、序列比對和RR-1基序分析Fig.2 Domain structure and alignment of LmAbd-5 and analysis of RR-1 motifs
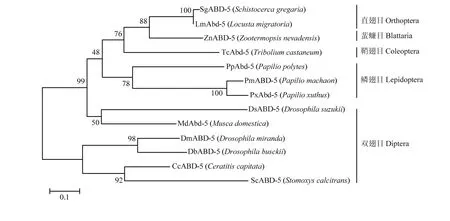
圖3 LmAbd-5與其他昆蟲Abd-5的系統聚類分析Fig. 3 The phylogenetic analysis of LmAbd-5 and Abd-5 from different insect species
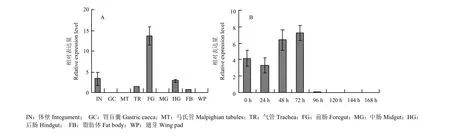
圖4 LmAbd-5在飛蝗 5 齡若蟲期不同組織部位和不同時期的表達Fig. 4 The relative expression of LmAbd-5 in different tissues and different stages of the 5th instar nymphs
2.4 基于RNAi的LmAbd-5生物學功能分析
為了探討LmAbd-5在飛蝗表皮形成中的作用,體外合成dsRNA并向5齡第2天若蟲分別注射等量的dsLmAbd-5和dsGFP(對照),正常飼養至蛻皮后,觀察成蟲表型并檢測沉默效率(圖5-A),發現注射dsLmAbd-5的5齡若蟲和對照組相同,均能正常蛻皮,盡管目的基因在成蟲第2天表達量顯著降低(處理組LmAbd-5表達量顯著降低了85.5%),但未出現肉眼可見的異常表型。分別取處理組和對照組蛻皮至成蟲第2天表皮進行超微結構觀察。結果表明與對照組相比,注射 dsLmAbd-5的成蟲內表皮片層結構疏松,導致內表皮片層變厚,使得整個內表皮變厚(圖5-B)。
3 討論
昆蟲表皮具有保護和防御等功能,在昆蟲生命過程中發揮著重要作用。昆蟲表皮由上表皮、外表皮、內表皮和細胞層構成,各層表皮的成分和機械性能不同[23],其中上表皮和外表皮主要在蛻皮前合成和沉積,內表皮是在蛻皮后合成和沉積。昆蟲表皮的主要成分是由幾丁質和大量表皮蛋白構成,其中CPR家族是最大的表皮蛋白家族,包括RR-1、RR-2 和RR-3等3種類型,在雙翅目、鱗翅目、鞘翅目、膜翅目、半翅目和直翅目等昆蟲中均有發現[6]。早在1988年,REBERS等[24]最先鑒定了CPR家族保守基序(R&R motif)G-x(8)-G-x(6)-Y-x-A-x-E-x-GY-x(7)-P-x(2)-P(x表示氨基酸,括弧內的數字為該處x的數目)。
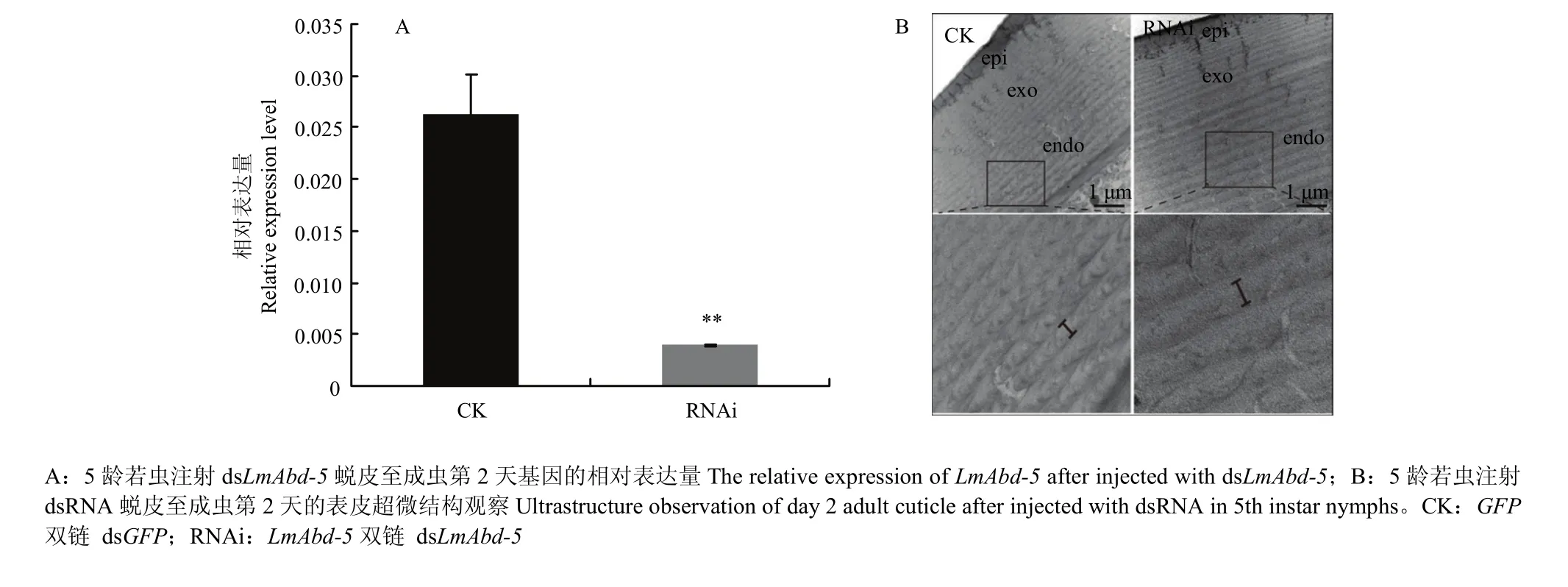
圖5 注射dsLmAbd-5后LmAbd-5的相對表達量和蛻皮至成蟲第2天的表皮超微結構觀察Fig. 5 The relative expression of LmAbd-5 and ultrastructure observation of day 2 adult cuticle after injected with dsRNA in 5th instar nymphs
20世紀90年代,NOHR等[25]利用雙向電泳技術,發現飛蝗內外表皮蛋白組成具有明顯的差異性。隨后,ANDERSEN[26]利用MALDI-MS技術,從沙漠蝗中分析鑒定出 8個內表皮蛋白,分別命名為 SgAbd-1、SgAbd-2、SgAbd-3、SgAbd-4、SgAbd-5、SgAbd-6、SgAbd-8和 SgAbd-9。隨著昆蟲基因組學的發展,越來越多昆蟲的Abd-5被鑒定,如雙翅目、鱗翅目、鞘翅目、蜚蠊目和直翅目等昆蟲。然而,在這些昆蟲中,Abd-5僅僅是序列被報道,具體功能尚未見深入研究。
本研究克隆獲得飛蝗內表皮蛋白基因LmAbd-5,其編碼的蛋白含有一個RR-1保守基序,屬于CPR家族中 RR-1亞類。組織分布研究發現,其主要在外胚層形成的組織中高表達,如前腸、后腸、氣管和體壁,而在胃盲囊、中腸、馬氏管、脂肪體和翅芽中低表達或不表達,表明 LmAbd-5可能參與表皮的形成;發育研究顯示其主要在蛻皮后72 h內具有高表達,而在96 h后表達急劇降低。飛蝗蛻皮后0—72 h是內表皮完全形成的時期,之后皮層出現溶離,新表皮開始合成。因此,LmAbd-5的表達與飛蝗內表皮合成和沉積時期一致,表明LmAbd-5可能是一種內表皮蛋白,參與昆蟲內表皮的形成。有研究發現,RR-1亞類的表皮蛋白主要存在于柔軟和具有韌性的水化角質層中,如雙翅目和鱗翅目昆蟲幼蟲表皮和節間膜以及飛蝗的內表皮[27],昆蟲內表皮中的幾丁質和表皮蛋白通過共價鍵與氫鍵結合,不被鞣化。CPR家族的保守RR基序具有幾丁質結合位點,能夠結合幾丁質,已在部分昆蟲研究中得到證實[11-13]。而飛蝗表皮蛋白LmAbd-5是否能結合幾丁質仍有待于進一步研究。
目前,RNAi技術是研究基因生物學功能的有效工具,筆者課題組利用RNAi在飛蝗幾丁質合成、降解和表皮蛋白家族等方面開展了大量工作[28-30]。飛蝗對RNAi極為敏感,本研究采用RNAi技術對飛蝗5齡第2天若蟲分別注射等量的dsLmAbd-5和dsGFP(對照),正常飼養觀察飛蝗表皮發育情況,發現注射dsLmAbd-5的5齡若蟲和對照組相同,均能正常蛻皮,盡管目的基因表達量顯著降低,但未發現可見的異常表型。分別取dsLmAbd-5注射組蛻皮后2 d的成蟲表皮(LmAbd-5表達量顯著降低)及對照組成蟲表皮進行透射電鏡觀察,發現與對照組相比,注射dsLmAbd-5的成蟲內表皮片層結構較為疏松,導致內表皮片層變厚,表明LmAbd-5在飛蝗內表皮的形成中具有重要作用。在飛蝗和其他昆蟲中,Abd是一個家族基因。根據節肢動物表皮蛋白數據庫[31]和飛蝗轉錄組數據庫,共發現包括LmAbd-5在內的8個Abd類型的表皮蛋白基因,然而其表達特性和功能以及它們相互之間是否具有協同或互補作用尚有待進一步的研究。
4 結論
經轉錄組數據庫分析獲得飛蝗LmAbd-5,其編碼的蛋白含有1個信號肽和1個幾丁質結合域ChtBD4,屬于CPR家族中的RR-1亞類;LmAbd-5主要在外胚層起源的組織中高表達,發育表達特性表明該基因主要在內表皮形成時期表達;沉默LmAbd-5后飛蝗未出現肉眼可見的表型,但超微結構觀察發現其參與飛蝗內表皮片層結構的形成。
[1] CHARLES J P. The regulation of expression of insect cuticle protein genes. Insect Biochemistry and Molecular Biology, 2010, 40(3): 205-213.
[2] CHAPMAN R F, SIMPSON S J, DOUGLAS A E. The Insects Structure and Function. 5th ed. Cambridge University Press, 2013.
[3] 王蔭長. 昆蟲生理學. 北京: 中國農業出版社, 2004. WANG Y C. Insect Physiology. Beijing: China Agriculture Press, 2004. (in Chinese)
[4] TETREAU G, DITTMER N T, CAO X, AGRAWAL S, CHEN Y R, MUTHUKRISHNAN S, HAOBO J, BLISSARD G W, KANOST M R, WANG P. Analysis of chitin-binding proteins from Manduca sexta provides new insights into evolution of peritrophin A-type chitinbinding domains in insects. Insect Biochemistry and Molecular Biology, 2015, 62: 127-141.
[5] REBERS J E, WILLIS J H. A conserved domain in arthropod cuticular proteins binds chitin. Insect Biochemistry and Molecular Biology, 2001, 31: 1083-1093.
[6] WILLIS J H. Structural cuticular proteins from arthropods: annotation, nomenclature, and sequence characteristics in the genomics era. Insect Biochemistry and Molecular Biology, 2010, 40(3): 189-204.
[7] KAROUZOU M V, SPYROPOULOS Y, ICONOMIDOU V A, CORNMAN R S, HAMODRAKAS S J, WILLIS J H. Drosophila cuticular proteins with the R&R consensus: Annotation and classification with a new tool for discriminating RR-1 and RR-2 sequences. Insect Biochemistry and Molecular Biology, 2007, 37(8): 754-760.
[8] GIBBS R A, BROWN S J, BEEMAN R W, WEINSTOCK1 G M. The genome of the model beetle and pest Tribolium castaneum. Nature, 2008, 452(7190): 949-955.
[9] FUTAHASHI R, OKAMOTO S, KAWASAKI H, ZHONG Y S, IWANAGA M, MITA K, FUJIWARA H. Genome-wide identification of cuticular protein genes in the silkworm, Bombyx mori. Insect Biochemistry and Molecular Biology, 2008, 38(12): 1138-1146.
[10] DITTMER N T, TETREAU G, CAO X, JIANG H, WANG P, KANOST M R. Annotation and expression analysis of cuticular proteins from the tobacco hornworm, Manduca sexta. Insect Biochemistry and Molecular Biology, 2015, 62: 100-113.
[11] DENG H M, LI Y, ZHANG J L, LIU L, FENG Q L. Analysis of expression and chitin-binding activity of the wing disc cuticle protein BmWCP4 in the silkworm, Bombyx mori. Insect Science, 2016, 23(6): 782-790.
[12] ARAKANE Y, ZHU Q, MUTHUKRISHNAN S, MATSUMIY M, KRAMER K J. Properties of catalytic, linker and chitin-binding domains of insect chitinase. Insect Biochemistry and Molecular Biology, 2003, 33: 631-648.
[13] WANG P, LI G, GRANADOS R R. Identification of two new peritrophic membrane proteins from larval Trichoplusia ni: structural characteristics and their functions in the protease rich insect gut. Insect Biochemistry and Molecular Biology, 2004, 34(3): 215-227.
[14] TAKEDA M, MITA K, QUAN G X, SHIMADA T, OKANO K, KANKE E, KAWASAKI H. Mass isolation of cuticle protein cDNAs from wing discs of Bombyx mori and their characterizations. Insect Biochemistry and Molecular Biology, 2001, 31(10): 1019-1028.
[15] NOH M Y, KRAMER K J, MUTHUKRISHNAN S, KANOST M R, BEEMAN R W, ARAKANE Y. Two major cuticular proteins are required for assembly of horizontal laminae and vertical pore canals in rigid cuticle of Tribolium castaneum. Insect Biochemistry and Molecular Biology, 2014, 53: 22-29.
[16] MUN S, YOUNG NOH M, DITTMER N T, MUTHUKRISHNAN S, KRAMER K J, KANOST M R, ARAKANE Y. Cuticular protein with a low complexity sequence becomes cross-linked during insect cuticle sclerotization and is required for the adult molt. Scientific Reports, 2015, 5: 10484.
[17] NOH M Y, MUTHUKRISHNAN S, KRAMER K J, ARAKANE Y. Tribolium castaneum RR-1 cuticular protein TcCPR4 is required for formation of pore canals in rigid cuticle. PLoS Genetics, 2015, 11(2): e1004963.
[18] ARAKANE Y, LOMAKIN J, GEHRKE S H, HIROMASA Y, TOMICH J M, MUTHUKRISHNAN S, BEEMAN R W, KRAMER K J, KANOST M R. Formation of rigid, non-flight forewings (Elytra) of a beetle requires two major cuticular proteins. PLoS Genetics, 2012, 8(4): e1002682.
[19] JESPERSEN S, HOJRUP P, ANDERSENT S O, ROEPSTORFF P. The primary structure of an endocuticular protein from two locust species, Locusta migratoria and Schistocerca gregaria, determined by a combination of mass spectrometry and automatic Edman degradation. Comparative Biochemistry and Physiology - Part B: Biochemistry & Molecular Biology, 1994, 109(1): 125-138.
[20] LIU S, WEI W, CHU Y, ZHANG L, SHEN J, AN C. De novo transcriptome analysis of wing development related signaling pathways in Locusta migratoria Manilensis and Ostrinia furnacalis (Guenée). PLoS ONE, 2014, 9(9): e106770.
[21] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔ Ctmethod. Methods, 2001, 25(4): 402-408.
[22] WANG X, FANG X, YANG P, JIANG X, JIANG F, ZHAO D, LI B, CUI F, WEI J, MA C, WANG Y, HE J, LUO Y, WANG Z, GUO X, GUO W, WANG X, ZHANG Y, YANG M, HAO S, CHEN B, MA Z, YU D, XIONG Z, ZHU Y, FAN D, HAN L, WANG B, CHEN Y, WANG J, YANG L, ZHAO W, FENG Y, CHEN G, LIAN J, LI Q, HUANG Z, YAO X, LV N, ZHANG G, LI Y, WANG J, WANG J, ZHU B, KANG L. The locust genome provides insight into swarm formation and long-distance flight. Nature Communications, 2014, 5: 2957.
[23] MOUSSIAN B, SEIFARTH C, MULLER U, BERGER J, SCHWARZ H. Cuticle differentiation during Drosophila embryogenesis. Arthropod Structure & Development, 2006, 35(3): 137-152.
[24] REBERS J E, RIDDIFORD L M. Structure and expression of a Manduca sexta larval cuticle gene homologous to Drosophila cuticle genes. Journal of Molecular Biology, 1988, 203(2): 411-423.
[25] NOHR C, ANDERSEN S O. Cuticular proteins from fifth instar nymphs of the migratory locust, Locusta migratoria. Insect Biochemistry and Molecular Biology, 1993, 23(4): 521-531.
[26] ANDERSEN S O. Amino acid sequence studies on endocuticular proteins from the desert locust, Schistocerca gregaria. Insect Biochemistry and Molecular Biology, 1998, 28: 421-434.
[27] NOH M Y, MUTHUKRISHNAN S, KRAMER K J, ARAKANE Y. Cuticle formation and pigmentation in beetles. Current Opinion Insect Science, 2016, 17: 1-9.
[28] 劉曉健, 崔淼, 李大琪, 張歡歡, 楊美玲, 張建珍. 飛蝗幾丁質合成酶2基因的表達特性、功能及調控. 中國農業科學, 2014, 47(7): 1330-1340. LIU X J, CUI M, LI D Q, ZHANG H H, YANG M L, ZHANG J Z. Expression, function and regulation of chitin synthase 2 gene in Locusta migratoria. Scientia Agricultura Sinica, 2014, 47(7): 1330-1340. (in Chinese)
[29] LI D, ZHANG J, WANG Y, LIU X, MA E, SUN Y, LI S, ZHU K Y, ZHANG J. Two chitinase 5 genes from Locusta migratoria: Molecular characteristics and functional differentiation. Insect Biochemistry and Molecular Biology, 2015, 58: 46-54.
[30] 王燕, 李大琪, 劉曉健, 李濤, 馬恩波, 范仁俊, 張建珍. 飛蝗表皮蛋白Obstructor家族基因的分子特性及基于RNAi的功能分析. 中國農業科學, 2015, 48(1): 73-82. WANG Y, LI D Q, LIU X J, LI T, MA E B, FAN R J, ZHANG J Z. Molecular characterization and RNAi-based functional analysis of Obstructor family genes in Locusta migratoria. Scientia Agricultura Sinica, 2015, 48(1): 73-82. (in Chinese)
[31] MAGKRIOTI C K, SPYROPOULOS I C, ICONOMIDOU V A, WILLIS J H, HAMODRAKAS S J. cuticleDB: a relational database of Arthropod cuticular proteins. BMC Bioinformatics, 2004, 5: 138.
(責任編輯 岳梅)
Expression and Functional Analysis of Endocuticle Structural Glycoprotein Gene LmAbd-5 in Locusta migratoria
ZHAO XiaoMing1, JIA Pan1,2, GOU Xin1,2, LIU WeiMin1, MA EnBo1, ZHANG JianZhen1
(1Research Institute of Applied Biology, Shanxi University, Taiyuan 030006;2College of Life Science, Shanxi University, Taiyuan 030006)
Locusta migratoria; cuticular proteins; LmAbd-5; RNAi; transmission electron microscopy
2016-11-28;接受日期:2017-03-24
國家自然科學基金(31640075,31672364)、山西省青年科學基金(201601D021102)、山西省高校科技創新基金(2016113)
聯系方式:趙小明,Tel:0351-7018871;E-mail:zxming@sxu.edu.cn。通信作者張建珍,Tel:0351-7018871;E-mail:zjz@sxu.edu.cn
Abstract:【Objective】The objective of this study is to obtain a cDNA sequence of endocuticle structural glycoprotein LmAbd-5 based on Locusta migratoria transcriptome, clarify its molecular characteristics and biological function, reveal its role in the formation of cuticle in L. migratoria, and provide a new molecular target for pest control. 【Method】The full length cDNA of LmAbd-5 was searched from transcriptome database of L. migratoria using bioinformatics method. The cDNA was cloned and sequenced. The signal peptide and function domain of deduced amino acid were analyzed by SignalP and SMART, respectively. Phylogenetic tree was constructed using the sequences of amino acid from different insect species by the MEGA 7.0 software with the neighbor-joining (NJ) method. Reverse transcription quantitative PCR (RT-qPCR) was applied to reveal the expression patterns of LmAbd-5 in different tissues on day 2 of 5th instar nymph and different developmental stages of integument. The effects of LmAbd-5 on locust growth development and the structure of cuticle were investigated by using RNA interference (RNAi) and transmission electron microscopy (TEM).【Result】The full length cDNA of LmAbd-5 was got from transcriptome database, which had 520 bp including ORF 303 bp. The gene structure analysis showed that LmAbd-5 has three exons. The deduced protein contains a signal peptide and one chitin binding domain 4 (ChtBD4) through the BLAST analysis, Abd-5 was highly conserved among insect species, and the sequence identity is as high as 81% between LmAbd-5 and SgAbd-5. Abd-5 belongs to the RR-1 class of CPR family by WebLogo analysis. The result of phylogenetic tree showed that LmAbd-5 has a close genetic relationship with SgAbd-5. RT-qPCR results showed that LmAbd-5 was predominately expressed in the tissues originated from ectoderm, such as the foregut, hindgut, trachea and integument, and lower expressed or not detected in the gastric caecum, midgut, Malpighian tube, fat body and wing pads. The expression at different stages showed that LmAbd-5 mainly expressed at early of 5th instar (0-72 h after ecdysis from 4th instar nymph), and up to the peak at 72 h after molting, then markedly decreased at 96-168 h. The expression pattern is related with the formation of endocuticle. Compared with dsGFP injected control, the nymphs with the injection of dsLmAbd-5 could normally molt, and no visible abnormal phenotypes was found although the expression of LmAbd-5 was decreased significantly after dsLmAbd-5 injection. However, compared to the control group, the lamellar structure from adult cuticle with injection of dsLmAbd-5 was loose, and lamellar became thicker, finally led to the endocuticle thickening.【Conclusion】LmAbd-5 was obtained from locust transcriptome database, which contains a signal peptide and ChtBD4, belonging to the RR-1 class of CPR family. LmAbd-5 mainly expressed in the tissues derived from ectoderm and in integument at early of 5th instar. Although there was no visible phenotypes after silencing LmAbd-5, but it was found that the lamellar structure of endocuticle is loose and endocuticle becomes thicken from ultrastructure by TEM, suggesting it may be participated in the formation of endocuticle in L. migratoria.

