SNP標記用于豬肉產品DNA溯源
吳 瀟,呂貝貝,王金斌,蔣 瑋,李 鵬,武國干,唐雪明*
SNP標記用于豬肉產品DNA溯源
吳 瀟,呂貝貝,王金斌,蔣 瑋,李 鵬,武國干,唐雪明*
(上海市農業科學院生物技術研究所,上海 201106)
采用基于個體基因組DNA序列的差異而進行個體識別的DNA溯源技術,建立DNA溯源系統對肉產品的質量安全進行控制。在實驗群體中(10 個品種,233 個個體)檢測了33 個新單核苷酸多態性(single nucleotide polymorphism,SNP)標記的遺傳多樣性,通過雜合度計算篩選出6 個SNP標記可用于豬肉產品DNA溯源。進一步在屠宰場采樣進行溯源模擬實驗,結果表明篩選的18 個SNP標記(6 個新SNP標記結合已有的12 個SNP標記)能有效區分100 頭豬個體,隨機抽取的10 個個體的組織樣品都能通過基因型比對找到對應的個體。本研究可為早日建立豬肉產品的DNA溯源系統提供一定的技術參考。
豬肉產品溯源;DNA溯源技術;SNP標記;個體識別
豬肉作為我國最主要的動物蛋白來源,其安全問題受到普遍關注。建立豬肉的溯源體系,可以實現對豬肉制品的可追溯管理,從而為消費者提供產品有關的詳細信息,包括養殖、運輸、屠宰、分割、銷售等各個環節,大大加強了政府相關部門對肉制品質量安全監管的力度[1-2]。
標簽溯源技術因為操作簡單、成本低,在我國得到廣泛的應用。但標簽存在易混淆、易脫落丟失,而且一旦豬肉產品間分割離開胴體就無法再進一步追溯。生物體的DNA存在于其任何組織的細胞中,而且個體的DNA序列具有特異性(同卵雙生除外)和穩定性,因此不受動物被分割的影響[3],通過鑒別個體DNA特征能夠準確地識別動物個體,因此DNA技術被用于肉產品的溯源中。
DNA溯源技術需要借助分子標記體現個體基因組序列的差異[4]。當前被認為是最具應用潛力的分子標記是SNP標記,已在多個國家得到應用。受飲食習慣影響,SNP標記主要在牛肉制品上應用較多,Heaton等[5]采用SNP標記對美國中西部17 個州進行了牛肉的追溯,在前期研究的基礎上將SNP標記數目縮小到20 個,這組標記實現了從在售牛肉樣品追溯到屠宰廠和農場[6]。日本為了杜絕瘋牛病牛肉疫情的傳播,Suekawa等[7]研究并使用SNP標記來篩選能區分日本本土黑牛、荷斯坦牛與進口的澳大利亞牛肉、美國牛肉,從而確保了日本牛肉食品的安全。韓國為了支持本國牛肉產業和維護消費者的知情權,Cheonga等[8]研發一組SNP標記用以區分韓國本地牛肉、進口牛肉和國產荷斯坦牛肉。比利時的Goffaux等[9]于就對豬基因的5’和3’非翻譯區的SNP標記進行研究,獲得一組21 個SNP標記,能有效區分比利時的主要商品豬品種。而我國豬肉產品溯源還停留在耳標等標簽技術階段。
在豬的長期育種工作中,已經積累了大量SNP標記,但只有至少滿足下列條件的標記才能用于溯源:1)變異度高,等位基因頻率較為接近;2)不同品種的等位基因分布差異小;3)雜合度要大于或等于0.3[2-3]。
由于現有SNP標記多為育種標記,跟某一類生產形狀相關,因此往往集中分布在某些染色體上,如1號染色體、12號染色體等,而另外部分染色體,如5號、8號、14號染色體等則缺乏SNP標記。因此為了滿足對豬肉產品進行DNA溯源的要求本研究率先進行了新SNP溯源標記的研究工作。
1 材料與方法
1.1 材料
實驗樣品采自上海崇明富農豬場,包括:皮特蘭(P)24 頭;申農(SN)32 頭;大申(DS)31 頭;皮申(PS)12 頭;長申(CS)26 頭;杜申(DuS)23 頭;皮大申(PDS)19 頭;長大申(CDS)25 頭;杜大申(DDS)7 頭;杜皮大申(DPDS)31 頭。
溯源模擬實驗的豬樣品采自上海復興屠宰場,隨機采集100 頭豬個體的耳組織樣品和其中10 頭豬個體的組織樣品(包括心臟、肝臟、胃部、腎臟、小腸、大腸、尾巴、肌肉樣品)。
1.2 方法
1.2.1 DNA的提取
采用酚/氯仿抽提法提取DNA,并稀釋到25 ng/μL,-20 ℃保存。提取方法參考《分子克隆實驗指南》。
1.2.2 PCR擴增和酶切
對缺乏SNP標記的豬4、5、14、15、16號和18號染色體,利用NCBI數據庫中豬的基因組序列作為模板,用Primer 5.0設計引物分離基因片段。PCR擴增體系為20 μL:其中Taq DNA聚合酶(5U/μL)0.20 μL,10×PCR Buffer 2.0 μL,dNTPs(2.5 mmol/L)0.40 μL,引物0.60 μL,DNA模板(稀釋后)3.0 μL,去離子蒸餾水13.20 μL,退火溫度依引物而定。酶切體系10~12 μL,其中擴增產物3~8 μL,酶切Buffer 1.2 μL,限制性內切酶5 U,反應6 h(各種酶的作用溫度不相同)。酶切產物用1%~2%的瓊脂糖電泳分析。

表1 19 對引物序列及片段長度Table 1 Sequences and length of the 19 primers used in this study
1.2.3 SNP標記的篩選
根據電泳圖片記錄個體的基因型,然后計算等位基因頻率(P)公式如下:

雜合度(heterozygosity,H)表示基因多樣性,群體的雜合度表示某位點為雜合子的概率,計算公式如下:

式中:Pi為i位點的等位基因頻率;n為等位基因數。
根據引物的擴增效率、瓊脂糖電泳的分辨率、等位基因頻率和雜合度是否大于0.3來進行SNP標記的篩選。
1.2.4 溯源模擬實驗
將采自屠宰場的100 頭豬個體的耳組織樣品按照1~100依次編號,其中10 頭豬只個體的樣品按照組織編號,如:L(肝臟樣品)、S(胃部樣品)、H(心臟樣品)、K(腎臟)、T(尾巴樣品)、I(大腸樣品)和M(肌肉樣品)。對于每一份樣品,檢測篩選的所有SNP標記的基因型,耳組織樣品的基因型代表個體的基因型,組織樣品的基因型代表豬分割肉的基因型,按照酶切帶型,1條帶記為1,出現2條帶記為2,3 條帶記為3,每一份樣品所有SNP的基因型形成一個類似條形碼的一串數字。將10 個組織樣品的基因型數字條碼和100 頭豬個體的基因型數字條碼進行比對。
2 結果與分析
2.1 SNP標記的挖掘
通過克隆子測序和序列比對分析,發現了33 個可以用PCR-RFLP方法識別的新SNP位點。SNP位點的堿基替代情況和內切酶如表2所示。
在實驗群體中進行33 個SNP標記的檢測,記錄每個位點的雜合子和純合子的個體數目。然后根據公式計算基因頻率和雜合度,其中有6 個SNP標記復合溯源標記的要求,詳見表3。6 個SNP標記等位基因分布均衡,雜合度最大為0.500 0,最小為0.244 9。雜合度低于0.3的有兩個,是SNP2和SNP3兩個位點在杜大申群體中的雜合度,這可能是因為杜大申樣本數太少的原因,除此之外,這兩個位點除了杜大申群體中雜合度稍低于0.3,其他群體中的雜合度都大于0.3,而且雜合度平均值分別為0.401 9和0.413 9,均大于0.3,所以這兩個位點也是符合溯源標記要求的。并且可以用于豬肉產品的溯源。

表2 新SNP位點及內切酶Table 2 New SNP sites and the corresponding endonucleases
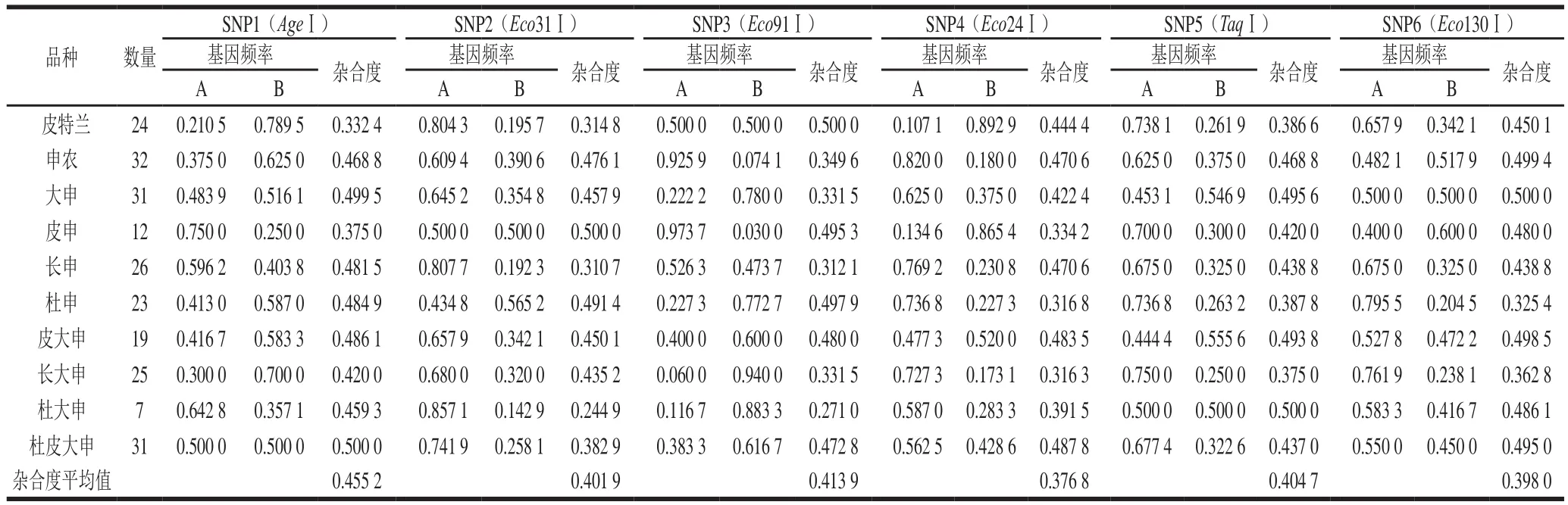
表3 6 個新SNP在11 個豬種中基因頻率及雜合度Table 3 Gene frequencies and heterozygosity of 6 new SNPs in 11 pig breeds
2.2 溯源模擬實驗結果
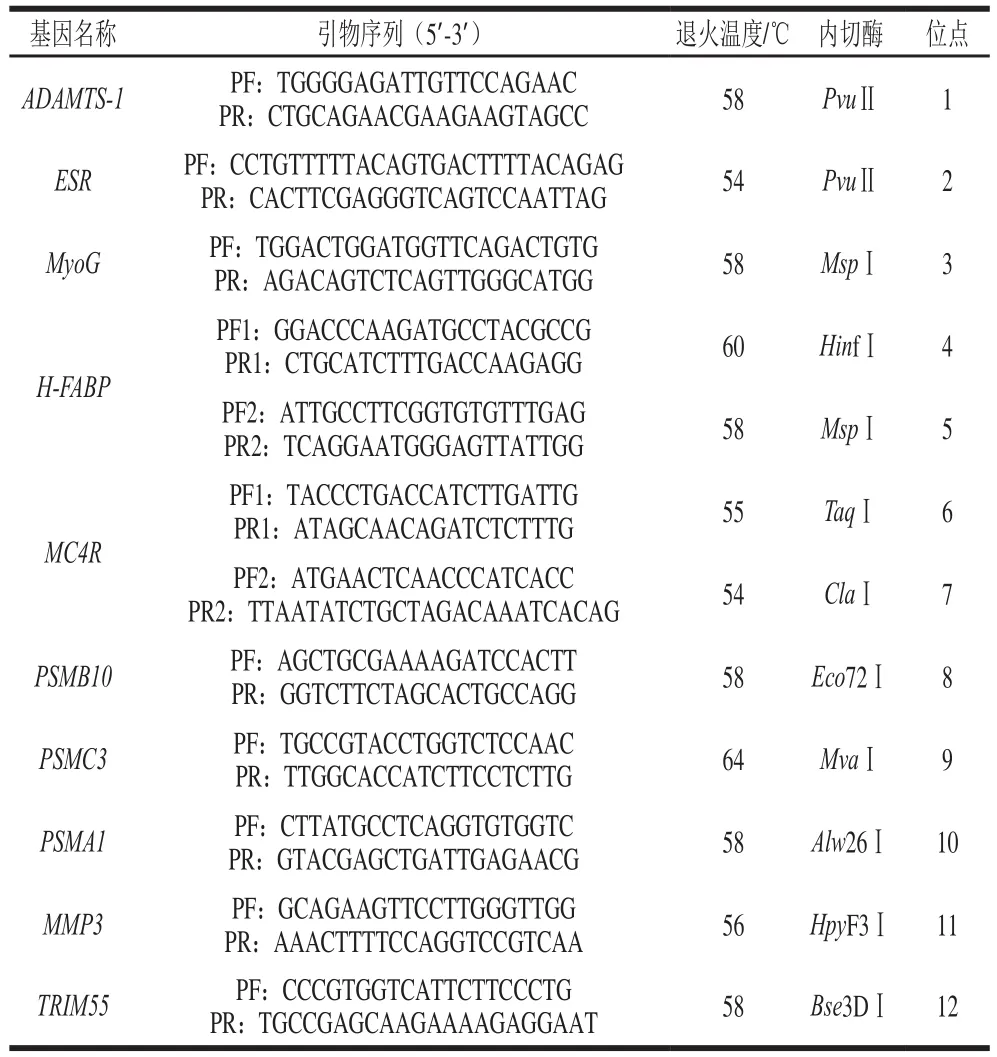
表4 12 個已有的可用于溯源的SNP標記[10-19]Table 4 Twelve existing SNP tags that can be used for traceability
為驗證篩選的SNP標記能否有效地進行豬肉產品溯源,本實驗在上海復興屠宰場進行了溯源模擬實驗。隨機采取了100 頭豬個體的耳組織樣,采集了其中10 頭個體的一處組織器官的樣品,代表市面上銷售的豬的分割產品。先在100 頭豬個體中檢測了18 個SNP的基因型,按照酶切帶型,記錄順序按照表4位點順序排列,再加表3中的6 個新SNP位點,個體的基因型可以表示為:212323222233113232。統計分析結果顯示這18 個SNP標記能鑒別出這100 頭豬個體。實驗同時測定了10 份組織器官樣品,同樣用數字表示基因型,如表5可知,通過查找和比對,10 個樣品都能在100 頭豬個體的基因型中找到基因型匹配的個體。
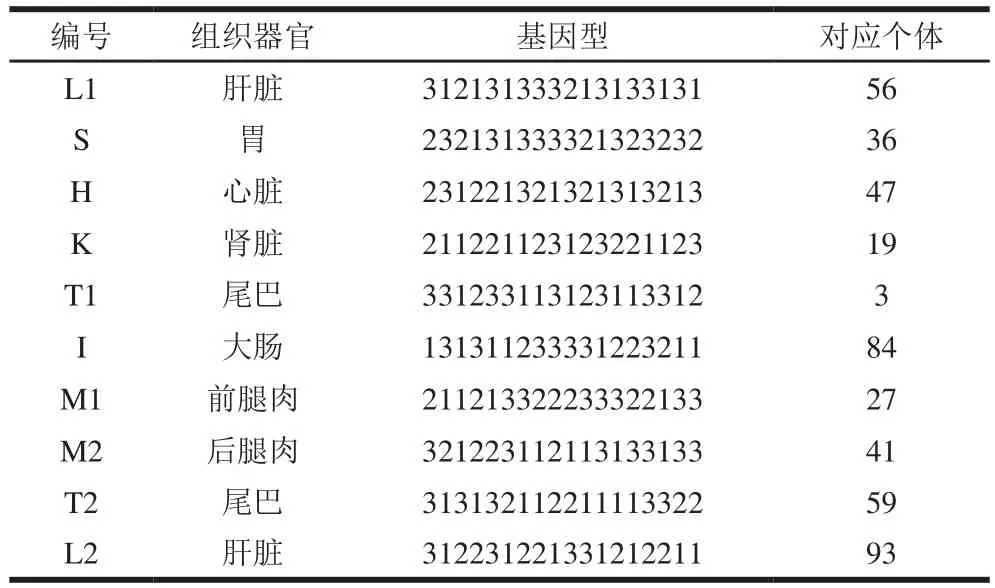
表5 10 份組織樣品基因型及對應的個體Table 5 Ten tissue samples and the corresponding individual genotypes
3 討論與結論
肉類食品安全問題關系到消費者的健康與生命安全[20-21],針對畜禽養殖、屠宰加工、產品貯運和銷售等環節存在的安全隱患,肉產品的追溯系統能起到一定的監管作用[22-24],有利于監管部門發現食品安全隱患后能迅速確定肉產品的來源及去向,及時召回受污產品,因此世界上許多國家已積極發展并建立肉類食品的追溯系統[2-3]。個體DNA存在于動物的每個細胞之中,不受動物發育期和器官的影響,而且每個個體的DNA是獨一無二的(同卵雙生除外)。研究者根據個體DNA的特異性和組織無差異性建立了DNA溯源技術,使得追溯不受動物分割的限制,因此DNA溯源技術被越來越多的國家廣泛應用到肉產品的溯源中[3]。
SNP標記是第3代分子標記,是單個核苷酸發生改變而產生多態性的DNA分子標記[25-27],屬于二等位基因多態,因此一個SNP理論上可以區分3 個動物個體,如果有n個位點,則能區分3n個個體。SNP在基因組內分布十分廣泛,如在哺乳動物中每500~1 000 bp序列就有可能包含一個SNP[4]。近年來,SNP標記作為分析生物遺傳多樣性的分子標記,已被廣泛用于豬的遺傳學[28-29]研究中。但是不是所有的SNP標記都可以用作溯源,因此需要建立群體對SNP標記進行篩選,只有符合品種內變異度高,品種間等位基因分布差異小,雜合度大于0.3的SNP標記才能用于DNA溯源。本實驗根據文獻[10-19]報道,檢索出大量的SNP標記,通過檢測分析,篩選出12 個可以用于溯源的SNP標記,如表4所示。同時,針對缺乏SNP溯源標記的染色體進行了新SNP標記的挖掘。根據NCBI數據庫豬的序列信息,設計了19 對引物,通過DNA池測得方法,發現了33 個可以用PCR-RFLP方法識別的新SNP位點。在采自上海崇明富農豬場的233 頭豬的實驗群體中檢測了這33 個SNP位點,通過等位基因頻率分析和雜合度計算,篩選到6 個可以用于溯源的SNP標記。將文獻中篩選獲得的12 個已有的SNP標記和6 個新SNP標記結合在一起形成一個SNP溯源標記的組合。
本研究為了驗證獲得的18個SNP溯源標記組合的溯源準確性,進行了豬肉產品的溯源模擬實驗。在上海復興屠宰場隨機采集了100 頭豬個體的耳組織樣品和其中10 頭豬個體的組織樣品,耳組織樣品的基因型代表個體的基因型,組織樣品的基因型代表分割肉的基因型。對于每一份樣品,檢測上步篩選的所有SNP標記的基因型,根據帶型記錄個體的基因型。溯源模擬結果顯示這18 個SNP標記能有效區分這100 頭豬個體。本研究同時檢測了采集的10 份組織器官樣品,同樣用數字表示基因型,通過查找和比對,10 個樣品都能在100 頭豬個體的基因型中找到基因型匹配的個體。阮泓越等[30]使用SNP標記對3 個國外引進豬品種(大白豬、長白豬和杜洛克豬)進行個體識別的研究,結果表明利用14 個SNP標記即可區分所有樣本,此外還進一步利用這些SNP標記對屠宰場隨機取樣的5 頭豬進行從肉樣到血液樣品的溯源研究,結果表明同一頭豬的肉樣和血液樣品的SNP座位是一一對應的。胡肄農等[31]使用7 對引物,擴增52 個SNP位點,經關聯性分析及雜合度過濾(雜合度不低于0.1)后,選留41 個SNP位點,用于蘇鐘豬個體身份識別,7 對引物擴增片段理論上可用于近5.0×106頭豬個體身份DNA識別的數字條形碼編制,基本可以滿足規模蘇鐘豬養豬場豬的個體身份識別。本實驗使用了18 個SNP標記,理論上有318個基因型體,能區分3.87×108個個體,因此18 個SNP標記是完全可以用于大規模的豬只個體識別和追溯。
采用DNA溯源技術進行豬肉產的溯源準確、可靠,但目前進行豬只個體DNA身份識別的成本比傳統的耳標等識別方法高,因此在我國難以普及,但是可以優先應用于一些生產品牌豬肉的大型養豬場。另外,針對豬的主要品種開發分辨率高、檢測位點少的檢測體系,可以針對地域性的主打品種進行SNP標記的篩選,隨著生物技術的發展,DNA溯源技術的成本會逐步降低,相信在不久的將來DNA溯源技術一定能廣泛的應用于我國動物個體識別和豬肉產品追溯中。
[1] 吳瀟, 潘玉春, 唐雪明. 肉制品的DNA溯源技術[J]. 豬業科學, 2009,26(3)∶ 105-106. DOI∶10.3969/j.issn.1673-5358.2009.03.031.
[2] 吳瀟, 張小波, 朱連龍, 等. 肉產品分子溯源標記的研究進展[J]. 食品科學, 2010, 31(7)∶ 308-311. DOI∶10.3969/j.issn.1673-5358.2009.03.031.
[3] 張小波, 吳瀟, 何慧, 等. 基于SNPs標記的豬肉DNA溯源技術的研究[J]. 中國農業科技導報, 2011, 13(3)∶ 85-91. DOI∶10.3969/i.issn.1008-0864.2011.03.14.
[4] 王淑新, 連林生. 單核苷酸多態性作為新一代分子標記的優越性[J].中國畜牧獸醫, 2008, 35(4)∶ 31-33.
[5] HEATON M P, HARHAY G P, BENNETT G L, et al. Selection and use of SNP markers for animal identification and paternity analysis in US beef cattle[J]. Mammalian Genome, 2002, 13(5)∶ 272-281.
[6] HEATON M P, JAMES E K, CLAWSON M L, et al. Use of bovine single nucleotide polymorphism markers to verify sample tracking in beef processing[J]. Journal of the American Veterinary Medical Association, 2005, 226(8)∶ 1311-1314.
[7] SUEKAWA Y, AIHARA H, ARAKI M D, et al. Development of breed identification markers based on a bovine 50K SNP array[J].Meat Science, 2010, 85(2)∶ 285-288.
[8] CHEONG H S, KIM L H, NAMGOONG S, et al. Development of discrimination SNP markers for Hanwoo (Korean native cattle)[J].Meat Science, 2013, 94(3)∶ 355-359.
[9] GOFFAUX F, CHINA B, DAMS L, et al. Development of a genetic traceability test in pig based on single nucleotide polymorphism detection[J]. Forensic Science International, 2005, 151(2/3)∶ 239-247.
[10] WU X, YANG S F, WANG J B, et al. Investigating identification and DNA traceability of pig by autosomal SNP and mitochondrial D-loop variant region markers[J]. Advanced Science Letters, 2013, 19(10)∶2995-2998.
[11] 黃敏瑞, 孫俊麗, 潘天彪, 等. 6 個SNP DNA標記在陸川豬群體中的遺傳多樣性分析[J]. 廣西畜牧獸醫, 2008, 24(2)∶ 67-69.
[12] 張桂香, 王志剛, 孫飛舟, 等. 56 個中國地方豬種SNP基因座的遺傳多樣性[J]. 遺傳學報, 2003, 30(3)∶ 225-233.
[13] 牛榮, 黃中波, 商海濤, 等. 廣西巴馬小型豬21 個SNP座位的DNA多態性分析[J]. 中國獸醫科技, 2002, 32(8)∶ 11-3. DOI∶10.3969/j.issn.1673-4696.2002.08.004.
[14] 吳迪, 張亞妮, 周占琴, 等. 合作豬和迪慶豬的SNP遺傳多樣性分析[J]. 甘肅農業大學學報, 2004, 39(2)∶ 106-109. DOI∶10.3969/j.issn.1003-4315.2004.02.002.
[15] 郭宏宇, 林家棟. 利用SNP DNA標記分析貴州3 個地方豬種的遺傳多樣性[J]. 安徽農業科學, 2009, 37(8)∶ 3453-3454. DOI∶10.3969/j.issn.0517-6611.2009.08.044.
[16] 徐玲玲, 吳艷花, 路靜, 等. 篩選適用于小型豬遺傳檢測的SNP位點[J]. 中國比較醫學雜志, 2009, 19(2)∶ 11-6. DOI∶10.3969/j.issn.1671-7856.2009.02.004.
[17] 公維華, 張寧波, 程佳月, 等. 小型豬SNP標記多重PCR體系的建立與應用[J]. 中國比較醫學雜志, 2009, 19(2)∶ 21-25. DOI∶10.3969/j.issn.1671-7856.2009.02.006.
[18] 曹果清, 李步高, 石建中, 等. 應用21 個SNP標記監測馬身豬遺傳多樣性變化趨勢[J]. 畜牧獸醫學報, 2010, 41(8)∶ 932-928.
[19] 霍金龍, 霍海龍, 苗永旺, 等. 利用76 個SNP標記分析滇南小耳豬的遺傳多樣性[J]. 云南農業大學學報, 2008, 23(5)∶ 638-643.DOI∶10.3969/j.issn.1004-390X.2008.05.012.
[20] 張秋, 陳慧. 肉類食品安全事件模擬演練設計與實施[J]. 肉類研究,2016, 30(11)∶ 43-47. DOI∶10.15922/j.cnki.rlyj.2016.11.009.
[21] 李天儀. 對肉類食品安全問題的思考[J]. 養殖與飼料, 2017(4)∶ 97-98.DOI∶10.13300/j.cnki.cn42-1648/s.2017.04.055.
[22] 王立方, 陸昌華, 胡肄農, 等. 新型生豬標識及肉產品可追溯系統的設計和實現[J]. 農業網絡信息, 2006(12)∶ 25-27. DOI∶10.3969/j.issn.1672-6251.2006.12.008.
[23] 楊亮, 羅清堯, 熊本海, 等. 豬肉質量安全可追溯系統屠宰環節的設計與實現[J]. 農業網絡信息, 2008(1)∶ 22-25. DOI∶10.3969/j.issn.1672-6251.2008.01.007.
[24] 謝菊芳, 陸昌華. 安全豬肉全程可追溯系統的設計與實現[J]. 黑龍江畜牧獸醫, 2007(4)∶ 94-96. DOI∶10.3969/j.issn.1004-7034.2007.04.049.
[25] 唐立群, 肖層林, 王偉平. SNP分子標記的研究及其應用進展[J]. 中國農學通報, 2012, 28(12)∶ 154-158. DOI∶10.3969/j.issn.1000-6850.2012.12.028.
[26] 李兆波, 吳禹, 王巖, 等. SNP標記技術及其在農作物育種中的應用[J]. 遼寧農業職業技術學院學報, 2010, 12(3)∶ 8-9. DOI∶10.3969/j.issn.1671-0517.2010.03.004.
[27] 卞光明, 胡則輝, 柴學軍, 等. SNP標記技術及其在水產動物遺傳學中的應用[J]. 浙江海洋學院學報(自然科學版), 2016, 35(4)∶346-353. DOI∶10.3969/j.issn.1008-830X.2016.04.014.
[28] 王彥平, 王懷中, 姜運良, 等. 影響豬生長和肉質性狀的顯著SNP在大約克和長白群體內的多態性分析[J]. 山東農業科學, 2016, 48(8)∶128-133. DOI∶10.14083/j.issn.1001-4942.2016.08.029.
[29] 曾治君, 劉晨龍, 楊慧, 等. 利用高密度SNP對豬血糖和糖基化血清蛋白性狀的全基因組關聯分析[J]. 中國農業科學, 2014, 47(18)∶3700-3707. DOI∶10.3864/j.issn.0578-1752.2014.18.018.
[30] 阮泓越, 宛煜嵩, 賀輝群, 等. 14 個SNP DNA 標記在豬個體識別和溯源中的應用研究[J]. 農業生物技術學報, 2010, 18(6)∶ 1129-1133.DOI∶10.3969/j.issn.1674-7968.2010.06.015.
[31] 胡肄農, 丁潛, 紀紅軍, 等. 邢光東蘇鐘豬個體身份SNP識別的數字條形碼編制[J]. 江蘇農業學報, 2014, 30(4)∶ 779-783.
Development and Application of Pork Traceability System Based on SNP Markers
WU Xiao, Lü Beibei, WANG Jinbin, JIANG Wei, LI Peng, WU Guogan, TANG Xueming*
(Biotechnology Research Institute, Shanghai Academy of Agricultural Sciences, Shanghai 201106, China)
DNA traceability technology is based on DNA sequence differences between individuals for individual identification and the tracing of meat back to an individual animal. Establishing a DNA-based traceability system is important for the safety and quality of meat products. The genetic diversity of 33 new single nucleotide polymorphism (SNP)markers was detected in a test group (233 individuals, 10 varieties) and 6 SNP markers, which are useful for pork product traceability, were obtained by heterozygosity calculation. A total of 18 SNP markers (6 new SNP markers in combination with 12 SNP markers) could effectively distinguish between 100 individuals collected from a local slaughter house in traceability simulation experiments. The corresponding individuals for tissue samples from 10 random individuals were found by genotype. This study has shown that SNP markers could be used for individual identification and pork traceability.
pork traceability; DNA traceability technology; SNP markers; individual identification
10.7506/spkx1002-6630-201724045
Q819
A
1002-6630(2017)24-0278-05
吳瀟, 呂貝貝, 王金斌, 等. SNP標記用于豬肉產品DNA溯源[J]. 食品科學, 2017, 38(24)∶ 278-282. DOI∶10.7506/spkx1002-6630-201724045. http∶//www.spkx.net.cn
WU Xiao, Lü Beibei, WANG Jinbin, et al. Development and application of pork traceability system based on SNP markers[J]. Food Science, 2017, 38(24)∶ 278-282. (in Chinese with English abstract) DOI∶10.7506/spkx1002-6630-201724045. http∶//www.spkx.net.cn
2016-10-20
閔行區產學研合作計劃項目(2016MH256)
吳瀟(1978—),女,副研究員,博士,研究方向為食品安全。E-mail:qwuxiao@126.com
*通信作者:唐雪明(1970—),男,研究員,博士,研究方向為食品安全。E-mail:xueming70@foxmail.com

