脊尾白蝦不同發育期線粒體基因組甲基化特征分析
薛 蓓,張 培,李志輝,趙 蓮,賴曉芳,閻斌倫,高 煥
( 1.淮海工學院 海洋生命與水產學院,江蘇省海洋生物技術重點實驗室,江蘇 連云港 222005;2.江蘇省海洋生物產業技術協同創新中心,江蘇 連云港 222001;3.江蘇省農業種質資源保護與利用平臺,江蘇 南京 210014 )
脊尾白蝦不同發育期線粒體基因組甲基化特征分析
薛 蓓1,2,3,張 培1,2,3,李志輝1,2,3,趙 蓮1,2,3,賴曉芳1,2,3,閻斌倫1,2,3,高 煥1,2,3
( 1.淮海工學院 海洋生命與水產學院,江蘇省海洋生物技術重點實驗室,江蘇 連云港 222005;2.江蘇省海洋生物產業技術協同創新中心,江蘇 連云港 222001;3.江蘇省農業種質資源保護與利用平臺,江蘇 南京 210014 )
為研究不同發育階段脊尾白蝦線粒體基因組表觀遺傳學特征,本研究在篩選硫化測序引物的基礎上,對不同發育期的脊尾白蝦線粒體基因組甲基化特征進行了分析。經引物篩選驗證,選取了4對引物在脊尾白蝦的受精卵、幼體期、仔蝦期和成蝦期共4個發育期進行甲基化特征的分析,結果表明,除ND5基因3′端序列(21#引物擴增區)無甲基化現象外,COX3基因的起始區序列(13#引物擴增區)、COX3基因的3′端序列(16#引物擴增區)和ND5基因5′端序列(19#引物擴增區)均存在不同程度的甲基化現象。其中,COX3基因的起始區序列僅在受精卵孵化期和幼體期存在甲基化現象,COX3基因3′端序列在除受精卵外的其他3個時期均存在甲基化現象,且以上甲基化率隨個體發育階段的提高呈下降趨勢;ND5基因5′端序列在4個不同發育期均存在甲基化現象,但甲基化率無顯著差異。甲基化主要發生在COX3基因和ND5基因位點上,推測脊尾白蝦在生長發育過程中通過調節線粒體基因組甲基化來調節機體能量代謝過程。本研究首次從整個發育周期角度探討了線粒體基因組甲基化特征,以期為進一步揭示甲殼類生長發育相關遺傳機制提供幫助。
脊尾白蝦;線粒體基因組;不同發育期;甲基化
在組蛋白修飾、DNA甲基化和染色質重塑等表觀遺傳學現象中,DNA甲基化是最為常見并且研究最為深入的[1]。在真核生物的細胞中,除核基因組DNA(nDNA)外,其線粒體細胞器中也含有DNA(mtDNA);mtDNA雖僅占細胞內總DNA的不到1%,但其轉錄產物對于發揮細胞正常功能是必不可少的[2-3]。以前針對DNA甲基化的研究多集中在nDNA上[4-5],而近年來關于mtDNA甲基化的研究也逐漸增多;但這些研究多集中在衰老[6]、神經退行性疾病[7]及腫瘤[8]等疾病方面,有關不同發育期下mtDNA甲基化的研究鮮見報道,在甲殼動物中更未見報道。
脊尾白蝦(Exopalaemoncarinicauda)是我國特有的養殖經濟種類,廣泛分布于我國沿海區域,尤以黃海、渤海區域最為豐富[9],產量可占我國東部沿海地區池塘混養模式產量的三分之一,且養殖規模有進一步擴大的趨勢[10]。因此,近年來為加強其生長發育及免疫等生理生化機制方面的了解,以其為對象的研究也逐漸增多[11-13]。在前期的研究中,已獲得脊尾白蝦線粒體基因組的全序列[14],并發現該蝦在應答饑餓時線粒體基因組會產生甲基化現象[15],但是,在其他情況下線粒體基因組的甲基化特征還不清楚,尤其在不同發育階段下,而這對于了解線粒體基因組在脊尾白蝦等甲殼類生物生長發育過程中的作用及其表達調控機制非常重要。因此,筆者希望通過對脊尾白蝦不同發育階段線粒體基因組甲基化特征的研究,為加深對甲殼類生長發育相關遺傳機制的認識提供幫助。
1材料與方法
1.1 試驗材料
自養殖池塘中選取性腺發育成熟的脊尾白蝦個體,保持溫度(27±1) ℃、鹽度29±1、pH 8.0±0.5的恒定養殖條件,在實驗室中進行養殖并取材。選取受精卵[16]、幼體期[17]、仔蝦期和成蝦期4個不同發育階段,每個發育期選取6個樣本。
1.2 線粒體基因組DNA提取及處理
利用基因組DNA提取試劑盒(MiniBEST Universal Genomic DNA Extraction Kit, TaKaRa)分別提取各發育期的總DNA(其中含有線粒體基因組DNA),受精卵、幼體期和仔蝦期樣本采用整體液氮研磨后提取,成蝦期樣本則取其腹部肌肉進行研磨后提取DNA。利用甲基化檢測試劑盒(EpiMarkTM, New England Biolabs Inc.)對提取的基因組進行重亞硫酸鹽處理以獲得處理后的線粒體基因組DNA,并以處理后的基因組DNA為PCR模板進行BSP引物的驗證和后續各不同發育期脊尾白蝦的甲基化特征分析。
1.3 硫化測序(BSP)引物的篩選及驗證
根據實驗室前期設計的引物[14],選取10對(引物3、8、11、12、13、16、19、21、33和48)擴增穩定的硫化測序引物(表1),用于對處理后基因組DNA的擴增,篩選擴增穩定的引物對脊尾白蝦線粒體基因組DNA的甲基化情況進行分析。引物名稱中的數字為線粒體基因組全序列中對應的序列區間。
采用如下PCR體系進行擴增:總體積25 μL,含10×PCR buffer 2.5 μL,Mg2+(25 mmol/L) 1.5 μL,dNTP (10 mmol/L) 0.5 μL,Taq DNA聚合酶(5 U/μL) 0.5 μL, Forward Primer (10 mmol/L) 1 μL,Reverse Primer (10 mmol/L) 1 μL,ddH2O 15 μL 及處理后的基因組DNA 3 μL。利用Touchdown PCR程序進行擴增:95 ℃ 30 s; 95 ℃ 15 s,55 ℃ 30 s,68 ℃ 45 s(其中退火溫度每個循環降低1 ℃,進行10個循環后再以45 ℃固定退火溫度進行30個循環);68 ℃ 5 min;4 ℃保存。
PCR產物經1.5%的瓊脂糖凝膠電泳驗證后,對有擴增條帶的片段進行切膠回收,利用pEASY-T3載體進行克隆轉化后,送上海生工生物工程有限公司測序,并通過對比測序序列與原序列確認是否為線粒體基因組的擴增產物。
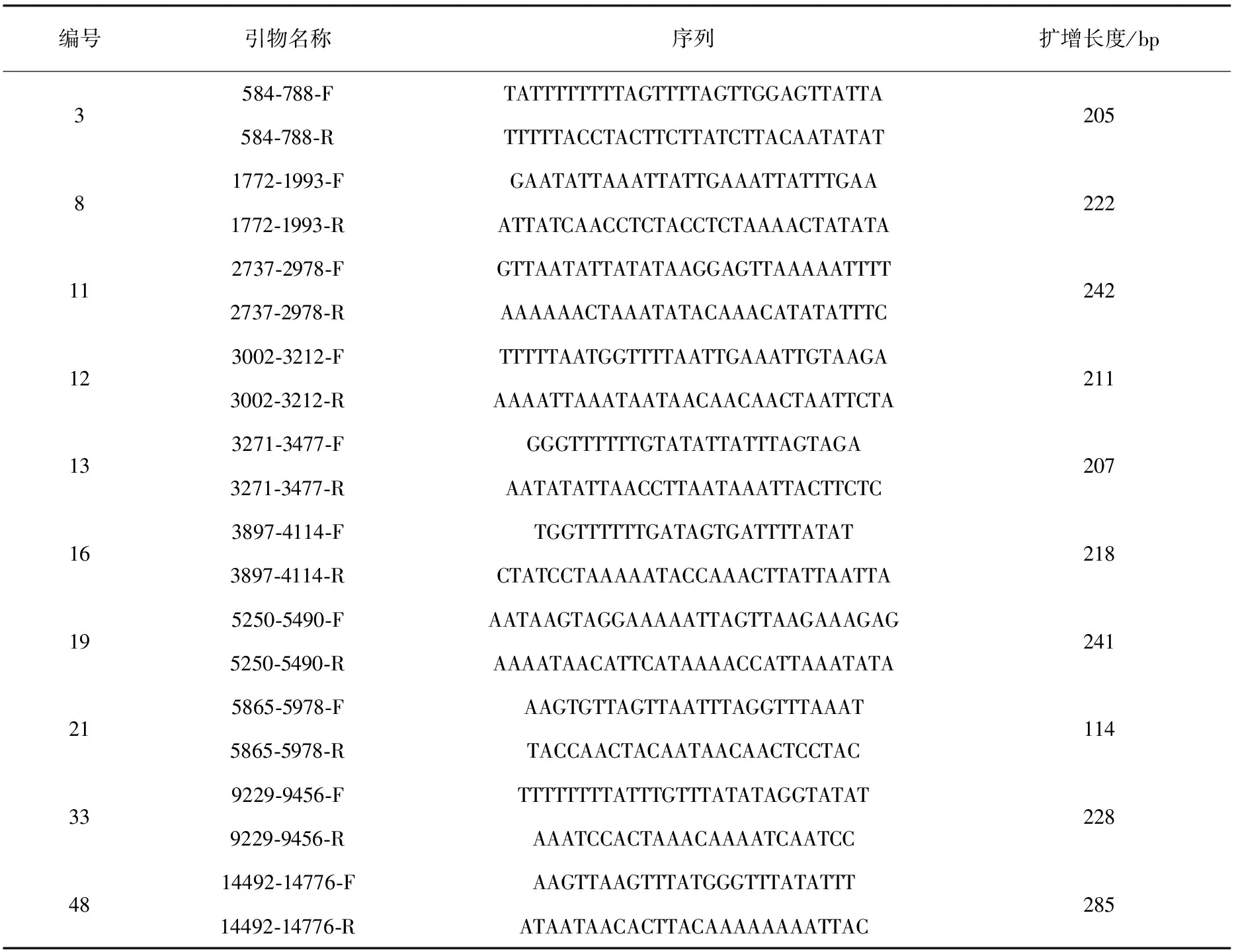
表1 脊尾白蝦線粒體基因組BSP引物
1.4 數據分析
將測序獲得的處理后線粒體基因組DNA序列與原序列通過在線軟件QUMA(http://quma.cdb.riken.jp/)進行比對分析以確認甲基化的有無及其相應特征,并計算相應的甲基化率,其中:
總甲基化率/%=(甲基化胞嘧啶位點個數/總胞嘧啶位點個數)×100%
CpG位點甲基化率/%=(甲基化CpG位點個數/總CpG位點個數)×100%
CpHs位點甲基化率/%=(甲基化CpHs位點個數/總CpHs位點個數)×100%
2 結 果
2.1 硫化測序引物的篩選結果
以各發育期混合DNA為模板的甲基化驗證后發現,10對硫化測序引物中,13、16、19和21共4對引物擴增穩定,這4對引物的具體遺傳信息特征如下:
引物13(13#)擴增區域位于COX3(細胞色素氧化酶亞基3)基因的起始區序列,序列全長207 bp,其中包含CpG位點4個,CpHs(CpA/CpT/CpC)位點46個;引物16(16#)擴增區域位于COX3基因的3′端序列和ND3基因的起始區序列,序列全長218 bp,其中包含CpG位點5個,CpHs位點46個;引物19(19#)擴增區域位于ND5(還原型輔酶Ⅰ脫氫酶亞基5)基因的5′端序列,序列全長241 bp,其中包含CpG位點5個,CpHs位點60個;引物21(21#)擴增區域位于ND5基因的3′端序列,序列全長114 bp,其中包含CpG位點4個,CpHs位點22個。
通過對不同發育期脊尾白蝦基因組DNA的硫化測序結果分析發現,在COX3基因的起始區序列和3′端序列以及ND5基因的5′端序列存在甲基化現象,而在ND5基因的3′端不存在甲基化現象。
2.2 不同發育期脊尾白蝦的甲基化特征
對4個不同發育期(受精卵、幼體期、仔蝦期和成蝦期)脊尾白蝦的甲基化特征分析發現,3對硫化測序引物的擴增序列的甲基化不僅發生在CpG位點,也發生在CpHs位點。但3個序列區間在具體的甲基化率等方面存在不同,具體如下:
COX3基因起始區序列(引物13#)僅在受精卵和幼體期各有1個個體存在甲基化,而其他發育期均未發現甲基化現象,受精卵的甲基化率高于幼體期(表2)。
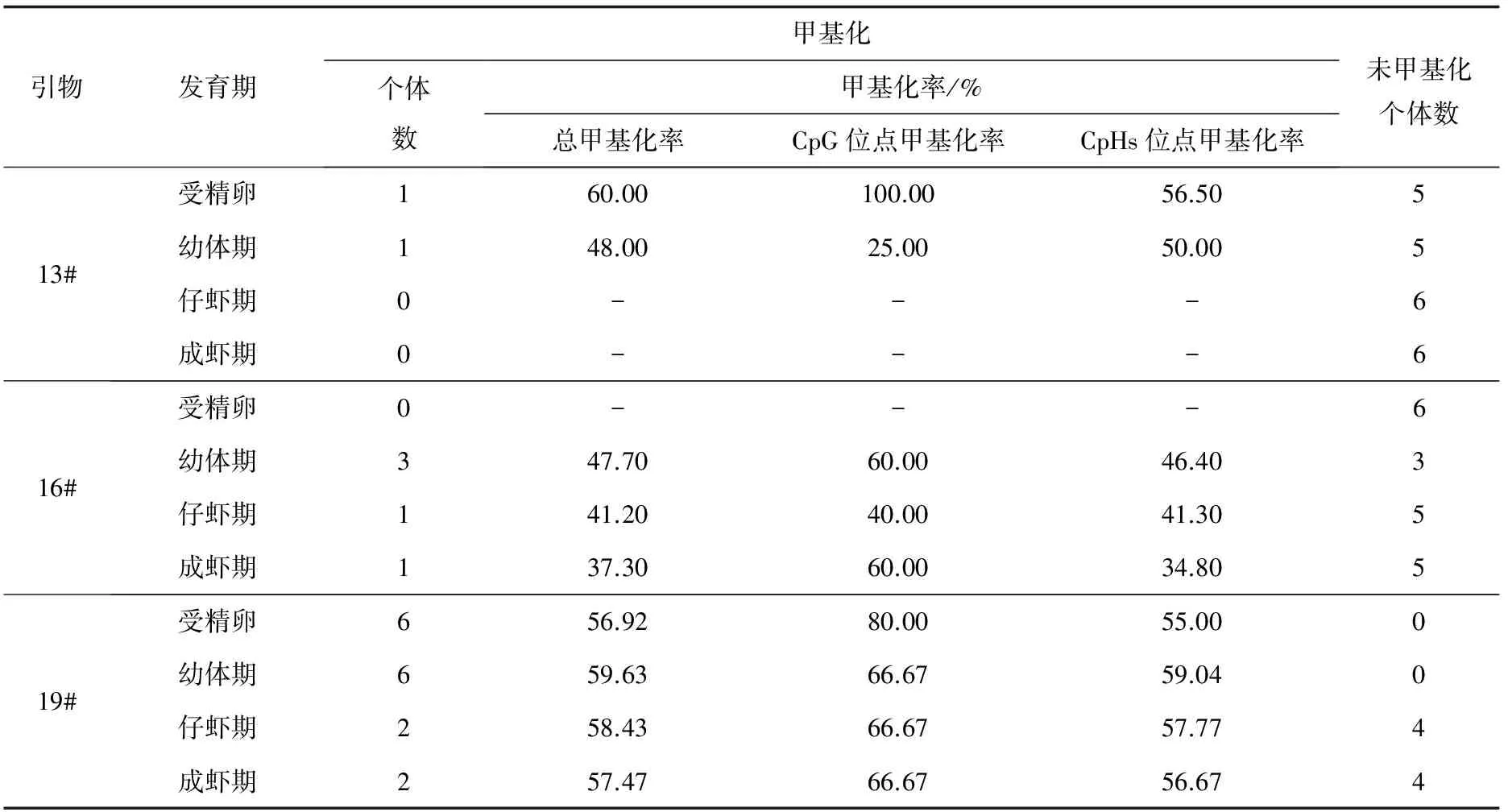
表2 不同發育期脊尾白蝦線粒體基因組甲基化特征
COX3基因的3′端序列(引物16#)在受精卵中未發現甲基化現象,在幼體期、仔蝦期和成蝦期分別有3個、1個和1個個體存在甲基化現象,且隨著脊尾白蝦的發育,甲基化率呈現下降的趨勢(表2);ND3基因的起始序列(引物16#)中未發現甲基化現象。
ND5基因5′端(引物19#)在4個不同發育期脊尾白蝦中均存在甲基化現象,只是在受精卵和幼體期取樣的6個個體均存在甲基化現象,而在仔蝦期和成蝦期各有2個個體存在甲基化現象。然而,19#引物的擴增序列在不同發育期的甲基化率不存在明顯的差異,單就甲基化位點來看并無明顯規律(表2)。
3 討 論
3.1 不同發育期脊尾白蝦線粒體基因組的甲基化水平
在生物體的生長發育過程中,DNA甲基化可以通過直接或間接的方式參與到基因的轉錄調控作用中,對基因表達具有重要的協同作用[18-19]。生物體內某些核基因在不同發育期的表達量存在差異[20-21],而這種差異與其甲基化水平存在一定的相關性[20]。那么,mtDNA在不同發育期中的甲基化水平是否存在差異?已有的研究表明,疾病[22]、衰老[7,23]、環境因子脅迫[24]和饑餓[15]等因素均可能影響mtDNA甲基化。本研究中通過對脊尾白蝦線粒體基因組甲基化水平的研究,發現其在不同發育期的甲基化水平存在差異,且隨著個體的發育,在甲基化個體數或總甲基化率水平上呈現降低的趨勢,這可能是由于隨著生物體的生長發育,其生命活動所需能量增多,而線粒體作為生物體的能量中心,其基因組甲基化水平的降低有利于相關基因的表達。
3.2 不同發育期脊尾白蝦COX3基因的甲基化情況
COX3基因的起始區序列(引物13#)及COX3基因的3′端序列(引物16#),在不同發育期的脊尾白蝦mtDNA的甲基化情況均表現為隨生物體發育甲基化程度降低的整體趨勢。細胞色素氧化酶是線粒體內膜呼吸鏈的重要組成[25],因此這也說明在脊尾白蝦的發育過程中,生物體通過調節自身mtDNA甲基化程度來調節能量代謝。但是,這兩個序列區間的甲基化情況還略有差異,COX3基因的起始序列在受精卵表現為較高的甲基化率,而COX3基因的3′端序列在該時期則并無甲基化現象;相反COX3基因的3′端序列在仔蝦期及成蝦期均存在甲基化現象,而COX3基因的起始序列則并無甲基化現象。說明同一基因的不同區間在同一發育階段的甲基化現象也存在不同,線粒體基因組甲基化的具體機理仍需進一步研究。
3.3 不同發育期脊尾白蝦ND5基因的甲基化情況
還原型輔酶Ⅰ脫氫酶是電子進入電子傳遞鏈的主要位點,為線粒體內三磷酸腺苷的形成提供約40%的質子,在氧化磷酸化系統中發揮著重要的作用[26-27]。但脊尾白蝦在4個不同發育期中ND5基因的5′端序列(引物19#)均表現為較高的甲基化率,且各發育期之間不存在顯著差異;而ND5基因的3′端序列(引物21#)在4個發育階段則均無甲基化現象。這進一步說明同一基因的不同區間在同一發育階段的甲基化現象存在不同。另外,已有的研究結果顯示[15],不同的饑餓期下ND5基因的5′端序列無甲基化現象,而ND5基因的3′端序列存在甲基化現象,這說明ND5基因可能在不同發育階段甲基化較為穩定,而饑餓脅迫應激后為甲基化現象的高發區。
此外,試驗中發現,同一發育期的6個個體在不同區間的甲基化發生情況存在較大的不同,一方面可能是因為取材時并未區分各發育期的具體Ⅰ~Ⅵ期,而同一發育期內的甲基化也存在變化,這仍需要進一步研究;另一方面基因組甲基化具有一定的可遺傳性[28],因而不同個體間可能存在遺傳上的差異,這需要進一步擴大樣品量并進行深入的研究。
[1] Lee T, Zhai J, Meyers B C. Conservation and divergence in eukaryotic DNA methylation[J]. Proceedings of the National Academy of Sciences, 2010, 107(20):9027-9028.
[2] 劉振山, 馮云, 薛志剛, 等. 線粒體DNA與DNA甲基化[J]. 生物學雜志, 2013, 30(5):77-80.
[3] Iacobazzi V, Castegna A, Infantino V, et al. Mitochondrial DNA methylation as a next-generation biomarker and diagnostic tool [J]. Molecular Genetics & Metabolism, 2013, 110(2):25-34.
[4] Laird P W. The power and the promise of DNA methylation markers [J]. Nature Reviews Cancer, 2003, 3(3):253-266.
[5] Dehan P, Kustermans G, Guenin S, et al. DNA methylation and cancer diagnosis:new methods and applications [J]. Expert Review of Molecular Diagnostics, 2009, 9(7):651-657.
[6] Mawlood S K, Dennany L, Watson N, et al. Quantification of global mitochondrial DNA methylation levels and inverse correlation with age at two CpG sites [J]. Aging, 2016, 8(2):1-6.
[7] Wong M, Gertz B, Chestnut B A, et al. Mitochondrial DNMT3A and DNA methylation in skeletal muscle and CNS of transgenic mouse models of ALS[J]. Front Cell Neurosci,2013(7):279.
[8] Feng S, Xiong L, Ji Z, et al. Correlation between increased ND2 expression and demethylated displacement loop of mtDNA in colorectal cancer[J]. Mol Med Rep, 2012, 6(1):125-130.
[9] 王緒峨. 脊尾白蝦繁殖生物學的初步觀察[J]. 動物學雜志, 1987, 22(1):7-10.
[10] Xu W, Xie J, Shi H, et al. Hematodinium infections in cultured ridgetail white prawns,Exopalaemoncarinicauda, in eastern China [J]. Aquaculture, 2010(300):25-31.
[11] Duan Y F, Jian L, Zhe Z, et al. Characterization of ADP ribosylation factor 1 gene fromExopalaemoncarinicauda, and its immune response to pathogens challenge and ammonia-N stress [J]. Fish & Shellfish Immunology, 2016(55):123-130.
[12] Zhang C S, Li F H, Xiang J H. Effect of salinity on growth and first sexual maturity ofExopalaemoncarinicauda(Holthuis, 1950) [J]. Chinese Journal of Oceanology & Limnology, 2014, 32(1):65-70.
[13] 梁俊平, 李健, 李吉濤, 等. 不同溫度對脊尾白蝦胚胎發育與幼體變態存活的影響[J]. 生態學報,2013, 33(4):1142-1152.
[14] Shen X, Sun M, Wu Z, et al. The complete mitochondrial genome of the ridgetail white prawnExopalaemoncarinicaudaHolthuis, 1950 (Crustacean:Decapoda:Palaemonidae) revealed a novel rearrangement of tRNA genes[J]. Gene, 2009, 437(1):1-8.
[15] 高煥, 趙蓮, 薛蓓, 等. 脊尾白蝦線粒體基因組應答饑餓的甲基化特征分析[J].水產學報, 2015, 39(7):953-960.
[16] 王緒峨.脊尾白蝦早期胚胎發育以及溫、鹽度與其孵化的關系[J]. 水產學報,1989,13(1):59-64.
[17] 梁象秋, 李亞娟, 周昭曼. 脊尾白蝦的幼體發育[J]. 水產學報, 1988,12(2):157-168.
[18] Grunau C, Renault E, Rosenthal A, et al. MethDB—a public database for DNA methylation data[J]. Nucleic Acids Research, 2001, 29(1):270-274.
[19] Jones P A. Functions of DNA methylation:islands, start sites, gene bodies and beyond[J]. Nature Reviews Genetics, 2012, 13(7):484-492.
[20] Huang Y Z, Zhan Z Y, Sun Y J, et al. Intragenic DNA methylation status down-regulates bovine IGF2, gene expression in different developmental stages[J]. Gene, 2014, 534(2):356-361.
[21] Wang R L, Li J, Staehelin C, et al. Expression analysis of two P450 monooxygenase genes of the tobacco cutworm moth (Spodopteralitura) at different developmental stages and in response to plant allelochemicals[J]. Journal of Chemical Ecology, 2015, 41(1):111-119.
[22] Pirola C J, Gianotti T F, Burgueo A L, et al. Epigenetic modification of liver mitochondrial DNA is associated with histological severity of nonalcoholic fatty liver disease [J]. Gut, 2013, 62(9):1356-1363.
[23] Dzitoyeva S, Hu C, Manev H. Effect of aging on 5-hydroxymethylcytosine in brain mitochondria [J]. Neurobiology of Aging, 2012, 33(12):2881-2891.
[24] Byun H M, Panni T, Motta V, et al. Effects of airborne pollutants on mitochondrial DNA methylation [J]. Particle & Fibre Toxicology, 2013, 10(1):18.
[25] Muntyan M S, Cherepanov D A, Malinen A M, et al. Cytochrome cbb3 of Thioalkalivibrio is a Na+-pumping cytochrome oxidase[J]. Proceedings of the National Academy of Sciences, 2015, 112(25):7695-7700.
[26] Hunte C, Zickermann V, Brandt U. Functional modules and structural basis of conformational coupling in mitochondrial complex I[J]. Science, 2010, 329(5990):448-451.
[27] Braun H P, Binder S, Brennicke A, et al. The life of plant mitochondrial complex I[J]. Mitochondrion, 2014(19):295-313.
[28] Eichten S, Borevitz J. Epigenomics:methylation′s mark on inheritance [J]. Nature, 2013, 495(7440):181-182.
MethylationProfileinMitochondrialGenomeofRidgetailWhitePrawnExopalaemoncarinicaudainResponsetoDifferentDevelopmentalStages
XUE Bei1,2,3, ZHANG Pei1,2,3, LI Zhihui1,2,3, ZHAO Lian1,2,3, LAI Xiaofang1,2,3,YAN Binlun1,2,3, GAO Huan1,2,3
( 1.Jiangsu Key Laboratory of Marine Biotechnology, College of Marine Life and Fisheries, Huaihai Institute of Technology, Lianyungang 222005, China; 2.Co-Innovation Center of Jiangsu Marine Bio-industry Technology, Lianyungang 222001, China; 3.The Jiangsu Provincial Platform for Conservation and Utilization of Agricultural Germplasm, Nanjing 210014, China )
BSP (Bisulfite sequencing PCR) primer pairs were screened in order to analyze the methylation profile of the mitochondria genome in response to different developmental stages in the ridgetail white prawnExopalaemoncarinicauda. Within ten synthesized BSP primer pairs, four primer pairs were selected for further studies because of their repeatable PCR amplification merits, including 13th (located in the 5′ end initiation zone of COX3), 16th (located in the 3′ sequence of COX3), 19th (located in the 5′ sequence of ND5) and 21st (located in the 3′ sequence of ND5). The methylation profile of different individuals at different developmental stages (fertilized eggs, larval, post larval and adult stages) were analyzed, using the above 4 primer pairs. The results showed that the methylation in both CpG sites and non-CpG sites were found in the 5′ and 3′ sequence of COX3 and the 5′ sequence of ND5, but not in the 3′ sequence of ND5. The 5′ sequence of COX3 was methylated at fertilized eggs and larval stage, while the 3′ sequence of COX3 was methylated at larval, post larval and adult stages. Additionally, the methylation ratio was decreased with the increasing of development stage for above two sequences of COX3. The 5′ sequence of ND5 was methylated in the all four developmental stages, and no significant differences for methylation ratio were found among different developmental stages. In view of the biology function of COX and ND genes which expression productions were responsible for maintaining the respiratory electron-transport chain in mitochondria, it is inferred that the function of methylation of mitochondrial genome in ridgetail white prawn is to regulate the energy metabolism during developmental stage.
Exopalaemoncarinicauda; mitochondrial genome; different developmental stage; methylation
10.16378/j.cnki.1003-1111.2017.05.015
2016-09-13;
2016-12-06.
江蘇高校優勢學科建設工程項目(PAPD);江蘇省高等學校自然科學研究重大項目(17KJA240001);江蘇省“六大人才高峰”創新人才團隊項目(2016-HYGC-CXTD-004);江蘇省2015年度普通高校研究生科研創新計劃項目(KYLX15_1486);連云港市產學研合作項目(CXY1517).
薛蓓(1992-),女,碩士研究生;研究方向:海洋生物繁殖與遺傳育種學.E-mail:malvsy@163.com.通訊作者:高煥(1976-),男,教授;研究方向:甲殼類種質資源開發及利用.E-mail:huanmr@163.com.
S917
A
1003-1111(2017)05-0628-06

