改良Trizol法提取富含花青素馬鈴薯塊莖的總RNA
劉芳 陳廣俠 馬蕾 楊煜 郭曉 楊曉慧 董道峰 楊元軍
摘要:富含花青素的馬鈴薯塊莖因含有大量多糖和多酚類次級代謝產物而難以大量提取高質量的總RNA。本研究以紫色和紅色馬鈴薯為材料,利用改良Trizol法實現了塊莖總RNA的快速高質量提取。試驗結果表明,在Trizol法中添加2%(V/V)-巰基乙醇、0.75%(W/V)十六烷基三甲基溴化銨(CTAB)和7.5%(W/V)醋酸鉀(KOAc),可以有效去除多糖、多酚類物質,獲得高產、完整的總RNA。改良Trizol法提取時間短、操作方便,提取的RNA通過了actin RT-PCR和18S rRNA RT-qPCR檢驗,可以用于后續的分子生物學試驗。
關鍵詞:馬鈴薯;塊莖;花青素;RNA提取;醋酸鉀;改良Trizol法
中圖分類號:S532.035.3文獻標識號:A文章編號:1001-4942(2018)12-0114-05
馬鈴薯是糧菜兼用作物,具有豐富的營養,有“地下蘋果”的美譽。紫色和紅色馬鈴薯除具有普通馬鈴薯的優良性狀,還含有大量花青素[1]。花青素具有殺菌、清除氧自由基和抗衰老的作用,可以降低心腦血管疾病、癌癥和糖尿病等的發生[2, 3]。隨著人們對富含花青素馬鈴薯重要性認識的增加,此類材料的分子生物學研究也逐漸開展起來。RNA提取是分子生物學研究的重要環節,RT-PCR、RT-qPCR、Northern雜交、cDNA文庫構建均需要高質量的RNA。目前,用于馬鈴薯塊莖RNA的提取方法主要有Trizol法[4, 5]、CTAB法、異硫氰酸胍法[6]、SDS-酚法[7]、LiCl法[8]。由于富含花青素的馬鈴薯塊莖含有大量多糖、酚類等次級代謝產物[9, 10],這些提取方法難以滿足此類馬鈴薯塊莖總RNA提取的需要。獲得大量高質量的塊莖總RNA成為富含花青素馬鈴薯分子生物學研究的迫切需要,因此本試驗采用改良Trizol法研究了紫色和紅色馬鈴薯塊莖RNA的高質量提取方法,以期為富含花青素馬鈴薯材料的分子生物學研究奠定基礎。
1?材料與方法
1.1?供試材料與試劑
供試材料:馬鈴薯四倍體材料SD92和SD140,其中SD92為紫皮紫肉類型,SD140為紅皮紅肉類型。SD92和SD140種植于溫室。70 d收獲,收獲后的塊莖洗凈后用吸水紙吸干水分,迅速切成片后用液氮速凍,置于-80℃保存備用。
試劑:焦碳酸二乙酯(DEPC)、交聯聚乙烯吡咯烷酮(PVPP)、十二烷基磺酸鈉(SDS)、三羥甲基氨基甲烷(Tris)、乙二胺四乙酸(EDTA)、氯化鋰(LiCl)、低熔點瓊脂糖(Agarose)、溴化乙錠(EB)均為 Sigma 公司產品;Trizol 試劑購自Ambion(USA);PrimeScriptⅡ 1st Strand cDNA Synthesis Kit為TaKaRa產品;DNaseⅠ購自Thermo(USA); RT-qPCR采用康為世紀公司UltraSYBR Mixture;Marker為DL1000 DNA Marker (TaKaRa),引物由上海生工合成;醋酸鉀(KOAc)、醋酸鈉(NaAc)等其他試劑均為國產分析純。
器皿處理:將所用的槍頭及離心管用0.1% DEPC滅菌水浸泡12 h,隨后121℃滅菌20 min,倒掉廢液,然后80℃烘8 h。對于所用的研缽和金屬藥勺,用錫箔紙封口后在 180℃烘烤8 h。所用試劑均用無RNase的0.1% DEPC滅菌水配制。
1.2?RNA提取方法
方法一:采用Trizol法和改良的Chan法提取RNA,Trizol法參照說明書,改良的Chan法具體步驟參照趙寶勰等的方法[9]。
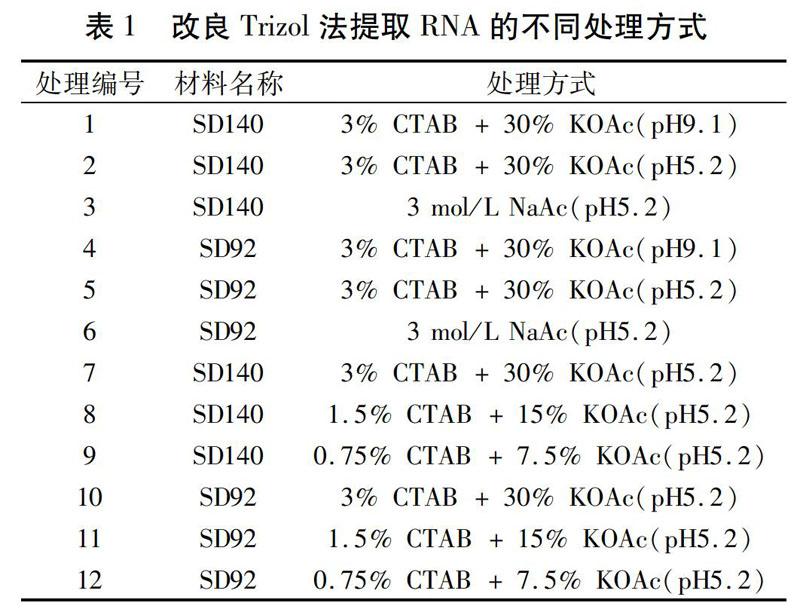
方法二:采用改良的Trizol法提取RNA,具體步驟如下:(1)取馬鈴薯塊莖適量,加液氮和少許PVPP充分研磨,然后取0.1 g研磨材料,加入1 mL的Trizol和2% (V/V) -巰基乙醇混勻,室溫放置10 min后,4℃、12 000 r/min離心10 min;(2)取上清于1.5 mL離心管中,加入200 μL氯仿混勻,室溫放置10 min后,4℃、12 000 r/min離心10 min;(3)取上清于1.5 mL離心管中,然后分別加入1/4氯仿和等于上清體積的其他溶液(表1)顛倒混勻,室溫放置10 min,12 000 r/min離心10 min;(4)取上清,加入等體積的異丙醇,顛倒混勻,室溫放置10 min,12 000 r/min離心10 min;(5)棄上清,加入1 mL的75%(V/V)預冷乙醇短暫離心,洗滌沉淀后,室溫晾干約5~10 min。(6)RNA干燥后用20 μL DEPC-H2O溶解,-80℃保存。140 V(6 V/cm)電泳15 min,溴化乙錠(EB)染色,Bio-Rad (USA)凝膠成像儀檢測RNA的完整性;利用 Nanophotometer P330 (IMPLEN,Germany)測定RNA的質量和濃度。
1.3?RNA的RT-PCR和RT-qPCR檢測
首先采用DNase Ⅰ(Thermo,USA)對RNA進行消化,具體步驟如下:取2 μL采用1.5% CTAB+15% KOAc(pH5.2)提取的RNA,1 μL 10buffer,1 μL DNaseⅠ,DEPC-H2O補足10 μL。37℃消化60 min,最后加入50 mmol/L EDTA 1 μL,65℃、10 min滅活DNaseⅠ。PCR檢測消化結果。取1 μL消化完全的RNA為模板進行反轉錄,反轉錄采用PrimeScriptⅡ 1st Strand cDNA Synthesis Kit(TaKaRa),具體步驟參照說明書。利用PCR擴增馬鈴薯肌動蛋白基因actin(XM_006351284.1)片段來檢測消化和反轉錄的結果。PCR反應體積為50 L,引物分別為actin F:5′-GATGGTGTCAGCCACAC-3′和actin R:5′-ATTCCAGCAGCTTCCATTCC-3′。PCR程序:94℃預變性3 min;95℃變性30 s,55℃退火30 s,72℃延伸1 min,40個循環;最后72℃延伸10 min。分別以1 L消化產物和反轉錄產物為模板進行PCR擴增actin片段,最后1% 瓊脂糖電泳檢測消化和反轉錄結果。
對反轉錄產物進行RT-qPCR檢測,50 μL反應體系,25 μL 2UltraSYBR Mixture (CWBIO)、1 μL cDNA、0.2 μmol/L引物和ddH2O。RT-qPCR反應在iCycler iQ (Bio-Rad,Hercules,CA,USA)中進行,流程為95℃ 15 s,60℃ 1 min,37個循環,3次重復。擴增基因為18S rRNA,引物分別為18S F:5′-CCTGGTCGGCATCGTTTA-3′,18S R:5′-CGAACAACTGCGAAAGCAT-3′。
2?結果與分析
2.1?Trizol法與改良Chan法提取RNA
Trizol法提取RNA時,未見紫色馬鈴薯和紅色馬鈴薯塊莖的RNA條帶,RNA未能成功提取,同時沒有DNA殘留。利用改良Chan法提取出的RNA有彌散帶,RNA降解嚴重,并且有大分子量的條帶,說明有DNA殘留。
2.2?改良Trizol 法提取RNA
在紫色馬鈴薯SD92和紅色馬鈴薯SD140塊莖中,3個處理均可以提取出RNA。而且3% CTAB + 30% KOAc(pH9.1)處理和3% CTAB + 30% KOAc(pH5.2)處理效果最佳,18S rRNA和28S rRNA條帶清晰完整,且RNA產量較高。而3 mol/L NaAc(pH5.2)處理提取的RNA產量明顯少于前兩個處理,尤其是在SD140中,效果更為明顯。
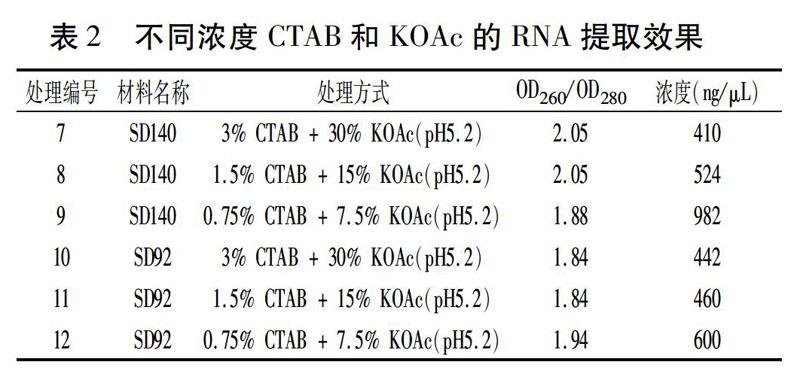
不同濃度CTAB和KOAc組合可以提取SD92 和SD140塊莖中完整的RNA,包括28S、18S、5S rRNA。從表2可以看出,在0.75%~3% CTAB和7.5%~30% KOAc時,CTAB和KOAc濃度越低,提取的RNA總量越大。添加3% CTAB和30% KOAc時,SD140和SD92的RNA提取量分別為410 ng/μL和442 ng/μL,而在0.75% CTAB和7.5% KOAc時,它們的提取量分別為982 ng/μL和600 ng/μL,后者的提取量是前者的2.40倍和1.36倍。另外,在SD92和SD140提取RNA的3種處理中,提取的RNA OD260/OD280都大于1.80,說明蛋白質的殘留量很低,RNA質量良好。
利用管家基因actin擴增檢測Trizol提取RNA的消化和反轉錄效果。通過PCR,RNA、cDNA和DNA均能擴增出actin條帶,消化后的RNA未擴增出條帶,說明DNA消化完全,提取的RNA質量優,雜質殘留量低,可用于后續的反轉錄等反應。
利用18S rRNA檢測改良Trizol法提取RNA反轉錄后cDNA的RT-qPCR效果。兩種材料中18S rRNA的擴增曲線是光滑的,兩個樣品的Ct值< 20,并且熔解曲線是單一峰,說明反轉錄的cDNA達到了熒光定量檢測的要求,表明改良Trizol法提取的RNA可以用于后續的熒光定量檢測。
3?討論與結論
富含花青素的馬鈴薯塊莖中含有多糖、酚類化合物,蛋白質和其他次級代謝產物,采用Trizol法和改良的Chan法不能從該類塊莖中提取出完整的RNA。Trizol法簡單方便、耗時較短,但不能有效除掉塊莖中的多糖。而RNA與多糖可以形成共沉淀[11],導致RNA難以分離。Chan法提取RNA時,使用3 mol/L NaAc有效去除塊莖中的多糖,但是步驟繁瑣、耗時較長,導致RNA出現了降解;并且該法利用LiCl選擇性沉淀RNA效果不佳,導致大量DNA殘留。
改良Trizol法提取RNA純度高、完整性好,可以用于后續的RT-PCR和RT-qPCR等分子生物學試驗。該法操作簡單方便,單樣品提取時間約90 min。改良Trizol法較Trizol法在提取過程中添加了PVPP、β-巰基乙醇、CTAB和KOAc。PVPP可以與酚類物質結合[12],從而除去塊莖中的酚類。β-巰基乙醇作為一種變性劑可以去除RNase和其他一些活性酶,有效阻止RNA的降解和酚類向醌類的轉化[13, 14]。由于在低鹽條件下,多糖會與核酸形成膠狀沉淀[15, 16],所以提取RNA時需要高鹽環境,而常用的NaCl對RNA提取的效果有限[17]。在本試驗中,采用高濃度的KOAc和NaAc去除多糖,與KOAc相比,NaAc提取RNA的產量較低,說明Na+對RNA的提取效果差于K+,采用KOAc去除多糖效果更好。CTAB作為一種很強的離子變性劑,可以使核酸從植物組織中充分分離[18, 19]。KOAc和CTAB共同使用,可以使RNA與多糖等其它代謝產物在充分分離的條件下,獲得高純度的RNA。
總之,改良Trizol法可以高效地提取富含花青素馬鈴薯塊莖的RNA,提取的RNA可以用于后續的基因表達分析等分子生物學試驗。此法對于其它富含多糖、多酚和花青素等代謝產物作物RNA的提取提供了參考。
參?考?文?獻:
[1]?Brown C R, Culley D, Bonierbale M, et al. Anthocyanin, carotenoid content, and antioxidant values in native South American potato cultivars[J]. HortScience, 2007, 42(7): 1733-1736.
[2] Liu Y H, Wang K L, Deng C, et al. Comparative transcriptome analysis of white and purple potato to identify genes involved in anthocyanin biosynthesis[J]. PLoS ONE, 2015,10(6):e0129148.
[3] Bontempo P, Carafa V, Grassi R, et al. Antioxidant, antimicrobial and anti-proliferative activities of Solanum tuberosum L. var. Vitelotte[J]. Food and Chemical Toxicology, 2013, 55: 304-312.
[4] 劉柏林, 尤佳, 張寧, 等. 3種商品化Trizol試劑提取高質量馬鈴薯塊莖總RNA的比較[J]. 甘肅農業大學學報, 2012, 2(1): 59-63.
[5] Wei Q, Wang Q L, Feng Z H, et al. Increased accumulation of anthocyanins in transgenic potato tubers by overexpressing the 3GT gene[J]. Plant Biotechnology Reports, 2012, 6:69-75.
[6] 王偉, 李立芹, 鄒雪, 等. 馬鈴薯塊莖總RNA提取方法比較[J]. 江蘇農業科學, 2013, 41(1): 38-40.
[7] 白斌, 張金文. 馬鈴薯塊莖RNA提取及RT-PCR法克隆酸性轉化酶基因[J]. 分子植物育種, 2005, 3(3): 479-484.
[8] Stushnoff C, Ducreux L J M, Hancock R D, et al. Flavonoid profiling and transcriptome analysis reveals new gene-metabolite correlations in tubers of Solanum tuberosum L.[J]. Journal of Experimental Botany, 2010, 61(4): 1225-1238.
[9] 趙寶勰, 程李香, 林必博, 等. 兩種馬鈴薯塊莖總RNA提取方法的比較[J]. 廣東農業科學, 2010(1):124-127.
[10]Lachman J,Hamouz K. ?Nutrition-a review[J]. Plant Soil and Environment, 2005, 51: 477-482.
[11]Gao J W, Liu J Z, Li B, et al. Isolation and purification of functional total RNA from blue-grained wheat endosperm tissues containing high levels of starches and flavonoids[J]. Plant Molecular Biology Reporter, 2001, 19(2): 185-186.
[12]Bridi R, Troncoso M J, Folch-Cano C, et al. A Polyvinylpolypyrrolidone (PVPP)-assisted folin-ciocalteu assay to assess total phenol content of commercial beverages[J]. Food Analytical Methods,2014,7(10): 2075-2083.
[13]Rashid A, Baldwin T, Gines M, et al. A high-throughput RNA extraction for sprouted single-seed barley (Hordeum vulgare L.) rich in polysaccharides[J]. Plants, 2017,6(1):1.
[14]Tong Z G, Qu S C, Zhang J Y, et al. A modified protocol for RNA extraction from different peach tissues suitable for gene isolation and real-time PCR analysis[J]. Molecular Biotechnology, 2012, 50: 229-236.
[15]Sabzevari A G, Hosseini R. A quick, efficient, and cost-effective method for isolating high-quality total RNA from tomato fruits, suitable for molecular biology studies[J]. Preparative Biochemistry & Biotechnology, 2014, 44: 418-431.
[16]Birtic S, Kranner I. Isolation of high-quality RNA from polyphenol, polysaccharide and lipid-rich seeds[J]. Phytochemical Analysis, 2006, 17(3):144-148.
[17]Deepa K, Sheeja T E, Santhi R, et, al. A simple and efficient protocol for isolation of high quality functional RNA from different tissues of turmeric (Curcuma longa L.)[J]. Physiological and Molecular Biology Plants, 2014, 20(2):263-271.
[18]Kim S H, Hamada T. Rapid and reliable method of extracting DNA and RNA from sweet potato, Ipomoea batatas (L). Lam[J]. Biotechnology Letters, 2005, 27(23): 1841-1845.
[19]Wang X C, Tian W M, Li Y X. Development of an efficient protocol of RNA isolation from recalcitrant tree tissues[J]. Molecular Biotechnology, 2008, 38(1): 57-64.

