侵染蠶豆ClYVV的鑒定及其衍生的小干擾RNA的深度測序鑒定研究
張趁華,鄭紅英,嚴丹侃,韓科雷,4,彭杰軍,魯宇文,林 林,章東方,陳劍平,*, 燕 飛,*
(1.福建農林大學 生命科學學院,福建 福州350002; 2.浙江省農業科學院 病毒學與生物技術研究所,浙江省和農業部植保生物技術重點實驗室,省部共建浙江省植物有害生物防控國家重點實驗室培育基地,浙江 杭州310021; 3.安徽省農業科學院 植物保護與農產品質量安全所,安徽 合肥230031; 4.南京農業大學 植物保護學院,江蘇 南京 210095)
馬鈴薯Y病毒屬(Potyviruses)病毒是一類重要的植物病原,是馬鈴薯Y病毒科(Potyviridae)的第一大屬,同時也是最大的RNA病毒屬。該屬病毒基因組有1條正單鏈RNA構成,基因組編碼一個多聚蛋白,并由自身蛋白酶裂解成多個具有不同功能的病毒蛋白,電鏡觀察下病毒粒子呈無包膜彎曲線狀。該屬病毒分布廣,能侵染豆科、茄科、菊科、莧科和藜科等多科植物,是大豆、馬鈴薯、煙草、玉米等重要農作物的主要病害,可造成多種作物明顯減產,對農業生產安全可造成嚴重威脅[1-2]。
三葉草黃脈病毒(Cloveryellowveinvirus, ClYVV)是馬鈴薯Y病毒屬的成員,基因組是正義單鏈RNA,全長約有10 kb,該基因編碼一個大的多聚蛋白,由自身蛋白酶裂解成11個成熟蛋白質[3-4]。ClYVV由英國的Hollings 和 Nariani在白三葉草中首次分離并報道[5]。目前,ClYVV是世界范圍內侵染豆科植物、觀賞植物和牧草的分布式病原體之一[6-13],可引起植株新鮮綠豆莢變形壞死、葉片早落、發育遲緩,嚴重的可引起植株全身壞死,扭曲變形壞死等多種病害癥狀,部分病例可致植株死亡,導致直接的經濟損失[14-15]。
在中國,僅在二十多年前的山東根據ClYVV生物學和血清學的特征檢測到幾例病樣[16-17],但到目前為止,這種病毒還沒有進一步的分子鑒定。本研究以安徽省合肥市具有明顯花葉癥狀的蠶豆新生葉片為研究對象,利用病毒來源的siRNA的重新測序來重建和測序侵染蠶豆的病毒,然后通過逆轉錄聚合酶鏈式反應(RT-PCR)驗證序列的準確性。此外,還分析了來自ClYVV的病毒sRNA讀取的表征。
1 材料與方法
1.1 試驗材料
在安徽省合肥市采集具有明顯花葉癥狀的蠶豆新生葉片,采集后迅速投于液氮中,置于-80 ℃冰箱保存備用。
1.2 試驗方法
1.2.1 蠶豆病葉組織RNA的提取和小RNA深度測序分析
應用總RNA純化試劑盒(LC Sciences, Cat. TRK-1001)從50 mg采集的蠶豆病葉組織中提取總RNA,對RNA進行定量并提交給LC-Bio Inc.(中國杭州)。用TruSeq小RNA樣品制備試劑盒制備小RNA文庫,并在HiSeq2000平臺(Illumina)上從兩端(配對末端)測序。序列讀取并修剪,以除去低質量和接頭序列,并使用Velvet Zerbino DR和PFOR Wu QF從頭組裝算法進行組裝。通過對GenBank數據庫的BLASTn和BLASTx搜索來分析組裝的重疊群,并且手動組裝重疊的重疊群。
1.2.2 蠶豆樣品雙鏈RNA的提取、cDNA的合成和ClYVV病毒序列擴增
為了進一步鑒定病毒,使用雙鏈RNA提取方法[18],從10 g蠶豆病葉樣品中提取病毒雙鏈RNA(dsRNA)。使用ReverTra Ace-α-TM(Toyobo)[19],用引物M4-T合成第一鏈cDNA。使用cDNA末端的快速擴增(RACE)PCR對病毒基因組的5′ 和3′ 末端進行PCR,并使用重疊的特異性引物對進行常規RT-PCR(引物見表1),擴增拼接了ClYVV-HF的全基因組序列,序列比較用CLUSTALW[20]完成。
表1ClYVV PCR擴增引物序列
Table1The PCR amplification primer sequence of ClYVV

引物名稱Primername引物序列Primersequence(5?3’)擴增片段長度Amplifiedfragmentlength/bpClYVV?1ClYVV?1(+):AAAATATAAAATCAATACAAGACAAATClYVV?1(-):CTCTTTATTACACCTCTCAC1023ClYVV?2ClYVV?2(+):GTGAGAGGTGTAATAAAGAGClYVV?2(-):ATGTATTTCGCTGCTTCCTG1067ClYVV?3ClYVV?3(+):CAGGAAGCAGCGAAATACATClYVV?3(-):GACAATCTGAGTGATGCGCTG1049ClYVV?4ClYVV?4(+):CAGCGCATCACTCAGATTGTCClYVV?4(-):CAAAGGAAAGTTGGTCTTGC1280ClYVV?5ClYVV?5(+):GCAAGACCAACTTTCCTTTGClYVV?5(-):ACTATGTAGCACGCTCAAGT1138ClYVV?6ClYVV?6(+):ACTTGAGCGTGCTACATAGTClYVV?6(-):ACTCTGCATCAATATCCTCATTC846ClYVV?7ClYVV?7(+):GAATGAGGATATTGATGCAGClYVV?7(-):CTCAGTGTCAACCAAGCCA1024ClYVV?8ClYVV?8(+):TGGCTTGGTTGACACTGAGClYVV?8(-):TGAGCAAGTCATCATAACCC1097ClYVV?9ClYVV?9(+):GGGTTATGATGACTTGCTCAClYVV?9(-):CTCGCTCTATAAAGATCAGA1223
1.2.3 ClYVV的生物學檢測和電鏡觀察
取適量的蠶豆病葉組織,在液氮中研磨至粉末狀,加入滅菌的1×PBS 溶液混合均勻后,摩擦接種本氏煙、豇豆、黃豆,四季豆和蠶豆,觀察是否出現病毒病病癥;若有病癥,采集病葉,進行RT-PCR檢測后(檢測引物為(+):CATTCCCACTTAAGCCAATTC; (-):AAGATCAGATTCACAACGAGG)用2%的磷鎢酸溶液負染,在電鏡下觀察。
2 結果與分析
2.1 蠶豆病葉組織小RNA深度測序初步分析結果
小RNA深度測序初步分析結果表明,除去宿主起源的重疊群之后,剩余的重疊群與馬鈴薯Y病毒屬中的病毒具有高度的序列相似性,總共1 361 555個重疊群被定位到ClYVV,組裝的病毒重疊群覆蓋了ClYVV模板的100%。
2.2 ClYVV病毒序列擴增和組裝
為了驗證由sRNA讀取組裝的ClYVV基因組,以表1中的引物通過RT-PCR,擴增到了ClYVV-HF基因組序列,證實了ClYVV的存在(圖1)。將擴增的PCR產物切膠回收,與載體連接檢測后送公司測序,將測序得到的片段拼接,獲得ClYVV-HF分離株完整的基因組全序列。獲得的ClYVV-HF分離株的完整的基因組排除3′ 末端的poly(A)尾巴外長度為9 585個核苷酸(nt),其中5′-UTR為191 nt;P1為906 nt,酶切位點位于F/S;Hc-Pro為1 371 nt,酶切位點位于F/S和G/G;P3為1 044 nt,酶切位點位于G/G和E/S;P3N-PIPO為179 nt,為移碼產生的蛋白;6K1為159 nt,酶切位點位于E/S和Q/S;CI為1 905 nt,酶切位點位于Q/S和E/S;6K2為159 nt,酶切位點位于E/S和Q/G;NIa-VPg為573 nt,酶切位點位于Q/G和E/S;NIa-Pro為729 nt,酶切位點位于E/S和Q/A;NIb為1 558 nt,酶切位點位于Q/A和Q/S;CP為811 nt,酶切位點位于Q/S(圖2)。該序列已上傳到GenBank數據庫中(登錄號為KU922565)。
2.3 ClYVV的生物學檢測和電鏡觀察
蠶豆病葉組織摩擦接種本氏煙、豇豆、黃豆、四季豆和蠶豆,3~4周后均表現出病毒病癥,其系統葉片表現出斑駁、褶皺、葉脈黃化等癥狀(圖4-a,b,d,e,f),取摩擦接種病毒植株的系統葉片首先進行RT-PCR檢測,擴增出的片段條帶單一,大小與預期結果相符(圖3),將擴增的PCR產物切膠回收,與載體連接檢測后送公司測序,所得序列為ClYVV特異性片段序列。將檢測后的帶毒病葉進行電鏡觀察,可觀察到絲狀病毒顆粒的存在(圖4-c),表明ClYVV可通過汁液摩擦接種多種豆科作物。
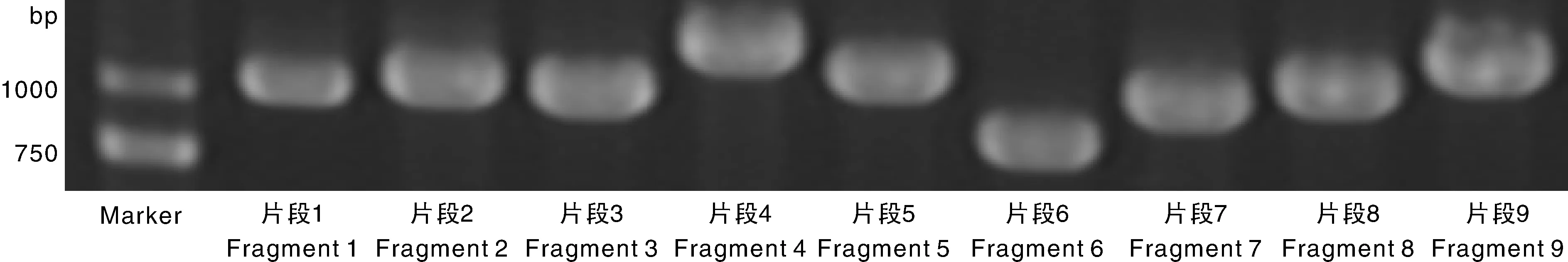
圖1 ClYVV-HF病毒基因序列的分段擴增Fig.1 Nine fragments amplified of CGMMV-HF

圖2 ClYVV-HF基因組結構示意圖Fig.2 Genome organization of ClYVV-HF
2.4 ClYVV小RNAs分析
進一步分析了ClYVV衍生的小干擾RNA的表征,來源于蠶豆樣本的ClYVV與其基因組相匹配的sRNAs序列主要分布在19~25 nt,其中以21 nt和22 nt核苷酸為主(圖5-A)。其中,vsiRNAs的大小為21 nt,占總RNA的66.84%,其次為22 nt,占總RNAs的26.76%(圖5-A),ClYVV vsiRNA的大小類別分析結果表明,21nt長的siRNA是其主要的抗病毒沉默組分,而DCL4是病毒siRNA的主要生產者。為了確定具有不同AGO復合物的vsiRNA之間的潛在相互作用,分析了其5′-末端核苷酸的vsiRNA的相對豐度。生物信息學分析揭示了來源于ClYVV的siRNA 5′-末端核苷酸腺苷(A)和尿苷(U)發生了強偏差(圖5-B),這個結果與先前報道的與優先結合親和力的AGO蛋白的小RNA 5′末端U(AGO1)和A(AGO2和AGO4)的報道一致[21-22]。為了探索vsiRNAs的起源,對vsiRNAs的極性分布進行了表征分析。比對結果顯示,來自ClYVV基因組正義鏈RNAs的vsiRNA(57.59%)高于源自ClYVV基因組反義鏈RNAs的vsiRNA(42.41%)(圖5-C),兩種不同來源的vsiRNA的比例為1.36︰1.0。其中,在ClYVV編碼的11個基因中,產生自CI編碼框的vsiRNA數量最多,其次是HC-Pro和NIb;另一方面,在ClYVV的所有基因中,正義鏈衍生的vsiRNA數量均多于負義鏈衍生的vsiRNA數量(1.41∶1.00),其中,來源于CP、6K1和VPg基因正義鏈衍生的vsiRNA數量分別比反義鏈衍生的vsiRNA數量分多71%、63%和55%,相比之下,在3′-UTR和5′-UTR區域,情況則相反(0.66∶1.00)(圖5-D)。為了鑒定來自ClYVV基因組的vsiRNA產生的熱點,將vsiRNA與ClYVV基因組序列進行了比對。在基因組中廣泛分布的ClYVV衍生的siRNA揭示了除了在ClYVV基因組中有超過800個讀段的7個熱點外,整個ClYVV基因組對ClYVV衍生的siRNA的形成提供了相對均勻的貢獻,在產生的這7個熱點中,21-nt vsiRNA占主導(圖5-E)。
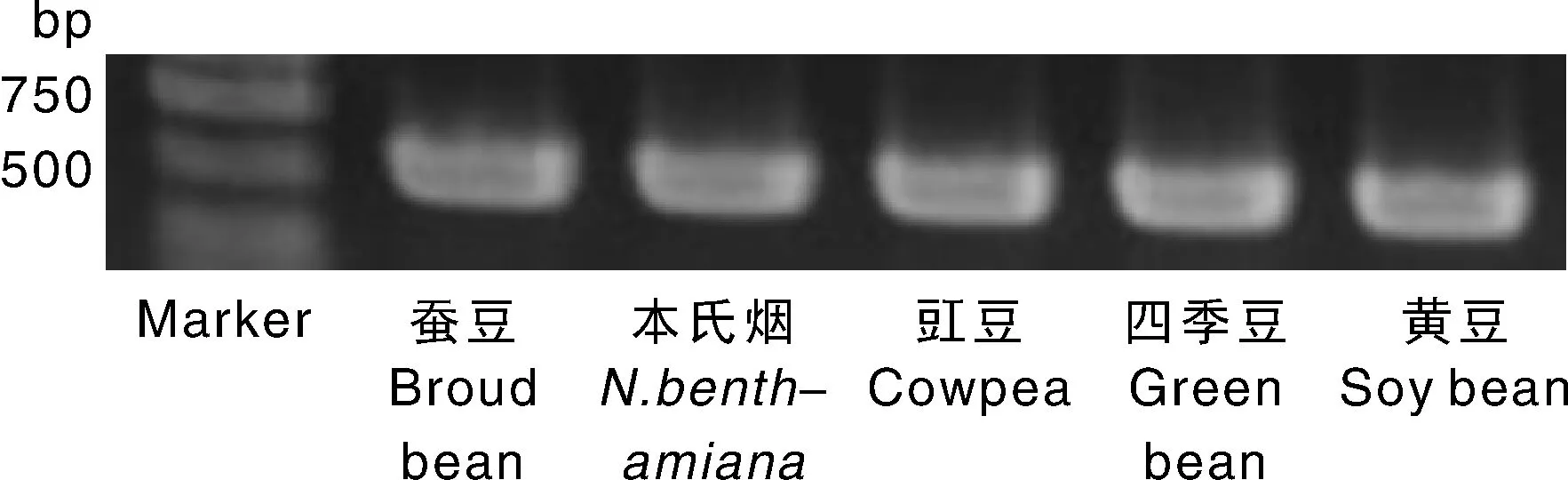
圖3 ClYVV摩擦接種相關寄主后的RT-PCR檢測結果Fig.3 The RT-PCR results of ClYVV infected hosts

a,蠶豆;b,本氏煙;c,電鏡圖片;d,豇豆;e,四季豆;f,黃豆。a, Broad bean; b, N.benthamiana; c, Electron microscopy picture; d, Cowpea; e, Green bean; f, Soybean.圖4 ClYVV摩擦接種相關寄主后的癥狀表現及電鏡觀察Fig.4 Symptoms and electron microscopic observation of ClYVV infected hosts
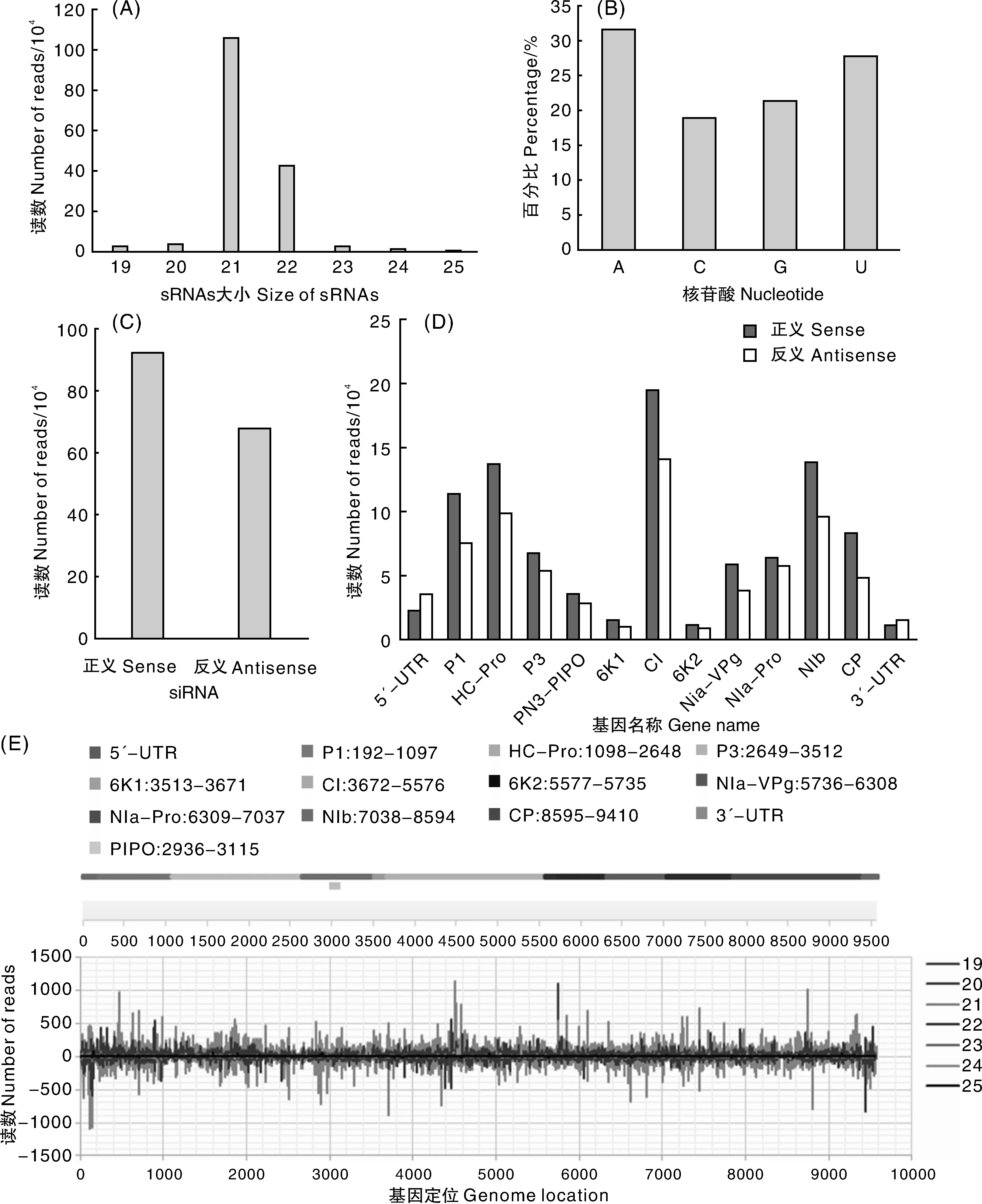
A,來源于蠶豆樣本的ClYVV與其基因組相匹配的病毒sRNA序列的分布;B,來源于ClYVV的siRNA 5′-末端核苷酸的豐度分析;C,來源于ClYVV基因組正義與反義siRNA統計分析;D,來源于ClYVV各組分和UTRs的正義和反義siRNA統計分析;E,與ClYVV基因組相關的ClYVV來源的siRNA簡介;軸上方的條表示衍生自病毒基因組(有義)鏈的siRNA,而軸下方的是衍生自互補(反義)鏈的siRNA。A, Distribution of virus sRNA sequences matching ClYVV genome from ClYVV infected broad bean; B, 5′-terminal nucleotide abundance of ClYVV-derived siRNA analyzed as unique sequences; C, Distinguished as sense (+) or antisense (-) polarity with respect to the ClYVV genome; D, Total sense versus antisense siRNA reads from individual genes and the UTRs of ClYVV genome; E, Profile of ClYVV-derived siRNAs along the ClYVV genome. Bars above the axis represent siRNAs derived from the viral genomic (sense) strand, and those below the axis are siRNAs derived from the complementary (antisense) strand.圖5 蠶豆來源的siRNAs簡要分析Fig.5 Genome organization of ClYVV Anhui isolates
3 討論
在病毒侵染植物中,對應于病毒基因組的小干擾RNA(siRNA)形成大部分小RNA群體。許多病毒已經通過從重疊siRNA序列中重組病毒序列被成功鑒定出來[23-24]。通過病毒衍生小RNA的深度測序和組裝,研究發現,安徽省合肥市蠶豆中存在ClYVV,并使用了重疊的特異性引物對其進行常規RT-PCR,擴增拼接了ClYVV-HF的全基因組序列,同時還分析了ClYVV-HF衍生的小干擾RNA的特征。
迄今為止,僅有3個ClYVV完整序列被報道,其中一個為韓國大豆來源(KF975894)[25],2個具有相同基因組,為日本的蠶豆來源(AB011819、NC_003536)[26-27]。本研究的ClYVV-HF全長核苷酸序列與日本和韓國分離株的核苷酸同源性分別為95.45%和95.10%,氨基酸同源性分別為100%和98.44%。考慮到這些菌株之間的保守性,本研究認為中國的ClYVV和日本的ClYVV可能屬于同一菌株。而對于植物病毒vsiRNA的研究,在先前很多報道中,21 nt和22 nt核苷酸在vsiRNA中所占比重較大[28-30],vsiRNA的大小分布反映了RNA沉默應對RNA病毒的途徑。
隨著農業經濟和運輸業的高速發展,植物病毒也呈現出一種高速蔓延的趨勢。我國是豆科作物的種植大國,本研究表明ClYVV可通過汁液摩擦接種在多種豆科植物上進行機械傳毒,豆科作物種植過程中的不當操作以及農產品的高速流通都有可能引起ClYVV的廣泛傳播,造成經濟損失。本研究是關于中國ClYVV分離株全部核苷酸序列的首次報道。研究獲得的序列數據將進一步為中國豆類植物和其他寄主上發生的ClYVV流行病學提供研究基礎和見解。
[1] 馬雪青, 王永剛, 周賢婧, 等. 馬鈴薯病毒研究新進展[J]. 食品工業科技, 2010, 31(10):429-434.
MA X Q, WANG Y G, ZHOU X J, et al. Research advances of potato virus[J].ScienceandTechnologyofFoodIndustry, 2010, 31(10):429-434.(in Chinese with English abstract)
[2] 尹明華, 劉燕, 郁雪婷,等. 懷玉山高山馬鈴薯莖尖再生苗6種病毒的DAS-ELISA檢測與分析[J]. 浙江農業學報,2017,29(10):1699-1705.
YIN M H, LIU Y, YU X T, et al. DAS-ELISA detection and analysis of six kinds of viruses in plantlets regenerated from Hua-iyushan high mountain potato shoot-tips[J].ActaAgriculturaeZhejiangensis, 2017,29(10):1699-1705. (in Chinese with English abstract)
[3] CHUNG B Y, MILLER W A, ATKINS J F, et al. An overlapping essential gene in the Potyviridae[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 2008, 105(15): 5897-5902.
[4] SASAYA T, SHIMIZU T, NOZU Y, et al. Biological, serological, and molecular variabilities of clover yellow vein virus[J].Phytopathology, 1997,87(10):1014-1019.
[5] HOLLINGS M, NARIANI T. Some properties of clover yellow vein, a virus fromTrifoliumrepensL.[J].AnnalsofAppliedBiology, 1965, 56(1): 99-109.
[6] FOSTER R, MUSGRAVE D. Clover yellow vein virus in white clover (Trifoliumrepens) and sweet pea (Lathyrusodoratus) in the North Island of New Zealand[J].NewZealandJournalofAgriculturalResearch,1985, 28(4):575-578.
[7] IREY M, ADKINS S, BAKER C. Clover yellow vein virus Identified in Ammi majus in Florida[J].PlantDisease, 2006, 90(3):380.
[8] KEHOE M A, COUTTS B A, BUIRCHELL B J, et al. Plant virology and next generation sequencing: experiences with a Potyvirus[J].PLoSOne, 2014, 9(8):e104580
[9] LARSEN R C, MIKLAS P N, EASTWELL K C, et al. A strain of Clover yellow vein virus that causes severe pod necrosis disease in snap bean[J].PlantDisease, 2008, 92(7):1026-1032.
[10] MORAN J, VAN RIJSWIJK B, TRAICEVSKI V, et al. Potyviruses, novel and known, in cultivated and wild species of the family Apiaceae in Australia[J].ArchivesofVirology, 2002, 147(10):1855-1867.
[11] ORTIZ V, CASTRO S, ROMERO J. First report of Clover yellow vein virus in grain legumes in Spain[J].PlantDisease, 2009, 93(1):106.
[12] PASEV G, KOSTOVA D, TURINA M. A New virulent isolate of Clover yellow vein virus onPhaseolusvulgarisin Bulgaria[J].JournalofPhytopathology, 2014, 162(11/12):703-711.
[13] YAMAMOTO H. Mosaic disease of bupleurum (Bupleurumgriffithii) caused by Clover yellow vein virus[J].AnnalsofthePhytopathologicalSocietyofJapan, 2003, 69(4):420-421.
[14] HART J P, GRIFFITHS P D. A series of eIF4E alleles at the Bc-3 locus are associated with recessive resistance to Clover yellow vein virus in common bean[J].TheoreticalandAppliedGenetics, 2013, 126(11):2849-2863.
[15] LARSEN R, MYERS J. A pod necrosis disease (chocolate pod) of snap bean (Phaseolusvulgaris) in Oregon caused by a strain of Clover yellow vein virus[J].Phytopathology, 2006, 96:S169.
[16] 李長松, 朱漢城. 侵染菜豆的三葉草黃脈病毒的鑒定[J]. 山東農業大學學報, 1989(4):223-226.
LI C S, ZHU H C. Identification of Clover yellow vein virus infectingPhaseolusvulgaris[J].JournalofShandongAgriculturalUniversity, 1989(4):223-226.(in Chinese with English abstract)
[17] 李長松. 侵染扁豆的三葉草黃脈病毒的鑒定[J]. 中國病毒學, 1991,6(3):223-226.
LI C S. Identification of Clover yellow vein virus infecting Dolichos lablab[J].VirologicaSinica, 1991,6(3):223-226.(in Chinese with English abstract)
[18] TZANETAKIS I E, MARTIN R R. A new method for extraction of double-stranded RNA from plants[J].JournalofVirologicalMethods, 2008, 149(1):167-170.
[19] CHEN J, ADAMS M. A universal PCR primer to detect members of the Potyviridae and its use to examine the taxonomic status of several members of the family[J].ArchivesofVirology, 2001, 146(4):757-766.
[20] LARKIN M A, BLACKSHIELDS G, BROWN N, et al. Clustal W and Clustal X version 2.0[J].Bioinformatics, 2007, 23(21):2947-2948.
[21] MI S, CAI T, HU Y, et al. Sorting of small RNAs intoArabidopsisargonautecomplexes is directed by the 5′ terminal nucleotide[J].Cell, 2008, 133(1):116-127.
[22] MONTGOMERY T A, HOWELL M D, CUPERUS J T, et al. Specificity of ARGONAUTE7-miR390 interaction and dual functionality in TAS3 trans-acting siRNA formation[J].Cell, 2008, 133(1):128-141.
[23] HAGEN C, FRIZZI A, KAO J,et al. Using small RNA sequences to diagnose, sequence, and investigate the infectivity characteristics of vegetable-infecting viruses[J].ArchivesofVirology, 2011, 156(7):1209-1216.
[24] KREUZE J F, PEREZ A, UNTIVEROS M, et al. Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs: a generic method for diagnosis, discovery and sequencing of viruses[J].Virology, 2009, 388(1):1-7.
[25] SHIN J C, KIM M K, KWAK H R, et al. First report of Clover yellow vein virus onGlycinemaxin Korea[J].PlantDisease, 2014, 98(9):1283.
[26] RAVELO G, KAGAYA U, INUKAI T, et al. Genetic analysis of lethal tip necrosis induced by Clover yellow vein virus infection in pea[J].JournalofGeneralPlantPathology, 2007, 73(1):59-65.
[27] UYEDA I, TAKAHASHI T, TAKAHASHI Y. A cDNA clone to clover yellow vein potyvirus genome is highly infectious[J].VirusGenes, 1997, 14(3):235-243.
[28] MITTER N, KOUNDAL V, WILLIAMS S, et al. Differential expression of tomato spotted wilt virus-derived viral small RNAs in infected commercial and experimental host plants[J].PloSOne, 2013, 8(10):e76276.
[29] PANTALEO V, SALDARELLI P, MIOZZI L, et al. Deep sequencing analysis of viral short RNAs from an infected Pinot Noir grapevine[J].Virology, 2010, 408(1):49-56.
[30] YAN F, ZHANG H, ADAMS M J, et al. Characterization of siRNAs derived from rice stripe virus in infected rice plants by deep sequencing[J].ArchivesofVirology, 2010, 155(6):935-940.

