青杄PwNAC42基因的克隆及表達模式分析
袁義杭 張鶴華 游韓莉 張凌云
(北京林業大學 省部共建森林培育與保護教育部重點實驗室,北京 100083)
轉錄因子是植物應對各種逆境的重要調控手段,通過與目標基因啟動子區的順式作用元件結合,調控靶基因的轉錄水平[1]。NAC轉錄因子是植物特有的一類轉錄因子,其名稱是矮牽牛(Petunia hybrida)的NAM基因、擬南芥(Arabidopsisthaliana)的ATAF1/2基因和CUC2基因的首字母縮寫[2]。NAC轉錄因子典型的結構模型由兩部分組成:存在于N端的高度保守的NAC結構域和存在于C端的高度多樣的轉錄調控區[3]。N端的NAC結構域由150-160個氨基酸殘基組成,分為A-E這5個亞結構域。其中,亞結構域C、D高度保守,含有核定位信號,與DNA結合[4]。C端的轉錄調控區由重復出現的簡單氨基酸組成,并富含谷氨酸、脯氨酸、絲氨酸、蘇氨酸等,具有激活或抑制轉錄的功能[5]。
NAC轉錄因子不僅參與植物生長發育的調控,還在植物應對環境脅迫時發揮了重要的作用[6]。擬南芥ORE1(ANAC092)與葉綠體發育關鍵基因GLK1/2互作,加速了葉綠體退化,促使葉片衰老[7]。酵母雙雜實驗證實香蕉(Musa acuminata)MaNAC1/2與乙烯信號組件的MaEIL5蛋白互作,推測MaNAC1/2基因通過乙烯信號通路參與香蕉果實的成熟調控[8]。擬南芥nac019突變體對高溫敏感,而過表達該基因增強了植物對高溫的抗性[9]。RhNAC3通過ABA依賴型途徑參與了月季(Rosa hybrida)對干旱脅迫的響應[10]。小麥(Triticum aestivum)的基因表達譜顯示TaNAC2響應干旱、鹽害、冷害和ABA處理,在擬南芥中過表達TaNAC2提高了植株對干旱、鹽和冷脅迫的耐受力[11]。
青杄(Picea wilsoniiMast.)為松科(Pinaceae)云杉屬(Picea)常綠喬木,是我國特有的木本針葉樹種,對干旱等惡劣環境具有非常強的適應能力,是進行抗逆基因研究的理想樹種。本文從轉錄組數據中獲得PwNAC42基因的cDNA全長序列,并對其進行生物信息學分析,預測理化性質及其蛋白結構等。采用實時熒光定量PCR檢測基因在青杄各組織中的表達量,以及在干旱、鹽害、冷脅迫和脫落酸(Abscisic Acid,ABA)處理下的表達模式。以期為研究木本植物中NAC轉錄因子的功能提供理論基礎,為林木遺傳育種提供分子依據。
1 材料與方法
1.1 材料
青杄球果、種子、花粉采集于中國科學院植物研究所和北京植物園,三年生青杄的根、莖和針葉用于組織特異性表達實驗。青杄種子在4℃春化后,放置于濕潤濾紙上,待其萌發后移植到營養土:蛭石:珍珠巖為2:1:1(V/V/V)的培養基質中培養。溫室光周期為16 h日照,溫度為21℃,相對濕度55%-65%,生長到8周的青杄幼苗用于逆境響應實驗。植物材料采集后用液氮處理,保存于-80℃備用。
1.2 方法
1.2.1 青杄PwNAC42基因編碼區的克隆 青杄cDNA文庫由Invitrogen(上海)公司利用Gateway技術構建[12]。以實驗室前期構建的均一化cDNA文庫為模板,設計引物NAC42-F和NAC42-R(表1)擴增PwNAC42的編碼區序列,膠回收后連接到pEASY-T1載體上,獲得PwNAC42單克隆。

表1 CDS克隆和RT-qPCR引物序列
1.2.2 生物信息學分析 將PwNAC42的cDNA序列導入DNAMAN軟件中,通過翻譯功能獲得其ORF序列及氨基酸序列。ProtParam在線工具(http://web.expasy.org/protparam/)用于預測蛋白分子質量、等電點、分子式及不穩定指數。ProtScale工具(http://web.expasy.org/protscale/)用于分析蛋白親/疏水性。NetPhos 3.1工具(http://www.cbs.dtu.dk/services/NetPhos/)用于預測蛋白可能存在的磷酸化位點。SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)用于預測蛋白是否存在信號肽結構域。TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM/)用于預測蛋白的跨膜區域。FoldUnfold工具(http://bioinfo.protres.ru/ogu/)用于預測蛋白無序化特征。GOR4在線工具(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)用于預測蛋白二級結構,SWISS-MODEL在 線 工 具(https://www.swissmodel.expasy.org/)用于預測蛋白三級結構。
通過 NCBI網站(https://www.ncbi.nlm.nih.gov/)的BLAST功能搜索PwNAC42氨基酸序列的同源序列,利用ClustalX軟件進行多序列比對分析,并用MEGA5軟件選擇鄰位相連法(Neighbour-joining)構建系統發育樹(bootstap檢測次數為1 000)。
1.2.3 組織特異性表達 利用北京艾德萊(Aidlab)公司的EASYspin Plus植物RNA快速提取試劑盒提取青杄球果、種子、花粉、根、莖和針葉的RNA,通過天根生化科技(北京)有限公司的FastQuant cDNA第一鏈合成試劑盒反轉錄成單鏈cDNA。cDNA模版濃度調成一致之后通過天根公司的熒光定量PCR試劑盒(SYBR)在StepOnePlusTMReal-Time PCR System(ABI公司)上進行RT-qPCR實驗,檢測PwNAC42在不同組織中的相對表達量。用于熒光定量PCR的引物為NAC42-RT-F和NAC42-RT-R,以青杄EF1-α作為內參基因,序列見表1[13]。試驗設置3次重復。
1.2.4 逆境響應分析 逆境響應試驗的處理方法參考周燕妮等[14],選取長勢相對一致的八周大的青杄幼苗。幼苗置于4℃處理0、3、6、12 h;再置于吸水紙上處理0、3、6、12 h;用200 mmol/L NaCl溶液處理0、3、6、12 h;用100 μmol/L脫落酸處理0、3、6、12 h。對照組為正常生長的植株。試驗設置3次重復,處理后的青杄幼苗液氮速凍,于-80℃備用。
2 結果
2.1 青杄PwNAC42全長cDNA的獲得
通過轉錄組數據獲得PwNAC42的cDNA序列全長,通過DNAMAN軟件推導其編碼的蛋白序列。PwNAC42基因cDNA序列全長共1 749 bp,其中編碼區1 140 bp,共編碼379個氨基酸。在91 bp處為起始密碼子ATG,1 228 bp處為終止密碼子TGA,1 741 bp處為Poly(A)9尾巴(圖1)。

圖1 PwNAC42全長cDNA的核苷酸序列及推導的氨基酸序列
2.2 PwNAC42結構及進化特征
2.2.1 功能結構域 用NCBI網站上的BLAST功能對PwNAC42蛋白進行功能域分析,結果(圖2-A)顯示PwNAC42蛋白有典型NAM(No apical meristem)結構域。
2.2.2 氨基酸組成及理化性質 ProtParam預測結果顯示,PwNAC42分子質量為42.92 kD,理論等電點為6.53,分子式為C1919H2934N522O582S9,其中絲氨酸(Ser)含量最高,為10.8%,其次為脯氨酸(Pro),含量為9.0%,并帶有負電殘基(Asp+Glu)48個,帶有正電氨基酸殘基(Arg+ Lys)46個,預測其半衰期約為30 h,不穩定指數46.78,預測蛋白不穩定。
PwNAC42蛋白疏水性分析結果顯示,76位精氨酸(Arg)分值最小,為-2.789,親水性最強,41位纈氨酸(Val)分值最大,為1.978,親水性最弱,預測蛋白為親水性蛋白(圖2-B)。磷酸化位點預測結果顯示,PwNAC42有31個絲氨酸磷酸化位點,有11個蘇氨酸磷酸化位點,有5個酪氨酸磷酸化位點(圖2-C)。SignalP 4.1預測發現該蛋白沒有信號肽結構域(結果未顯示)。蛋白跨膜結構域預測結果顯示,PwNAC42整條多肽鏈都位于細胞膜外,不存在跨膜結構域(圖2-D)。蛋白無序化特征預測結果顯 示,PwNAC42在 1-12、76-87、168-229、295-318處的氨基酸處于無序化區域(圖2-E)。

圖2 PwNAC42蛋白結構域及理化性質分析
2.2.3 蛋白質二級結構和三級結構 PwNAC42蛋白的二級結構預測結果(圖3-A)發現,α-螺旋含量為16.89%,延伸鏈為12.14%,無β-轉角結構,無規則卷曲結構含量達到70.98%。PwNAC42蛋白三級結構預測結果(圖3-B)顯示,蛋白無規則卷曲結構較多。
2.2.4 多序列比對和系統進化 在NCBI網站上用BLAST功能對PwNAC42進行同源蛋白搜索,尋找到下列物種的同源蛋白:黑云杉(Picea mariana,PmNAC,AAC32123.1)、北美云杉(Piceasitchensis,PsNAC,ABK26029.1)、 油 棕(Elaeis guineensis,EgNAC,XP_010922868.1)、海棗(Phoenix dactylifera,PdNAC,XP_008792531.1)、 鳳 梨(Ananas comosus,AcNAC,OAY70955.1)、 石 刁 柏(Asparagus officinalis,AoNAC,XP_020254522.1)、蓮(Nelumbo nucifera,NnNAC,XP_010257746.1)、水稻(Oryza sativa,OsNAC,BAA89798.1)、小果野蕉(Musa acuminata,MaNAC,XP_009394855.1)、小 蘭 嶼 蝴 蝶 蘭(Phalaenopsis equestris,PeNAC,XP_020578930.1)、 鐵 皮 石 斛(Dendrobiumcatenatum,DcNAC,XP_020673301.1)、玉米(Zea mays,ZmNAC,ADK25055.1)、短花藥野生稻(Oryza brachyantha,ObNAC,XP_015693359.1)、 擬 南 芥(AtNAC,AT1G01720.1)。利用 ClustalX工具進行多序列比對,發現PwNAC42與其它物種的NACs在N端相似性較高,在C端則僅與同為云杉屬的黑云杉和北美云杉相似性較高(圖4-A)。用MEGA5鄰接法構建系統進化樹,結果(圖4-B)顯示,PwNAC42與云杉屬NACs聚為一類,擬南芥和其它植物則為另兩類。
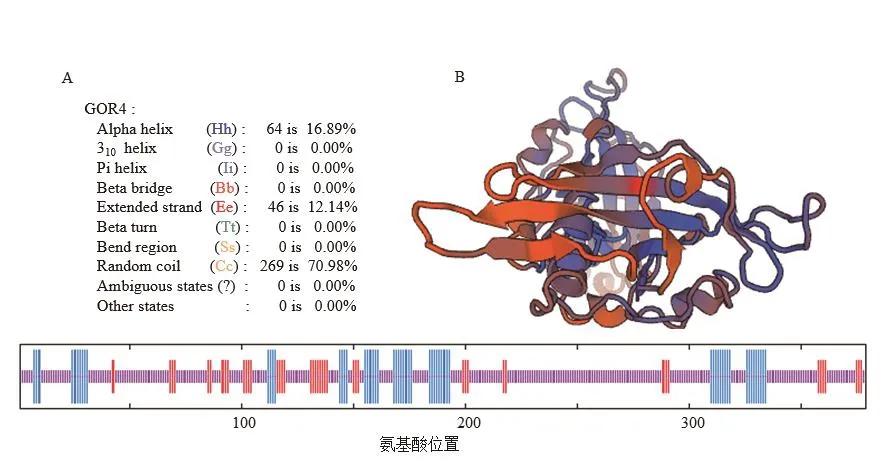
圖3 PwNAC42蛋白二級結構及三級結構分析
2.3 PwNAC42表達特征
PwNAC42在各個組織器官中均有表達,但主要在球果中表達,球果中的表達量約為花粉中表達 量 的 41倍( 圖 5)。PwNAC42對 NaCl、 干 旱、4℃、ABA均有響應。200 mmol/L NaCl溶液處理下,PwNAC42表達量隨著時間增加顯著,在12 h時表達水平達到了對照組的88倍左右(圖6-A)。在干旱條件下,PwNAC42表達量變化顯著,6 h前呈現上升的趨勢,6 h和12 h的表達量則沒有顯著差異(圖6-B)。4℃處理下,PwNAC42表達量呈現上升的趨勢,在處理12 h時基因的表達水平達到了對照組表達量的18倍(圖6-C)。ABA處理也顯著提高了PwNAC42的表達量,表達量隨時間持續上升,表達水平在12 h時約為對照組的14倍(圖6-D)。
3 討論
NAC轉錄因子最早從矮牽牛中發現,被命名為NAM[15],之后越來越多的NAC轉錄因子被分離鑒定出來。全基因組分析發現,在擬南芥、水稻、煙 草(Nicotiana tabacum)、 大 豆(Glycine max)、楊樹(Populus trichocarpa)和中國白菜(Brassica campestris)等植物中分別有至少117、151、152、152、163和 188個 NAC 轉錄因子家族成員[16-17]。本試驗從青杄中克隆得到PwNAC42基因,通過BLAST工具顯示,PwNAC42的N端存在典型的NAM結構域。多序列比對發現,PwNAC42與其它物種的NACs在N端相似性較高,而C端序列僅與云杉屬NACs的相似性較高,與其它草本、木本植物相似性較低(圖4)。從而推測PwNAC42的C端序列在進化上較為保守。
前人的研究表明,NAC轉錄因子參與了植物果實成熟的調控過程。香蕉中的MaNAC1/2通過與乙烯信號組件相互作用,可能參與了香蕉果實的成熟調控[8]。番茄(Solanum lycopersicum)SlNAC3基因在果實的綠熟期和破色期中表達量較高,而干涉該基因表達后,類胡蘿卜素的合成受到抑制,果實成熟的進程明顯延遲[18]。本研究的組織特異性表達試驗發現,青杄PwNAC42主要在球果中表達,推測其可能與球果成熟有關,但具體調控植物果實成熟的途徑仍有待研究。

圖4 PwNAC42蛋白多序列比對(A)及系統進化樹分析(B)

圖5 PwNAC42在青杄各組織中的表達量

圖6 PwNAC42在逆境處理下表達量的變化
以往的研究發現,NAC轉錄因子在模式植物擬南芥和小麥、水稻、玉米、番茄等作物類植物響應逆境脅迫中發揮了重要功能[9、11、19-21]。本研究結果顯示,鹽脅迫、冷脅迫及干旱脅迫處理后PwNAC42表達量隨時間變化而明顯上升(圖6),推測其可能在植物響應這些逆境的過程中發揮了作用。ABA信號轉導途徑是植物響應逆境脅迫的重要途徑之一,NAC轉錄因子既依賴于ABA途徑發揮作用,也獨立于ABA信號途徑。Zhu等[21]研究了番茄中的SlNAC4基因,發現該基因的沉默能提高植物對鹽和干旱的抗性,但基因的表達受到MeJA(Methyl Jasmonate)而不是ABA的誘導,從而推測SlNAC4可能通過不依賴ABA的途徑參與植物對逆境的響應。前人對陸地棉(Gossypium hirsutum)中NACs進行了研究,發現GhNAC7響應乙烯、ABA、干旱、鹽分、低溫等多種處理,其啟動子區存在ABREs(ABA response cis-element)元件,進而推斷GhNAC7通過ABA信號通路參與了陸地棉對逆境的響應[22]。在本實驗中,外源施加ABA時PwNAC42表達量顯著上升,因此推測PwNAC42可能依賴于ABA信號通路響應環境脅迫,但具體的調控機制仍有待研究。
4 結論
從青杄cDNA文庫中克隆了一個NAC家族的轉錄因子PwNAC42。組織特異性表達結果表明PwNAC42主要在球果中表達。逆境響應實驗結果發現,該基因響應多種非生物脅迫處理包括NaCl、干旱、低溫及ABA,說明其參與了青杄對逆境脅迫的響應。
[1] Singh KB, et al. Transcription factors in plant defense and stress responses[J]. Curr Opin Plant Biol, 2002, 5(5):430-436.
[2]Aida M, Ishida T, Fukaki H, et al. Genes involved in organ separation inArabidopsis:an analysis of the cup-shaped cotyledon mutant[J]. The Plant Cell, 1997, 9(6):841-857.
[3]Shen H, Yin Y, Chen F, et al. A bioinformatic analysis of NAC genes for plant cell wall development in relation to lignocellulosic bioenergy production[J]. Bioenergy Research, 2009, 2(4):217-232.
[4]Ooka H, Satoh K, Doi K, et al. Comprehensive analysis of NAC family genes inOryza sativaandArabidopsis thaliana[J]. DNA Research, 2003, 10(6):239-247.
[5]Wang Z, Dane F. NAC(NAM/ATAF/CUC)transcription factors in different stresses and their signaling pathway[J]. Acta Physiologiae Plantarum, 2013, 35(5):1397-1408.
[6]Olsen AN, Ernst HA, Leggio LL, et al. NAC transcription factors:structurally distinct, functionally diverse[J]. Trends Plant Sci,2005, 10(2):79-87.
[7]Rauf M, Arif M, Dortay H, et al. ORE1 balances leaf senescence against maintenance by antagonizing G2-like-mediated transcription[J]. Embo Reports, 2013, 14(4):382-388.
[8]Shan W, Kuang J, Chen L, et al. Molecular characterization of banana NAC transcription factors and their interactions with ethylene signalling component EIL during fruit ripening[J]. J Exp Bot, 2012, 63(14):5171-5187.
[9]Guan Q, Yue X, Zeng H, et al. The protein phosphatase RCF2 and its interacting partner NAC019 are critical for heat stress-responsive gene regulation and thermotolerance inArabidopsis[J]. Plant Cell,2014, 26(1):438-453.
[10] Jiang G, Jiang X, Lü P, et al. The rose(Rosa hybrida)NAC transcription factor 3 gene, RhNAC3, involved in ABA signaling pathway both in rose andArabidopsis[J]. PLoS One, 2014, 9(10):e109415.
[11]Mao X, Zhang H, et al. TaNAC2, a NAC-type wheat transcription factor conferring enhanced multiple abiotic stress tolerances inArabidopsis[J]. J Exp Bot, 2012, 63(8):2933-2946.
[12]張盾, 劉亞靜, 李長江, 等. 青杄均一化cDNA文庫構建及EST序列分析[J]. 生物技術通報, 2012(6):71-76.
[13]Yu Y, Li Y, Huang G, et al. PwHAP5, a CCAAT-binding transcription factor, interacts with PwFKBP12 and plays a role in pollen tube growth orientation in Picea wilsonii[J]. J Exp Bot,2011, 62(14):4805-4817.
[14]周燕妮, 李艷芳, 張通, 等. 青杄PwUSP2基因的克隆和表達分析[J]. 植物生理學報, 2015, 51(8):1307-1314.
[15]Souer E, Houwelingen AV, Kloos D, et al. The no apical meristem gene of petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J]. Cell, 1996, 85(2):159-170.
[16]Puranik S, Sahu PP, Srivastava PS, et al. NAC proteins:regulation and role in stress tolerance[J]. Trends Plant Sci, 2012, 17(6):369-381.
[17]Ma J, Wang F, Li MY, et al. Genome wide analysis of the NAC transcription factor family in Chinese cabbage to elucidate responses to temperature stress[J]. Scientia Horticulturae, 2014,165(3):82-90.
[18]韓芹芹. 番茄SlNAC3基因通過延遲果實軟化和改變類胡蘿卜素的合成調控果實成熟[D]. 武漢:華中農業大學, 2011.
[19]Jeong JS, Kim YS, et al. Root-specific expression of OsNAC10 improves drought tolerance and grain yield in rice under field drought conditions[J]. Plant Physiol, 2010, 153(1):185-197.
[20]Lu M, Ying S, Zhang DF, et al. A maize stress-responsive NAC transcription factor, ZmSNAC1, confers enhanced tolerance to dehydration in transgenicArabidopsis[J]. Plant Cell Reports,2012, 31(9):1701-1711.
[21]Zhu M, Chen G, Zhang J, et al. The abiotic stress-responsive NAC-type transcription factor SlNAC4 regulates salt and drought tolerance and stress-related genes in tomato(Solanum lycopersicum)[J]. Plant Cell Reports, 2014, 33(11):1851-1863.
[22] Shah ST. 陸地棉中NAC家族與葉片衰老和脅迫應答相關功能的遺傳學分析[D]. 北京:中國農業科學院, 2013.

