成都地區鴨源沙門氏菌的分離鑒定及其耐藥特征、毒力基因分析
, ,, ,, ,3,,,,3
沙門氏菌(Salmonellaspp.)屬腸桿菌科,革蘭氏陰性直桿菌,是常見的食源性人獸共患病原菌,以家畜、家禽和野生動物帶菌量較多。目前已有2 610個沙門血清型被報道[1],其中部分血清型能引起人體傷寒、副傷寒、胃腸炎、腹瀉和敗血癥等疾病。
國內外學者的報道[2-3]表明,動物性食品在生產中極易受到沙門氏菌的污染。據不完全統計,我國70%~80%的細菌性食物中毒事件是由沙門氏菌引起,其中90%以上是通過動物性食品感染所致[4],嚴重影響了公眾健康;在全球范圍,每年大約有9 400萬人感染沙門氏菌,約155 000人死于急性感染,其中85%是由動物性食品感染引起[5]。而近年來,由于抗菌藥物在養殖業和醫療行業的大量使用,導致沙門氏菌的耐藥性逐漸增強,這無疑會加大沙門氏菌感染的防治難度,從而使感染和致死人數增加。就目前相關研究[6]來看,沙門氏菌主要是通過粘附、定植、入侵和釋放毒力因子等機制來感染宿主并導致疾病,多種毒力基因參與表達,主要分布在毒力島(SPI)、鞭毛(Fimbrial)和質粒(Plasmid)上,毒力基因攜帶率越高,表明沙門氏菌的潛在致病性越強。
我國是鴨養殖和鴨制品生產大國,2005年鴨存欄量就超過了7.25億只,占世界存欄量的72%左右,一旦沙門氏菌在肉鴨生產鏈中暴發,并由此傳播到人群,將會給食品安全、公眾衛生和經濟生產帶來嚴重后果。因此,對鴨源沙門氏菌進行深入研究具有重要的衛生學意義,但目前國內外的相關研究主要集中在豬肉源、牛肉源和雞肉源上,而鮮有關于鴨源沙門氏菌的系統性報道。
本研究旨在調查肉鴨屠宰鏈中沙門氏菌的污染分布和耐藥狀況,并從分子層面對其毒力基因的攜帶情況進行調查,為研究沙門氏菌在肉鴨屠宰加工過程中的流行、傳播及其風險評估提供基礎數據,并對相關生產提出建議。
1 材料與方法
1.1材料
1.1.1樣品來源及采樣方法樣品采集于四川省成都市某肉鴨屠宰加工公司,屠宰對象為6周齡櫻桃谷肉鴨,分別來源于大邑、崇州、新津、邛崍、金堂等地的5家養殖場。根據沙門氏菌的生物學特性與污染途徑,確定采樣對象為糞樣、胴體表面、水樣及肉樣,采集方式如下:用無菌剪刀剪取凈膛后的完整盲腸,置于保鮮袋中,其內容物代表屠宰前糞樣;沿屠宰和宰后各環節,隨機無菌采集棉拭子樣(胴體頸部、背部)、水樣(沖淋水、浸池水、車間用水)和肉樣(鴨腳、鴨翅)。采集完畢后將所有樣品編號并放入有冰袋的隔熱泡沫箱中,4 h內低溫運回實驗室進行下一步處理。
1.1.2菌株腸炎沙門氏菌(Salmonellaenteritidis)CICC21482,大腸桿菌(Escherichiacoli)ATCC25922,均由四川農業大學食品微生物實驗室保存。
1.1.3試劑緩沖蛋白胨水(BPW),四硫磺酸鈉煌綠(TTB)增菌液,亞硒酸鹽胱氨酸(SC)增菌液,木糖賴氨酸脫氧膽鹽(XLD)瓊脂,法國科馬嘉沙門氏菌顯色培養基,水解酪蛋白(MH)瓊脂等均購于杭州微生物試劑有限公司;三糖鐵(TSI)瓊脂,靛基質試劑,尿素瓊脂,賴氨酸脫羧酶試驗培養基等購自北京奧博星生物技術有限責任公司;DL-2000 DNA Marker、GoldviewTM核酸染料、Premix Taq Version 2.0、瓊脂糖購自寶生物工程(大連)有限公司;沙門氏菌屬診斷血清(60種)試劑盒購自寧波天潤生物藥業有限公司。
1.1.4主要儀器BSC—1300ⅡA2 型生物安全柜(蘇州安泰空氣技術有限公司);電熱恒溫培養箱(上海一恒科學儀器有限公司);Bio-Rad C1000梯度PCR擴增儀、Bio-Rad GelDoc XR凝膠成像系統、水平電泳槽(美國Bio-Rad公司)。
1.1.5抗菌藥物青霉素類:氨芐西林(AMP,10 μg/片)、阿莫西林/克拉維酸(AMC,20/10 μg/片);頭孢類:頭孢曲松(CRO,30 μg/片);氨基糖苷類:慶大霉素(GEN,10 μg/片)、卡那霉素(KAN,30 μg/片);四環素類:四環素(TET,30 μg/片)、米諾環素(MH,30 μg/片);磺胺類:甲氧芐啶/磺胺甲噁唑(SXT,1.25/23.75 μg/片);喹諾酮類:萘啶酸(NAL,30 μg/片)、環丙沙星(CIP,5 μg/片)。均購于杭州微生物試劑有限公司。
1.2方法
1.2.1菌株分離純化參照GB 4789.4-2010[7]的方法對樣品進行處理和增菌。用無菌接種環取增菌液劃線接種于XLD瓊脂平板,在培養箱中37 ℃條件培養18~24 h。根據GB 4789.4-2010[7]中的判斷方法,判定并挑取可疑菌落劃線接種于科馬嘉沙門氏菌顯色培養基,于37 ℃條件下培養18~24 h,根據菌落形態挑取紫色、藍紫色或淺紫色圓形光滑菌落進行分離純化。
1.2.2生化鑒定按照GB 4789.4-2010[7]的方法,對分離株進行革蘭氏鏡檢、三糖鐵瓊脂培養和賴氨酸脫羧酶試驗,并按標準判定沙門氏菌疑似菌株。
1.2.3雙重PCR鑒定參照邵碧英等[8]的方法進行。針對沙門氏菌invA基因和hut基因設計兩對特異性引物,由寶生物工程(大連)有限公司合成。PCR反應體系為:2×PCR Mix 12.5 μL,invA基因和hut基因上下游引物0.2 μL、0.6 μL,DNA模板 2 μL,補滅菌超純水使最終反應體系為25 μL。將PCR產物于1.5%瓊脂糖(0.5×TBE)凝膠電泳30 min后,凝膠成像系統分析結果,能同時擴增出invA基因和hut基因特異性片段的菌株判定為沙門氏菌陽性菌株。
1.2.4血清型分型按照GB 4789.4-2010[7]的方法,用沙門氏菌屬診斷血清(60種)試劑盒對PCR陽性菌株進行血清分型。
1.2.5藥敏試驗按照CLSI推薦的紙片擴散法[9],對陽性菌株進行10種常用抗菌藥物敏感性試驗,以大腸桿菌ATCC25922為質控菌株。記錄各類藥物對陽性菌株產生的抑菌圈直徑,并根據CLSI標準[8]對試驗結果進行判定。
1.2.6毒力基因檢測根據文獻設計沙門氏菌毒力島(avrA[10]、ssaQ[11]、mgtC[11]、siiD[11]、sopB[11])、鞭毛(bcfC[12])、質粒(spvR[13]、spvB[13]、spvC[14])三大類毒力基因引物,由寶生物工程(大連)有限公司合成。PCR反應體系為:2×PCR premix 12.5 μL、DNA模板2 μL、上下游引物各1 μL,補滅菌超純水使最終反應體系為25 μL。PCR產物于1.5%瓊脂糖(1.0×TAE)凝膠電泳30 min后,凝膠成像系統分析結果。
2 結 果
2.1沙門氏菌分離情況結合鏡檢觀察和生化試驗,從343份樣品中總共分離純化得到99株沙門氏菌疑似菌株(表1),宰前樣品沙門疑似菌株檢出率(45.71%)最高。沿著脫毛環節向下,疑似沙門氏菌檢出率總體有所升高,其中車間用水中有沙門疑似菌株檢出。雙重PCR鑒定結果顯示,99株疑似沙門氏菌均能同時擴增出284 bp(invA)和495 bp(hut)兩條特異性條帶(圖1),陽性率為100%。
表1肉鴨屠宰鏈沙門氏菌分離情況
Tab.1Prevalence of Salmonella spp.from the duck slaughter chain

SourceSampleNSalmonellaspp.(%)BeforeslaughteringCecumcontent3516(45.71)DepilationstageSurfaceswab&rinsewater9722(22.68)EviscerationstageSurfaceswab&rinsewater8922(24.72)DuckmeatDuckfeet&duckwing6023(38.33)AfterslaughteringSurfaceswab&products&soakingwater&plantwater6216(25.81)Total34399(28.86)
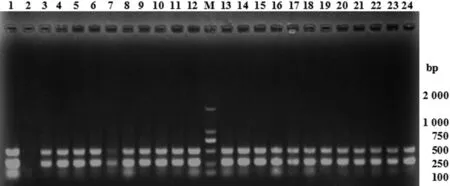
M: DL 2000 DNA Marker; 1: S.enteritidis CICC21482; 2: negative control; 3-12,13-24: part of samples圖1 部分沙門氏菌雙重PCR電泳圖Fig.1 Electrophoresis of duplex PCR amplified products from part of the isolated Salmonella spp.
2.2沙門氏菌分離菌株血清分型99株沙門氏菌陽性分離株有91株可以定型,5株只能定群,剩余3株產生了自凝現象不能分型。已定型的菌株共產生了7種血清型(表2),以印第安納沙門氏菌(S.Indiana)、紐蘭沙門氏菌(S.Newlands)和鴨沙門氏菌(S.Anatum)為優勢血清型,優勢血清群為B群和E1群。其中車間用水分離株產生了紐蘭和鴨沙門氏菌兩種血清型。在屠宰鏈生產鏈中,印第安納沙門氏菌檢出量沿屠宰鏈逐漸減少,而紐蘭沙門氏菌則呈上升趨勢;鴨沙門氏菌在分割肉樣中檢出較多,傷寒沙門氏菌(S.Typhi)只在宰前和屠宰環節有檢出,阿貢納沙門氏菌(S.Agona)只在屠宰環節有檢出,鼠傷寒沙門氏菌(S.Typhimurium)只在分割肉樣中有檢出。
表2肉鴨屠宰鏈沙門氏菌分離株血清分型結果
Tab.2Serotyping of Salmonella spp.from the duck slaughter chain
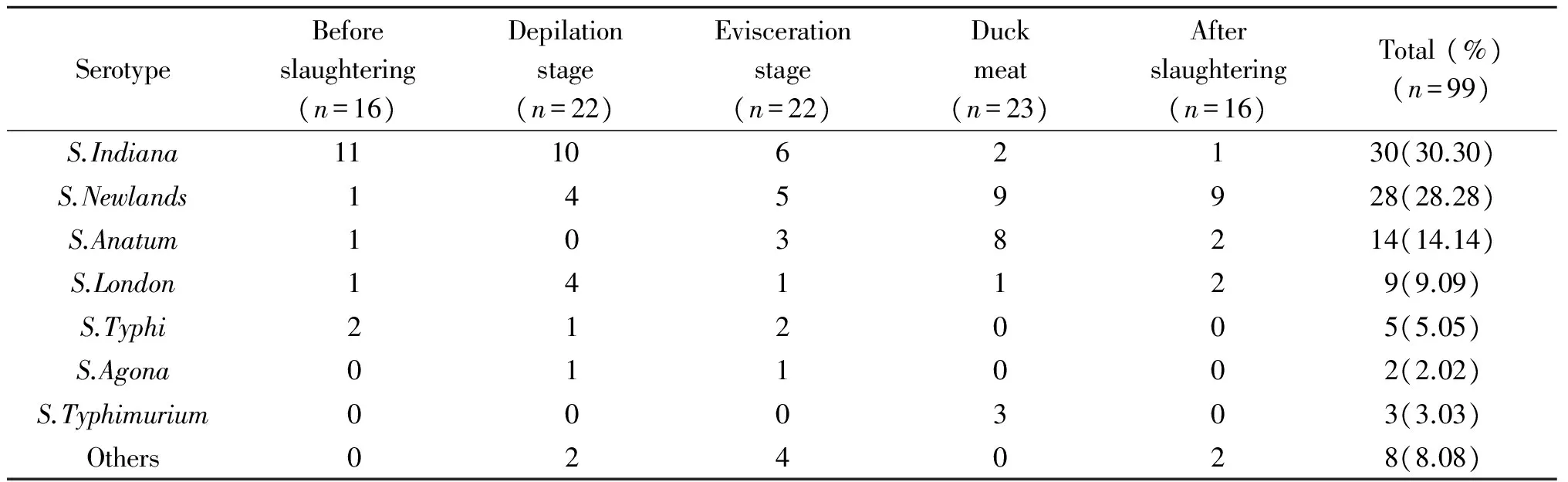
SerotypeBeforeslaughtering(n=16)Depilationstage(n=22)Eviscerationstage(n=22)Duckmeat(n=23)Afterslaughtering(n=16)Total(%)(n=99)S.Indiana111062130(30.30)S.Newlands1459928(28.28)S.Anatum1038214(14.14)S.London141129(9.09)S.Typhi212005(5.05)S.Agona011002(2.02)S.Typhimurium000303(3.03)Others024028(8.08)
2.3沙門氏菌分離株藥敏試驗
2.3.1沙門氏菌分離株耐藥率99株沙門氏菌對10種常見抗菌藥的耐藥率統計如表3所示,96.97%的菌株具有耐藥性。分離株對NAL的耐藥率最高,達到了91.92%。不同血清型產生了不同的耐藥特征,其中傷寒和鼠傷寒沙門氏菌對AMP、TET、NAL的耐藥率均達到了100%,印第安納沙門氏菌對8種抗菌藥的耐藥率都在50%以上,耐藥性較為突出;紐蘭和鴨沙門氏菌對NAL的耐藥率較高,對其他抗菌藥的耐藥率則處于較低水平。
表3鴨源沙門氏菌對不同抗菌藥物的耐藥率(%)
Tab.3Resistance rates of duck originated Salmonella spp.against 10 antimicrobial agents
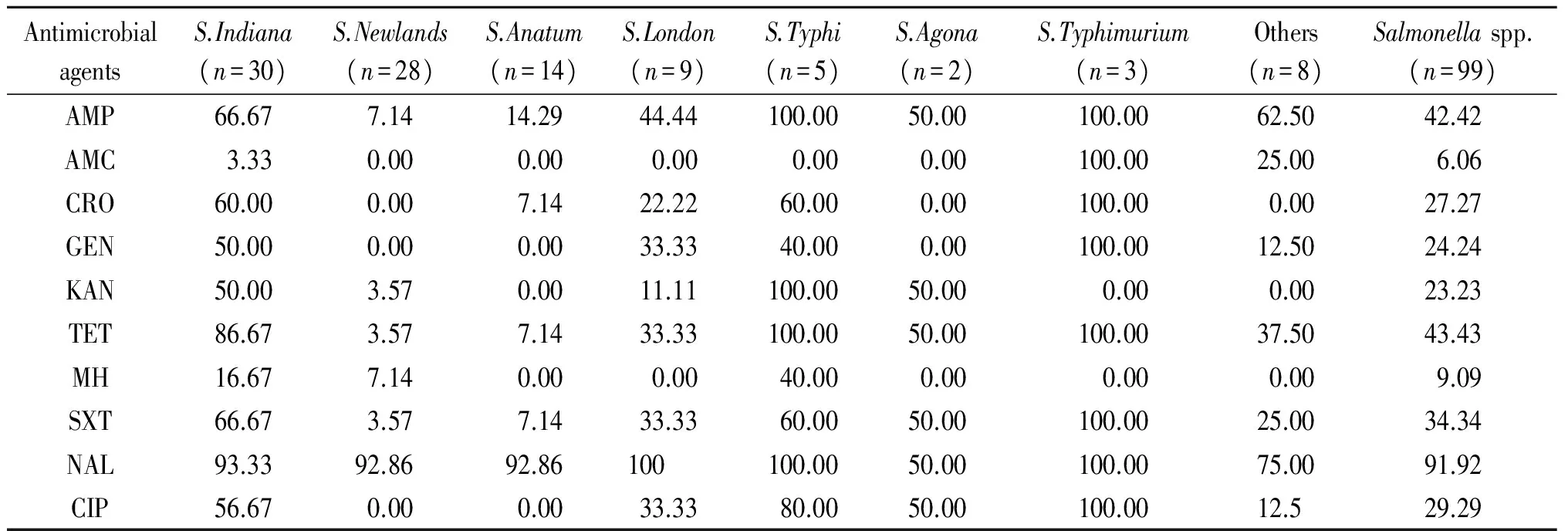
AntimicrobialagentsS.Indiana(n=30)S.Newlands(n=28)S.Anatum(n=14)S.London(n=9)S.Typhi(n=5)S.Agona(n=2)S.Typhimurium(n=3)Others(n=8)Salmonellaspp.(n=99)AMP66.677.1414.2944.44100.0050.00100.0062.5042.42AMC3.330.000.000.000.000.00100.0025.006.06CRO60.000.007.1422.2260.000.00100.000.0027.27GEN50.000.000.0033.3340.000.00100.0012.5024.24KAN50.003.570.0011.11100.0050.000.000.0023.23TET86.673.577.1433.33100.0050.00100.0037.5043.43MH16.677.140.000.0040.000.000.000.009.09SXT66.673.577.1433.3360.0050.00100.0025.0034.34NAL93.3392.8692.86100100.0050.00100.0075.0091.92CIP56.670.000.0033.3380.0050.00100.0012.529.29
2.3.2沙門氏菌分離株多重耐藥率及耐藥譜沙門氏菌分離株中多重耐藥菌株(耐3種及以上抗菌藥物)占47.47%(圖2),其中耐4種(10.10%)、7種(11.11%)及8種(9.09%)抗生素的菌株較多,未出現同時耐10種抗菌藥物的菌株。傷寒沙門氏菌(S.Typhi)和鼠傷寒沙門氏菌(S.Typhimurium)的多重耐藥率達到了100%(表5),其中傷寒沙門氏菌集中在7耐(80.00%),鼠傷寒沙門氏菌集中在8耐(100.00%);印第安納沙門氏菌(S.Indiana)多重耐藥率高達93.33%,其中4耐(20.00%)和8耐(20.00%)菌株較多,并有9耐(6.67%)菌株出現;倫敦沙門氏菌(S.London)多重耐藥率為44.44%,相對較低,主要集中在7耐(33.33%);紐蘭沙門氏菌(S.Newlands)和鴨沙門氏菌(S.Anatum)的多重耐藥率最低,分別為3.57%和7.14%,多數菌株只耐NAL一種藥物。
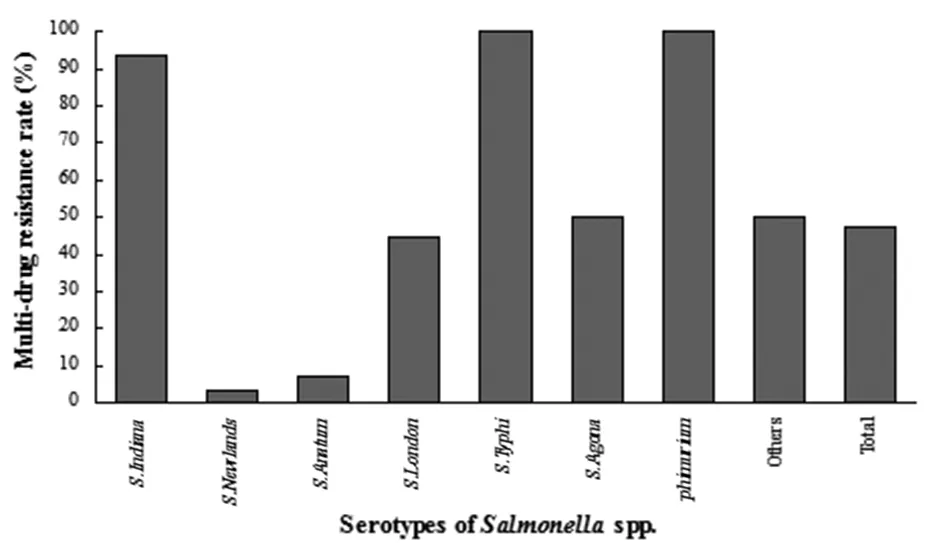
圖2 鴨源沙門氏菌多重耐藥分布情況Fig.2 Multi-drug resistance distribution of duck originated Salmonella spp.
99株沙門氏菌對10種抗菌藥物產生了34種耐藥譜,其中優勢譜型為NAL,占到了40.40%。印第安納沙門氏菌產生了19種耐藥譜,優勢譜型為AMP-CRO-GEN-KAN-TET-SXT-NAL-CIP(16.67%);紐蘭沙門氏菌產生了5種耐藥譜,優勢譜型為NAL(82.14%);鴨沙門氏菌產生了3種耐藥譜,優勢譜型為NAL(85.71%);倫敦沙門氏菌產生了4種耐藥譜,優勢譜型為NAL(55.56%);傷寒沙門氏菌產生了4種耐藥譜,優勢譜型為AMP-CRO-GEN-KAN-TET-NAL-CIP(40.00%);鼠傷寒沙門氏菌只產生了AMO-AMC-CRO-GEN-TET-SXT-NAL-CIP一種耐藥譜。
2.4沙門氏菌分離株毒力基因檢測99株沙門氏菌均有毒力基因檢出(表4),其中avrA、ssaQ、spvR的檢出率最高,分別為93.94%、90.91%、94.95%;mgtC、siiD、sopB、bcfC的檢出率較高,分別為87.88%、77.78%、83.84%、80.81%;spvB和spvC的檢出率最低,分別為29.29%和11.11%。鼠傷寒沙門氏菌對9種毒力基因的檢出率均達到了100.00%(圖3);傷寒沙門氏菌的檢測結果表現出了一定差異,對avrA、ssaQ、mgtC、siiD、sopB和bcfC的檢出率分別為40.00%、60.00%、20.00%、40.00%、40.00%和40.00%,明顯低于其他血清型。
M: DL 2000 DNA Marker; 1,3,5,7,9,11,13,15: PCR products of avrA,ssaQ and siiD,mgtC,sopB,bcfC,spvR,spvB,spvC gene; 2,4,6,8,10,12,14,16: negative control of avrA,ssaQ and siiD,mgtC,sopB,bcfC,spvR,spvB,spvC gene圖3 鼠傷寒沙門氏菌毒力基因PCR檢測電泳圖Fig.3 Electrophoresis of virulence genes PCR amplified products from S.Typhimurium
表4鴨源沙門氏菌毒力基因檢測結果(%)
Tab.4Detection results of virulence genes carried by Salmonella spp.in duck slaughterhouse
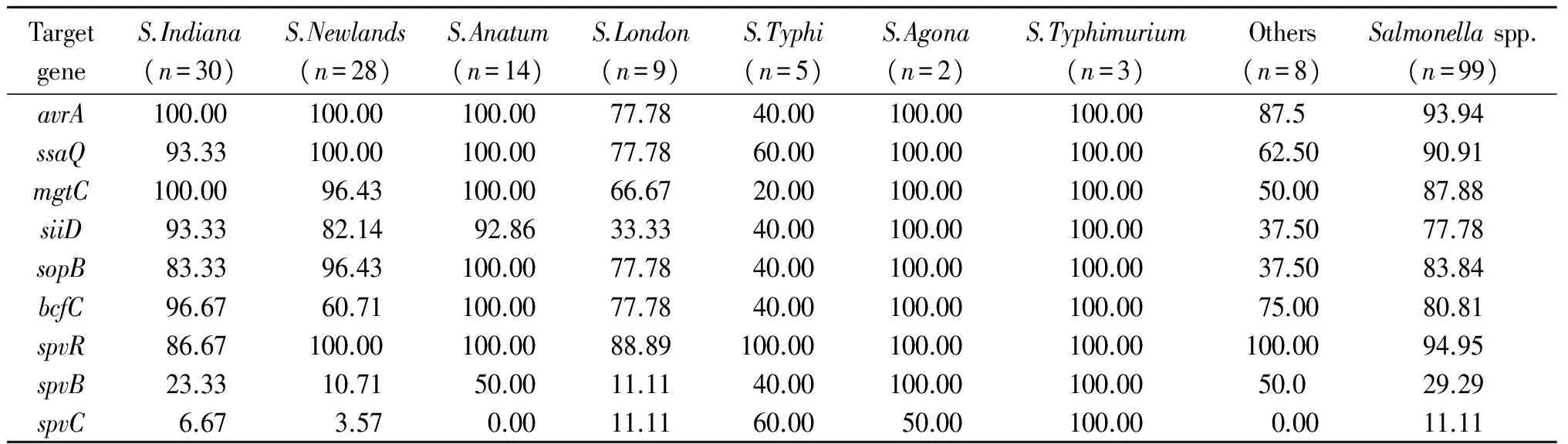
TargetgeneS.Indiana(n=30)S.Newlands(n=28)S.Anatum(n=14)S.London(n=9)S.Typhi(n=5)S.Agona(n=2)S.Typhimurium(n=3)Others(n=8)Salmonellaspp.(n=99)avrA100.00100.00100.0077.7840.00100.00100.0087.593.94ssaQ93.33100.00100.0077.7860.00100.00100.0062.5090.91mgtC100.0096.43100.0066.6720.00100.00100.0050.0087.88siiD93.3382.1492.8633.3340.00100.00100.0037.5077.78sopB83.3396.43100.0077.7840.00100.00100.0037.5083.84bcfC96.6760.71100.0077.7840.00100.00100.0075.0080.81spvR86.67100.00100.0088.89100.00100.00100.00100.0094.95spvB23.3310.7150.0011.1140.00100.00100.0050.029.29spvC6.673.570.0011.1160.0050.00100.000.0011.11
3 討 論
本研究沙門氏菌總體分離率為28.86%,與Li等[15](26.9%)的報道較為一致,低于Cha等[16](43.4%)的報道,而高于Dong等[17](6.5%)的報道,出現該結果的原因可能是不同地區沙門氏菌的流行情況不同,也可能與采樣方式、采樣季節、分菌方法等有關。沿屠宰鏈觀察,宰前樣品沙門氏菌檢出率最高;沿脫毛環節向下,沙門氏菌檢出率有所上升,與Wang等[2]的研究結果不同,結合各環節沙門氏菌分離株的血清型和耐藥表型分析,推測受沙門氏菌污染的車間用水通過沖淋、浸泡等方式對脫毛后的樣品造成了污染。此外,宰后樣品的沙門氏菌檢出率達到了25.81%,一定程度上影響了鴨制食品的食用安全。
已定型的沙門氏菌所產生的7種血清型均為國內沙門氏菌常見血清型,優勢血清群為B群和E1群,與李郁等[4]研究一致。本次研究無常見的腸炎沙門氏菌(S.Enteritidis)及德爾卑沙門氏菌(S.Derby)檢出,而印第安納沙門氏菌和紐蘭沙門氏菌的檢出率較高,與Imen等[18]、Yang等[19]學者的研究有所差異。沿屠宰鏈觀察,印第安納沙門氏菌檢出量呈下降趨勢,這與樣品的熱燙和沖淋有關;紐蘭和鴨沙門氏菌檢出量呈上升趨勢,經推測是樣品被車間用水污染所造成;其他劣勢血清型在屠宰鏈的分布無規則性,其中傷寒、阿貢納和鼠傷寒沙門氏菌只在個別環節有檢出,可能是工作人員和屠宰環境對屠宰對象形成了交叉污染。所檢出的7種血清型中,倫敦沙門氏菌、阿貢納沙門氏菌和鼠傷寒沙門氏菌在我國均有人群感染致病事件出現,說明鴨源沙門氏菌對公眾健康具有潛在的威脅。
藥敏試驗結果顯示分離株對萘啶酸的耐藥率最高,對阿莫西林/克拉維酸的耐藥率最低,與Cha等[16]的報道較為一致。而分離株對其他抗菌藥的耐藥率普遍低于國內外學者的報道[20-21],差異性明顯,這與不同國家和地區在養殖業中常用的抗菌藥物種類不同有關,也與耐藥性在沙門氏菌中的水平傳播機制有一定聯系。本研究中鴨源沙門氏菌的多重耐藥率為47.47%,高于Vo AT等[22](29%)的報道,而低于Pan等[20](81.5%)的報道,除去地域和時間因素外,抗菌藥物的選擇和水體污染(車間用水分離出的紐蘭和鴨沙門氏菌只耐NAL)也會對該結果有一定影響。不同沙門氏菌血清型表現出了不同的耐藥特征,共產生了34種耐藥譜,其中以印第安納沙門氏菌、傷寒沙門氏菌和鼠傷寒沙門氏菌的耐藥性最為突出,與Yang等[19]的研究結果比較一致。一旦具有多重耐藥性的沙門氏菌通過鴨肉傳播到人群,無疑會給沙門氏菌感染的防治帶來嚴重的挑戰,因而必須加強抗菌藥物的管控和合理使用。
毒力基因檢測結果顯示沙門氏菌分離株對spvR、avrA、ssaQ、mgtC、siiD、sopB、bcfC的攜帶率較高,說明鴨源沙門氏菌具有較強的潛在致病性。但毒力基因的總體攜帶率與Graciela等[3]、Li等[23]的研究相比存在一定差異,這可能與菌株來源或血清型的不同有關。此外本研究還發現鼠傷寒沙門氏菌對9種毒力基因的攜帶率均達到了100.00%,與該血清型的強致病性對應明顯。
本次研究選擇將肉鴨作為研究對象,明確了肉鴨屠宰生產鏈中沙門氏菌的污染分布、耐藥狀況及毒力基因分布情況,結果顯示肉鴨屠宰生產鏈容易受到不同血清型、不同耐藥表型,且毒力基因攜帶率高的沙門氏菌污染,一旦失控將會對食品安全和公眾健康造成嚴重的后果。對此,相關食品公司應當改善加工環境,加強水源保護,合理使用抗菌藥物并隨時關注工作人員的健康狀況,同時相關部門也應當加強衛生監督,合力保障食品安全和公眾健康。
參考文獻:
[1] Mezal EH,Sabol A,Khan MA,et al.Isolation and molecular characterization ofSalmonellaentericaserovar Enteritidis from poultry house and clinical samples during 2010[J].Food Microbiol,2014,38: 67-74.DOI: 10.1016/j.fm.2013.08.003
[2] Wang H,Ye K,Wei X,et al.Occurrence,antimicrobial resistance and biofilm formation ofSalmonellaisolates from a chicken slaughter plant in China[J].Food Ctrl,2013,33(2):378-384.DOI: 10.1016/j.foodcont.2013.03.030
[3] Graciela L,Geovana M,Marisa C,et al.Antimicrobial resistance and class 1 integron-associated gene cassettes inSalmonellaentericaserovar Typhimurium isolated from pigs at slaughter and abattoir environment[J].Vet Microbiol,2016,194: 84-92.DOI:10.1016/j.vetmic.2016.04.020
[4] Li Y,Jiao XA,Wei JZ,et al.Analysis on the serotypes and drug-susceptibility ofSalmonellaisolates from pig carcass[J].Chin J Zoonoses,2008,24(1): 67-70.(in Chinese)
李郁,焦新安,魏建忠,等.屠宰生豬沙門氏菌分離株的血清型和藥物感受性分析[J].中國人獸共患病學報,2008,24(1):67-70.DOI: 10.3969/j.issn.1002-2694.2008.01.017
[5] Yang XJ,Huang JH,Wu QP,et al.Prevalence,antimicrobial resistance and genetic diversity ofSalmonellaisolated from retail ready-to-eat foods in China[J].Food Ctrl,2016,60(2): 50-56.DOI: 10.1016/j.foodcont.2015.07.019
[6] Cao TX,Jiang WC,He WC,et al.Research progress on virulence factors ofSalmonella[J].Chin J Prevent Vet Med,2014,36(4): 331-334.(in Chinese)
曹恬雪,蔣文燦,何文成,等.沙門氏菌毒力因子的研究進展[J].中國預防獸醫學報,2014,36(4): 331-334.DOI: 10.3969/j.issn.1008-0589.2014.04.19
[7] National Health and Family Planning Commission of the People’s Republic of China.GB 4789.4-2010.National standards for food safety,food microbiology testing,Salmonellaspp [S].Beijing: China Standard Press,2010.(in Chinese)
中華人民共和國國家衛生和計劃生育委員會.GB 4789.4-2010.食品安全國家標準,食品微生物學檢驗,沙門氏菌檢驗[S].北京: 中國標準出版社,2010.
[8] Shao BY,Chen B,Tang MY,et al.Development of multiplex PCR detection method forSalmonella[J].Food Sci,2007,28(10): 489-492.(in Chinese)
邵碧英,陳彬,湯敏英,等.沙門氏菌多重PCR檢測方法的建立[J].食品科學,2007,28(10): 489-492.DOI: 10.3321/j.issn:1002-6630.2007.10.124
[9] CLSI.M100-S19 Performance standards for antimicrobial susceptibility testing[S].Wayne: Clinical and Laboratory Standards Institute,2016.
[10] Rita P,Susanne M,Erhard T,et al.Prevalence and polymorphism of genes encoding translocated effector proteins among clinical isolates ofSalmonellaenterica[J].Int J Med Microbiol,2000,290: 605-617.DOI: 10.1016/S1438-4221(00)80009-0
[11] Sara MS,Irene R,Rodicio MR,et al.Detection of virulence determinants in clinical strains ofSalmonellaentericaserovar Enteritidis and mapping on macrorestriction profiles[J].Int J Med Microbiol,2006,55: 365-373.DOI: 10.1099/jmm.0.46257-0
[12] Stephan H,Roberto M,Muna A,et al.Virulotyping and antimicrobial resistance typing ofSalmonellaentericaserovars relevant to human health in Europe[J].Foodborne Pathog Dis,2010,7: 523-535.DOI: 10.1089/fpd.2009.0447
[13] Cheng Q,Pang RL,Wang RC,et al.Comparative study on pathogenicity ofSalmonellaisolates from different sources of laboratory mice and the detection of their virulence genes[J].Chin J Zoonoses,2013,29(5): 460-465.(in Chinese)
程瓊,龐瑞亮,王若晨,等.不同源沙門氏菌對小鼠致病力的比較與毒力基因檢測[J].中國人獸共患病學報,2013,29(5): 460-465.DOI: 10.3969/cjz.j.issn.1002-2694.2013.05.008
[14] Fatemeh F,Mohammad D,Masoumeh D,et al.Genetic diversity and virulence genes ofSalmonellaentericasubspeciesentericaserotype Enteritidis isolated from meats and eggs[J].Microbial Pathogenesis,2017,107: 451-456.DOI: 10.1016/j.micpath.2017.04.026
[15] Li R,Lai J,Wang Y,et al.Prevalence and characterization ofSalmonellaspecies isolated from pigs,ducks and chickens in Sichuan Province,China[J].Intl J Food Microbiol,2013,163(1): 14-18.DOI: 10.1016/j.ijfoodmicro.2013.01.020
[16] Cha SY,Kang M,Yoon RH,et al.Prevalence and antimicrobial susceptibility ofSalmonellaisolates in Pekin ducks from South Korea[J].Comparat Immunol Microbiol Infect Dis,2013,36(5): 473-479.DOI: 10.1016/j.cimid.2013.03.004
[17] Dong P,Zhu L,Mao Y,et al.Prevalence and profile ofSalmonellafrom samples along the production line in Chinese beef processing plants[J].Food Ctrl,2014,38(4): 54-60.DOI: 10.1016/j.foodcont.2013.09.066
[18] Imen F,Amal BH,Noel G,et al.Genetic diversity of food-isolatedSalmonellastrains through Pulsed Field Gel Electrophoresis (PFGE) and Enterobacterial Repetitive Intergenic Consensus (ERIC-PCR)[J].PLoS One,2013,8(12): E81315.DOI: 10.1371/journal.pone.0081315
[19] Yang B,Qu D,Zhang X,et al.Prevalence and characterization ofSalmonellaserovars in retail meats of marketplace in Shaanxi,China[J].Intl J Food Microbiol,2010,141(1): 63-72.DOI: 10.1016/j.ijfoodmicro.2010.04.015
[20] Pan Z,Wang X,Zhang X,et al.Changes in antimicrobial resistance amongSalmonellaentericasubspecies enterica serovar Pullorum isolates in China from 1962 to 2007[J].Vet Microbiol,2009,136(3): 387-392.DOI: 10.1016/j.vetmic.2008.11.015
[21] Oueslati W,Rjeibi MR,Mhadhbi M,et al.Prevalence,virulence and antibiotic susceptibility ofSalmonellaspp.strains,isolated from beef in Greater Tunis (Tunisia)[J].Meat Sci,2016,119: 154-159.DOI: 10.1016/j.meatsci.2016.04.037
[22] Vo AT,Van DE,Fluit AC,et al.Antibiotic resistance,integrons andSalmonellagenomic island 1 among non-typhoidalSalmonellaserovars in the Netherlands[J].Intl J Antimicrob Agents,2006,28(3): 172-179.DOI: 10.1016/j.ijantimicag.2006.05.027
[23] Li K,Ye S,Walid QA,et al.Antimicrobial susceptibility,virulence gene and pulsed-field gel electrophoresis profiles ofSalmonellaentericaserovar Typhimurium recovered from retail raw chickens,China[J].Food Ctrl,2017,72: 36-42.DOI: 10.1016/j.foodcont.2016.07.032

