生物凈水柵對凡納濱對蝦腸道菌群組成的影響
裴鵬兵,吳潔瓊,梁宏豪,杜 虹,2,4
( 1.汕頭大學 理學院 生物系,廣東 汕頭 515063;2.汕頭大學 理學院,海洋生物研究所,廣東省海洋生物技術重點實驗室,廣東 汕頭 515063;3.福建省水產研究所,福建省海洋生物增養殖與高值化利用重點實驗室,福建 廈門 361013;4.汕頭大學 理學院,汕頭大學—馬爾凱理工大學藻類聯合研究中心,廣東 汕頭 515063 )
凡納濱對蝦(Litopenaeusvannamei)又稱南美白對蝦,具有個體大、生長快、營養需求低、抗病力強等優點,是目前世界上三大養殖對蝦中單產量最高的對蝦品種之一。近年來,在凡納濱對蝦養殖過程中,病害尤其是細菌性疾病、水質惡化等現象頻發[1],像白斑病、紅腿病、軟殼病等[2-3]。水質的好壞直接影響著對蝦腸道菌群組成,近幾年的研究表明[4-5],細菌在蝦腸道中的作用受到越來越多的關注,其對宿主動物的腸道發育、營養、免疫應答和疾病抗性發揮著重要作用。對蝦腸道內的一些有益共生菌對疾病有一定的抗病性[6-7],同時也存在著一些條件致病菌,二者共同維持著腸道菌群的動態平衡[8]。因此,研究對蝦腸道菌群動態分布對了解對蝦的健康和可持續養殖有著積極作用。楊鶯鶯等[9]利用純化培養法,從人工飼料飼養的對蝦腸道和養殖水體中共分離出71株細菌,而環境中99%的細菌是不可培養的,所以純化培養法不能全面地反映對蝦腸道菌群組成及多樣性。劉淮德等[10]應用變性梯度凝膠電泳技術在凡納濱對蝦腸道中檢測出了12種細菌。王春忠等[3]采用構建16S rRNA基因克隆文庫的方法對長毛對蝦(Penaeuspenicillatus)海水養殖環境以及蝦腸道微生物群落組成進行研究,檢測到對蝦腸道細菌優勢種群為厚壁細菌(75.79%)、梭桿菌(13.68%)和γ-變形桿菌(10.53%)。Illumina Miseq高通量測序作為新一代測序技術,具有速度快、測序容量大、精確度高等特點[11],能夠較為真實、全面地反映樣品中微生物群落組成的基本特征,廣泛地應用于各個領域。
生物凈水柵——以聚酯為材質通過纏繞成結而形成線性附著基質作為微生物附著的載體[12],微生物在其表面不斷聚集、生長、繁殖,逐漸形成一層結構復雜的生物膜,生物凈水柵在池塘水產養殖上受到廣泛關注[13-14]。筆者以傳統池塘養殖(對照組)和添加生物凈水柵池塘養殖(試驗組)的凡納濱對蝦腸道為試驗對象,采用高通量測序技術對兩組對蝦腸道的菌群組成進行分析,比較兩組對蝦腸道菌群組成的差異,以期為生物凈水柵應用于池塘養殖提供參考意見。
1 材料與方法
1.1 試驗設計
試驗實施的地點位于廣東省汕頭市西郊牛田洋養殖基地,設置一個添加生物凈水柵池塘(試驗組)和一個傳統池塘(對照組),對照組未添加生物凈水柵,其他條件均一致。兩個池塘在養殖期間均不投藥和添加抗生素。對蝦的養殖時間為2015年6月23日至2015年12月19日,樣品采集時間是2015年10月31日,采樣日停止投喂飼料。凡納濱對蝦起始規格為(2.0±0.1) cm,兩個池塘投苗量均為200尾/m2。
每日7:00開始充氧,充氧3~4 h,并于10:00投喂飼料,投喂量視具體情況而定,前3周日投喂2次(第2次于18:00投喂),之后每日投喂1次。當遇到惡劣天氣,如酷熱或臺風暴雨天氣時,分別采取補充水源(附近的溝渠水源)和及時排出雨水等措施。
1.2 試驗試劑、儀器
DNA提取試劑盒(E.Z.N.A.? Stool DNA Kit)購自Omega公司、DNA Marker(λ-Hind Ⅲ digest)購自Takara公司、DNA Marker(D2000)購自Tiangen公司、Eppendorf高速冷凍離心機(5424R型)購自廈門市諾源泰儀器設備有限公司,Eppendorf舒適型恒溫混勻器(5355型)購自上海創奕科教設備有限公司,Scientz-48型高通量組織研磨器購自寧波新芝生物科技股份有限公司。
1.3 試驗材料
1.3.1 生物凈水柵
生物凈水柵由汕頭市阿科蔓環保科技有限公司生產,材料為PET材質。該生物凈水柵由多條線性生物基(直徑約5 cm)固定于單一繩子上而一排一排自然懸浮于水面上,使其遍布整個池塘。每組生物凈水柵之間間隔1 m,每組有20條生物基,自然懸浮高度為1.5 m,并且于投苗前將其安置池塘內(圖1)。
圖1 生物凈水柵鋪設
1.3.2 凡納濱對蝦
試驗所用凡納濱對蝦取自廣東省汕頭市西郊牛田洋養殖基地,從蝦塘隨機捕撈體色正常、體表無損傷、大小均一的成熟期凡納濱對蝦,置于冰盒中保存,帶回實驗室立即處理。每組隨機取30尾蝦,隨機分為3個重復,分別命名為試驗組1、試驗組2、試驗組3、對照組1、對照組2、對照組3。在無菌條件下,先用無菌水漂洗試驗用凡納濱對蝦3次,再用75%的無水乙醇沖洗蝦體表3次,用滅菌的解剖刀剖開蝦體腔,用滅菌的鑷子小心取出蝦腸道,再置于0.9%的無菌生理鹽水中漂洗3次,隨后分別置于無菌離心管中,所有樣品均放置-80 ℃超低溫冰箱凍存備用。取相同量的蝦腸道,置于Scientz-48高通量組織研磨器中充分研磨,研磨后用無菌生理鹽水沖洗得到蝦腸道勻漿。
1.4 試驗方法
采用Illumina Miseq高通量測序技術(委托深圳華大基因科技服務有限公司進行測序和生物信息學分析),根據細菌16S rRNA基因V4區序列設計引物如下:正向引物515F 5′-GTGCCAGMGCCGCGGTAA-3′;反向引物806R 5′-GGACTACHVGGGTWTCTAAT-3′。
1.5 數據分析
所獲得的原始數據經過數據過濾,濾除低質量的序列,剩余高質量的整理過的數據方可用于后期分析。通過序列之間的重疊關系將序列拼接成標簽,利用uparse(v7.0.1090)軟件在97%相似度下將標簽聚成1個可操作分類單元,得到可操作分類單元的代表序列。然后通過可操作分類單元與數據庫比對,對可操作分類單元進行物種注釋,基于可操作分類單元和物種注釋結果進行樣品物種復雜度分析以及組間物種差異分析。樣品多樣性分析包括物種指數、Chao指數、Ace指數、Shannon指數以及Simpson指數。利用SPSS 19.0軟件對數據做差異顯著性分析,設置差異顯著性水平α=0.05,當P<0.05時表示差異顯著。
2 結 果
2.1 物種分類及豐度統計
2.1.1 可操作分類單元及其豐度統計
6個樣品獲得的有效序列條數分別為31 512(試驗組1)、31 351(試驗組2)、31 255(試驗組3)、31 411(對照組1)、30 367(對照組2)和31 329(對照組3),共產生187 225條序列(表1)。6個樣品各產生的可操作分類單元數分別為214(試驗組1)、125(試驗組2)、118(試驗組3)、172(對照組1)、109(對照組2)和83(對照組3),試驗組樣品平均可操作分類單元數約150,對照組樣品的平均可操作分類單元數約120(表1)。
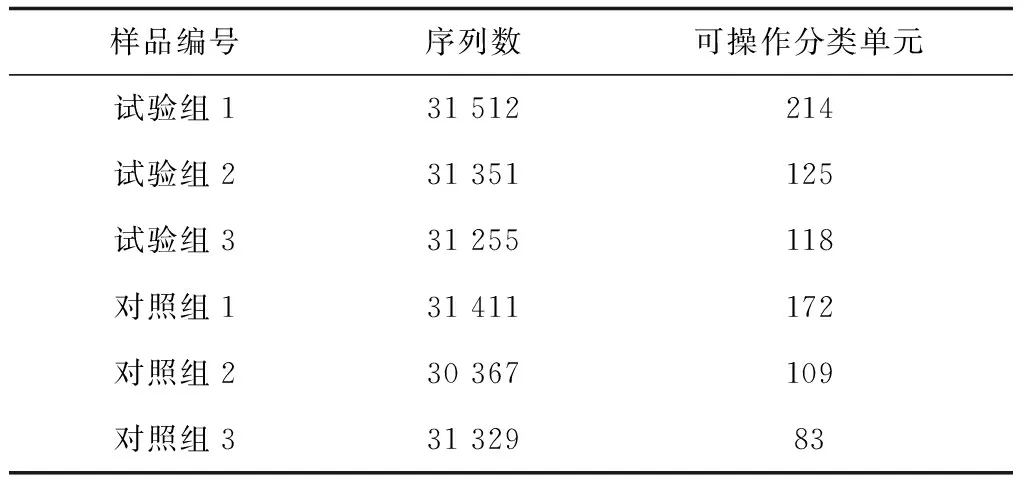
表1 樣品可操作分類單元數統計
2.1.2 可操作分類單元維恩圖
基于試驗組和對照組樣品中所用的可操作分類單元做維恩圖,比較兩組對蝦腸道菌群的物種豐富度差異。兩組對蝦腸道樣品有效可操作分類單元總數為418個,共有的可操作分類單元數為117個,占試驗組樣品可操作分類單元總數的39.53%,占對照組樣品可操作分類單元總數的48.95%。其中試驗組樣品獨有可操作分類單元數為179個,對照組樣品獨有可操作分類單元數為122個,試驗組物種的獨有率達到60.47%,而對照組物種的獨有率為51.05%(圖2)。數據表明,兩組對蝦腸道菌群的物種豐富度存在差異,試驗組樣品的物種豐富度要高于對照組,即添加生物凈水柵池塘凡納濱對蝦腸道菌群豐富度比傳統池塘凡納濱對蝦腸道菌群豐富度高。所得可操作分類單元結果用R語言工具統計和作圖軟件做等級豐度曲線圖(圖3)。等級豐度曲線結果顯示,6個樣品中的試驗組1的物種豐富度最高,均勻度最低,表明該樣品中物種組成最豐富,但樣品中各物種所占比例差異較大。試驗組樣品中試驗組1、試驗組2、試驗組3的物種豐富度分別高于對照組樣品中對應的對照組1、對照組2、對照組3,而試驗組1的均勻度最低(圖3)。整體上看,試驗組樣品的物種豐富度要比對照組樣品高,且均勻度要高于對照組(表2)。
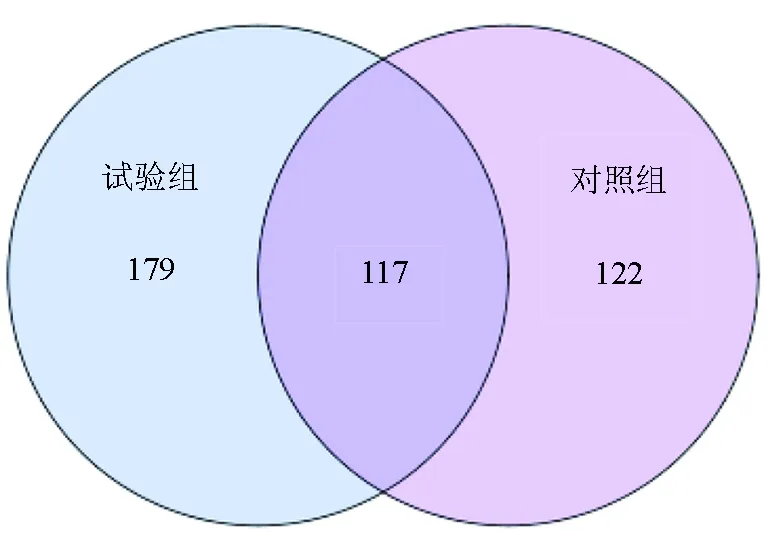
圖2 兩組樣品的維恩圖
2.2 細菌多樣性指數
從各組樣品的多樣性指數(表2)可見,試驗組樣品的各項指數均高于對照組樣品,其中,試驗組樣品的Chao指數和Ace指數分別高于對照組樣品的26.34%、24.49%(P>0.05),說明試驗組蝦腸道的細菌菌群豐富度高于對照組蝦腸道。試驗組樣品的Shannon指數高于對照組樣品的13.33%(P>0.05),說明試驗組蝦腸道菌群多樣性高于對照組。
2.3 細菌菌群組成
對兩組樣品的有效序列進行歸類操作分析,統計不同分類單元所對應的細菌門類及其相對豐度。試驗結果表明,兩組樣品之間的菌群結構相似,結構組成所占比例上存在一定的差異性。在門水平上,添加和未添加生物凈水柵池塘凡納濱對蝦腸道菌群結構主要由13個細菌門組成,分別為變形菌門、擬桿菌門、軟壁菌門、放線菌門、綠彎菌門、浮霉菌門、厚壁菌門、疣微菌門、藍藻門、梭桿菌門、綠菌門、酸桿菌門和硝化螺旋菌門。其中,試驗組樣品和對照組樣品均以變形菌門含量最高,分別占試驗組樣品中細菌總數的58.97%和對照組樣品中細菌總數的40.74%(表3,圖4a)。兩組樣品中另外兩大優勢門類分別是擬桿菌門和軟壁菌門,兩者分別占試驗組樣品中細菌總數的26.90%和11.69%,在對照組樣品中的占比分別為28.46%和29.34%(表3)。梭桿菌門和綠菌門均在試驗組樣品中鑒定到,而在對照組樣品中并未鑒定出這兩個細菌門類,這可能跟添加生物凈水柵有關。另外,硝化螺旋菌門出現在了對照組樣品中而未出現在試驗組樣品中,硝酸螺旋菌門下的硝化螺旋菌屬作為硝化細菌,可將亞硝酸鹽氧化成硝酸鹽。
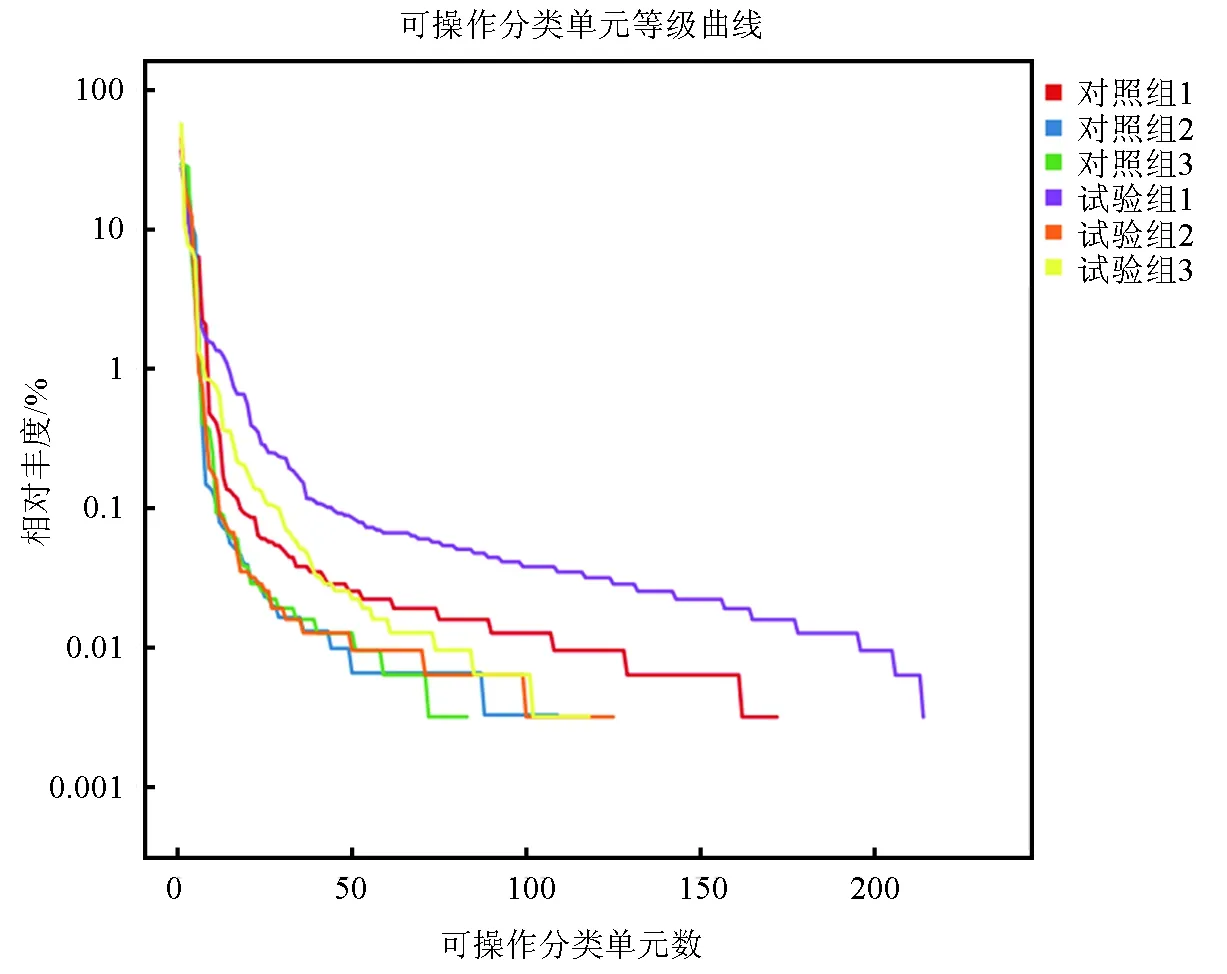
圖3 各樣品之間物種多樣性比較
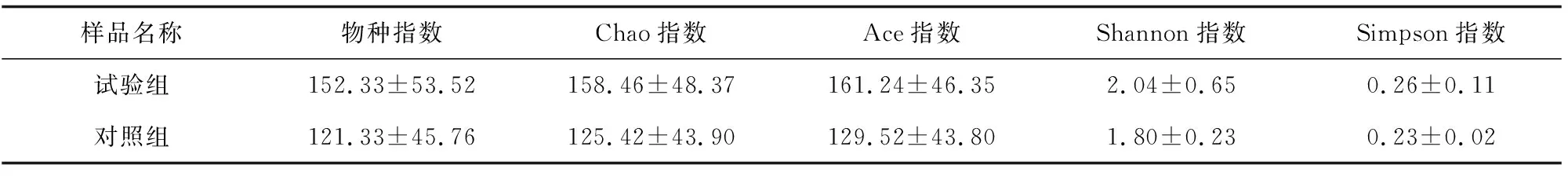
樣品名稱物種指數Chao指數Ace指數Shannon指數Simpson指數試驗組152.33±53.52158.46±48.37161.24±46.352.04±0.650.26±0.11對照組121.33±45.76125.42±43.90129.52±43.801.80±0.230.23±0.02
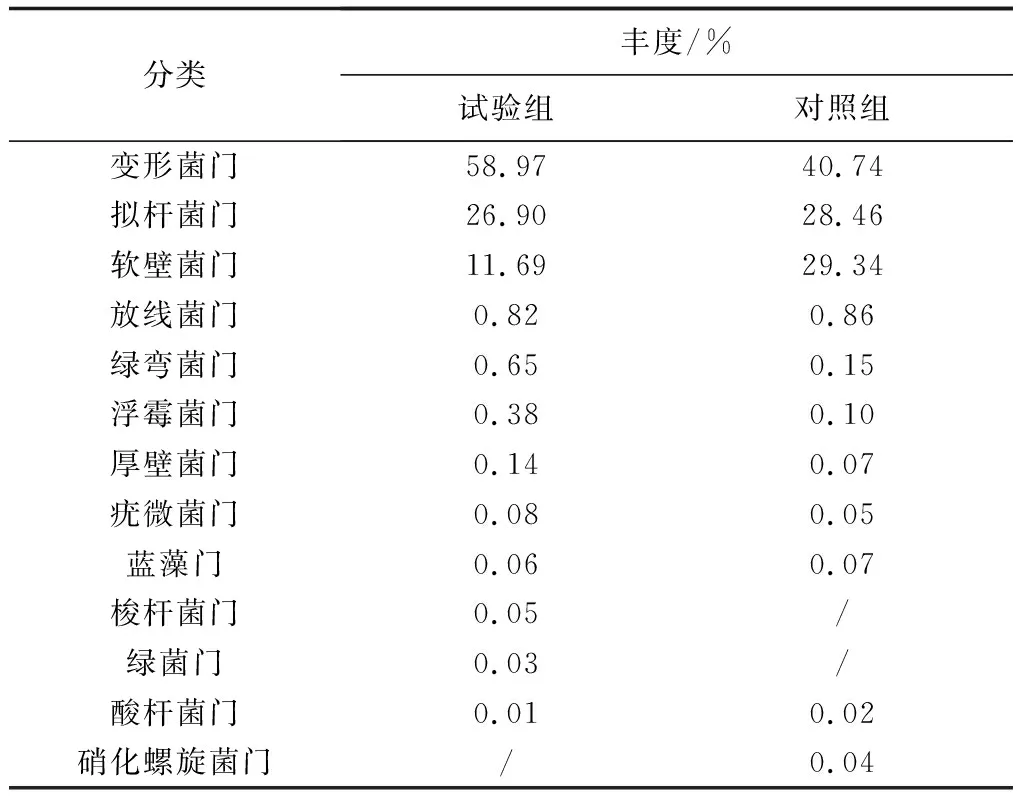
表3 兩組樣品優勢細菌門類及其相對豐度
注:表格中“/”表示未鑒定出.下同.
在屬水平上,添加和未添加生物凈水柵池塘凡納濱對蝦腸道菌群結構主要由弧菌屬(Vibrio)、希瓦氏菌屬(Shewanella)、Cloacibacterium、黃桿菌屬(Flavobacterium)、紅桿菌屬(Rhodobacter)、Herbaspirillum、噬氫菌屬(Hydrogenophaga)、短根瘤菌屬(Bradyrhizobium)、蒼白桿菌屬(Ochrobactrum)、Lutibacterium等細菌屬組成(表4,圖4b)。將兩組樣品中占比>1%的細菌屬定義為該組樣品的優勢菌屬。在兩組樣品中,優勢菌屬的構成及所占比例有明顯不同,試驗組樣品的優勢菌屬有弧菌屬、希瓦氏菌屬、Cloacibacterium、黃桿菌屬、紅桿菌屬,分別占該組樣品的70.83%、18.29%、1.97%、1.38%和1.06%,這5種優勢菌屬共占據該組樣品的93.53%。對照組樣品的優勢菌屬僅有弧菌屬和希瓦氏菌屬,分別占該組樣品的58.51%和37.67%,兩種優勢菌屬共占據該組樣品的96.18%。在試驗組樣品中發現的優勢菌屬Cloacibacterium和紅桿菌屬在對照組樣品中并未發現,兩組樣品中共有的優勢菌屬分別為弧菌屬和希瓦氏菌屬(表4)。可以看出,試驗組樣品中菌群結構比對照組樣品中菌群結構復雜,且優勢菌屬也比對照組樣品多,這表明,添加生物凈水柵池塘凡納濱對蝦腸道菌群結構比傳統池塘凡納濱對蝦腸道菌群結構復雜,應證了添加生物凈水柵池塘凡納濱對蝦腸道菌群多樣性高于傳統池塘凡納濱對蝦腸道菌群多樣性(表2)。
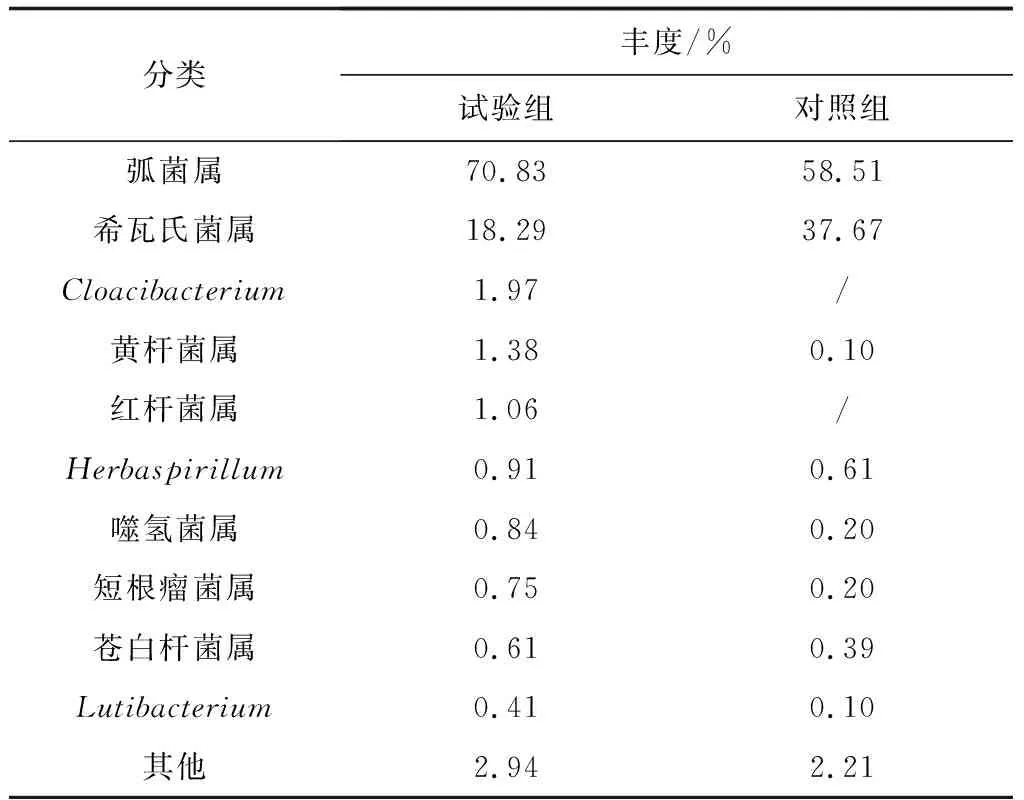
表4 兩組樣品優勢細菌屬類及其相對豐度
3 討 論
3.1 細菌菌群豐富度分析
近十幾年來,一些學者對動物腸道菌群結構進行了一系列研究。現有的技術中,純化培養法[8]只能研究1%可培養的細菌,99%不可培養的細菌將無法研究[18],PCR-DGGE法[15-16]和16S rRNA基因克隆文庫法[17]研究菌群結構的信息量不夠充分,也就不能全面反映腸道區系內的菌群結構及多樣性。近幾年興起的高通量測序技術(如Illumina測序和454測序)[19-20]則克服了這些缺陷,比較全面地揭示了腸道區系內的菌群結構及多樣性。本研究通過高通量測序技術,從兩組池塘凡納濱對蝦腸道中共檢測出16個細菌門105個細菌屬。
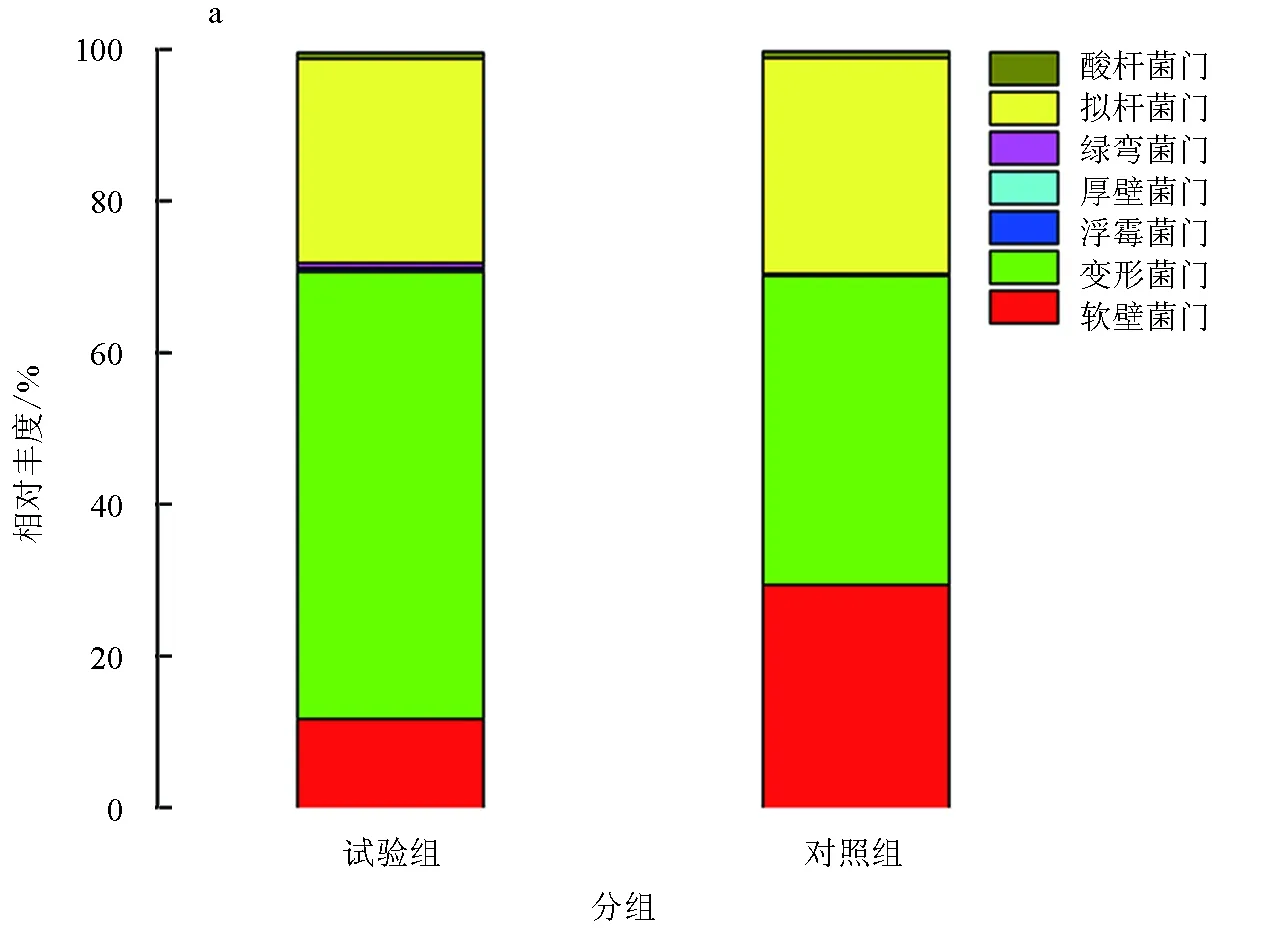
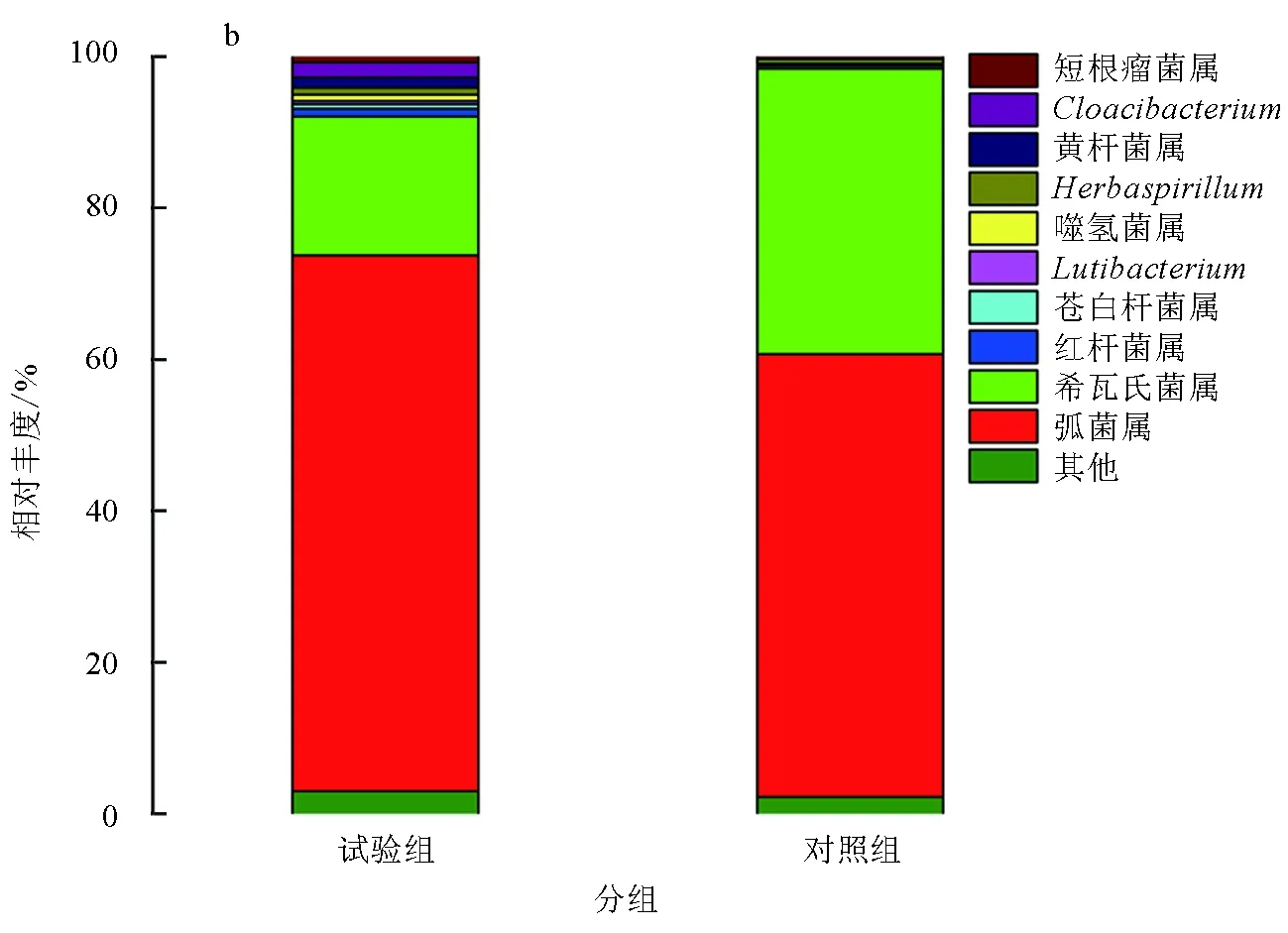
圖4 兩組樣品在門和屬水平上細菌菌群結構及分布a表示兩組樣品在門水平上細菌菌群結構及分布;b表示兩組樣品在屬水平上細菌菌群結構及分布.
關于對蝦腸道菌群結構及養殖環境微生物菌群結構已有諸多報道[21-24],而添加生物凈水柵池塘凡納濱對蝦腸道菌群結構還未見報道。筆者前期的研究結果顯示[12],生物凈水柵作為一種附著基質,能夠富集一些微生物和細菌,這些微生物和細菌聚集在一起形成一層生物膜。本研究中,試驗組凡納濱對蝦腸道菌群豐富度高于對照組。有研究表明[13],凡納濱對蝦會以一些附著基質作為棲息場所,并以該附著基質上的微生物作為額外的食物來源。生物凈水柵上的微生物大多來源于水體環境[25],水體環境微生物豐富度遠遠高于對蝦腸道菌群豐富度[26]。試驗組凡納濱對蝦腸道菌群有較高的豐富度,可能其腸道內含有外界微生物。依托于包括生物凈水柵在內的各種基質或載體而發展起來的生物膜技術大多用于污水處理中[27],目前在水產養殖上也有著廣泛應用。研究表明[28],養殖水體中的氮、磷元素有賴于水體中大量的固氮細菌、硝化細菌和反硝化細菌,依靠生物凈水柵表面附著的生物膜建立了去除氮、磷元素的氨氧化細菌、硝化細菌等有益微生物。生物凈水柵不僅穩定了水質,而且增加了凡納濱對蝦腸道菌群豐富度。
3.2 細菌多樣性分析
在本研究中,試驗組對蝦腸道菌群多樣性高于對照組,添加生物凈水柵將有助于提高對蝦腸道菌群結構及多樣性。有研究表明[29-30],由于生物基載體具備可供附著的棲息環境,水體中一部分營附著生活的細菌逐漸在生物膜上形成優勢種群,生物膜上出現了難以在水體中形成優勢種群的細菌。生物凈水柵作為可供微生物、細菌附著的生物基載體,其表面可能積累了一些優勢細菌。而生物凈水柵又為凡納濱對蝦生長提供了棲息場所,那些優勢種群的細菌就會成為凡納濱對蝦額外的食物來源,這就解釋了試驗組凡納濱對蝦腸道菌群多樣性高于對照組。
3.3 細菌菌群組成分析
有研究顯示[31],不同養殖季節下,凡納濱對蝦腸道菌群中主要優勢菌門類為放線菌門、未分類細菌門、變形菌門和擬桿菌門,分別占細菌總數的53.5%、22.4%、18.8%和4.32%。本試驗結果顯示,兩組凡納濱對蝦腸道菌群結構中均以變形菌門、擬桿菌門和軟壁菌門為優勢細菌門類,分別占試驗組樣品細菌總數和對照組樣品細菌總數的58.97%、26.90%、11.69%和40.74%、28.46%、29.34%。本研究與前人的研究比較發現,對蝦腸道菌群結構組成有一定的相似性,但在組成比例上存在差異,這可能與對蝦的養殖季節、棲息地不同有關,也可能與對蝦的生理狀態不同有關[32]。兩組凡納濱對蝦腸道菌群結構均以變形菌門為最大優勢細菌門類,所占比例上試驗組高于對照組。變形菌門是細菌中最大的一門,試驗組凡納濱對蝦腸道內存在的外界微生物可能大部分都屬于變形菌門類。
凡納濱對蝦腸道微生物菌群結構和功能在對蝦生長中具有重要作用,是維持機體腸道內環境穩定的關鍵因素[33]。相關研究表明[23],工廠化養殖凡納濱對蝦腸道內優勢菌屬主要是不可培養細菌、芽孢桿菌屬(Bacillus)和弧菌屬。Wang等[34]從野生的中國明對蝦(Fenneropenaeuschinensis)中分離出47株菌屬,弧菌屬和發光桿菌屬(Photobacterium)在整個腸道內為優勢菌屬。本試驗對池塘養殖的凡納濱對蝦腸道菌群結構研究顯示,兩組凡納濱對蝦腸道均以弧菌屬和希瓦氏菌屬為主要優勢菌,研究的差異可能在于養殖模式、對蝦品種和養殖環境的不同,但均有弧菌屬的存在,這就表明弧菌屬是凡納濱對蝦腸道內較為常見的菌屬。且有研究指出[9],對蝦腸道菌群結構組成受外部環境影響較大,以弧菌屬為常見菌屬。本研究中,試驗組凡納濱對蝦腸道內優勢菌屬為弧菌屬、希瓦氏菌屬、Cloacibacterium、黃桿菌屬和紅桿菌屬,對照組凡納濱對蝦腸道內優勢菌屬為弧菌屬和希瓦氏菌屬,兩組凡納濱對蝦腸道內共有的優勢菌為弧菌屬和希瓦氏菌屬。紅桿菌屬能夠抑制致病菌的存活,其廣泛地存在于生物絮凝團水體中[35,19],在試驗組凡納濱對蝦腸道內發現紅桿菌屬,而在對照組中并未發現。試驗組凡納濱對蝦腸道內弧菌屬所占比例要高于對照組,有可能試驗組凡納濱對蝦腸道內弧菌屬下大多都是有益菌,一些致病菌可能被抑制生長。造成兩組凡納濱對蝦腸道內菌群結構差異的可能因素是跟添加生物凈水柵有關。
4 結 論
以高通量測序技術為手段,比較了添加生物凈水柵池塘和傳統池塘凡納濱對蝦腸道細菌群落結構及多樣性,了解了凡納濱對蝦在這兩種養殖條件下對蝦腸道菌群的一般結構、優勢菌群及相應的比例。通過比較分析發現,添加生物凈水柵池塘凡納濱對蝦腸道菌群豐富度比傳統池塘組高,其菌群多樣性也高于傳統池塘。凡納濱對蝦腸道菌群結構及多樣性與添加和不添加生物凈水柵有著密切聯系,這也為今后池塘對蝦養殖提供了參考意見,為蝦塘的健康可持續發展提供理論依據。
[1] 溫崇慶, 何瑤瑤, 薛明,等.高通量測序分析DNA提取引起的對蝦腸道菌群結構偏差[J].微生物學報,2016,56(1):130-142.
[2] 鄭天倫, 孔蕾, 朱凝瑜.浙江省南美白對蝦病害流行情況及防控對策[J].科學養魚,2011,3(12):48-49.
[3] 王春忠, 林國榮, 嚴濤,等.長毛對蝦海水養殖環境以及蝦腸道微生物群落結構研究[J].水產學報,2014,38(5):706-712.
[4] Chaiyapechara S, Rungrassamee W, Suriyachay I,et al.Bacterial community associated with the intestinal tract ofP.monodonin commercial farms[J].Microbial Ecology,2012,63(7):938-953.
[5] Schryver P D, Vadstein O.Ecological theory as a foundation to control pathogenic invasion in aquaculture[J].ISME Journal,2014,8(12):2360-2368.
[6] Round J L, Mazmanian S K.The gut microbiota shapes intestinal immune responses during health and disease[J].Nature Reviews Immunology,2009,9(5):313-323.
[7] Sonnenburg J L, Chen C T, Gordon J I.Genomic and metabolic studies of the impact of probiotics on a model gut symbiont and host[J].PloS Biology,2006,4(12):2213-2226.
[8] 李繼秋, 譚北平, 麥康森.白斑綜合征病毒與凡納濱對蝦腸道菌群區系之間關系的初步研究[J].上海水產大學學報,2006,15(1):109-113.
[9] 楊鶯鶯, 李卓佳, 林亮,等.人工飼料飼養的對蝦腸道菌群和水體細菌區系的研究[J].熱帶海洋學報,2006,25(3):53-56.
[10] 劉淮德, 王雷, 王寶杰,等.應用PCR-DGGE分析南美白對蝦腸道微生物多樣性[J].飼料工業,2008,29(20):55-58.
[11] 秦楠, 栗東芳, 楊瑞馥.高通量測序技術及其在微生物學研究中的應用[J].微生物學報,2011,51(4):445-457.
[12] 裴鵬兵, 杜虹, 黃曉穎,等.PET凈水柵對蝦池養殖生態環境的影響研究[J].南方水產科學,2017,13(4):42-51.
[13] 江興龍, 鄧來富.凡納濱對蝦(Litopenaeusvannamei)池塘生物膜低碳養殖技術研究[J].海洋與湖沼,2013,44(6):1536-1543.
[14] 江興龍.日本鰻鱺(Anguillajaponica)土池生物膜原位修復低碳養殖技術的研究[J].海洋與湖沼,2012,43(6):1134-1140.
[15] 張云鵬, 朱立穎, 肖朝耿,等.南美白對蝦體表與腸道細菌菌群結構的DGGE分析[J].中國食品學報,2016,16(8):218-224.
[16] Liu H D, Wang L, Liu M,et al.The intestinal microbial diversity in Chinese shrimp (Fenneropenaeuschinensis) as determined by PCR-DGGE and clone library analyses[J].Aquaculture,2011,317(1/4):32-36.
[17] 李可, 鄭天凌, 田蘊,等.南美白對蝦腸道微生物群落的分子分析[J].微生物學報,2007,47(4):649-653.
[18] 楊坤杰, 王欣, 熊金波,等.健康和患病凡納濱對蝦幼蝦消化道菌群結構的比較[J].水產學報,2016,40(11):1765-1773.
[19] Cardona E, Gueguen Y, Magre K,et al.Bacterial community characterization of water and intestine of the shrimpLitopenaeusstylirostrisin a biofloc system[J].BMC Microbiology,2016,16(1):1-9.
[20] Wu S G, Tian J Y, Gatesoupe F J,et al.Intestinal microbiota of gibel carp (Carassiusauratusgibelio) and its origin as revealed by 454 pyrosequencing[J].World Journal of Microbiology and Biotechnology,2013,29(9):1585-1595.
[21] 吳金鳳, 熊金波, 王欣,等.腸道菌群對凡納濱對蝦健康的指示作用[J].應用生態學報,2016,27(2):611-621.
[22] 孫振麗, 宣引明, 張皓,等.南美白對蝦養殖環境及其腸道細菌多樣性分析[J].中國水產科學,2016,23(3):594-605.
[23] 李玉宏, 柴鵬程, 胡修貴,等.應用RFLP和DGGE技術分析工廠化養殖凡納濱對蝦腸道微生物群落特征[J].漁業科學進展,2014,35(2):83-89.
[24] Rungrassamee W, Klanchui A, Chaiyapechara S,et al.Bacterial population in intestines of the black tiger shrimp (Penaeusmonodon) under different growth stages[J].PloS One,2013,8(4):e60802.
[25] Zhao P, Huang J, Wang X H,et al.The application of bioflocs technology in high-intensive, zero exchange farming systems ofMarsupenaeusjaponicus[J].Aquaculture,2012,354/355(2):97-106.
[26] Wang C Z, Lin G R, Yan T,et al.The cellular community in the intestine of the shrimpPenaeuspenicillatusand its culture environments[J].Fisheries Science,2014,80(5):1001-1007.
[27] 劉晴, 楊蘭, 王東田,等.凈水污泥為載體的生物膜法在印染廢水處理中的應用[J].環境工程學報,2017,11(1):188-196.
[28] 孫寓姣, 趙軒, 王蕾,等.灃河水系脫氮微生物群落結構研究[J].生態環境學報,2014,23(9):1451-1456.
[29] Qiu C S, Zhang D D, Sun L P,et al.Purification of high ammonia wastewater in a biofilm airlift loop bioreactor with microbial communities analysis[J].World Journal of Microbiology and Biotechnology,2015,31(1):49-57.
[30] Pang S, Zhang S H, Lv X Y,et al.Characterization of bacterial community in biofilm and sediments of wetlands dominated by aquatic macrophytes[J].Ecological Engineering,2016,97(1):242-250.
[31] Tang Y Y, Tao P Y, Tan J G,et al.Identification of bacterial community composition in freshwater aquaculture system farming ofLitopenaeusvannameireveals distinct temperature-driven patterns[J].International Journal of Molecular Sciences,2014,15(8):13663-13680.
[32] Xiong J B, Wang K, Wu J F,et al.Changes in intestinal bacterial communities are closely associated with shrimp disease severity[J].Applied Microbiology and Biotechnology,2015,99(16):6911-6919.
[33] 張家松, 段亞飛, 張真真,等.對蝦腸道微生物菌群的研究進展[J].南方水產科學,2015,11(6):114-119.
[34] Wang X H, Li H R, Zhang X H,et al.Microbial flora in the digestive tract of adult penaeid shrimp (Penaeuschinensis)[J].Journal of Ocean University of Qingdao,2000,30(3):492-498.
[35] Cytryn E, Van R J, Schramm A,et al.Identification of bacteria potentially responsible for oxic and anoxic sulfide oxidation in biofilters of a recirculating mariculture system[J].Applied and Environmental Microbiology,2005,71(10):6134-6141.

