小麥小花細胞核蛋白質雙向電泳體系的優化
宋歸華,張迎新,2,侯澤豪,孫坤坤,方正武,馬東方,張改生,王書平
(1.長江大學農學院/湖北省澇漬災害與濕地農業重點實驗室,湖北荊州 434025;2. 中國科學院遺傳與發育生物學研究所, 北京 100101;3. 西北農林科技大學農學院/小麥育種教育部工程研究中心,陜西楊凌 712100)
在真核細胞當中,細胞核是一個重要的亞細胞器,更是遺傳信息的主要儲存場所,包含著絕大多數DNA,是細胞遺傳與代謝的活動中心[1]。而細胞核蛋白質在基因表達調控、信號傳導、染色質重塑、轉錄調節等方面具有決定性的作用[2]。全細胞蛋白質中約10%~20%的蛋白質位于細胞核當中,在復雜的代謝網絡和生理過程中扮演著重要的角色,暗示著細胞核具有的功能多樣性與這些蛋白質密切相關[3-4]。此外,高等植物中,除韌皮部成熟的篩管等極少數細胞外,其他細胞都具有細胞核[5]。因此,研究細胞核的蛋白質組成及其動態變化,對于深入解析植物生長發育與逆境脅迫應答過程中基因表達調控的分子機制具有至關重要的意義。
蛋白質組學對于廣泛分析蛋白質表達、轉錄后翻譯以及蛋白質之間的相互作用具有主導性作用。但由于高等真核細胞的復雜性,對其全細胞蛋白質組進行分析,受到諸多因素的限制,例如蛋白質豐度、大小、疏水性及其他電泳特性等。而將全細胞蛋白質組依據細胞的大分子結構(包括亞細胞隔室、亞細胞器、大分子結構、多蛋白復合物等)分解成一系列亞細胞蛋白質組[6],這不僅降低了全細胞分析的復雜性,而且為我們提供了蛋白質定位及通路劃分方面的重要信息[7],目前已經在小麥線粒體[8-9]、葉綠體[10]、質膜[11]等亞細胞器中得到了廣泛的應用。近年來,隨著植物蛋白質組學技術平臺的日趨成熟、各類植物基因組測序工作的不斷完成以及細胞核蛋白質數據庫的逐漸完善,極大地促進了細胞核蛋白質組學在植物中的應用。目前,有關植物細胞核蛋白質組學的研究主要集中在雙子葉植物的擬南芥(Arabidopsisthaliana)[12-14]、蘋果(Maluspumila)[15]、鷹嘴豆(Cicerarietinum)[16-18]、大豆(Glycinemax)[19]、番茄(Solanumlycopersicum)[20]以及單子葉植物的水稻(Oryzasativa)[2, 21-23]、洋蔥(Alliumcepa)[24]、維柯薩(Xerophytaviscosa)[25-27]、玉米(Zeamays)[28]等,而在其他植物上,尤其是小麥細胞核蛋白質組學方面的研究鮮見報道。眾所周知,針對于亞細胞蛋白質組學,如何獲得高純度、完整性好的細胞核是進行其蛋白質組學研究的前提之一。迄今為止,已經報道并建立了擬南芥[12-13]、水稻[2, 21-22]、維柯薩[25-27]等細胞核蛋白質雙向電泳體系,涉及懸浮細胞[13, 21-22]、葉片[12, 15, 25-27]、幼苗[2, 16-18]、根尖分生組織[2]等不同的組織器官。由于植物間細胞核在細胞大小、蛋白質含量、次生代謝物、亞細胞組分相關聯程度等方面存在顯著的差異,因此探索不同植物細胞核及其蛋白質的最佳提取方法,建立和優化蛋白質2-DE技術對提高特異組織細胞核的分辨率仍是亟待解決的問題[9]。
本研究以小麥三核期小花為材料,對細胞核蛋白質組學中細胞核的分離與純化、蛋白質提取方法、SDS-PAGE膠濃度、上樣量等方面進行了優化,探索出一套適合于小麥小花細胞核分離及純度鑒定的方法,建立了分辨率高和重復性好的細胞核蛋白質2-DE的分析體系,以期對今后開展小麥細胞核蛋白質組學研究提供理論和技術支撐,也為其他同科、屬植物的細胞核2-DE蛋白質研究提供參考。
1 材料和方法
1.1 供試材料與處理
供試材料為正常發育小麥品種西農1376,在散粉前,通過株型、穗型結合鏡檢來判斷花藥小孢子發育時期,4 ℃剝取三核期小花(去芒和穎片)。
1.2 細胞核的分離與純化
稱取小麥小花(去芒和穎片)60 g,于含有0.3% PVP的液氮中快速研磨成粉末。將研磨好的粉末置于600 mL預冷的蔗糖緩沖液(0.5 mol·L-1蔗糖, 80 mmol·L-1KCl, 10 mmol·L-1EDTA, 1 mmol·L-1亞精胺, 1 mmol·L-1精胺, 10 mmol·L-1Tris-HCl, pH 9.5)中,冰浴20 min;輕柔混勻后加入0.15% β-Me和0.5% Triton X-100,4 ℃下裂解20 min,每隔2 min輕柔攪拌一次,每次20 s。均勻后,用2層紗布和1層Miracloth過濾,濾液于4 ℃下1 800 r·min-1離心20 min。棄上清,沉淀用軟毛筆懸浮于150 mL蔗糖緩沖液中;2層Mirocloth過濾,4 ℃、57 r·min-1離心2 min。取上清,4 ℃、1 800 r·min-1離心15 min;沉淀經懸浮后,4 ℃、1 800 r·min-1離心10 min。棄上清,最后得到的沉淀即為細胞核。
1.3 細胞核蛋白質的制備、定量及SDS-PAGE檢測
依據陳 征等[9]和宋齊魯等[11]的方法,略有改動,提取蛋白質樣品。在細胞核樣品中加入預冷的TCA-丙酮溶液(10% TCA,0.07% β-ME,1 mmol·L-1PMSF),在-20 ℃下過夜沉淀蛋白質。4 ℃下25 000 r·min-1離心30 min,棄上清,沉淀用預冷的丙酮溶液(0.07% β-ME, 1 mmol·L-1PMSF)洗滌,-20 ℃下靜置1 h后,4 ℃下25 000 r·min-1離心30 min,重復離心、洗滌至沉淀物為純白色。加入預冷的80%丙酮,重懸沉淀,-20 ℃下靜置1 h;4 ℃下25 000 r·min-1離心30 min,沉淀冷凍成干粉。
將細胞核蛋白質干粉溶解于裂解緩沖液[9 mol·L-1尿素,4% CHAPS,65 mmol·L-1DTT,0.5% IPG(pH 4~7)緩沖液,0.001%溴酚藍]中,分別于液氮與32 ℃水浴中交替凍融3次,22 ℃下25 000 r·min-1離心15 min,上清液為蛋白質樣品,Bradford法[29]測定樣品液蛋白質含量。
1.4 細胞核純度檢測
分別在細胞器和蛋白質水平上對分離到的細胞核進行純度檢測。細胞核的顯微觀察采用DAPI熒光染色法[30]。分離過程中各個組分葉綠素含量的測定參考Dean和Miskiewicz的方法[31]進行,包括勻漿液、首次沉淀、最終的上清液及細胞核沉淀。在1 mL懸浮樣品中分別加入8 mL丙酮和1 mL ddH2O,室溫下避光萃取過夜,1 000 r·min-1離心5 min,立即在652 nm波長下測定吸光值。
細胞核蛋白質經12%的PAGE-SDS電泳分離后進行PVDF轉膜,以TBST(含5% BSA)溶液封閉后,依次經一抗(Anti-histone core antibody;Abcam公司,ab7832)和二抗(Abcam公司)孵育后,最后用堿性磷酸酶底物顯色試劑盒顯色。
1.5 固相pH梯度雙向電泳
參照宋齊魯等[11]的方法進行。取適量蛋白質樣品與水化液共350 μL充分混勻后,加入聚焦槽內。將17 cm(pH 4~7) IPG膠條膠面朝下置于樣品溶液上,被動吸收1 h后,加3 mL礦物油覆蓋膠條,置于IPG-phor等電聚焦儀進行等電聚焦,設置極限電流為50 μA。等電聚焦結束后,依次用含2% DTT和2.5%碘乙酰胺的平衡緩沖液(6.0 mol·L-1尿素,2%SDS,0.375 mol·L-1Tris-HCl pH 8.8,20%甘油)分別平衡15 min。然后將IPG膠條轉移到SDS-PAGE凝膠上,用低熔點瓊脂糖封膠液密封排除氣泡,電流20 mA。其中,上樣量參考陳 征等[9]和王書平等[10]的方法進行,分別為200 μg、230 μg和250 μg;膠濃度設定為11%、12%和13%[11]。待溴酚藍前沿距玻璃板下緣0.5 cm時停止電泳,凝膠染色按改良后的硝酸銀染方法操作,見表1。

表1 蛋白質硝酸銀染步驟Table 1 Sliver staining protocol for proteins
1.6 凝膠圖像分析
銀染后的2-DE凝膠用光密度掃描儀UMAX PowerLook 2100XL掃描。在PDQuest 2DE 8.0.1分析軟件的輔助下,對圖像進行背景扣除、強度校正、標準化、斑點檢測、獲取斑點位置坐標等分析。
1.7 統計分析
利用Microsoft Excel 2010進行數據整理和表格繪制,采用OriginPro 2017軟件進行統計分析及繪圖。
2 結果與分析
2.1 小麥小花細胞核的分離純度
利用高滲蔗糖緩沖液分離出了小麥小花細胞核,并通過反復的差速離心技術去除了其他細胞器的污染,富集到了高濃度的細胞核。通過對葉綠素含量的測定和比較,葉綠素在其他分離組分中含量較高,為99.8%,而在富集純化后的細胞核中僅為1.8%(圖1A)。進一步研究發現,細胞核經DAPI染色后,呈現出較為均勻、清晰的細胞輪廓,細胞核完整性較高。經統計分析,完整細胞核的分離效率達到95%以上(圖1B)。為了進一步對細胞核蛋白質的富集情況進行分析,采用梯度SDS-PAGE進行蛋白質的分離(圖1C)及用細胞核組蛋白特異性抗體進行蛋白質免疫印跡檢測(圖1D)。結果表明,所制備的細胞核蛋白質條帶分布均勻、清晰,組蛋白H1、H3、H2B顯示全面完整。綜合以上結果,分離獲得的細胞核污染少,純度較高,所制備的細胞核蛋白質組次生代謝物質含量較低,且較為完整。
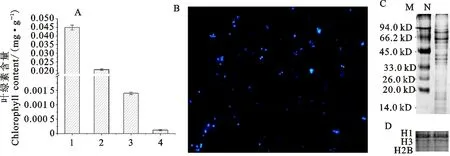
A:不同分離純化階段葉綠素含量測定;1:粗提液;2:沉淀;3:上清液;4:細胞核;B:細胞核DAPI染色;C:細胞核蛋白質SDS-PAGE(12%);M:Marker;N:細胞核蛋白質;D:組蛋白免疫印跡分析。
A:Determination of chlorophyll content at different stages of purification; 1:Crude homogenate; 2:Pellet; 3:Supernatant; 4:Nuclear fraction; B:The purified nuclear fraction was stained with DAPI; C:SDS-PAGE(12%) Silver-stained image of extracted nuclear proteins;M:Marker; N:Nuclear fraction;D:Western blot analysis of extracted nuclear proteins with anti-histone core.
圖1細胞核的分離提取及純度鑒定
Fig.1Isolationandpuritydeterminationofthenuclearfraction
2.2 不同SDS-PAGE凝膠濃度對2-DE圖譜的影響
SDS-PAGE凝膠濃度決定了細胞核蛋白質組的分離效果,并有效確保高、低分子量蛋白質點在2-DE圖譜上的均勻分布。從圖2來看,當凝膠濃度為11%時,高分子量蛋白質點(>26 kD)得到了相對較好的分離,但低分子量蛋白質點(<20 kD)由于超出了凝膠的分離范圍,部分蛋白質點未能在圖譜上檢測到,此時檢測到細胞核蛋白質組數量為241個(圖2A)。改用13%的凝膠后,蛋白質點在圖譜上的分布情況恰恰相反,表現為高分子量蛋白質點高度凝聚,部分蛋白質點不能有效識別,進而降低了分辨率(圖2C),共檢測到223個蛋白質點。而經12%凝膠進行分離后,整個細胞核蛋白質組得到了較好的分離效果(圖2B),蛋白質點分布均勻且清晰圓潤,共檢測獲得264個蛋白質點。這表明采用凝膠濃度為12%更能對細胞核蛋白質組進行有效地分離。
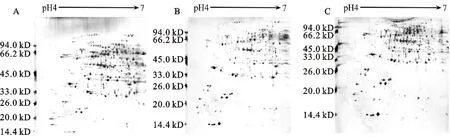
A:11% SDS-PAGE;B:12% SDS-PAGE;C:13% SDS-PAGE. 圖2 不同SDS-PAGE凝膠濃度的2-DE圖譜比較分析Fig.2 Comparison of 2-DE maps of different SDS-PAGE gel concentration
2.3 不同上樣量對2-DE圖譜的影響
蛋白質的上樣量大小直接決定了2-DE圖譜上蛋白質點的數量。結果表明,上樣量為200 μg時,2-DE圖譜上低豐度蛋白質較弱或無顯示,蛋白點數量較少,共檢測到212個蛋白質點(圖3A)。隨著上樣量的增加,蛋白點的數量呈上升趨勢。當上樣量增至250 μg時,高豐度蛋白質明顯聚集,堿性端出現過飽和重疊現象,2-DE圖譜上縱橫條紋明顯,背景加深,點與點之間難以分離,不利于后期檢測和分析,此時,共檢測到241個蛋白質點(圖3C)。上樣量為230 μg時,低豐度蛋白質點增加,無橫條紋干擾,圖譜清晰,蛋白質點獨立清晰,共檢測到264個蛋白質點(圖3B)。
2.4 小麥小花細胞核蛋白質組在2-DE圖譜上的分布特征
等電點分析結果表明,在pI 4.0~4.5區域內表達蛋白質點最少,為16±1個,pI 6.0~6.5區域內表達蛋白質點最多,為71±5個,其他等電點區域表達蛋白質點分別為43±4個(pI 4.5~5.0)、50±3個(pI 5.0~5.5)、55±4個(pI 5.5~6.0)、29±2個(pI 6.5-7.0)(圖4A)。在分子量方面,14.4 kD以下區域內表達的蛋白質點最少,為3±1個,45.0~66.2 kD區域內表達的蛋白質點最多,為85±4個,其他分子量區域內表達的蛋白質點分別為20±2個(14.4~20.0 kD)、16±2個(20.0~26.0 kD)、10±1個(26.0~33.0 kD)、42±3個(33.0~45.0 kD)、38±2個(66.2~94.0 kD)、50±3個(>94.0 kD)(圖4B)。
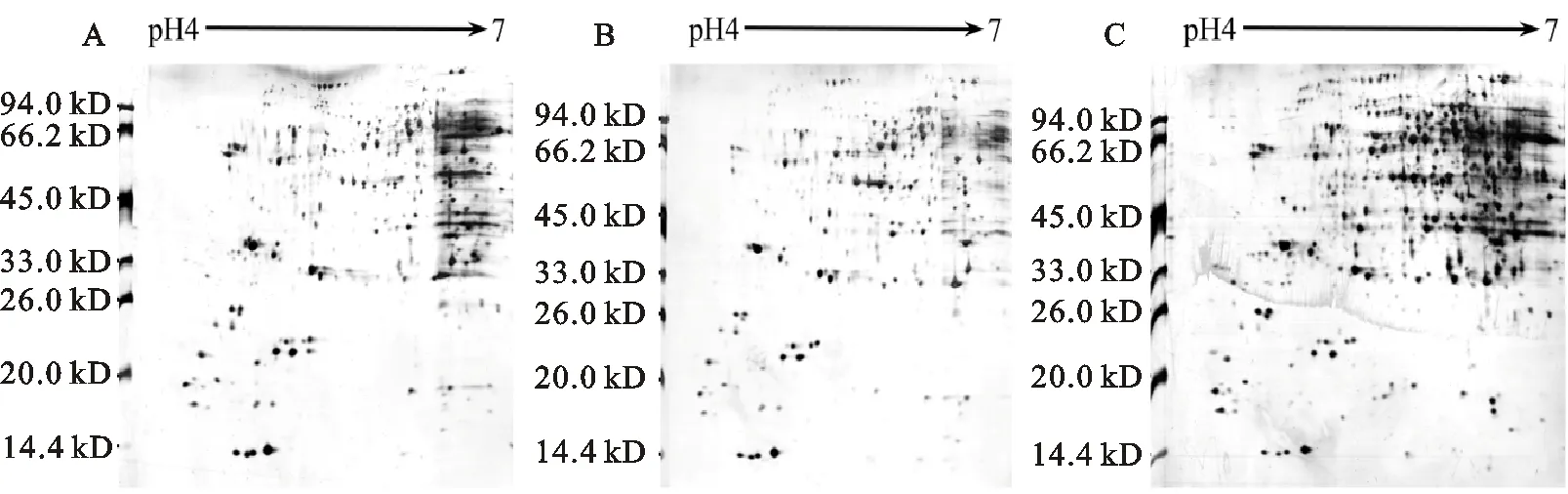
A:上樣量為200 μg的2-DE圖譜;B:上樣量為230 μg的2-DE圖譜;C:上樣量為250 μg的2-DE圖譜。
A:2-DE maps obtained with loading proteins samples of 200 μg; B:2-DE maps obtained with loading proteins samples of 230 μg;C:2-DE maps obtained with loading proteins samples of 250 μg.
圖3不同上樣量的2-DE圖譜比較
Fig.3Comparisonof2-DEmapsofdifferentloadingquantity
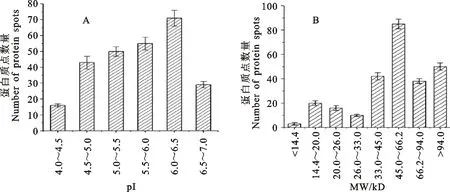
A:蛋白質的等電點分布特征;B:蛋白質的分子量分布特征。
A:Distribution of protein spots according to isoelectric point(pI);B:Distribution of protein spots according to and molecular weight(MW).
圖4細胞核蛋白質點的分布特征
Fig.4Distributionofnuclearproteinsspots
3 討 論
植物與動物和微生物相比,具有細胞壁、葉綠體、次生代謝物多等特點,因此植物的細胞核提取也就相對較為困難。亞細胞蛋白質組學的上游研究技術是亞細胞結構的提取和純化即細胞的分級分離,一般葉綠體沉降系數為3×105 s,密度為1.21 g·cm-3;線粒體沉降系數為1~1.7×104s,密度為1.18~1.19 g/cm3;而細胞核沉降系數為7×106s左右,密度為1.3 g·cm-3[10]。因此,各細胞組分依據浮力密度的不同可逐級有效分離。目前的植物細胞核蛋白質組學研究主要是從植物葉片、幼苗或懸浮培養細胞中提取細胞核,再利用勻漿機破碎或細胞壁水解酶除去細胞壁,然后通過改變細胞內外滲透壓破碎原生質體,并利用不同的離心方式以富集細胞核[1]。本研究中,通過在小麥小花研磨過程中加入0.3% PVP,不僅有效去除了多糖多酚等次生代謝物的污染,同時也減少了細胞核蛋白質的修飾。而在蔗糖溶液中加入適當濃度的Triton X-100(0.5%),能夠在短時間內裂解細胞膜,避免了對核膜的損傷。在細胞核分離過程中,本研究采用的差速離心法簡單快捷,已經在其他作物上得到了應用[2,16-20, 24, 28]。就小麥三核期小花而言,其含有更多的多糖、淀粉粒及部分未破裂的花粉粒等物質。依據這一特性,本研究在離心過程中適當的進行了優化,在增加過濾次數(2次)的同時也相應增加了離心洗滌次數,通過不同的離心速率最大程度地去除了質膜、葉綠體、線粒體、細胞質等的污染。
亞細胞純度鑒定是確保后續研究順利實施的前提。本研究分別采用三種方法來進行細胞核純度檢測,首先測定細胞核制備液中葉綠素含量,以確定葉綠體在細胞核中的污染程度;其次通過對獲取的細胞核進行DAPI染色,經顯微鏡觀察以直觀的判斷亞細胞結構的形態學特征,觀察是否有其他組分的存在及研究對象的完整性;最后制備細胞核蛋白質,以組蛋白為細胞核標記蛋白質,通過免疫印跡技術來檢測細胞核蛋白質的純度。經比較分析,本研究所提取的細胞核純度較高,所制備的細胞核蛋白質組次生代謝物質含量較少,較完整。這為雙向電泳的順利開展奠定了材料基礎。
植物細胞核是儲藏遺傳信息的主要場所,其蛋白質組的動態變化直接影響基因的表達調控,進而調節植物生長發育與環境應答過程[4-5]。因此,建立和健全細胞核蛋白質組學研究的方法和體系將為解析植物發育與逆境應答的分子機制提供重要信息。而為了獲得更多的蛋白質信息,要求在進行植物蛋白質樣品制備時,盡量提高樣品蛋白質的溶解性并減少蛋白質的損失和降解,盡可能保留較多的蛋白質種類。樣品質量的好壞直接決定雙向電泳的分辨率、重復性和蛋白質質譜的結果[9]。在前期的研究過程中,已經采用TCA-丙酮法對小麥小花葉綠體、線粒體以及葉片質膜的蛋白質進行提取,構建并優化了相應的蛋白質雙向電泳體系。本研究在這些研究結果的基礎上,繼續采用成熟的TCA-丙酮法來制備細胞核蛋白質,同時結合細胞核中核酸含量高、疏水性核膜蛋白質難以溶解等特點,對該制備方法進行了進一步優化。利用超聲波對細胞核裂解液進行間隔冰浴破碎,使大量核酸分子形成小片段,降低蛋白質樣品的粘度,有助于細胞核蛋白質的溶解。離心時加大轉速,有效去除蛋白樣品中的鹽分,減少2-DE圖譜中橫豎條紋干擾的現象。此外,聚焦過程中,通過在聚焦槽陽極端架設鹽橋,不僅可以避免因鹽離子過高造成的燒膠條現象,同時還可以減少2-DE圖譜上的橫條紋。
凝膠濃度和上樣量直接影響雙向電泳的分辨率和重復性[9,11]。因此,在蛋白質組學研究中,針對目標蛋白質的不同,應對分離膠的濃度進行適當調整。本研究中11% SDS-PAGE的凝膠孔隙較大,凝膠阻力小,蛋白質運動速度較快,2-DE圖譜上部產生空白;13% SDS-PAGE的凝膠孔隙相對較小,凝膠阻力變大,高分子量的蛋白質在中上部凝聚,不利于蛋白質有效的分離;12% SDS-PAGE凝膠濃度對小麥細胞核蛋白質的分離效果最佳,條帶清晰,無拖尾、條紋等現象發生。上樣量的大小是直接影響2-DE圖譜質量和可靠性的重要因素之一,過低的上樣量不利于低豐度蛋白質點的檢測,過高時則高豐度蛋白質點易于掩蓋其他蛋白質點,且橫條紋和拖尾較嚴重。本研究通過對不同上樣量的比較分析,確定在230 μg時,能獲得很好的2-DE圖譜效果,2-DE膠背景干凈,大大地提高了細胞核蛋白質的分離效率和分辨率。
參考文獻:
[1] ERHARDT M,ADAMSKA I,FRANCO O L.Plant nuclear proteomics-inside the cell maestro [J].FEBSJournal,2010,277(16):3295.
[2] CHOUDHARY MK,BASU D,DATTA A,etal.Dehydration-responsive nuclear proteome of rice(OryzasativaL.) illustrates protein network,novel regulators of cellular adaptation,and evolutionary perspective [J].Molecular&CellularProteomics,2009,8(7):1579.
[3] KUMAR A,AGARWAL S,HEYMAN J A,etal.Subcellular localization of the yeast proteome [J].Genes&Development,2002,16(6):707.
[4] NARULA K,DATTA A,CHAKRABORTY N,etal.Comparative analyses of nuclear proteome:extending its function [J].FrontiersinPlantScience,2013,4:100.
[5] 王思寧,趙 琪,戴紹軍.植物細胞核蛋白質組學研究進展[J].現代農業科技,2013(5):225.
WANG S N,ZHAO Q,DAI S J.Research advances on plant cell nucleus proteomics [J].ModernAgriculturalScience&Technology,2013(5):225.
[6] JUNG E,HELLER M,SANCHEZ J C,etal.Proteomics meets cell biology:The establishment of subcellular proteomes [J].Electrophoresis,2000,21(16):3369.
[7] WOJCIK C,DEMARTINO G N.Intracellular localization of proteasomes [J].InternationalJournalofBiochemistry&CellBiology,2003,35(5):579.
[8] WANG S P,ZHANG G S,ZHANG Y X,etal.Comparative studies of mitochondrial proteomics reveal an intimate protein network of male sterility in wheat(TriticumaestivumL.) [J].JournalofExperimentalBotany,2015,66(20):6191.
[9] 陳 征,王書平,張改生,等.小麥小花高純度線粒體的分離及其蛋白質雙向電泳體系建立[J].中國生物化學與分子生物學報,2013,29(10):983.
CHEN Z,WANG S P,ZHANG G S,etal.Isolation of high purity mitochondria from wheat floret for two-dimensional electrophoresis [J].ChineseJournalofBiochemistry&MolecularBiology,2013,29(10):983.
[10] 王書平,張改生,葉景秀,等.殺雄劑SQ-1誘導小麥生理型雄性不育小花完整葉綠體差異蛋白質的鑒定[J].中國生物化學與分子生物學報,2010,26(9):854.
WANG S P,ZHANG G S,YE G X,etal.Differential proteomics analysis of floret intact chloroplasts proteins in wheat physiological male sterile line induced by CHA-SQ-1 [J].ChineseJournalofBiochemistry&MolecularBiology,2010,26(9):854.
[11] 宋齊魯,王書平,張改生,等.小麥旗葉高純度質膜的提取及蛋白質組學雙向電泳體系的建立[J].中國生物化學與分子生物學報,2013,29(7):690.
SONG Q L,WANG S P,ZHANG G S,etal.Preparation of plasma membrane with purity from wheat flag leaves for 2-DE proteomics [J].ChineseJournalofBiochemistry&MolecularBiology,2013,29(7):690.
[12] BAE M S,CHO E J,CHOI E Y,etal.Analysis of the Arabidopsis nuclear proteome and its response to cold stress [J].PlantJournal,2003,36(5):652.
[13] CALIKOWSKI T T,MEULIA T,MEIER I.A proteomic study of theArabidopsisnuclear matrix [J].JournalofCellularBiochemistry,2003,90(2):361.
[14] JONES A M,MACLEAN D,STUDHOLME D J,etal.Phosphoproteomic analysis of nuclei-enriched fractions fromArabidopsisthaliana[J].JournalofProteomics,2009,72(3):439.
[15] SIKORSKAITE-GUDZIUNIENE S,HAIMI P,GELVONAUSKIENE D,etal.Nuclear proteome analysis of apple cultivar 'Antonovka' accessions in response to apple scab(Venturiainaequalis) [J].EuropeanJournalofPlantPathology,2017,148(4):771.
[16] PANDEY A,CHOUDHARY M K,BHUSHAN D,etal.The nuclear proteome of chickpea(CicerarietinumL.) reveals predicted and unexpected proteins [J].JournalofProteomeResearch,2006,5(12):3301.
[17] PANDEY A,CHAKRABORTY S,DATTA A,etal.Proteomics approach to identify dehydration responsive nuclear proteins from chickpea(CicerarietinumL.) [J].Molecular&CellularProteomics,2008,7(1):88.
[18] SUBBA P,KUMAR R,GAYALI S,etal.Characterisation of the nuclear proteome of a dehydration-sensitive cultivar of chickpea and comparative proteomic analysis with a tolerant cultivar [J].Proteomics,2013,13(12-13):1973.
[19] COOPER B,CAMPBELL K B,FENG J,etal.Nuclear proteomic changes linked to soybean rust resistance [J].MolecularBiosystems,2011,7(3):773.
[20] HOWDEN A J M,STAM R,MARTINEZ HEREDIA V,etal.Quantitative analysis of the tomato nuclear proteome duringPhytophthoracapsiciinfection unveils regulators of immunity [J].NewPhytologist,2017,215(1):309.
[21] KHAN M M,KOMATSU S.Rice proteomics:recent developments and analysis of nuclear proteins [J].Phytochemistry,2004,65(12):1671.
[22] MUJAHID H,TAN F,ZHANG J,etal.Nuclear proteome response to cell wall removal in rice(Oryzasativa) [J].ProteomeScience,2013,11(1):26.
[23] AKI T,YANAGISAWA S.Application of rice nuclear proteome analysis to the identification of evolutionarily conserved and glucose-responsive nuclear proteins [J].JournalofProteomeResearch,2009,8(8):3912.
[24] GONZLEZ-CAMACHO F,MEDINA F J.Identification of specific plant nucleolar phosphoproteins in a functional proteomic analysis [J].Proteomics,2004,4(2):407.
[25] ABDALLA K O,THOMSON J A,RAFUDEEN M S.Protocols for nuclei isolation and nuclear protein extraction from the resurrection plantXerophytaviscosafor proteomic studies [J].AnalyticalBiochemistry,2009,384(2):365.
[26] ABDALLA K O,BAKER B,RAFUDEEN M S,etal.Proteomic analysis of nuclear proteins during dehydration of the resurrection plantXerophytaviscosa[J].PlantGrowthRegulation,2010,62(3):279.
[27] ABDALLA K O,RAFUDEEN M S.Analysis of the nuclear proteome of the resurrection plantXerophytauiscosain response to dehydration stress using iTRAQ with 2DLC and tandem mass spectrometry [J].JournalofProteomics,2012,75(8):2361.
[28] FERREIRA B S,BRANCO A T,DE OLIVEIRA M A,etal.Methodological improvements on extraction of nuclear proteins and its preliminary analysis during the maize(ZeamaysL.) endosperm development [J].Protein&PeptideLetters,2006,13(10):981.
[29] ZUO S S,LUNDAHL P.A micro-bradford membrane protein assay [J].AnalyticalBiochemistry,2000,284(1):162.
[30] WANG S P,ZHANG G S,SONG Q L,etal.Abnormal development of tapetum and microspores induced by chemical hybridization agent SQ-1 in wheat [J].PLoSOne,2015,10(3):e0119557.
[31] DEAN R L,MISKIEWICZ E.Rates of electron transport in the thylakoid membranes of isolated,illuminated chloroplasts are enhanced in the presence of ammonium chloride [J].Biochemistry&MolecularBiologyEducation,2010,31(6):410.

