大豆α-生育酚的遺傳與QTL分析
梁慧珍,余永亮,許蘭杰,楊紅旗,董薇,譚政偉,李磊,裴新涌,劉新梅
?
大豆α-生育酚的遺傳與QTL分析
梁慧珍1,余永亮1,許蘭杰1,楊紅旗1,董薇1,譚政偉1,李磊1,裴新涌2,劉新梅1
(1河南省農業科學院芝麻研究中心,鄭州 450002;2河南省農業科學院農業經濟與信息研究所,鄭州 450002)
【目的】通過對大豆α-生育酚進行遺傳和QTL分析,研究其遺傳機制,定位其主效QTL,為高α-生育酚含量的大豆品種選育奠定遺傳學基礎。【方法】以栽培大豆晉豆23為母本、山西農家品種大豆灰布支黑豆(ZDD02315)為父本雜交衍生的447個RIL作為供試群體構建遺傳圖譜,試驗群體及親本分別于2011年、2012年和2015年夏季在河南省農業科學院原陽試驗基地種植,冬季在海南省三亞南繁基地種植。田間試驗采取隨機區組設計,2次重復。從6個環境中每個家系選取15.00 g籽粒飽滿,大小一致的大豆種子,利用高效液相色譜法定性、定量測定樣品中的α-生育酚含量。采用主基因+多基因混合遺傳分離分析法和WinQTLCart 2.5復合區間作圖法,對大豆α-生育酚含量進行主基因+多基因混合遺傳分析和QTL定位。【結果】基于主基因+多基因混合遺傳分離分析法,α-生育酚受4對主基因控制,遺傳基因分布在雙親中。4對主基因間加性效應值中3對為正值,表明這些基因來源于母本晉豆23;1對為負值,表明該對基因來源于父本灰布支黑豆;4對主基因之間相互作用的上位性效應表現為正值和負值的各有3對,說明不同基因間上位性效應對α-TOC的影響方向并不完全一致。環境因素引起的變異為0.13%—4.05%。表明α-TOC主要受4對主基因影響,受環境因素影響較小。采用WinQTLCart 2.5復合區間作圖(CIM)共檢測到17個影響α-生育酚的QTL,分布于第1、2、5、6、8、14、16、17共8條染色體中,單個QTL的貢獻率8.35%—35.78%,QTL主要表現為加性效應。q同時在2011年原陽、2012年原陽和三亞、2015年原陽4個環境下檢測到,且均定位在第1染色體Satt320—Satt254標記區間19.79 cM處,解釋的表型變異分別為12.55%、12.01%和11.89%、12.61%,加性效應值0.119-0.132,增加α-TOC含量的等位基因來自母本晉豆23;q同時在2011年原陽和三亞、2015年原陽3個環境下檢測到,且均定位在第8染色體Sat_129—Satt377標記區間44.53 cM處,解釋的表型變異分別為23.18%和22.56%、23.01%,加性效應值-0.195—-0.180,增加α-TOC含量的等位基因來自父本灰布支黑豆。q和q2個QTL能夠穩定遺傳。【結論】α-生育酚最適遺傳模型符合4MG-AI,即4對具有加性上位性效應的主基因遺傳模型。其遺傳主要受4對主基因影響,受環境因素影響較小。檢測到α-生育酚的2個穩定主效QTL,Satt320—Satt254和Sat_129—Satt377是共位標記區間。
大豆;α-生育酚;主基因+多基因;遺傳;QTL
0 引言
【研究意義】大豆維生素E是脂溶性維生素,參與生物體內信號轉導、基因表達調控、抗低溫脅迫等多種代謝途徑,具有提高機體免疫力、抗不育、抗癌及預防心血管疾病的作用[1-3]。天然維生素E中,只有α-生育酚能被人體很好地吸收和代謝[4],而且α-生育酚的生物活性高,因此維生素E主要以α-生育酚含量為指標[5]。近年來,天然維生素E以每年10%的需求增長,但是,中國國內產量較少,生產幾乎被美國、德國、日本等國家壟斷[6]。因此,選育富含高α-生育酚的大豆品種有重要意義。【前人研究進展】目前,α-生育酚QTL定位研究主要集中在玉米[7-8]、油菜[9]、大麥[10]、向日葵[11-12]和芥菜[13],大豆α-生育酚的遺傳機制和QTL定位較少。Dwiyanti等[14]用SSR標記對大豆F2和F3群體中α-生育酚含量進行了遺傳分析,發現K連鎖群對α-生育酚含量影響較大。Shaw等[15]利用Bayfield×Shire重組自交系群體(RIL),檢測到6個控制α-生育酚的QTL,分別定位在第1、2、4、5、12和20染色體上。Li等[16-17]利用Bayfield×Hefeng 25大豆RIL群體檢測到4個控制α生育酚的QTL,分別定位在第2、6、8和20染色體上。張紅梅等[18]利用Essex×ZDD2315大豆RIL群體分別檢測到2個和4對控制大豆α-生育酚的加性和互作QTL。【本研究切入點】目前,關于大豆α-生育酚QTL被檢測到的報道較少,由于遺傳背景和環境條件的不同,表現穩定的更少,大豆α-生育酚分子標記輔助育種受到限制。【擬解決的關鍵問題】本研究以晉豆23×灰布支黑豆衍生的447個RIL為試驗材料,利用WinQTLCart2.5[19]復合區間模型分析方法,對大豆α-生育酚進行QTL定位分析;采用主基因+多基因混合遺傳分離分析方法[20],分析α-生育酚含量遺傳規律,為高α-生育酚含量的大豆品種選育奠定遺傳學基礎。
1 材料與方法
1.1 試驗材料與設計
供試材料為山西省農業科學院經濟作物研究所配制的晉豆23×灰布支黑豆雜交組合。該組合包含447個家系的重組自交系群體,即每一F2單株衍生成F2家系,每一F2家系再選株建成家系,至2011年是F14代。親本晉豆23和灰布支黑豆分別來自山西省栽培大豆和農家品種。試驗群體及親本分別于2011年、2012年和2015年夏季在河南省農業科學院原陽試驗基地種植,冬季在海南省三亞南繁基地種植。田間試驗采取隨機區組設計,2次重復,行長4.0 m,行距0.4 m。按照大田模式進行田間管理,四周分設保護行,一播全苗。
1.2 α-生育酚含量測定及數據分析
分別從6個環境下每個重復、每個家系各選取15.00 g大小一致、籽粒飽滿的大豆種子,用旋風磨(Retsch ZM100,Φ = 1.0 mm,Rheinische,Germany)磨粉。取樣粉碎后的豆粉樣品2.5 g,加入30 mL無水乙醇混合均勻,再加入5 mL濃度10%抗壞血酸溶液和10 mL氫氧化鉀溶液,用恒溫水浴皂化30 min;然后提取皂化液,采用100 mL正己烷分3次進行,棄去水層;最后,用無水硫酸鈉過濾提取液,在旋轉蒸發瓶中旋轉蒸發,將殘留液用氮氣吹干,再用2.5 mL甲醇溶液進行溶解,通過0.45 μm濾膜,4℃保存備用。
利用高效液相色譜技術(high performance liquid chromatography,HPLC),采用外標法對α-生育酚異構體進行定量分析。色譜柱為DIKMA公司產品,色譜柱填料為symmetry,鉆石C18,5 μm,柱規格為250.0 mm×4.6 mm;熒光檢測器激發波長290 nm,發射波長330 nm;選用甲醇為流動相,流速1.5 mL·min-1。柱溫40℃,進樣量20 μL,檢測時間10 min。以α-生育酚(Alpha tocopherol,以下簡稱α-TOC)峰面積代入回歸方程進行定量分析。α-生育酚含量的計量單位是:mg·(100 g)-1
利用SAS V9.2軟件的PROC ANOVA程序對6種環境下α-生育酚含量進行聯合方差分析和描述統計。遺傳變異系數GCV(100%)=g/×100。其中g為遺傳方差的標準差;為群體平均數,均由試驗數據估計。遺傳率2=g2/[g2+(ge2/)+ (2/)],其中:g2為遺傳方差;ge2為基因型×年份(環境)方差;2為誤差方差;為重復數;為年份數。
1.3 遺傳連鎖圖譜的構建與QTL分析
利用Mapmaker/Exp 3.0構建連鎖圖譜,圖譜總長度2 047.6 cM。包括27個連鎖群、227個SSR標記,平均圖距8.8 cM(該圖譜在在H(Chr.12)染色體出現了1個間隙,在K(Chr.9)、F(Chr.13)和J(Chr.16)3條染色體上均出現了2個間隙,形成了27個連鎖群)。經與Song等[21]整合的圖譜相比對,標記排序相對一致。引物信息、標記試驗過程及作圖過程見參考文獻[22-23]。
QTL定位軟件為WinQTLCart 2.5,命名方式參照McCouch等[24]方法。定位采用軟件中復合區間作圖法(CIM)Zmapqtl方法的Model 6,設置該窗口之外的5個標記作為余因子、步移速度2 cM,向前回歸法全基因組掃描,以確定各性狀QTL數目及其在染色體上的位置。選取臨界閾值LOD=2.5(0.05顯著水平),檢測各環境下的QTL效應。當LOD≥2.5時,認為該位置上QTL存在,臨近位點間圖距小于5 cM時,認定為同一個QTL。
1.4 數量性狀主基因+多基因混合遺傳模型分析
采用主基因+多基因混合遺傳分離分析法[20]中極大似然法和IECM估算晉豆23×灰布支黑豆及其衍生的RIL各世代、各成分分布的參數,,通過AIC準則和一組適合性測驗,選擇最適遺傳模型;利用每一個成分的分布參數通過最小二乘法估計相應的遺傳參數。主基因遺傳率mg2(%) =mg2/p2,多基因遺傳率pg2(%) =pg2/p2,環境遺傳率e2(%) =e2/p2。各遺傳方差間的關系為p2=mg2+pg2+e2。其中:p2為群體表型方差;mg2為主基因遺傳方差;pg2為多基因遺傳方差;e2為多環境遺傳方差。
2 結果
2.1 大豆α-生育酚含量的表型分析
表1和圖1列出了親本和RIL群體中α-生育酚含量的表型變異情況。原陽和三亞2個地點3年試驗中α-生育酚親本差分別為-0.68和-0.72、-0.70和-0.64、-0.71和-0.79,平均差分別為-0.70和-0.72。經值檢測,雙親均值在各年度、各地點均差異顯著。2個親本在后代分離群體中表現出較大的分離程度,不同環境下,RIL群體的最小值和最大值之間差異明顯,存在較大幅度超親分離,表明親本間位點互補,大豆α-生育酚遺傳基因分布在雙親中,通過雜交可以得到超親分離的株系。α-生育酚含量呈現出近似正態的連續分布。同一性狀同一地點不同年份間遺傳變異系數差別不大。α-生育酚3年間2點的遺傳率為62.03%—77.35%。表2為3年聯合方差分析結果。α-生育酚基因型在組分間存在顯著差異,其余變異包括不同環境之間、重復間、年份×地點、年份×基因、地點×基因、年份×地點×基因差異不顯著,表明大豆α-生育酚含量的表達主要受基因的影響,環境影響較小。
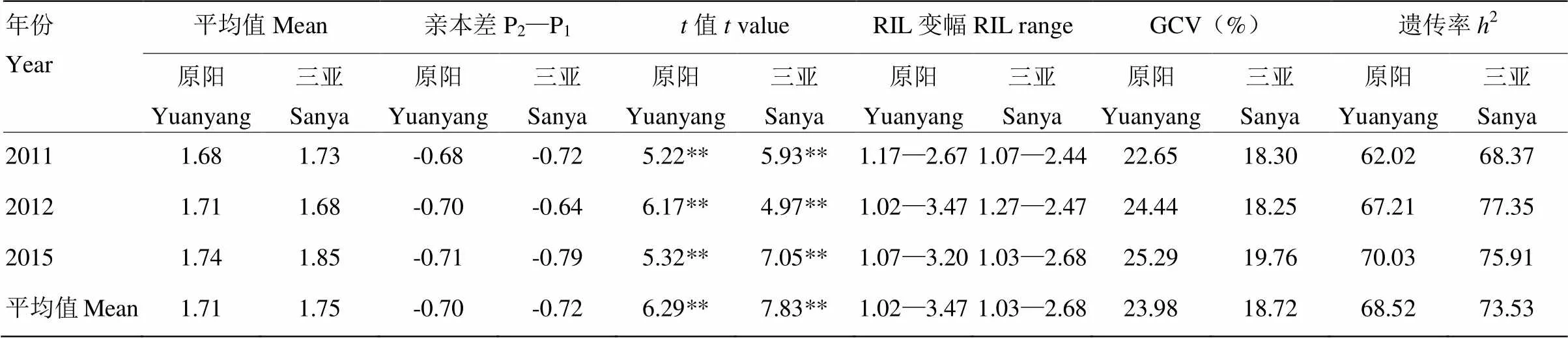
表1 RIL群體大豆α-生育酚含量表型變異
**表示在0.01水平差異極顯著;值=雙親均值差異顯著性(值)
** significance at the 0.01 levels;value=Significant test for difference in parents means (value)
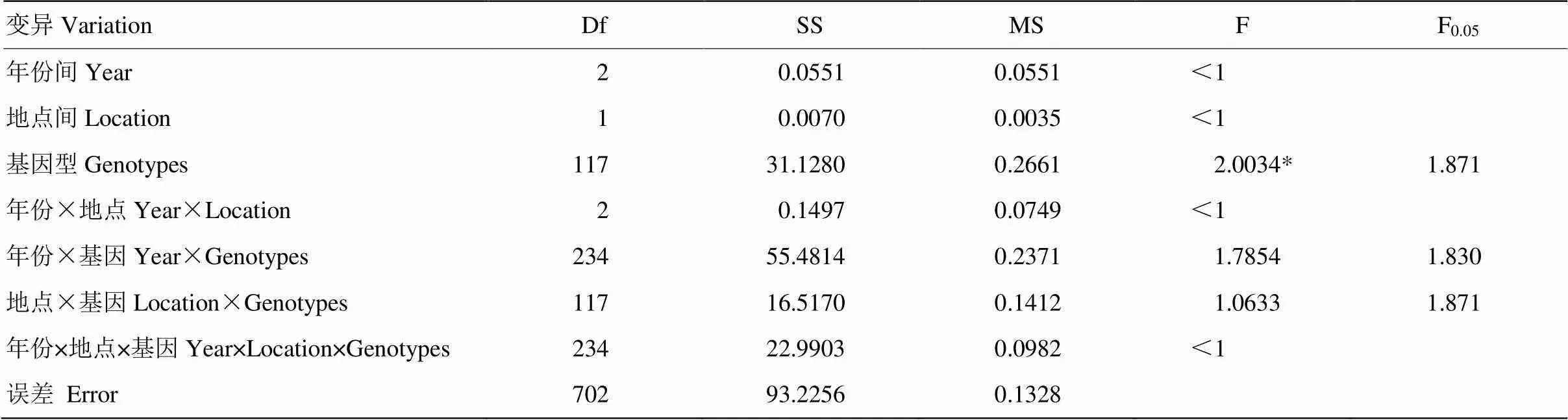
表2 大豆種子α-生育酚含量方差分析
*表示在0.05水平差異顯著
*significance at the 0.05 levels
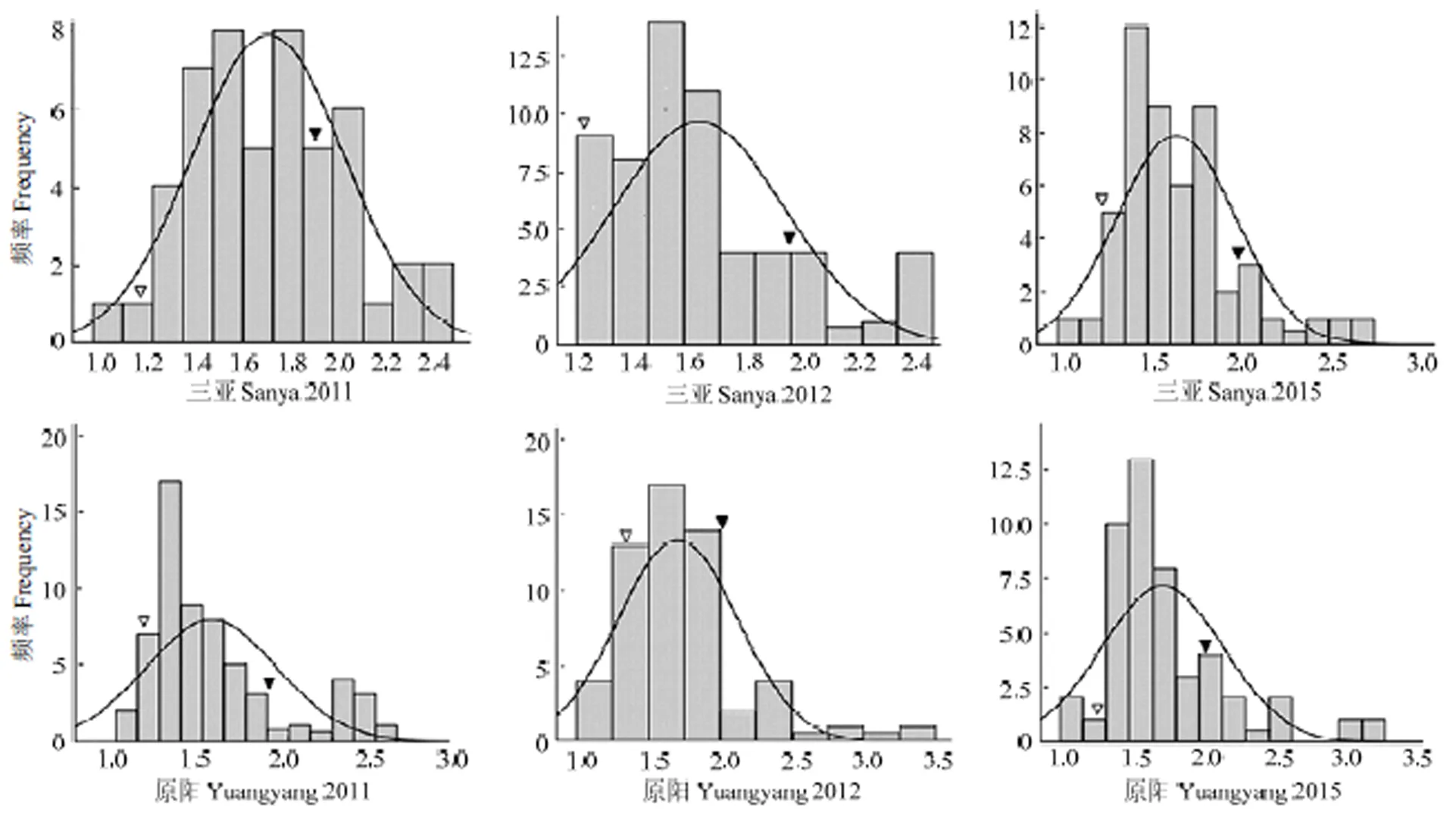
:灰布支黑豆α-生育酚含量α-TOC content of Huibuzhiheidou;:晉豆23 α-生育酚含量α-TOC content of Jindou23
2.2 α-生育酚的主基因+多基因混合遺傳分析
分別選用2011年、2012年和2015年各環境下試驗數據的平均值進行主基因+多基因混合遺傳分析,根據AIC值和適合性測驗(表3),α-TOC最適遺傳模型符合4MG-AI,即4對具有加性上位性效應的主基因遺傳模型。4對主基因間加性效應值中3對為正值,表明基因來源于母本晉豆23;1對為負值,表明基因來源于父本灰布支黑豆;4對主基因之間相互作用的上位性效應表現為正值和負值的各有3對,說明不同基因間上位性效應對α-TOC的影響方向并不完全一致。3年6個環境中均表現出較大的遺傳力,達到95.95%—99.87%,沒有檢測到多基因效應。環境因素引起的變異為0.13%—4.05%,效應值較小。說明α-TOC的遺傳主要受4對主基因影響,環境影響較小。
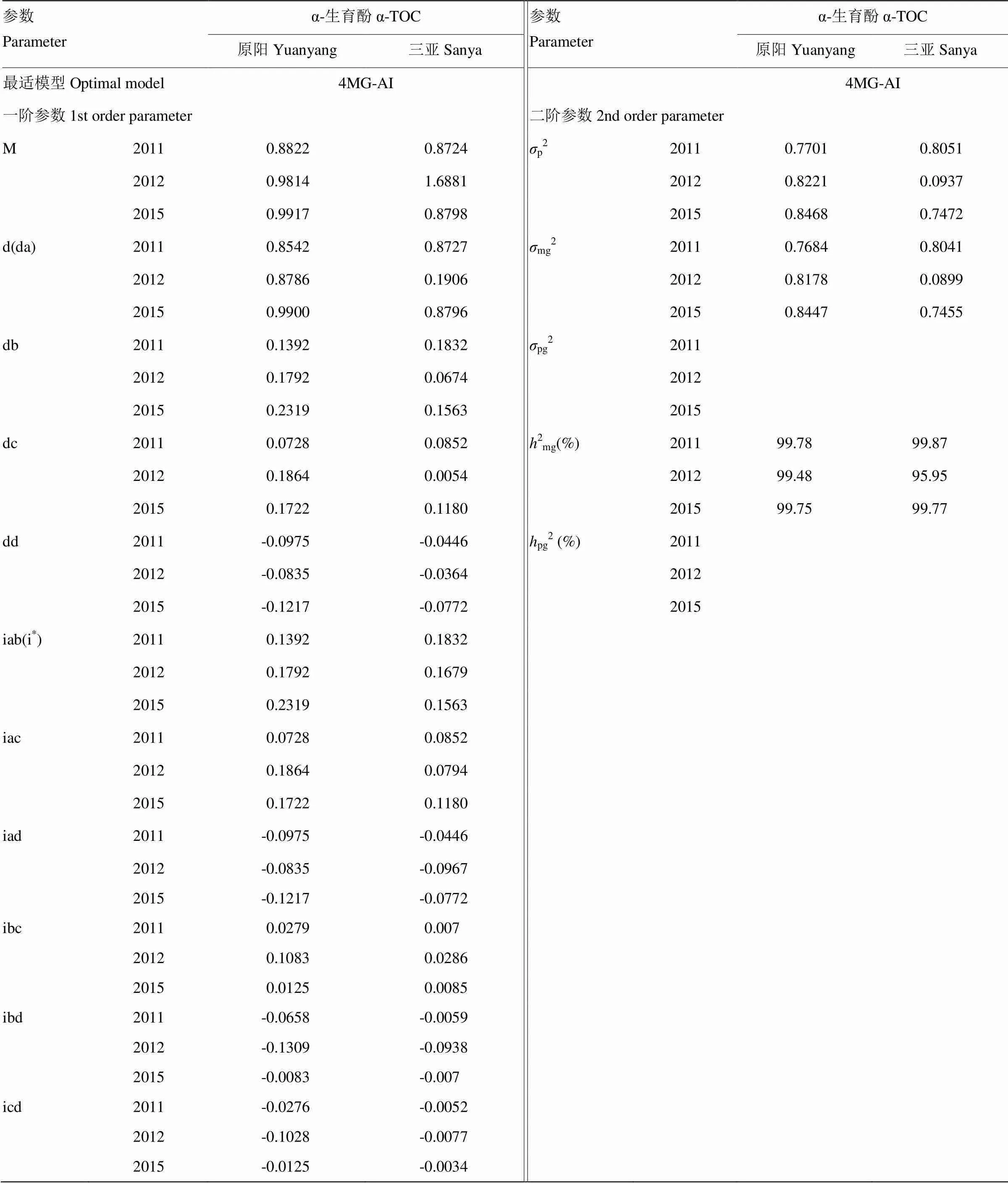
表3 α-生育酚最適模型及遺傳參數估計結果
2.3 大豆α-生育酚的QTL定位
根據WinQTLCart 2.5的定位結果,共在8個連鎖群上檢測到17個α-生育酚的QTL(表4,圖2),分別位于A1(Chr.5)、A2(Chr.8)、B2(Chr.14)、C2(Chr.6)、D1a(Chr.1)、D1b(Chr.2)、D2(Chr.17)和J_2(Chr.16)染色體上,解釋的表型變異范圍為8.35%—35.78%。位于A1(Chr.5)染色體Satt545—Satt511標記區間的q,表型貢獻率為35.78%,解釋的表型變異最大。其中,q同時在2011年原陽、2012年原陽和三亞、2015年原陽4個環境下檢測到,且均定位在D1a(Chr.1)染色體Satt320—Satt254標記區間19.79 cM處,解釋的表型變異分別為12.55%、12.01%和11.89%、12.61%,說明這是一個能夠穩定遺傳的QTL。加性效應值0.119—0.132,增加α-TOC含量的等位基因來自母本晉豆23;同時,在qq均被定位在D1a(Chr.1)染色體上,但2個QTL位置相距較遠,應該不是同一個QTL,說明D1a(Chr.1)染色體上可能存在多個與α-TOC相關的基因。q同時在2011年原陽和三亞、2015年原陽3個環境下檢測到,且均定位在A2(Chr.8)染色體Sat_129—Satt377標記區間44.53 cM處,解釋的表型變異分別為23.18%和22.56%、23.01%,說明這是一個能夠穩定遺傳的QTL。加性效應值-0.195—-0.180,增加α-TOC含量的等位基因來自父本灰布支黑豆。加性效應值正負表現不一,加性效應值為正,說明該QTL對大豆α-生育酚含量起正向作用,等位基因來自于母本晉豆23;加性效應值為負,說明說明該QTL對大豆α-生育酚含量起負向作用,等位基因來自于父本灰布支黑豆。
3 討論
3.1 大豆α-生育酚的遺傳機制
本研究采用混合遺傳模型分離分析方法研究大豆α-生育酚在河南省和海南省2個地點3個年份共6個環境中的遺傳;采用WinQTLCart 2.5軟件中CIM方法進行QTL定位分析。研究表明,α-生育酚的遺傳最適遺傳模型符合4MG-AI,即4對具有加性上位性效應的主基因遺傳模型。3年6個環境中的遺傳力達95.95%—99.87%,環境因素引起的變異僅為0.13%—4.05%。說明α-TOC的遺傳主要受到4對主基因影響,受環境因素影響較小。同時,本研究在8條染色體中檢測到17個α-生育酚的QTL。2種分析結果相比較,發現QTL定位數目與遺傳模型預測的數目并不完全一致。2011年原陽和2012年三亞各檢測出4個QTL,與α-生育酚最適遺傳模型4MG-AI一致,但是其余環境中檢測出的QTL數目為2—3個,與遺傳模型預測的數目不統一。出現這種現象的可能原因有:1)與QTL定位圖譜有關。與標準圖譜相比較,本研究選用的圖譜本身還需要進一步完善;2)與混合遺傳模型有關。本研究選用的是4對主基因+多基因混合遺傳模型[25],該模型只分析了主基因間無連鎖的情況,簡化了主基因間的連鎖作用。同時,該遺傳模型成分分布數和待估參數比較多,分析過程受到限制,估算結果的準確性有待提高;3)控制α-生育酚含量的表達在時空上存在差異。因此,對于α-生育酚含量的遺傳機理研究,在本研究的基礎上尚需進一步深化。

表4 α-生育酚QTL位置及其參數
3.2 大豆α-生育酚含量的QTL分析及與前人定位結果的比較
為了提高QTL定位的精度,通常采取多年多點數 據分析方法,增大QTL檢測強度,進而挖掘出表現穩定的QTL[26]。本研究分析了3年2個地點共6個環境中α-生育酚含量,4個環境下在D1a(Chr.1)染色體Satt320—Satt254標記區間19.79 cM處均檢測到q,3個環境下在A2(Chr.8)染色體Sat_129—Satt377標記區間44.53 cM處均檢測到q,表明q和q為α-生育酚的穩定主效QTL,與之相對應的2個區間也是相對準確可靠。下一步研究將對該共位標記區間實施精細定位,通過將關聯的目標區間內的主效QTL剖分為幾個連鎖的多效性QTL,搜索可能的候選基因,為高α-生育酚含量分子標記輔助育種奠定基礎。
生物的遺傳和進化過程中,廣泛存在多因一效和一因多效現象。這兩種基因作用方式從不同角度共同促進生命活動的發現[27]。多因一效促成了生物多樣性。不同的生物過程參與到同一種表型上,降低了單因素所帶來的局限性,出現了多態性。一因多效將多種過程聯系起來,共同參與基因表達過程,形成一系列生物軸,一方面促進行為的統一性,一方面形成正負反饋,避免生理生化過程的失穩。本研究發現,在8個連鎖群上共檢測到17個α-生育酚的QTL,加性效應值正負均有表現,這些等位基因一方面來自于母本晉豆23,一方面來自于父本灰布支黑豆,推測多因一效可能存在于α-生育酚的形成過程。同時也發現,本研究中穩定表達的與α-生育酚含量相關標記Sat_129和Satt377,在以往研究中,Sat_129與影響大豆生育期[28]脂肪含量[29]和染料木素[30]的基因位點位于同一區域;與影響大豆染料木苷[30]、生育期[31]和耐澇性[32]基因位點位于同一區域;Satt041與影響大豆側根數[33]、Satt267與影響大豆茸毛[34]和側根數[35]的基因位點位于同一區域。表明控制這些性狀的基因可能存在一因多效現象。這些基因位點之間是否存在表達調控機制并通過QTL間的上位性反映出來的?不同研究中這些發揮作用的位點又如何影響到α-生育酚的差異呢?需要進行深入研究與探討。
與以往研究相比,盡管各研究選用的群體、圖譜和環境條件不同,仍有一部分α-生育酚定位結果相吻合。與Shaw等[15]相比較,均定位在A1、D1a和D1b染色體上,且在A1染色體上均定位在Satt545標記處;與Li等[16-17]相比較,均定位在B2、C2和D1b染色體上;與張紅梅等[18]相比較,均定位在A1和C2染色體上;與李海燕[36]等相比較,均定位在A2、C2和D1b染色體上。但是,本研究中檢測出的q和q2個穩定主效QTL,前人研究中雖然在這兩條染色體上均檢測出α-生育酚QTL,但是均不再同一個標記區間,分析其原因可能受到定位群體、定位圖譜、遺傳背景或環境差異等諸多因素影響。除此之外,本研究中定位出的其他QTL,均未見公開報道。
3.3 遺傳圖譜對QTL定位的影響
遺傳圖譜的構建在基因定位與圖位克隆、遺傳多樣性及遺傳標記輔助選擇育種等方面都具有重要意義。遺傳圖譜的應用價值,與圖譜的飽和度、標記數量的多少、標記在圖譜上定位的準確性、標記在圖譜上分布的均勻程度等有關。一個基本的連鎖框架圖要求標記間平均間距不超過20 cM。主基因定位時,其平均距離要求在10—20 cM或更小[37]。如果連鎖群標記較少,圖譜上標記的密度較低,會對QTL的檢出效率造成影響,可能檢測不到效應值較小的QTL,影響定位的準確性[38]。同時,從另一角度分析,此時依然能夠被檢測到的QTL,可能是效應較大的主效QTL,對數量性狀基因的圖位克隆和分子標記輔助選擇更具實際價值。但是,因為前者降低了QTL定位的準確性,其價值與提高LOD的閾值來檢測主效QTL相比依然不可同日而語。本研究圖譜是由海南省熱帶農業資源開發利用研究所方宣鈞研究員提供的(論文中致謝部分有說明),圖譜總長度2 047.6 cM,平均圖距8.8 cM[22],總體上看圖譜平均間距尚能滿足定位要求。下一步工作中,希望能進一步提高圖譜分子標記密度和圖譜飽和度,不斷提高定位結果的精準度。盡管如此,利用該圖譜定位了一批大豆數量性狀的QTL,在國內外期刊發表,研究結果仍然具有一定的理論和現實意義。
本研究所得定位結果與前人相比,多個QTL定位均在相同的染色體上,但定位的標記區間不盡相同,定位位置是否重疊尚不能確定。因為,不同研究中選用的圖譜不盡相同,圖譜不盡完善,所定位的標記區間是否重疊還需要隨著研究手段和使用圖譜的不斷進步和完善逐步得到驗證。Li等[39]認為,由于作圖群體所用親本不同,長期的遺傳演化過程中,親本材料染色體結構會發生變異,相同序列所處的區段在不同的親本中發生改變,導致在不同的研究者之間即便采用相同的分子標記構建的連鎖圖譜之間,分子標記在連鎖群上的排列順序也不盡相同。分子標記用于遺傳圖譜構建是遺傳學領域的重大進展之一,隨著新的標記技術的發展,圖譜上標記的密度也將越來越高。目前的工作可以為將來更加精細的定位提供參考
目前,國內外研究中,QTL定位結果精細程度依然不足。下一步科學研究工作的方向可以通過對大豆α-生育酚性狀進行QTL精細作圖定位,重點跟蹤研究多環境、多分析方法均能檢測到的QTL區域,發掘控制α-生育酚的功能基因。
4 結論
α-生育酚最適遺傳模型符合4MG-AI,即4對具有加性上位性效應的主基因遺傳模型。其遺傳主要受4對主基因影響,受環境因素影響較小。檢測到α-生育酚的2個穩定主效QTL,Satt320—Satt254和Sat_129—Satt377是共位標記區間。
致謝:本研究連鎖圖譜和群體材料由海南省熱帶農業資源開發利用研究所方宣鈞研究員和山西省農業科學院經濟作物研究所劉學義研究員提供,在此表示感謝。
[1] RIMBACH G, MOEHRING J, HUEBBE P. Gene-regulatory activity of alpha-tocopherol., 2010, 15: 1746-1761.
[2] ABBASI A R, HAJIREZAEI M, HOFIUS D, SONNEWALD U, VOLL L M. Specific roles of α- and γ-tocopherol in abiotic stress responses of transgenic tobacco., 2007, 143: 1720-1738.
[3] HINCHA D K. Effects of α-tocopherol (vitamin E) on the stability and lipid dynamics of model membranes mimicking the lipid composition of plant chloroplast membranes., 2008, 582: 3687-3692.
[4] KANWISCHER M, PORFIROVA S, BERGMULLER E, D?RMANN P. Alterations in tocopherol cyclase activity in transgenic and mutant plants ofaffect tocopherol content, tocopherol composition, and oxidative stress., 2005, 137: 713-723.
[5] TAVVA V S, KIM Y H, KAGAN I A, DINKINS R D, KIM K H, COLLINS G B. Increased alpha-tocopherol content in soybean seed overexpressing thegamma-tocopherol methyltransferase gene., 2007, 26(1): 1-70.
[6] 李海燕, 隋美楠, 聶騰坤, 史帥, 韓英鵬, 李文濱. 大豆維生素E五個相關基因表達模式分析. 東北農業大學學報, 2016, 47(5): 15-22.
LI H Y, SUI M N, NIE T K, ShI S, HAN Y P, LI W B. Expression analysis of five relative genes of soybean vitamin E., 2016, 47(5): 15-22. (in Chinese)
[7] FENG F, DENG F, ZHOU P, YAN J, WANG Q, YANG R, LI X. QTL mapping for the tocopherols at milk stage of kernel development in sweet corn., 2013, 193(3): 409-417.
[8] XU S, ZHANG D, CAI Y, ZHOU Y, TRUSHAR S, FARHAN A, LI Q, LI Z, WANG W, LI J, YANG X, YAN J. Dissecting tocopherols content in maize (L.), using two segregating populations and high-density single nucleotide polymorphism markers.,2012, 12(1): 201.
[9] WANG X, ZHANG C, LI L, FRITSCHE S, ENDRIGKEIT J, ZHANG W, LONG Y, JUNG C, MENG J. Unraveling the genetic basis of seed tocopherol content and composition in rapeseed (L.)., 2012, 7(11): e50038.
[10] GRAEBNER R C, WISE M, CUESTA-MARCOS A, GENIZA M, BLAKE T, BLAKE V C, BUTLER J, CHAO S, HOLE D J, HORSLEY R, JAISWAL P, OBERT D, SMITH K, ULLRICHL S, HAYESL P M. Quantitative trait loci associated with the tocochromanol (vitamin E) pathway in barley., 2015, 10(7): e0133767.
[11] HADDADI P, EBRAHIMI A, LANGLADE N B, YAZDI-SAMADI B, BERGER M, CALMON A, NAGHAVI M R, VINCOURT P, SARRAFI A. Genetic dissection of tocopherol and phytosterol in recombinant inbred lines of sunflower through quantitative trait locus analysis and the candidate gene approach., 2012, 29(3): 717-729.
[12] MORAL L D, FERNANDEZ-MARTINEZ J M, VELASCO L, PEREZVICH. Quantitative trait loci for seed tocopherol content in sunflower., 2012, 52 (2): 786-794.
[13] GUPTA S, SANGHA M K, KAUR G, BANGA S, GUPTA M, KUMAR H, BANGA S S. QTL analysis for phytonutrient compounds and the antioxidant molecule in mustard (L.)., 2015, 201(3): 345-356.
[14] DWIYANTI M S, UJIIE A, THUY L T B, YAMDA T, KITAMURA K. Genetic analysis of high α-tocopherol content in soybean seeds., 2007, 57: 23-28.
[15] SHAW E, RAJCAN I. Molecular mapping of soybean seed tocopherols in the cross ‘OAC Bayfield’בOAC Shire’., 2017, 136: 83-93.
[16] LI H, WANG Y, HAN Y, TENG W, ZHAO X, LI Y, LI W. Mapping quantitative trait loci (QTLs) underlying seed vitamin E content in soybean with main, epistatic and QTL×environment effects., 2016, 135(2): 208-214.
[17] LI H, LIU H, HAN Y, WU X, TENG W, LIU G, LI W. Identification of QTL underlying vitamin E contents in soybean seed among multiple environments., 2010, 120(7): 1405-1413.
[18] 張紅梅, 李海朝, 文自翔, 顧和平, 袁星星, 陳華濤, 崔曉艷, 陳新, 盧為國. 大豆籽粒維生素E含量的QTL分析. 作物學報, 2015, 41(2): 187-196.
ZHANG H M, LI H C, WEN Z X, GU H P, YUAN X X, CHEN H T, CUI X Y, CHEN X, LU W G. Identification of QTL associated with vitamin E content in soybean seeds., 2015, 41(2): 187-196.
[19] WANG S C, BASTEN C J, ZENG Z B. Windows QTL cartographer 2.5 user manual. Department of Statistics, North Carolina State University, Raleigh, NC, 2005.
[20] 曹錫文, 劉兵, 章元明. 植物數量性狀分離分析Windows軟件包SEA的研制. 南京農業大學學報, 2013, 36(6): 1-6.
CAO X W, LIU B, ZHANG Y M. SEA: a software package of segregation analysis of quantitative traits in plants., 2013, 36(6): 1-6. (in Chinese)
[21] SONG Q J, MAREK L F, SHOEMAKER R C, LARK K G, CONCIBIDO V C, DELANNAY X, SPECHT J E, CREGAN P B. A new integrated genetic linkage map of the soybean., 2004, 109: 122-128.
[22] 梁慧珍. 大豆子粒性狀的遺傳及QTL分析[D]. 楊凌: 西北農林科技大學, 2006.
LIANG H Z. Genetic analysis and QTL mapping of seed traits in soybean ((L.) Merr)[D]. Yangling: Northwest A&F University, 2006. (in Chinese)
[23] 王珍. 大豆SSR遺傳圖譜構建及重要農藝性狀QTL分析[D]. 南寧: 廣西大學, 2004.
WANG Z. Construction of soybean SSR based map and QTL analysis important agronomic traits[D]. Nanning: Guangxi University, 2004. (in Chinese)
[24] MCCOUCH S R, CHO Y G, YANO M, PAUL E, BLINSTRUB M, MORISHIMA H, KINOSHITA T. Report on QTL nomenclature., 1997, 14: 11–14.
[25] 王金社, 李海旺, 趙團結, 蓋鈞鎰. 重組自交家系群體4對主基因加多基因混合遺傳模型分離分析方法的建立. 作物學報, 2010, 36(2): 191-201.
WANG J S, LI H W, ZHAO T J, GAI J Y. Establishment of segregation analysis of mixed inheritance model with four major genes plus polygenes in recombinant inbred lines population., 2010, 36(2): 191-201. (in Chinese)
[26] JANSEN R C, VAN OOIJIEN J M, STAM P, LISTER C, DEAN C. Genotype-by-environment interaction in genetic mapping of multiple quantitative trait loci., 1995, 91: 33-37.
[27] HAGIWARA W E, ONISH K, TAKAMURE I, SANO Y. Transgressive segregation due to linked QTLs for grain characteristics of rice., 2006, 150: 27-35.
[28] WANG Y, CHENG L, LENG J, WU, C, SHAO G, HOU W, HAN T. Genetic analysis and quantitative trait locus identification of the reproductive to vegetative growth period ratio in soybean ((L.) Merr.)., 2015, 201(2): 275-284.
[29] LIANG H, YU Y, WANG S, LIAN Y, WANG T, WEI Y, GONG P, LIU X, FANG X, ZHANG M. QTL mapping of isoflavone, oil and protein contents in soybean (L. Merr.) [D]., 2010, 9(8): 1108-1116.
[30] 梁慧珍, 余永亮, 楊紅旗, 許蘭杰, 董薇, 牛永光, 張海洋, 劉學義, 方宣鈞. 大豆異黃酮及其組分含量的遺傳分析與QTL檢測. 作物學報, 2015, 41(9): 1372-1383.
LIANG H Z, YU Y L, YANG H Q, XU L J, DONG W, NIU Y G, ZHANG H Y, LIU X Y, FANG X J. Genetic analysis and QTL mapping of isoflavone contents and its components in soybean., 2015, 41(9): 1372-1383. (in Chinese)
[31] RIZAL G, KARKI S, WANG Y, CHENG L, LENG J, WU C, SHAO G, HOU W, HAN T. Genetic analysis and quantitative trait locus identification of the reproductive to vegetative growth period ratio in soybean ((L.) Merr.)., 2015, 201(2): 275-284.
[32] RIZAL G, KARKI S. Alcohol dehydrogenase (ADH) activity in soybean ((L.) Merr.) under flooding stress., 2011, 2(1): 50-57.
[33] 梁慧珍, 董薇, 許蘭杰, 余永亮, 楊紅旗, 譚政偉, 許陽, 陳鑫偉. 不同氮磷鉀處理大豆苗期主根長和側根數的QTL定位分析. 中國農業科學, 2017, 50(18): 3450-3460.
LIANG H Z, DONG W, XU L J, YU Y L, YANG H Q, TAN Z W, XU Y, CHEN X W. QTL mapping for main moot length and lateral root number in soybean at the seedling stage in different N, P and K environments., 2017, 50(18): 3450-3460.
[34] DU W, YU D, FU S. Analysis of QTLs for the trichome density on the upper and downer surface of leaf blade in soybean [(L.) Merr.]., 2009, 8(5): 529-537.
[35] 梁慧珍, 余永亮, 楊紅旗, 張海洋, 董薇, 崔暐文, 鞏鵬濤, 方宣鈞. 幼苗期大豆根系性狀的遺傳分析與QTL檢測. 中國農業科學, 2014, 47(9): 1681-1691.
LIANG H Z, YU Y L, YANG H Q, ZHANG H Y, DONG W, CUI W W, GONG P T, FANG X J. Genetic and QTL analysis of root traits at seedling stage in soybean [(L.) Merr.]., 2014, 47(9): 1681-1691. (in Chinese)
[36] 李海燕. 大豆維生素E含量的遺傳分析及QTL定位[D]. 哈爾濱: 東北林業大學, 2010.
LI H Y. Genetic and QTL analysis of the content of vitamin E in soybean[D]. Harbin: Northeast Forestry University, 2010. (in Chinese)
[37] 方宣鈞, 吳為人, 唐紀良. 作物DNA標記輔助育種. 北京: 科學技術出版社, 2001.
FANG X J, WU W R, TANG J L.. Beijing: Science Press, 2001. (in Chinese)
[38] SONG K, SLOCUM M K, OSBORN T C. Molecular marker analysis of genes controlling morphological variation in(syn. campestris)., 1995, 90: 1-10.
[39] LI Z, PINSON S R M, STANSEL J W. Identification of quantitative trait loci (QTLs) for heading date and plant height in cultivated rice (L.)., 1995, 91: 374-381.
Inheritance and QTL mapping for α-tocopherol in Soybean
LIANG HuiZhen1, YU YongLiang1, XU LanJie1, YANG HongQi1, DONG Wei1, TAN ZhengWei1, LI Lei1, PEI XinYong2, LIU XinMei1
(1Sesame Research Center, Henan Academy of Agricultural Sciences, Zhengzhou 450002;2Institute of Agricultural Economy and Information, Henan Academy of Agricultural Sciences, Zhengzhou 450002)
【Objective】 Inheritance and main QTL for α-tocopherol were detected by genetic analysis and QTL mapping. the results lay a genetic foundation for the selection of soybean varieties with high α-tocopherol content in soybean.【Method】The 447 RILs were derived from a cross between Jindou23 of commercial cultivar as the female parent and Huibuzhi of farm variety from Shanxi Province (ZDD02315) as the male parent that construct SSR genetic linkage map. The parent lines and the RILs were cultivated in summer at Yuanyang testing ground of Academy of Agricultural Sciences, and in Winter at Sanya of Hainan province in 2011, 2012, 2015. A complete random design with two replications was used in this study. Each plot of a single genotype provided 15.00 g big-plump seeds with same size in six environmental conditions. α-tocopherol content was detected quantitatively and qualitatively by High Performance Liquid Chromatography (HPLC). Major gene plus polygene mixed inheritance and QTL mapping for α-tocopherol were detected by major gene plus polygene mixed inheritance analysis and composite interval mapping with WinQTLCart 2.5.【Result】The results showed that α-tocopherol was controlled by four pairs of main genes by major gene plus polygene mixed inheritance analysis. and the four pairs of main genes distributed in two parents. Three main genes shared the same direction with positive additive effect and involved novel alleles from the same parent, Jindou23; one main gene has negative additive effects and donated by Huibuzhi of black beans. Three pairs of genes shared the different direction of positive or negative epistatic effects shared the different direction to α-tocopherol contribution. The phenotypic variation explained by QTL by environment interaction ranged from 0.13 to 4.05%, and indicated that α-tocopherol was significantly affected by four pairs of main genes, more than by environment. Seventeen QTLs for α-tocopherol were mapped on 8 chromosomes 1, 2, 5, 6, 8, 14, 16, and 17, separately; the variation accounted for by each of these seventeen QTLs ranged from 8.35% to 35.78%; and QTL showed additive effect.was all located in marker intervals between Satt320-Satt254 (19.79 cM) on chromosomes 1 in four environmental conditions of 2011 at Yuanyang, 2012 at Yuanyang and Sanya, and 2015 at Yuanyang. and explained 12.55%, 12.01%, 11.89%, 12.61% of the phenotypic variation. It had an additive effect of 0.119-0.132 donated by Jinbean23.was all located in marker intervals between Sat_129-Satt377 (44.53 cM) on chromosomes 8 in three environmental conditions of 2011 at Yuanyang and Sanya, 2015 at Yuanyang. and explained 23.18%, 22.56%, 23.01% of the phenotypic variation. It had an additive effect of -0.195--0.180 donated by Huibuzhi. qand qcan be stably expressed in different genetic backgrounds.【Conclusion】α-tocopherol was controlled by four pairs of additive epistatic effect major genes genetic model (4MG-AI), and it less affected by environmental factor. The two stable main QTL of Satt320-Satt254 and Sat_129-Satt377 were co-localization marker intervals in soybean.
soybean; α-tocopherol; major gene plus polygene; genetic mechanisms; QTL
10.3864/j.issn.0578-1752.2019.01.002
2018-06-25;
2018-08-07
國家現代農業產業技術體系建設專項資金(CARS-21)、河南省藥用植物遺傳改良創新型科技團隊、國家農業科研杰出人才及其創新團隊(特種油料品質改良)、河南省科技攻關計劃(182102310062)、河南省重大科技專項(181100110300)
梁慧珍,Tel:0371-65751589;E-mail:lhzh66666@163.com
(責任編輯 李莉)

