CaMV35S啟動子及其在轉基因作物中的應用和檢測
湯 婷,謝實龍,祝 旋,徐俊鋒,唐 俊,汪小福,*
(1.阜陽師范學院 生物與食品工程學院,安徽 阜陽 236037; 2.浙江省農業科學院 農產品質量標準研究所,浙江 杭州 310021)
從1996年美國最早開始商業化生產和銷售轉基因作物開始,轉基因作物的商業化在爭論中得到迅速發展。目前,共有40個國家開展了轉基因作物的商業化種植。而在轉基因作物中,外源目的基因被整合進宿主細胞時,1個能夠高效啟動目的基因表達的啟動子是必不可少的。啟動子是RNA聚合酶能夠識別并與之結合,從而促使基因轉錄的一段DNA序列,是決定基因表達時間、部位、強度等的主要調控元件,是完成基因表達的表達元件中至關重要的一部分,通常位于基因上游。一個典型的啟動子包括CAAT-box和TATA-box,它們分別是依賴DNA的RNA聚合酶的識別和結合位點,一般位于轉錄起始位點上游幾十個堿基處。在核心啟動子上游通常會有一些特殊的DNA序列,即順式作用元件,轉錄因子與之結合從而激活或抑制基因的轉錄。一旦RNA聚合酶定位并結合在啟動子上即可啟動基因轉錄,因此,啟動子是基因表達調控的重要元件,它與RNA聚合酶及其他蛋白輔助因子等反式作用因子的相互作用是啟動子調控基因轉錄的實質。根據啟動子的轉錄模式可將其分為3類:組成型啟動子、組織或器官特異性啟動子和誘導型啟動子[1-2]。其中,組成型啟動子能在所有組織中都啟動基因表達,具有持續性,不表現時空特異性,RNA和蛋白質表達量也是相對恒定的。從結構上看,大多數組成型啟動子轉錄起始位點上游幾百個核苷酸處,都存在核苷酸序列TGACTG[3]。基因工程中,花椰菜花葉病毒(CaMV35S)啟動子被廣泛地使用,其能夠在許多植物物種的幾乎所有發育階段及所有組織中高效表達,被廣泛應用于轉基因植株,如玉米、棉花、大豆、番茄等的構建[4]。
1 CaMV35S啟動子簡介
1.1 結構
花椰菜花葉病毒(Cauliflowermosaicvirus,CaMV)是一種感染十字花科成員的DNA病毒。該病毒長度大約8 kb,它的完整核苷酸序列已經被確定[5]。在病毒的生命周期中,35S啟動子開啟病毒DNA的轉錄,產生一個8 kb的轉錄本,稱為35S RNA[6]。35S啟動子起始于CaMV 35S RNA轉錄起始點-941至+208的Bg l Ⅱ片段,在位于CaMV 35S DNA中的-46至-105區段存在增強子序列,該啟動子包括TATA盒、倒轉重復序列和CAAT盒、核心序列4個部分(圖1-A)為CaMV35S序列[7]。1985年,Odell等[8]首次展示了CaMV35S的結構和功能,Jefferson等[9]和Sanders等[10]利用CaMV35S啟動子測量了其在轉基因植物葉莖根花中對報告基因的轉錄促進能力。Odell等[8]認為,CaMV35S啟動子啟動基因轉錄要依賴其周圍臨近序列,包括一個TATA框,然而,轉錄的速度卻由其上游大約300 bp的分散DNA 序列決定。Simpson等[11]認為,這個區域是一個增強子(能夠提高轉錄速率的DNA序列)。Odell等[8]的研究表明,-343至-46的上游片段是35S啟動子的主要部分。在之后的研究中,Fang等[12]發現35S啟動子可以劃分為2個區域:從-90 bp至+8 bp為A區域,主要負責基因在胚根、胚乳及根組織內表達;區域B(-343至-90 bp)主要控制基因在胚的子葉、成熟植株的葉組織及維管組織內表達。圖1-B展示了CaMV35S序列劃分的不同區域。在B區域內的增強子序列可以提高表達水平,若是刪除-343至-208區域,基因的轉錄強度減少50%。如果35S啟動子中存在2個B區將使35S啟動子活性提高10倍,對異源啟動子也有作用,但B區對某些啟動子起表達阻遏因子作用。

A,35S啟動子從-343到+8的DNA序列,填充三角形表示子域的斷點位置。在方框中,位置-99是ORF VI的終止密碼子 (TGA),另外2個TGACG和TATA序列。B,CaMV35S的區域B、區域A和TATA盒的示意圖。A, The DNA sequence of 35S promoter from-343 to+8, the break point position of the subdomain represented by the triangle. In the box, position-99 was the termination codon (TGA) of ORF VI, and the other two boxes were TGACG and TATA sequences, respectively. B, Schematic diagram of area B, area A and TATA boxes in CaMV35S.圖1 CaMV35S序列及其劃分區域Fig.1 CaMV35S sequence and its partition region
1.2 CaMV35S啟動子的功能
20世紀80年代初,Chua和洛克菲勒大學的合作者在感染了花椰菜花葉病毒(CaMV)的植物中分離出了一種啟動子,該啟動子負責花椰菜花葉病毒(CaMV)的整個基因組的轉錄表達,它的沉降系數是35S,因此,這個啟動子被命名為CaMV35S啟動子,它是目前廣泛使用的組成型啟動子之一。已被實驗證實的組成型病毒啟動子有2種,分別為19S和35S。其中,19S啟動子負責轉錄CaMV病毒部分基因,但是沒有35S應用廣泛。35S RNA,除了作為翻譯的模板,它的直接末端重復序列,通過逆轉錄酶也可以作為病毒DNA合成的中間產物[13]。另外,Guilley等[6]研究表明,在受感染的細胞中,病毒快速復制需要大量的35S RNA,35S RNA的高水平生產證明了這個啟動子的強度。Kuhlemeier等[14]根據35S轉錄起始位點的信息及克隆CaMV序列的可用性,鼓勵使用35S啟動子作為植物基因表達研究的模型系統。Morelli等[15]和Nagy等[16]對轉基因煙草細胞或轉基因矮牽牛花[17]相對轉錄水平的定量測定顯示,35S啟動子比NOS啟動子強至少30倍。同時,CaMV35S啟動子具適度的天然啟動能力。因此,在轉基因植物中,CaMV35S啟動子被大量地用于外源基因調控。實驗表明,將包含35S上游序列400~1 000 bp的CaMV片段插入煙草[18-19]或是矮牽牛花[17]的基因,可以顯示出活性。此外,啟動子還可以在幾個雙子葉植物和單子葉植物的原生質體中表達[20-23]。
1.3 CaMV35S啟動子的改造
在植物轉基因研究中,使用天然啟動子往往不能得到滿意的結果,尤其是在特異表達或誘導表達時,表達水平不夠理想。因此,選擇合適的啟動子或對啟動子進行改造,是增強外源基因特異表達的重要手段。Lv等[24]將35S最小啟動子結合在特異表達啟動子GRPp的5′端形成雙向啟動子,這些雙向啟動子能夠指導GUS和GFP基因在所有獨立的轉基因煙草的維管特異表達。轉基因植物的維管特異性雙元雙向啟動子可用于同時調控某些維管特異性功能基因的表達。這一方法還可用于其他植物中極性啟動子的生物技術研究。張秀春等[25]利用植物防御基因中的病原誘導響應元件和最小35S啟動子(-62至+1),人工合成了啟動子SAR,并以GUS基因為報告基因,在轉基因擬南芥中研究人工啟動子的表達特性。結果表明,SAR啟動子在子葉、毛刺、根莖交接處和根系中優勢表達,在老葉的表達量高于幼葉,說明SAR啟動子具有組織和發育時期表達特異性。彭舒[26]將篩選得到的F-box、GST1-box、S-box、W-box作用元件進行隨機串聯后與35S最小啟動子進行組合,成功構建了人工病原物誘導型啟動子和植物表達載體。Xie等[27]將CaMV35S啟動子上游反向連接了51 bp的Pmini啟動子,改造后的這個啟動子具有雙向表達功能,并且2個方向表達強度相同,同時改造后的雙向啟動子在擬南芥中的遺傳性和穩定性表現良好。
2 CaMV35S啟動子在轉基因作物中的應用及其檢測方法
2.1 應用
自1986年世界上第1例轉基因作物問世以來,轉基因作物迅猛發展[28]。轉基因作物具有抗蟲、抗病、抗逆、高產、優質等優點,使其得到世界范圍的廣泛關注。根據全球農業生物技術應用咨詢署(ISAAA)提供的統計資料,2016年,美國、阿根廷、加拿大等28個以上國家種植了大豆、玉米、棉花、油菜、馬鈴薯等轉基因作物。雖然,轉基因技術給人們帶來了巨大的經濟效益,但轉基因作物及其食品的環境釋放安全性及食用安全性也存在巨大爭議。為保護貿易、明確消費,各國政府均對安全監管提出了更高的要求[29]。作為轉基因作物安全性監管的支撐,轉基因產品檢測技術也成為研究的熱點。常用的轉基因檢測方法主要有基于蛋白質的檢測技術和基于DNA的檢測技術。如酶聯免疫吸附檢測(ELISA)、聚合酶鏈式反應(PCR)、Southern雜交、基因芯片和DNA電化學傳感器技術等。其中,以PCR為主的基于DNA序列的轉基因檢測方法是普遍接受的轉基因檢測技術,其有能力從高度加工的材料中擴增特定的DNA片段,其主要依據是轉基因作物中特異性外源DNA片段。用于植物轉化的外源DNA片段可以分為通用元件、目的基因、外源載體序列3大類,根據不同的外源DNA片段,檢測策略可以分為4個層次:篩選檢測、基因特異性檢測、載體特異性檢測和轉化事件特異性檢測[30]。其中,最常用的一個元件就是CaMV35S啟動子。數據表明,2013年,有來自27種不同物種的336種轉基因作物被商業化[31],隨著越來越多的轉基因作物進入田間試驗,這一數字還在不斷增加,在此過程中,僅開發了部分品種的轉化事件特性檢測方法。一般來說,在轉基因檢測過程中對所有可能發生的轉化事件進行PCR檢測是不可行的。一種常見的做法是對許多轉化事件常見的目標進行篩選,而CaMV35S啟動子就是這樣的一個常見目標。運用轉基因作物數據庫(http://ceragmc.org/index.php?action5gm_crop_database)中可用的轉基因信息,對轉基因作物中CaMV35S和TNOS進行了初步的統計分析,結果見表1。
由表1可知,經過批準的商業轉基因轉化事件的65.7%(67/102)包含了CaMV35S啟動子,53.49%(55/102)包含NOS,以及81.4%(83/102)包含任一個或者兩者。在重要的商業轉基因作物中,CaMV35S和TNOS存在比例更高。在轉基因玉米(Zeamays)的28個商業轉化事件中,只有1個沒有這2個元件,而在16個商業的油菜籽(Brassicanapus)中,只有2個例外。一些轉基因的轉化事件,如轉基因玉米BT11、T25和轉基因棉花MON531,它們的改造結構中包含了2個CaMV35S序列。
2.2 檢測方法
由于CaMV35S啟動子對轉基因作物的檢測具有重要意義,因此,研究者們建立了大量的轉基因篩選方法,其中一些方法已被國際標準化組織(ISO)、歐洲聯盟(歐盟)、中國及其他國家和地區采用,并作為轉基因檢測的標準方法[32-36]。表2列舉了來自不同文獻和檢測標準的25種檢測CaMV35S啟動子方法[37],分別命名為M1~M25。其中,M1被ISO 2156915通過,M13被ISO 2157032通過,M1、M14、M11通過了中國的國家標準,M1、M2、M15、M16通過了中國的工業標準,而M1、M4、M12、M14被歐盟數據庫收集,用于轉基因成分分析。這25種方法包含了10種傳統PCR方法和15種實時定量PCR方法。用于實時定量PCR方法的部分引物對,如M1、M3、M4、M5、M12和M14,也可以用于常規的定性PCR檢測。另外,表2中也列出了每種方法的引物/探針相對于CaMV序列的位置和擴增子的序列大小。CaMV35S啟動子是GMO中最常修改的元件之一,在不同的基因和載體上,由于來源不同的菌株或在載體構建過程中的修飾或突變,CaMV35S啟動子序列可能存在差異[34]。由寡核苷酸序列的比對結果可知,轉化事件176、BT11、T25、MON810和DLL2517中的CaMV35S啟動子序列來源于CaMV基因組[38](圖2)。本課題組收集了玉米、大豆等一些常見轉基因作物的不同轉化事件中的CaMV35S序列,利用Vector NIT軟件將這些序列進行比對,結果顯示,這些轉化事件中CaMV35S序列存在一定差異,所有比對的CaMV35S序列都不完全一致(圖2)。因此,許多基于CaMV35S啟動子的檢測方法可用于轉基因檢測,但是只有部分方法通過了必要的驗證過程和實驗室內對少量的轉基因轉化事件方法驗證。Morisset等[39]在TC1507玉米的CaMV35S序列中發現了一個單核苷酸多態性(SNP),檢測引物/探針的結合位點出現SNP導致CaMV35S擴增效率不高。國際生命科學研究所(ILSI)請求100多個實驗室使用CaMV35S和TNOS來檢測轉基因作物,一些實驗室在CaMV35S測試中遇到了方法學缺陷,如靈敏度低、重現性低、誤報或否定。Holden等[40]用8種玉米參考材料測試了5種基于CaMV35S的方法的適用性,結果表明,2種方法在一些測試材料中存在線性回歸參數的缺陷和多重PCR擴增的缺陷。如果在轉換載體或育種過程中,測試樣品攜帶的CaMV35S序列被改變,那么該方法的差異性可能會導致不同的測試結果。ILSI的調查顯示,大部分參與實驗的實驗室都希望有一種標準化的方法,這種方法可以產生一致的檢測結果,而且實驗的重現性更好。綜合比較現有方法,還沒有最優的方法可用。
表1轉基因作物中含有的CaMV35S和TNOS數據統計
Table1Data statistics of CaMV35S and TNOS included in transgenic crops
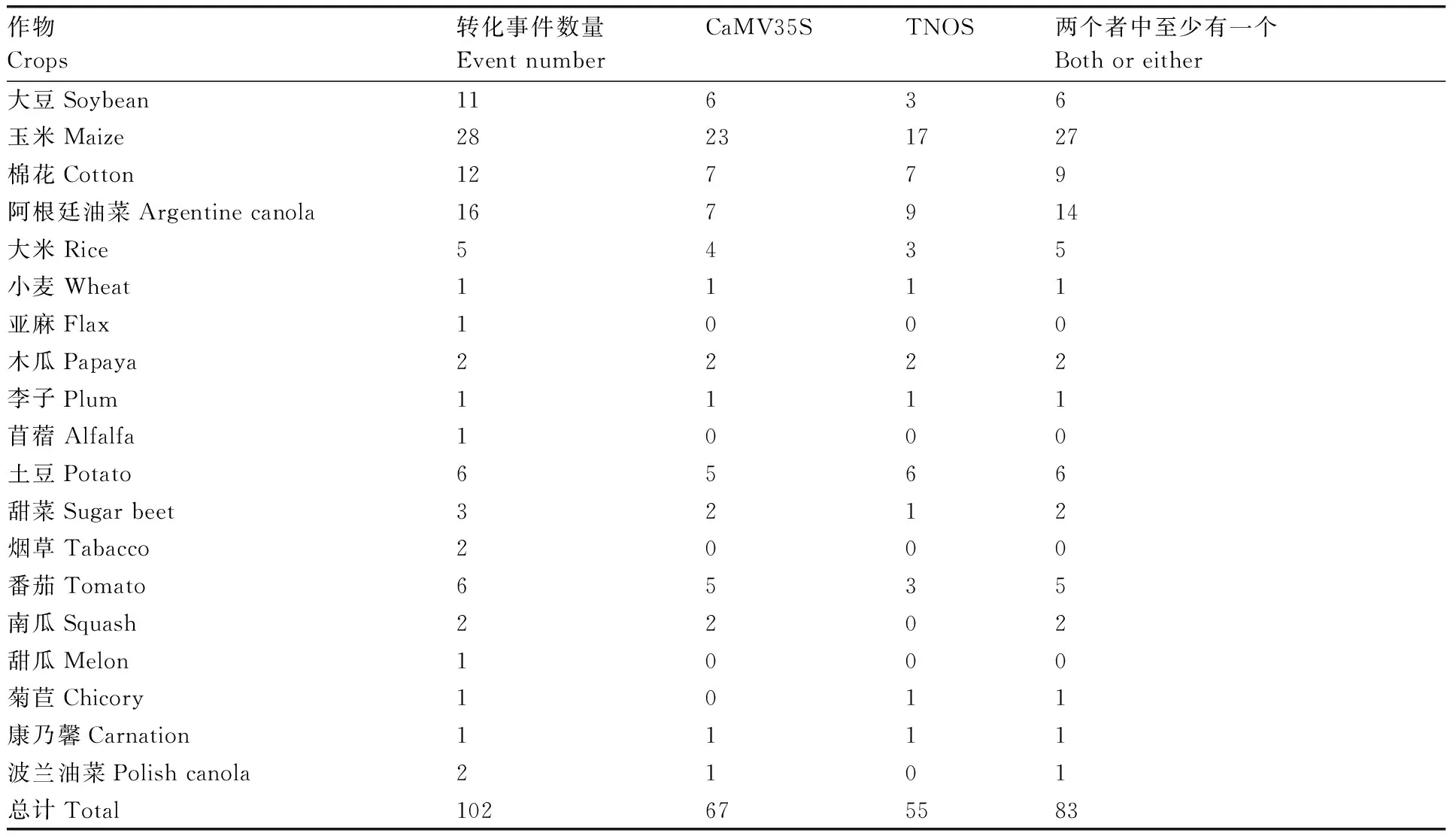
作物Crops轉化事件數量Event numberCaMV35STNOS兩個者中至少有一個Both or either大豆Soybean11636玉米Maize28231727棉花Cotton12779阿根廷油菜Argentine canola167914大米Rice5435小麥Wheat1111亞麻Flax1000木瓜Papaya2222李子Plum1111苜蓿Alfalfa1000土豆Potato6566甜菜Sugar beet3212煙草Tabacco2000番茄Tomato6535南瓜Squash2202甜瓜Melon1000菊苣Chicory1011康乃馨Carnation1111波蘭油菜Polish canola2101總計Total102675583
數據來源于文獻[40].
Data came from literature [40].
表2用于檢測CaMV35S啟動子的定性和實時定量PCR系統中使用的引物和熒光探針
Table2Primers and fluorescent probes used in detection of CaMV35S promoter in qualitative and real-time quantitative PCR systems
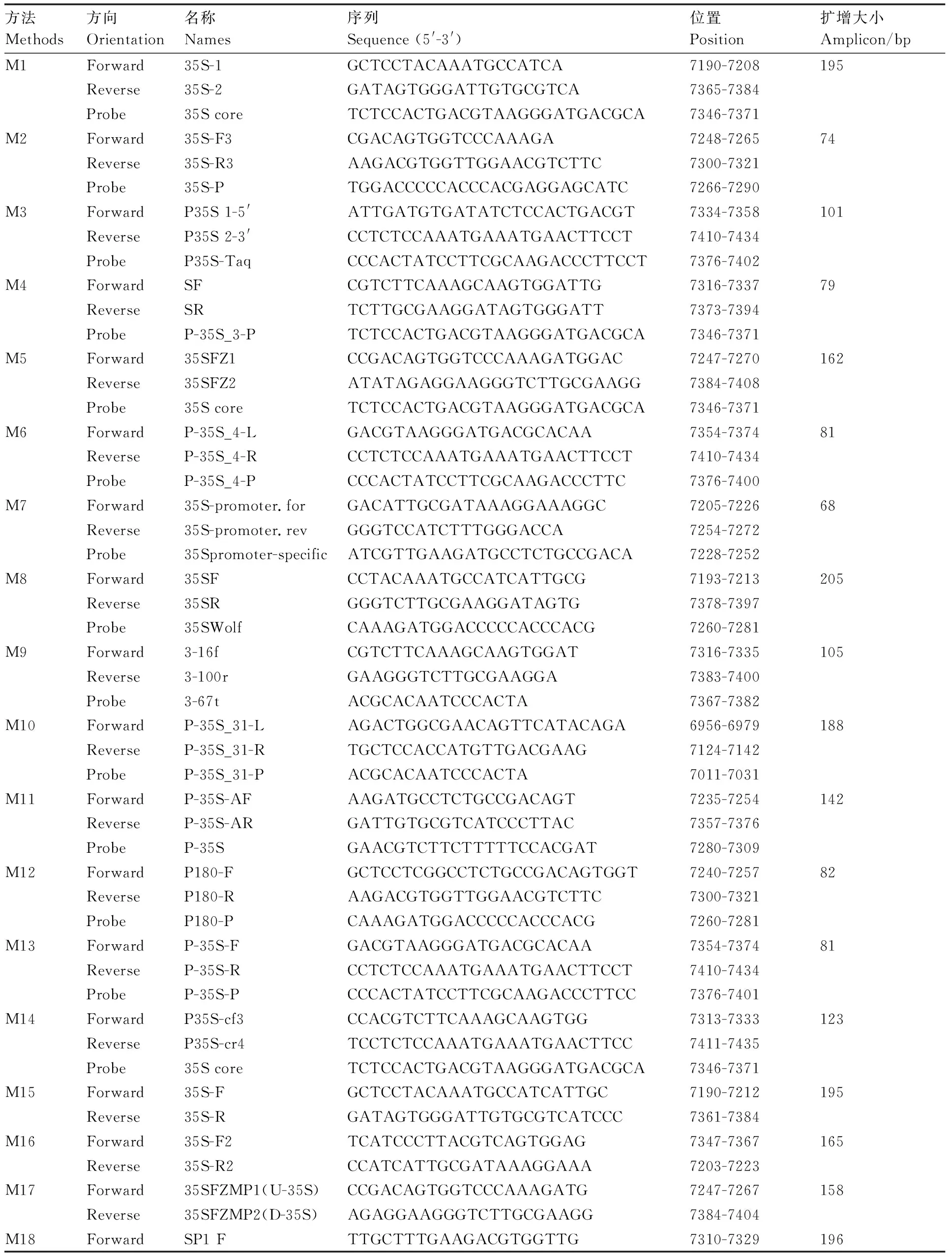
方法Methods方向Orientation名稱Names序列Sequence (5'-3')位置Position擴增大小Amplicon/bpM1Forward35S-1GCTCCTACAAATGCCATCA7190-7208195Reverse35S-2GATAGTGGGATTGTGCGTCA7365-7384Probe35S coreTCTCCACTGACGTAAGGGATGACGCA7346-7371M2Forward35S-F3CGACAGTGGTCCCAAAGA7248-726574Reverse35S-R3AAGACGTGGTTGGAACGTCTTC7300-7321Probe35S-PTGGACCCCCACCCACGAGGAGCATC7266-7290M3ForwardP35S 1-5'ATTGATGTGATATCTCCACTGACGT7334-7358101ReverseP35S 2-3'CCTCTCCAAATGAAATGAACTTCCT7410-7434ProbeP35S-TaqCCCACTATCCTTCGCAAGACCCTTCCT7376-7402M4ForwardSFCGTCTTCAAAGCAAGTGGATTG7316-733779ReverseSRTCTTGCGAAGGATAGTGGGATT7373-7394ProbeP-35S_3-PTCTCCACTGACGTAAGGGATGACGCA7346-7371M5Forward35SFZ1CCGACAGTGGTCCCAAAGATGGAC7247-7270162Reverse35SFZ2ATATAGAGGAAGGGTCTTGCGAAGG7384-7408Probe35S coreTCTCCACTGACGTAAGGGATGACGCA7346-7371M6ForwardP-35S_4-LGACGTAAGGGATGACGCACAA7354-737481ReverseP-35S_4-RCCTCTCCAAATGAAATGAACTTCCT7410-7434ProbeP-35S_4-PCCCACTATCCTTCGCAAGACCCTTC7376-7400M7Forward35S-promoter.forGACATTGCGATAAAGGAAAGGC7205-722668Reverse35S-promoter.revGGGTCCATCTTTGGGACCA7254-7272Probe35Spromoter-specificATCGTTGAAGATGCCTCTGCCGACA7228-7252M8Forward35SFCCTACAAATGCCATCATTGCG7193-7213205Reverse35SRGGGTCTTGCGAAGGATAGTG7378-7397Probe35SWolfCAAAGATGGACCCCCACCCACG7260-7281M9Forward3-16fCGTCTTCAAAGCAAGTGGAT7316-7335105Reverse3-100rGAAGGGTCTTGCGAAGGA7383-7400Probe3-67tACGCACAATCCCACTA7367-7382M10ForwardP-35S_31-LAGACTGGCGAACAGTTCATACAGA6956-6979188ReverseP-35S_31-RTGCTCCACCATGTTGACGAAG7124-7142ProbeP-35S_31-PACGCACAATCCCACTA7011-7031M11ForwardP-35S-AFAAGATGCCTCTGCCGACAGT7235-7254142ReverseP-35S-ARGATTGTGCGTCATCCCTTAC7357-7376ProbeP-35SGAACGTCTTCTTTTTCCACGAT7280-7309M12ForwardP180-FGCTCCTCGGCCTCTGCCGACAGTGGT7240-725782ReverseP180-RAAGACGTGGTTGGAACGTCTTC7300-7321ProbeP180-PCAAAGATGGACCCCCACCCACG7260-7281M13ForwardP-35S-FGACGTAAGGGATGACGCACAA7354-737481ReverseP-35S-RCCTCTCCAAATGAAATGAACTTCCT7410-7434ProbeP-35S-PCCCACTATCCTTCGCAAGACCCTTCC7376-7401M14ForwardP35S-cf3CCACGTCTTCAAAGCAAGTGG7313-7333123ReverseP35S-cr4TCCTCTCCAAATGAAATGAACTTCC7411-7435Probe35S coreTCTCCACTGACGTAAGGGATGACGCA7346-7371M15Forward35S-FGCTCCTACAAATGCCATCATTGC7190-7212195Reverse35S-RGATAGTGGGATTGTGCGTCATCCC7361-7384M16Forward35S-F2TCATCCCTTACGTCAGTGGAG7347-7367165Reverse35S-R2CCATCATTGCGATAAAGGAAA7203-7223M17Forward35SFZMP1(U-35S)CCGACAGTGGTCCCAAAGATG7247-7267158Reverse35SFZMP2(D-35S)AGAGGAAGGGTCTTGCGAAGG7384-7404M18ForwardSP1 FTTGCTTTGAAGACGTGGTTG7310-7329196
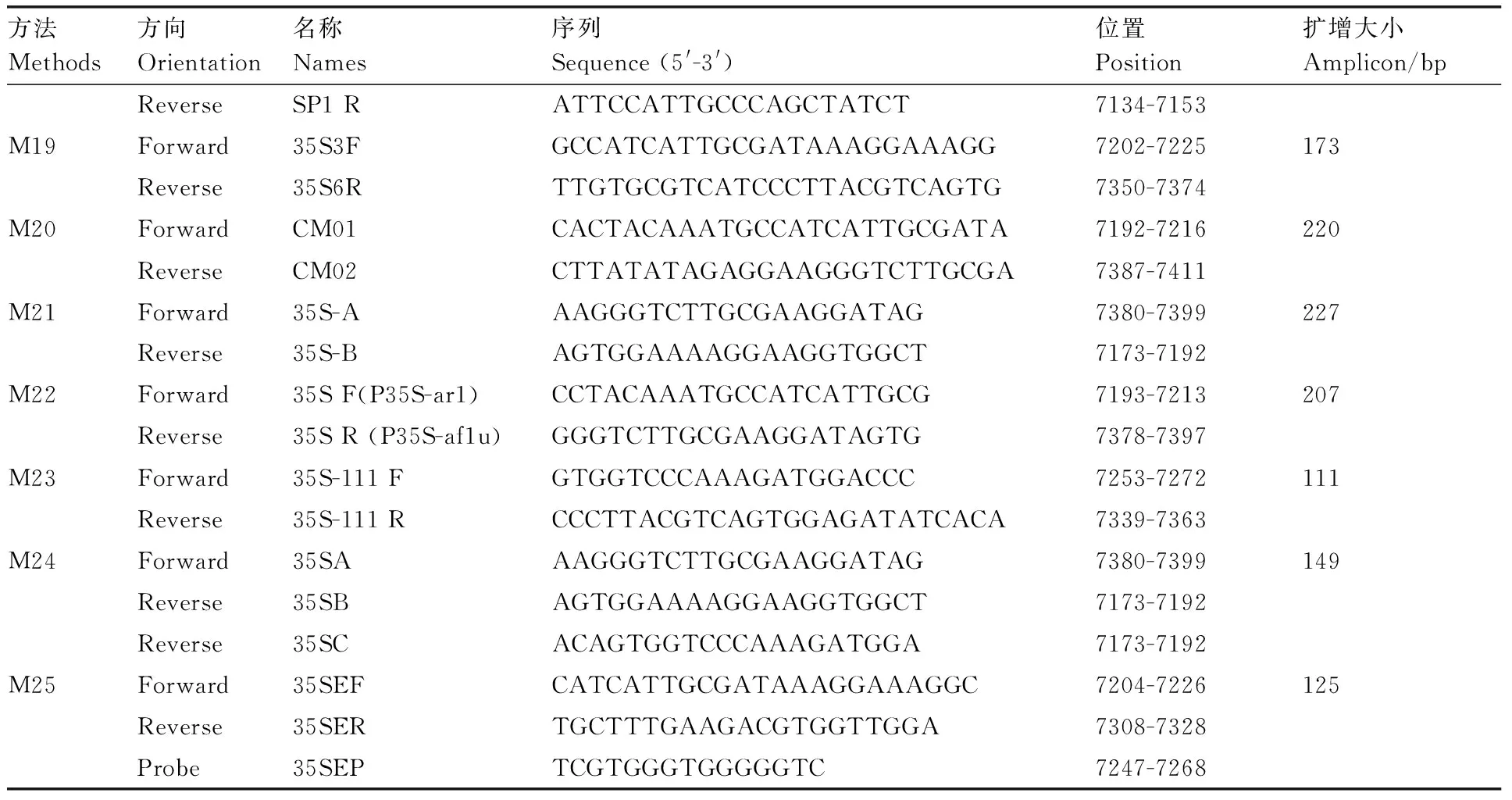
續表2
目前,針對CaMV35S的轉基因成分快速篩查檢測,迫切需要找到不同CaMV35S序列中的保守序列,根據這這段保守序列,設計相應的引物進行檢測,能夠同時滿足不同轉基因作物中CaMV35S的檢測需要,避免由于CaMV35S序列差異而造成的不同檢測結果,從而建立檢測CaMV35S的統一方法。

圖2 十七個轉化事件中CaMV35S序列比對結果Fig.2 Alignment results of CaMV35S sequence in 17 conversion events
3 展望
轉基因食品及其安全問題近年來已經成為全球關注的熱點[41-43],世界各國紛紛出臺政策,要求對轉基因產品進行限量標識。我國于2001年和2002年先后發布了《農業轉基因生物安全管理條例》《農業轉基因生物標識管理辦法》等配套管理辦法,明確規定對進入我國流通市場的轉基因生物及產品實施標識制度。目前,基于DNA的PCR技術是轉基因成分檢測的最主要手段。根據檢測目標及特異性的差異可將PCR技術分為:篩查檢測、基因檢測、構建檢測及轉化體特異性檢測。其中,篩查檢測的目標主要是轉基因作物中常用的一些共用元件,如啟動子、終止子及標記基因,雖然其特異性不強,但因為針對的是共用元件的檢測,其檢測的覆蓋率最高。在日常檢測中,面對大量的檢測樣品,篩查檢測是最主要的前期檢測方法,CaMV35S啟動子是轉基因成分篩查檢測的主要元件之一。
目前,檢測CaMV35S啟動子的PCR方法中存在以下問題:首先,針對CaMV35S啟動子的檢測引物繁多,只有一部分是來自ISO標準,或相應國家或地區的標準,經過了實驗室研究和實驗之間的驗證過程,多數引物是各個實驗室內部開發的;這些引物針對的CaMV35S啟動子序列不同,所以會產生不一致的檢測結果,從而給轉基因成分檢測的結果及后續的監測和監管造成巨大影響;尤其是處在全球貿易的今天,針對進出口產品的檢測結果差異會造成一定的貿易爭端。其次,面對這些多而雜的檢測方法,給檢測者造成了選擇困難;針對不同的檢測樣品,在眾多方法中選擇一種簡便準確的檢測方法具有一定的難度。所以,建立標準化的CaMV35S啟動子檢測方法很重要。但是,由于目前檢測機構或研究室之間的實驗數據還沒有做到全面共享和互用。目前,還沒有一個數據庫包含了目前所有轉基因作物的全部序列信息,包括CaMV35S啟動子序列信息,因此很難建立一種標準化的檢測方法。在今后的研究中,針對CaMV35S啟動子檢測方法的研究,相關轉基因作物的序列信息及檢測數據需要進行交流和共享,從而使檢測人員了解相應轉化體中CaMV35S啟動子的應用情況,以及設計的檢測引物的覆蓋率。同時各個檢測機構或實驗室之間需要展開數據共享與共同驗證,建立針對CaMV35S啟動子的相對統一的標準檢測方法。

