陸海高代回交群體抗黃萎病QTL定位
郭志軍,趙云雷,陳偉,王紅梅,龔海燕,桑曉慧,崔艷麗,趙佩
(中國農業科學院棉花研究所/棉花生物學國家重點實驗室,河南安陽455000)
我國棉花黃萎病是由大麗輪枝菌引起的土傳維管束病害,實際生產中很難徹底根治,已成為我國棉花生產中最嚴重的病害之一,給棉花生產造成了極大損失[1-2]。長期棉花生產實踐表明,選育并種植抗病品種是最有效的防治措施。然而,由于陸地棉栽培種抗源匱乏,而且傳統育種方法選擇效率低、周期長,抗病育種進展較慢。通過連鎖分析定位與抗病基因緊密連鎖的分子標記,進而用于分子標記輔助選擇育種,可有效加快抗病新品種培育進程[3-5]。
目前,國內外開展了大量的棉花抗黃萎病QTL(Quantitative trait loci)定位方面的研究,定位群體也逐漸由陸陸群體和初級群體向陸海群體和高級群體演變,并取得了很大的進展,定位了大量的抗病QTL[6-10]。但直接應用于抗病遺傳改良并應用于生產實踐的尚未見報道,因此需要進一步發掘和鑒定抗黃萎病QTL,以加快分子標記輔助選擇抗病育種進程。本研究通過構建以陸地棉栽培種魯棉研28為遺傳背景的陸海高代回交群體,定位與黃萎病抗性相關的QTL,以期為分子標記輔助選擇育種提供標記。
1 材料與方法
1.1 試驗材料
選用高抗黃萎病海島棉品種“海7124”和高感黃萎病陸地棉標準系 “TM-1”配制抗感組合F1,再以陸地棉主栽品種魯棉研28為輪回親本進行連續回交、自交,最終分別得到137個BC4F1和BC4F2:3株系。
1.2 試驗設計
2013年分別在河南安陽中國農業科學院棉花研究所病圃(以下簡稱“病圃”)和試驗農場東場(以下簡稱“大田”),對 137 個 BC4F2:3株系進行抗病性鑒定。病圃環境下,設3次重復,單行區,行長8 m,株距0.25 m,0.8 m等行距,隨機區組排列。病圃是人工感染的水泥池,長為20 m,寬為2.5 m,所接病原菌為具中等致病力的安陽菌系。每個病池均設抗病對照和感病對照,分別以中植棉2號和冀棉11為抗病和感病對照。大田環境下,設3次重復,兩行區,6 m行長,株距0.25 m,0.8 m等行距,隨機區組排列,其他管理同大田。
大田環境下于9月12號、9月25號,病圃環境下于8月5號、8月28號,按照全國統一病情分級標準GB/T 22101.5―2009《葉棉花抗病蟲性評價技術規范第5部分:黃萎病》[11](詳見表1)進行病害調查,計算病情指數和相對(矯正)病情指數,并采用Excel 2010統計分析抗病鑒定結果,并進行正態分布檢驗。
病情指數=(∑級數×每級的病株數/調查總株數×4)×100;
相對病情指數=實測病指×K.
其中K值是校正系數,用全國統一規定的感病對照病情指數50.0,除以本期鑒定感病對照病指實測值。

表1 棉花黃萎病Ⅴ級劃分標準Table 1 Grading stand for cotton Verticillium wilt with five levels
1.3 分子檢測
采用改良的CTAB法提取海7124、TM-1和魯棉研28以及BC4F1群體的DNA[12]。利用2 010對SSR引物進行多態性檢測,再利用多態性引物對BC4F1群體進行擴增檢驗。PCR(Polymerase chain reaction)擴增體系、反應程序和聚丙烯酰胺凝膠電泳參照張軍等[13]的方法。
1.4 遺傳圖譜構建及QTL定位
參考Yu Jing等[14]構建的整合遺傳連鎖圖譜,將在親本間和群體上均表現出多態性的Sim-ple sequence repeats(SSR)標記直接錨定在染色體上,用Mapchart2.2生成遺傳連鎖圖。
采用WinQTLCart V2.5[15]軟件以復合區間作圖法檢測QTL,設置步長為1 cM(centi Morgan),用似然比值即LR值進行顯著性檢驗,當LOD≥2.5時,則認為存在QTL。QTL命名參照本課題已有的命名方法[16]。
2 結果與分析
2.1 多態性SSR引物篩選以及在BC4F1群體中擴增效果
采用2010對SSR引物進行親本間多態性篩選,挑選擴增條帶清晰而且在海7124和TM-1、魯棉研28之間具有多態性的引物,最終共篩選出494對多態性SSR引物,多態性比率約為24.6%。繼而,采用494對多態性SSR引物對BC4F1群體進行擴增檢驗,494對多態性引物中僅有243對引物具有群體多態性,占多態性引物的比率約為50%。
2.2 遺傳圖譜的構建
將243對具有群體多態性的SSR引物與Yu Jing等[14]構建的整合遺傳連鎖圖譜進行比對,直接將標記定位到相應的染色體上,并采用Mapchart2.2軟件繪制遺傳連鎖圖(圖1)。最終共將216個多態性SSR標記錨定在26條染色體上,另有27個標記未能進入連鎖群。每條染色體上多態性標記分布2~25個不等,其中Chr3、Chr10、Chr12、Chr13等四條染色體上標記最少只有2個,第16條染色體上標記最多為25個。采用進入連鎖群的216個多態性位點繪制連鎖圖,圖譜可覆蓋棉花基因組全長3 880 cM,標記間平均距離15.77 cM。

圖1 整合遺傳連鎖圖譜和定位的QTL在圖譜上的圖示Fig.1 The integrated genetic linkage map and QTL mapped in the linkage map

圖1(續)Fig.1 (Continued)
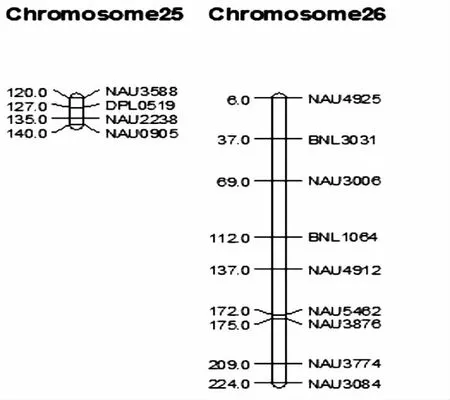
圖1(續)Fig.1 (Continued)
2.3 不同試驗條件下病情指數變異
棉花黃萎病抗性屬數量性狀,由微效多基因控制,易受環境影響。數量性狀表現型是基因型與環境互作的結果,應服從正態分布。因此,在進行QTL定位前,首先要對群體表型進行正態分布檢驗。對大田和病圃不同調查時期的BC4F2:3家系病情指數進行正態分布統計,結果表明大田和病圃兩個環境下,只有病圃環境下8月28日調查時期下BC4F2家系的峰度略大于1,其余時期的BC4F2家系病指斜度和峰度絕對值均小于1,服從正態分布,調查結果可用于QTL定位(表 2)。
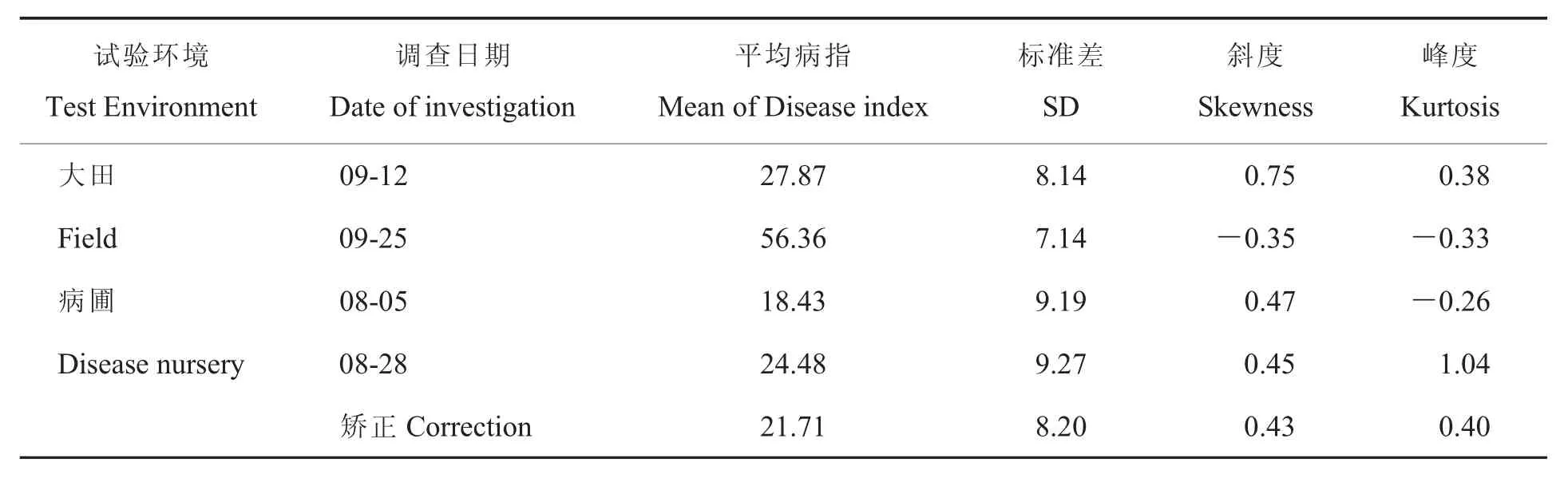
表2 BC4F2家系病指基本統計分析Table 2 The descriptive analysis of the disease index to Verticillium wilt by the BC4F2families
2.4 QTL定位分析
采用WinQTLCart V2.5軟件以復合區間作圖法定位QTL,共定位到6個與黃萎病抗性相關的 QTL(表 3)。
大田環境下,在9月12日這一時期檢測到2個 QTLs即 qnV14-2和 qnV14-3,2個 sQTL均位于第14條染色體上,均與分子標記NAU3312相連鎖,貢獻率分別為19.98%和14.84%;大田9月25日調查時期未檢測到QTL。
病圃環境下,共定位到4個QTLs。其中在8月5日這一時期定位到2個QTLs即qdV1-1和qdV15-1,分別位于Chr1和Chr15染色體上,分別與分子標記NAU5107和NAU3346相連鎖,貢獻率分別為14.15%和20.26%;病圃8月28日調查時期未檢測到QTL。
采用相對病情指數,也定位到2個QTLs即qdV5-1和qdV14-1,分別位于Chr5和Chr14染色體上,分別與分子標記JEPPR0050和NAU3913相連鎖,貢獻率分別為8.56%和11.11%。
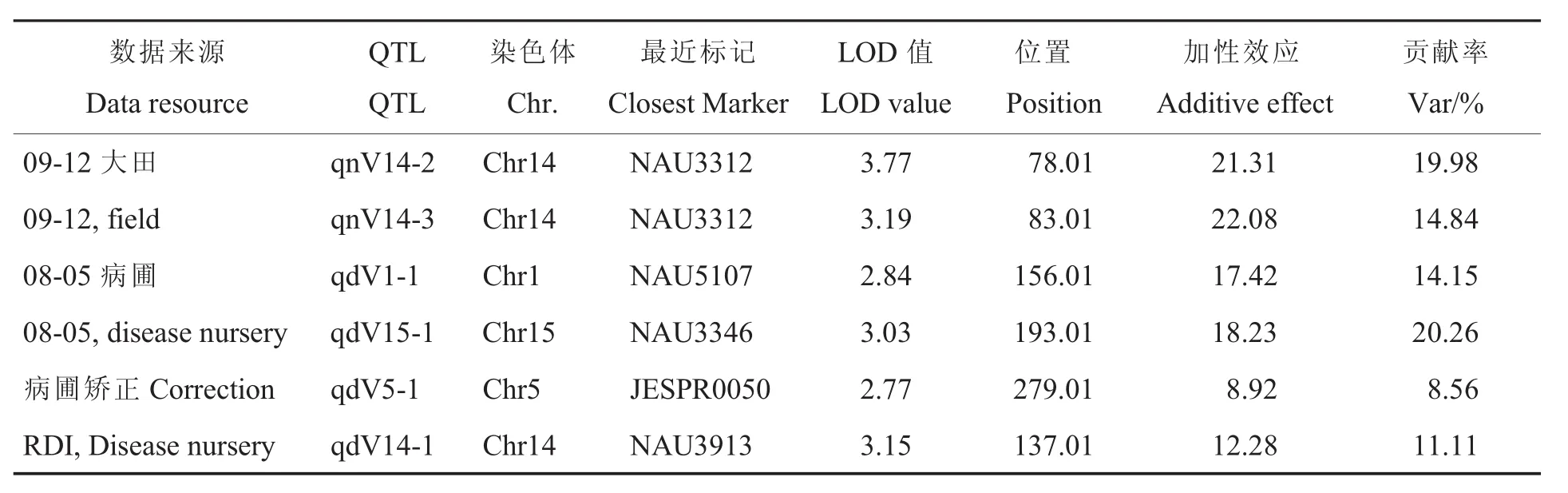
表3 復合區間作圖法定位抗黃萎病QTLTable 3 Mapping QTL for Verticillium wilt resistance by composite interval mapping method
3 討論
3.1 關于作圖群體的選擇
作圖群體不僅是構建遺傳連鎖圖的根本,也是QTL定位的基礎,其中構建群體時所選擇的親本及群體的類型是決定群體優劣的重要因素。本研究首先選用高抗黃萎病的海島棉品種海7124和高感黃萎病的陸地棉標準系TM-1做抗、感親本,配制雜交組合F1,以確保分離后代的黃萎病抗性表現型具有明顯的差異。同時,為了創制能夠更好的應用于生產實踐的抗病新品系,自F1之后將輪回親本更換為生產上綜合性狀優良、大面積推廣的陸地棉栽培種魯棉研28。經4輪回交,BC4F1群體遺傳背景大都趨近于魯棉研28,可有效地削弱QTL與遺傳背景的互作效應,可提高定位的精度。但是連續回交,也造成作圖時標記偏分離現象嚴重,前人研究中同樣出現[17-19]。偏分離位點的存在勢必會對作圖的精度造成一定的影響,然而若僅僅是簡單的刪除這些偏分離位點,將會導致遺傳連鎖群覆蓋率偏低,也會遺漏很多重要的信息。因此采用較高的LOD值或采用比較作圖或抽樣群體作圖是處理偏分離標記的比較好的方法[20]。本研究在采用較高LOD值時能夠進入連鎖群的標記較少,且大多數標記被定位在同一連鎖群上,究其原因,主要在于經連續回交,BC4F1群體所攜帶的海島棉片段很少,因此參考Yu Jing等[14]的高密度遺傳連鎖圖來定位QTL。
近年來,陸海漸滲系逐漸被應用到棉花相關性狀的QTL定位。戎福喜等[21]采用陸海漸滲系定位了與棉花吐絮期葉綠素含量、熒光參數及相關性狀的QTL,共計44個。趙君等[10]采用染色體片段代換系定位了一個抗病主效QTL,位于D4染色體,可解釋表型變異64.8%。本研究中,對BC4F1群體進行擴增時發現,BC4F1群體中只滲透有少數的海島棉染色體片段,最少的為1個,最多的為38個,雜合居多,純合的海島棉染色體片段僅有12個,而且其中7個片段聚合在同一份材料中。因此,本群體可為今后開展以陸海漸滲群體或CSSL群體抗病QTL精細定位奠定基礎。
3.2 QTL定位
棉花黃萎病是一種土傳真菌維管束病害,其發生和發展受環境影響很大,因此,抗病性鑒定準確性至關重要。為提高試驗精準度,本研究設置大田和病圃兩個環境進行抗病性鑒定。
采用復合區間作圖法在兩個環境下共檢測到6個QTLs,其中大田環境下2個,病圃環境下4個。大田環境下檢測到的QTLs相對較少,可能與2013年氣溫偏高有關,氣溫偏高在一定程度上會抑制黃萎病害的發生和發展,對鑒定結果有一定的影響。定位到的6個QTLs中除qdV5-1以外,其余5個QTLs貢獻率均大于10%。其中,QTL qdV15-1位于第15條染色體上,張保才[22]定位 到 的 QTLs DImean-1-BC2F1、DIf-1-BC1S1 和DI825-1-BC2F1以及王沛政[23]定位到的QTL qVW-Chr.15-1同樣位于第15條染色體上。病圃環境下定位到的QTL qdV5-1與張保才[22]定位的DImean-3-BC2F1、DIf-5-BC2F1以 及 楊 昶[24]定 位 的qVV-A5-1RIL592、qVL-A5-1RIL592 和 qVL-A5-1RILBP2和寧志怨[25]定位的qVW-A5-1均位于第5條染色體上。本研究中在大田和病圃兩個環境下,在第14條染色體上共檢測到3個QTLs分別為 qnV14-2、qnV14-3、 qdV14-1,張保才[24]在第14條染色上檢測到 2個 QTLs DIf-4-BC2F1、DIf-3-BC2F1,同時寧志怨[25]也在第 14條染色體(D2)上檢測到 QTL qVWD2-1。同時,本研究在病圃條件下,在第1條染色體上新檢測到1個QTL qdV1-1。
由于不同的QTL定位研究所采用的材料、標記均有所不同,因此,不同研究之間所共有的標記較少,很難將已定位的QTL與前人相比較。通過與前人相比較,也只能明確本研究所定位到的QTL與前人的在染色體水平上相同。然而,隨著生物信息學和比較基因組學的發展及其在玉米相關性狀基因比較定位上的成功應用[26-27],為通過參考高密度遺傳連鎖圖譜進行QTL定位和整合提供參考。本研究進行QTL定位時,參考Yu Jing等[14]整合的遺傳連鎖圖,經與Guo等[28]的高密度遺傳連鎖圖譜相比對,雖然標記之間的距離有所不同,但在染色體上的位置和順序基本一致。因此,本研究所定位的QTL應該是準確可靠的,與之相關聯的分子標記可以嘗試應用于分子標記輔助選擇育種。
4 結論
本研究定位到了6個抗黃萎病QTLs,分別位于6條染色體上,第14條染色體上最多為4個。與6個QTLs相連鎖的分子標記NAU3312、NAU5107、NAU3346、JESPR0050、NAU3913 可嘗試應用于分子標記輔助選擇抗病育種。

