基于TCGA數據庫分析TET1在肺腺癌的表達及其相關分析
馬晨陽 楊勇*
肺癌是常見的惡性腫瘤,其發病率和病死率居惡性腫瘤首位,且在中國上升趨勢明顯[1]。目前對肺癌的防治主要為控煙和早期診斷篩查,其中肺癌早期診斷篩查處于關鍵地位。若能早期篩查到肺癌病變,并手術切除,肺癌患者的5年生存率可得到較大提高[2]。近年來隨著表觀遺傳學的發展,5-甲基胞嘧啶(5-methylcytosine,5mC)在腫瘤的早期診斷和防治方面扮演了重要角色,而由其在DNA甲基胞嘧啶雙加氧酶1(Ten-eleven translocation methylcytosine dioxygenase 1,TET1)調控腫瘤機制成為新的熱點。研究表明,TET1作為一個重要的催化酶參與了DNA的主動去甲基化過程[3],并且與胃癌、結直腸癌、肝癌、肺癌和乳腺癌等多種癌癥的發生發展密切相關。由于肺癌早治療、高生存的優勢,尋求可能的判斷早期肺癌,檢測復發和轉移的分子標記具有較高價值。本研究利用TCGA公共數據,探討TET1在肺腺癌(LUAD)中的表達和預后情況,并預測其參與LUAD發生發展的作用機制。
1 材料與方法
1.1 研究對象 本研究所采用的數據均來源于腫瘤 基 因 組 圖 譜(Cancer Genome Atlas,TCGA)( 美國National Cancer Institute和National Human Genome Research Institute聯合啟動項目),數據收集了截至2019年1月的LUAD組織樣本及臨床預后數據(包括mRNA表達數據和臨床資料)。通過多種在線TCGA數據分析工具,包括可視化工具 cBioPortal[4]和 GEPIA[5]等,對TET1在LUAD中的表達情況進行分析。排除標準:(1)患有其他腫瘤病史;(2)存在基本資料缺失患者。
1.2 研究方法 本研究首先通過GEPIA分析TCGA的LUAD數據庫,比較TET1基因在LUAD和正常組織中的表達差異,并對其臨床病理分期和總生存預后進行相關分析,確定TET1基因在LUAD中的醫學研究價值。然后通過cBioPortal分析比較TET1在TCGA數據庫多個數據集的LUAD基因轉錄組(RVAseqV2,raw count)的總體表達水平,排除個體與組織取樣時間的差異。研究TET1基因拷貝數變異、甲基化水平與其mRNA表達水平的相關性,推測TET1基因在LUAD發生作用的機制。同時檢測TET1基因與臨床常見LUAD驅動基因EGFR、TP53的相關性,并對TET1、EGFR、TP53三個基因通過cBioPortal的Network功能和STRING[6]進行蛋白相互作用網絡分析,確認TET1區別于EGFR、TP53在LUAD中的獨特作用,即作為不同于傳統LUAD驅動基因的新型基因靶點地位。在獲取TET1的共表達基因后,與來源于TCGA和GEO的LUAD數據集所共有的高表達基因(P-value<0.001,|FoldChange|>2.02)進行venn圖分析,找出確定的基因組,并將基因組集導入DAVID分析工具[7]進行GO基因集富集分析和通路分析,并使用funrich3.1.3導出效果圖。
1.3 統計分析 采用SPSS 20.0統計軟件。生存分析使用Kaplan-Meier和Log-rank檢驗,P<0.05為差異有統計學意義。其它基因表達與CNV的相關性以及共表達蛋白的相關性使用Spearman(相關系數:1代表完全線性正相關,0不相關,-1完全線性負相關)和Pearson檢驗相關系數(0.8~1.0極強相關,0.6~0.8強相關,0.4~0.6中等程度相關,0.2~0.4弱相關,0.0~0.2極弱相關或無相關),R>0.3且P<0.05認為有統計學相關。富集功能分析采用Fisher檢驗,P<0.05為功能富集有統計學差異。
2 結果
2.1 TET1在LUAD及正常肺組織中的表達情況 GEPIA分析TET1在LUAD、肺鱗癌(LUSC)與相鄰正常肺組織的表達情況,結果見圖1。圖1A表明在TCGA數據庫中,TET1基因在LUAD組和LUSC組的mRNA表達情況,結果顯示TET1在LUAD和LUSC的表達水平均顯著高于相鄰正常組織(P<0.05)。圖1B通過oncomine在線分析工具對TET1在非小細胞肺癌及正常肺組織的表達情況再次分析,以對GEPIA的結果驗證,結果顯示TET1在LUAD的表達量仍高于正常肺組織。因此,由以上重復驗證可推斷TET1在LUAD的表達量確實高于正常肺組織(P<0.05)。
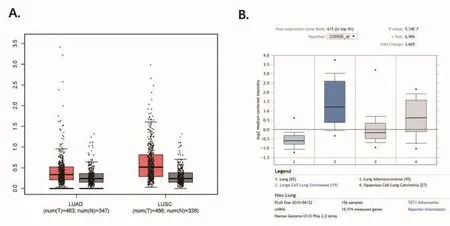
圖1 TET1基因在不同非小細胞肺癌的表達情況[A:TET1在LUAD、LUSC與正常肺組織表達水平的情況;B:TET1在正常肺(1)、LUAD(2)、大細胞肺癌(3)、LUSC(4)與正常肺組織表達水平的情況]
2.2 TET1表達水平對LUAD患者預后的影響 GEPIA分析在LUAD中TET1高低表達水平對患者的病理分期及其生存預后的情況,結果見圖2。圖2A表明隨著LUAD臨床病理的不斷惡化,TET1的表達水平呈現上升趨勢,而LUADⅠ期的TET1表達水平雖然最大值與后期表達水平相當,但其中位數仍低于后期表達水平,這可能是由于樣本個體數據所造成的偏倚。可見在不同的病理總分期之間的TET1表達水平具有統計學差異(P=0.04),且隨病理分期的增加TET1基因的表達水平逐漸上升。圖2B表明LUAD患者的總生存期與TET1基因表達水平的相關性,與TET1基因高表達(n=239)相比,TET1基因低表達(n=226)的LUAD患者總生存率[P=0.045;HR=1.4,P(HR)=0.046]明顯降低,可見TET1高表達患者的生存時間較短,預后較差。
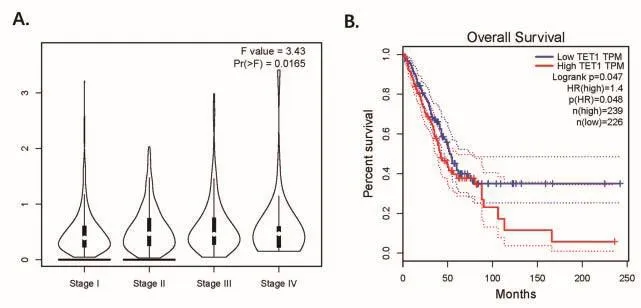
圖2 TET1基因在LUAD病理分期的情況及生存預后(A:TET1在不同LUAD病理分期的表達情況;B:TET1表達水平與LUAD患者生存時間的相關性)
2.3 納入分析的人群基本特征 cBioPortal分析選 擇TCGA數 據 集Lung Adenocarcinoma(TCGA,Provisional),共納入的586例LUAD樣本。其中男242例(41.4%),女280例(47.9%);除去年齡未知的數據,平均年齡(65.619±9.914)歲;LUAD病理分期Ⅰ期(包括Ⅰ、ⅠA、ⅠB)有279例(47.8%),Ⅱ期(包括Ⅱ、ⅡA、ⅡB)有124例(21.3%),Ⅲ期(包括ⅢA、ⅢB)有85例(14.6%),Ⅳ期有26例(4.5%),此外分期未知70例(12.0%);除去未知數據外,平均腫瘤直徑(1.18±0.515)cm。
2.4 TET1基因的拷貝數變異、甲基化水平與其mRNA表達水平的相關性 cBioPortal在線分析工具可對TCGA數據庫LUAD(TCGA,Provisional)數據集中TET1基因拷貝數變異、甲基化水平與其mRNA表達水平的相關性情況進行分析。拷貝數變異(Copy number variation,CNV)指>1kb DNA大片段的缺失(Deletion)、增加(Duplication)或倒置(Inversion),作為一種基因組多樣性形式,在癌癥發展過程中起著重要作用。TET1基因的拷貝數變異(CNV)主要集中于 Diploid、Gain和 Shallow Deletion,其中 Diploid為二倍體,即沒有拷貝數變異,Shallow Deletion是拷貝數的輕度丟失,Gain是輕度拷貝數擴增。隨著DNA甲基化水平的不斷上升,TET1基因的表達量逐漸下降,兩者呈負相關,相關性較弱(Spearman:r=-0.33,Pearson:r=-0.13,P<0.001)。總之,TET1 mRNA Sqe分析表明TET1基因在LUAD中雜合缺失、低水平基因擴增的頻率較高,而其與DNA甲基化相關性較小。
2.5 TET1基因與臨床常見LUAD驅動基因的相關性 目前臨床常用的檢測LUAD驅動基因主要為EGFR、TP53,其與患者的治療、預后密切相關。作為近年熱門的免疫治療,EGFR、TP53基因突變是治療的優勢靶點。本研究將結合EGFR、TP53基因,通過cBioPortal在線分析工具分析在TCGA數據庫LUAD(TCGA,Provisional)數據集中TET1基因與兩者的相關性。結果顯示,EGFR與TP53基因共表達趨勢顯著(P<0.001,q<0.001),TET1與 TP53基因的存在共表達趨勢(P=0.012),存在顯著的共表達趨勢,而其與EGFR基因則無顯著相關性(P=0.061)。通過GEPIA對TET1與EGFR、TP53兩者在LUAD的相關性再次進行驗證,結果顯示,在整合所有TCGALUAD數據后,發現TET1與EGFR呈弱相關性(R=0.3,P=3.4e-11),而TET1與TP53兩者呈極弱相關性(R=0.11,P=0.017)。這與cBioPortal單數據集分析結果較一致,綜合兩者分析,可以認為TET1與EGFR、TP53基因的相關性較弱,可能是不同于傳統EGFR、TP53基因的新型LUAD靶點基因。
2.6 蛋白相互作用網絡分析(PPI) 通過cBioPortal的Network功能和STRING對TET1、EGFR、TP53三個基因進行蛋白相互作用網絡分析,確認TET1區別于EGFR、TP53在LUAD中的獨特位置。通過cBioPortal的Network功能,共篩選出TET1、EGFR、TP53基因的共表達基因如下:ACTB、ACTG1、AGO2、AKT1、AKT2、AKT3、BAZ1B、CCNK、CDH10、CDH12、CDH18、CDH6、CDH9、CDKN2A、COP1、DDR2、DROSHA、DVL3、DYRK1A、EGFR、FCGR1A、FCGR3A、FOXA1、GAB2、GDNF、GRB2、HGS、MAPK14、MAPKAPK、MCL1、MDM4、MET、NDRG1、NF1、PDP1、PIK3CA、PIP4K2B、PITPNA、PRKAB2、PRKD1、PRKDC、PTK2、S100A2、SHC1、SKP2、SPTA1、STK11、STK17A、TERT、TET1、TP53、TP53INP1、TRIO。將基因納入Network網絡后,EGFR和TP53在PPI網絡中處于中心地位,與絕大多數的共表達基因節點相連接,而TET1由于聯系較弱,并未出現在Network網絡中。通過STRING對以上共表達基因進行再次驗證后,發現TET1基因處于PPI網絡的邊緣位置,與EGFR、TP53基因無直接連接節點,間接連接節點僅AKT1。綜合相關性分析和PPI網絡分析,說明TET1確實與傳統LUAD驅動基因EGFR、TP53無明顯相關,可能是作為一種獨立的驅動基因對LUAD的發生發展起作用的。
2.7 TET1的共表達基因 通過cBioPortal分析方法對TET1在LUAD的共表達基因進行篩選,結果的P值從小到大進行排序,選取P值最小的前100個共表達基因。同時通過Cancer RNA-Seq Nexus[8]下載TCGA和GEO的LUAD基因數據集組,對高表達基因進行篩選去重處理(P-value<0.001,|FoldChange|>2.02),得到三組基因集,分別為早期LUAD與正常肺組織對照所得的高表達基因,中晚期LUAD與正常肺組織對照所得的高表達基因以及GEO數據庫(GES40419)中LUAD與正常肺組織對照所得的高表達基因,通過Venn圖進行分析后確定共表達的基因組。將共表達基因組與cBioPortal分析方法所得的TET1共表達基因組再次進行Venn圖分析,確定TET1在LUAD有13個共表達基因,分別為TET1、DNMT3A、MEX3A、PAPOLA、SMC6、XPO5、ATAD2B、ZNF146、ZNF260、HMOX2、CCAR1、TRADD、TMEM219。
2.8 TET1基因的PPI網絡分析 在獲取TET1的共表達基因后,通過cBioPortal的Network功能將共表達基因導入,進行PPI網絡分析。結果顯示:基因 MRPL54、TRADD、CTSD、CD74、MAPKAPK3、PRPF4B、RPS6KA1、GNA1S、CLTB、PSMB10、SOS1、SPSF6、TREM2、LGALS3、OAZ1處 于 調控網絡的中心地位,有多個Node相連接。其中,RPS6KA1、MAPKAPK3、CTSD、CD74、LGALS3 為現有藥物的靶點。
2.9 TET1基因集富集分析及通路預測 通過對TET1基因相關共表達基因分析發現,TET1基因與一些基因表達水平存在明顯的關聯,對包括TET1相關表達GO功能富集分析(見表1),發現這些共表達基因的細胞組分主要集中在細胞核和細胞質,生物學功能主要集中于調控堿基、核苷功能,分子功能富集主要在DNA連接和轉錄活動的調控。因此,TET1相關共表達基因主要通過遺傳轉錄調控方面調節腫瘤的發生發展。表1表明三條通路 HSA-212165、HSA-74160、HSA-5334118與TET1基因具有相關性。提示對TET1基因在LUAD的機制分析可從這三條通路考慮,且此三條通路的功能與go注釋分析結果,遺傳轉錄調控調節腫瘤相一致。

表1 TET1的通路分析
3 討論
LUAD作為惡性程度較高的呼吸道腫瘤,若早期未發現,常會錯過最佳治療時期,放療、化療等綜合治療對患者生存時間的改善甚微,其治療的首選方案為手術,但長期的臨床實踐表明,對于LUAD單純手術的治療效果有限[2]。因此,早期發現并診斷LUAD成為目前迫在眉睫的問題。通過對LUAD基因的調查,發現了以前的基因研究尚不完善。在過去的幾十年里,有一些分子標志物被研究出來,作為LUAD早期診斷的基因標志物。
DNA甲基化是腫瘤發生發展過程中的重要角色,其通過沉默抑癌基因表達而影響腫瘤進程,而DNA去甲基化則可使抑癌基因再表達。TET1基因作為DNA去甲基化酶在許多腫瘤的發生、發展中有著不同的改變,其對于腫瘤的密切關系與日俱增。LUAD患者存在TET1基因突變頻率增加的情況,并且LUAD中TET1大多數的變異是基因缺失,TET1基因主要是通過腫瘤細胞周期抑制劑調控腫瘤進展。在胃癌、結直腸癌、肝癌、肺癌和乳腺癌等多種癌癥中發現腫瘤組織中TET1表達水平的降低常伴隨5hmC水平降低[9],表明5hmC也可能在TET1蛋白的作用下通過去甲基化作用抑制抑癌基因的表達。在實驗動物腫瘤種植模型上,有研究者觀察到小鼠TET1基因的敲除會強化腫瘤細胞的侵襲能力、生長速度及轉移能力[10],并發現5hmC水平的相應變化。這可能是由于TET1表達降低導致抑癌基因沉默,最終使正常細胞惡變并促其增殖。但TET1基因還存在一些不足,其在非實體腫瘤的檢測中也有升高,如在白血病中等等。所以,通過認識、了解TET1基因的改變特點,有可能為LUAD的早期診斷及治療提供新的思路和方法。
本研究結果表明,在LUAD中TET1基因表達上升是一種預后較差的因素。利用 TCGA數據庫分析發現TET1基因在LUAD中特異性高表達,且TET1高表達的LUAD患者總生存預后較差。對TET1基因的CNV、甲基化水平與其mRNA的表達水平的相關性研究,發現TET1基因在LUAD中雜合缺失、低水平基因擴增的頻率較高,而其與DNA甲基化相關性較小。臨床常檢測驅動基因TP53、EGFR在LUAD中的表達,相關性分析發現TET1基因與兩者無顯著相關,而PPI網絡分析也驗證了這一結果,可見TET1基因可能作為一種獨立的LUAD驅動基因對LUAD的發生、發展起到作用。
再次對TET1基因功能的研究通過共表達基因的方式進行,對TET1共表達基因的PPI網絡分析發現DNMT3A、DNMT3L、CCAR1與TET1基因存在明顯的上下游關系。結合TET1及其共表達基因的KEGG-GO功能富集分析,發現這些共表達基因的功能主要集中于一些遺傳轉錄調控方面的信號通路,可確認TET1在LUAD產生作用主要是通過遺傳轉錄調控方面調節腫瘤的發生發展。
綜上所述,本研究依托TCGA數據庫,對LUAD患者進行大樣本數據分析和臨床資料對比分析,探討TET1基因表達水平對患者預后的影響,可確定TET1基因可作為不同于傳統EGFR、TP53的LUAD驅動基因,對LUAD的發生發展起到促進作用。對TET1在LUAD的后續機制和相關共表達基因的研究,有利于臨床新型的LUAD預后評估分子標志物和治療作用靶點的發現和應用。

